È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Utilizzo In Vitro Fluorescence Resonance Energy Transfer per studiare la dinamica dei complessi della proteina ad una scala di tempo di millisecondo
In questo articolo
Riepilogo
Interazioni proteina-proteina sono critiche per sistemi biologici, e studi di cinetica di legame forniscono intuizioni la dinamica e la funzione dei complessi della proteina. Descriviamo un metodo che quantifica i parametri cinetici di una proteina complessa che utilizza il trasferimento di energia di risonanza di fluorescenza e la tecnica di flusso interrotto.
Abstract
Le proteine sono i principali operatori dei sistemi biologici, e di solito interagiscono con altri macro - o piccole molecole per svolgere le loro funzioni biologiche. Tali interazioni possono essere altamente dinamiche, significato le subunità interagenti sono costantemente associate e dissociate a determinate tariffe. Mentre l'affinità di legame utilizzando tecniche quali discesa quantitativa rivela la forza dell'interazione, studiare la cinetica di legame di misurazione fornisce intuizioni su come veloce si verifica l'interazione e per quanto tempo ogni complesso può esistere. Inoltre, la cinetica di un'interazione in presenza di un fattore supplementare, ad esempio un fattore di scambio di proteina o un farmaco, di misura aiuta a rivelare il meccanismo mediante il quale l'interazione è regolata da un altro fattore, fornendo conoscenze importanti per la avanzamento della ricerca biologica e medica. Qui, descriviamo un protocollo per la misurazione la cinetica di legame di un complesso proteico che ha un tasso di alta associazione intrinseco e possa essere dissociato rapidamente da un'altra proteina. Il metodo utilizza il trasferimento di energia di risonanza di fluorescenza per segnalare la formazione del complesso della proteina in vitro, e consente il monitoraggio veloce associazione e dissociazione del complesso in tempo reale su un fluorimetro flusso interrotto. Usando questa analisi, le costanti di velocità di associazione e dissociazione del complesso proteico sono quantificate.
Introduzione
Attività biologiche sono in ultima analisi, effettuate dalle proteine, più di cui interagire con gli altri per corrette funzioni biologiche. Utilizzando un approccio computazionale, l'importo totale delle interazioni proteina-proteina in essere umano è stimato essere ~ 650.0001e rottura di queste interazioni spesso conduce a malattie2. A causa del loro ruolo essenziale nel controllo dei processi cellulari e organismi, sono stati sviluppati numerosi metodi per lo studio delle interazioni proteina-proteina, quali lievito-due-ibrido, complementazione bimolecolare fluorescenza, Spalato-luciferasi 3analisi di complementazione e co-immunoprecipitazione. Mentre questi metodi sono bravi a scoprire e confermando le interazioni proteina-proteina, sono solitamente non quantitativi e così fornire informazioni limitate sull'affinità tra i partner di proteine interagenti. Quantitativi di pull-down può essere utilizzato per misurare l'affinità di legame (ad esempio, la costante di dissociazione Kd), ma non misura la cinetica dell'associazione, né può essere applicata quando il Kd è molto bassa a causa di un'inadeguata rapporto segnale-rumore4. Surface plasmon resonance (SPR) spettroscopia quantifica la cinetica di legame, ma richiede una superficie specifica e l'immobilizzazione di un reattivo sulla superficie, che potenzialmente può modificare la proprietà di associazione del reagente5. Inoltre, è difficile per SPR misurare veloce associazione e dissociazione tariffe5, e non è opportuno utilizzare SPR per caratterizzare l'evento di scambio delle unità secondarie della proteina in un complesso proteico. Qui, descriviamo un metodo che permette di misurare il tasso di proteine complesse montaggio e smontaggio in una scala di tempo di millisecondo. Questo metodo è essenziale per determinare il ruolo di Cullin -unesso -Nedd8 -dissociated della proteina 1 (Cand1) come il F-box protein cambio fattore6,7.
Cand1 regola la dinamica della ligasi E3 di Skp1•Cul1•F-casella proteina (SCF), che appartengono alla grande famiglia della ligasi di ubiquitin Cullin-anello. SCFs sono costituiti il cullin Cul1, che lega la proteina di dominio anello Rbx1, e una proteina F-box intercambiabile, che recluta substrati e lega Cul1 attraverso la proteina dell'adattatore Skp18. Come una E3 ligasi, SCF catalizza la coniugazione dell'ubiquitina al suo substrato ed è attivato quando il substrato è reclutato dalla proteina F-box, e quando Cul1 viene modificato dalla proteina ubiquitina-come Nedd89. Cand1 associa Cul1 non modificato e legandosi, disturba sia l'associazione di proteine Skp1•F-box con Cul1 e la coniugazione di Nedd8 a Cul110,11,12,13. Di conseguenza, Cand1 è sembrato essere un inibitore di attività SCF in vitro, ma Cand1 carenza negli organismi ha causato i difetti che suggerisce un ruolo positivo di Cand1 nel regolare l'attività SCF in vivo14,15,16 , 17. questo paradosso infine è stato spiegato da uno studio quantitativo che ha rivelato le interazioni dinamiche tra proteine Cul1, Cand1 e Skp1•F-box. Usando le fluorescenza risonanza energy transfer (FRET) analisi che rilevano la formazione dei complessi SCF e Cul1•Cand1, l'associazione e dissociazione rate costanti (ksu e koff, rispettivamente) sono stati misurato singolarmente. Le misurazioni hanno rivelato che sia Cand1 e Skp1•F-box forma estremamente stretta complesso proteico con Cul1, ma la kfuori di SCF è aumentata drammaticamente di Cand1 e la kfuori del Cul1•Cand1 è aumentato drammaticamente da Skp1•F-casella proteina6,7. Questi risultati forniscono il supporto iniziale e fondamentale per definire il ruolo di Cand1 come un fattore di scambio di proteina, che catalizza la formazione di nuovi complessi SCF attraverso il riciclo Cul1 dai vecchi complessi SCF.
Qui, presentiamo la procedura di sviluppare e utilizzare il tasto test per studiare la dinamica del complesso Cul1•Cand17, e lo stesso principio può essere applicato per lo studio della dinamica delle varie biomolecole. FRET si verifica quando un donatore è eccitato con la lunghezza d'onda appropriata, e un accettore con spettro di eccitazione sovrapposizione lo spettro di emissione del donatore è presente all'interno di una distanza di 10-100 Å. Lo stato eccitato viene trasferito al ricettore, quindi diminuendo l'intensità di donatore e aumentando l' intensità di accettore18. L'efficienza della FRET (E) dipende dalla funzione del raggio di Förster (R0) e la distanza tra il donatore e accettore fluorofori (r) ed è definito da: E = R06/ (R0 6 + r6). Il raggio di Förster (R0) dipende da alcuni fattori, tra cui l'orientamento angolare di dipolo, la sovrapposizione spettrale della coppia donatore-accettore e la soluzione usata19. Per applicare l'analisi FRET su un fluorimetro flusso interrotto, che rileva la variazione dell'emissione dei donatori in tempo reale e permette di eseguire misure di veloce ksu e kfuori, è necessario stabilire FRET efficiente che Risultati in una significativa riduzione dell'emissione di donatore. Di conseguenza, progettazione efficiente FRET scegliendo la coppia appropriata di coloranti fluorescenti e siti su proteine bersaglio per fissare i coloranti è importante e sarà discusso in questo protocollo.
Protocollo
1. progettazione del dosaggio FRET.
- Scaricare il file di struttura dei Cul1•Cand1 complessi da Protein Data Bank (file 1U6G).
- Visualizzare la struttura della Cul1•Cand1 complesso in PyMOL.
- Utilizzare la funzione di misurazione nel menu di creazione guidata di PyMOL per stimare la distanza tra il primo amminoacido di Cand1 e l'ultimo amminoacido di Cul1 (Figura 1).
- Il visualizzatore online spettri di carico (Vedi Tabella materiali) e visualizzare contemporaneamente l'eccitazione e spettri di emissione di 7-amino-4-methylcoumarin (AMC) e FlAsH (Figura 2). Si noti che AMC è il donatore FRET e FlAsH è l'accettore FRET.
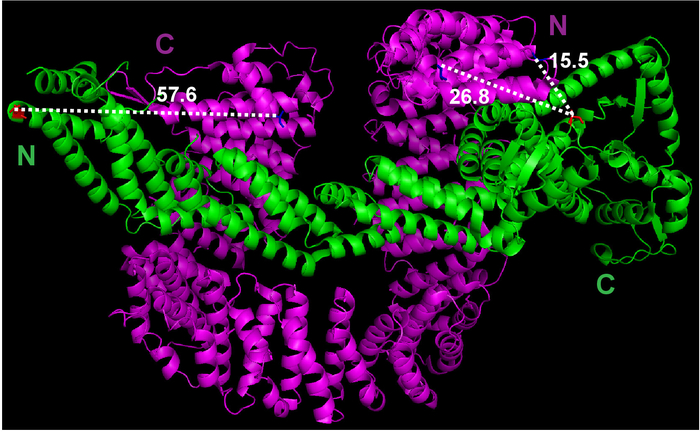
Figura 1: struttura cristallina della misurazione della distanza tra potenziale etichettatura siti e Cul1•Cand1. Il file di struttura di cristallo è stato scaricato da Protein Data Bank (File 1U6G) e hanno visto in PyMOL. Misure tra atomi selezionati sono state fatte di PyMOL. Clicca qui per visualizzare una versione più grande di questa figura.
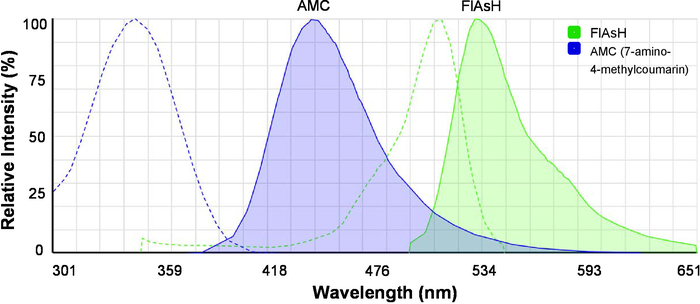
Figura 2: l'eccitazione e spettri di emissione delle tinture fluorescenti per FRET. Sono mostrati gli spettri di AMC (7-ammino-4-methylcoumarin) e FlAsH. Le linee tratteggiate indicano gli spettri di eccitazione, e linee continue indicano spettri di emissione. L'immagine originalmente è stato generato dalla SpectraViewer di fluorescenza e fu modificata per una maggiore chiarezza. Clicca qui per visualizzare una versione più grande di questa figura.
2. preparazione del Cul1AMC•Rbx1, la proteina di donatore FRET
- Plasmidi per l'espressione umana Cul1sortasi•Rbx1 in cellule di Escherichia coli . Si noti che i due plasmidi cheesprimono Cul1•Rbx1 umano in cellule di Escherichia coli sono descritte in dettaglio in una precedente relazione20.
- Aggiungere una sequenza di DNA codifica "LPETGGHHHHHH" (sortasi-His6 tag) all'estremità 3' della sequenza di codificazione Cul1 attraverso standard di PCR e clonazione metodi,,2122.
- Il nuovo plasmide per confermare l'inserimento del gene è preciso di sequenza.
- Co-express Cul1sortasi•Rbx1 in cellule di Escherichia coli . Il metodo è derivato da una precedente relazione20.
- Mix 100 ng ogni dei due plasmidi con BL21 (DE3) cellule chimicamente competenti per co-trasformazione utilizzando il calore shock metodo23. Crescere le cellule sulla piastra di agar LB contenente 100 µ g/mL ampicillina e cloramfenicolo 34 µ g/mL a 37 ° C durante la notte.
- Inoculare in 50 mL di coltura LB con colonie appena trasformati e crescere durante la notte a 37 ° C con agitazione di 250 giri/min. Questo dà una coltura starter.
- Inoculare 6 beute, ognuna con 1 L di LB medium, con coltura starter di 5 mL ciascuna e far crescere a 37 ° C con 250 giri/min agitazione fino a quando il OD600 è ~ 1.0. Raffreddare la cultura a 16 ° C e aggiungere isopropilico-β-D-thiogalactoside (IPTG) a 0,4 mM. Mantenere la cultura a 16 ° C durante la notte con l'agitazione di 250 giri/min.
- Raccogliere le cellule di Escherichia coli mediante centrifugazione a 5.000 x g per 15 min e recuperare il pellet cellulare in provette coniche da 50 mL.
Nota: Il pellet di cellule possa essere elaborato per la purificazione della proteina o essere congelato a-80 ° C prima di procedere con le fasi di purificazione della proteina.
- Purificazione della Cul1sortasi•Rbx1 complesso. Questo metodo è derivato da una precedente relazione20.
- Aggiungere 50 mL di tampone di lisi (30 mM Tris-HCl, 200 mM NaCl, 5 mM DTT, 10% glicerolo, 1 compressa di cocktail, inibitore della proteasi pH 7,6) per il pellet di cellule di e. coli che esprimono Cul1sortasi•Rbx1.
- Lisare le cellule su ghiaccio con sonicazione ad ampiezza di 50%. Si alternano tra 1 secondo e 1 secondo spegnere ed eseguire per 3 min.
- Ripetere il punto 2.3.2 x 2-3.
- Trasferire il lysate delle cellule in una provetta da centrifuga da 50 mL e rimuovere i detriti cellulari mediante centrifugazione a 25.000 x g per 45 min.
- Incubare le cellule chiare lisata con 5 mL di glutatione perline a 4 ° C per 2 h.
- Centrifugare la miscela perline-lisato a 1.500 x g per 2 min a 4 ° C. Eliminare il surnatante.
- Lavare le perle con 5 mL di tampone di lisi (con senza inibitori di proteasi) e rimuovere il supernatante dopo centrifugazione a 1.500 x g per 2 min a 4 ° C.
- Ripetere il punto 2.3.7 2x.
- Aggiungere 3 mL di tampone di lisi per le perline lavate e trasferire i residui della perla in una colonna vuota.
- Aggiungere 5 mL di tampone di eluizione (50 mM Tris-HCl, 200 mM NaCl, 10 mM ridotto glutatione, pH 8.0) nella colonna. Incubare per 10 minuti e raccogliere l'eluato.
- Ripetere il passaggio 2.3.10 x 3-4.
- Aggiungere 200 µ l di trombina di 5 mg/mL (Vedi Tabella materiali) per l'eluato dalle perle di glutatione e incubare per una notte a 4 ° C.
Nota: Il protocollo può essere messo in pausa qui. - Diluire il campione della proteina con tampone A (25mm HEPES, 1 millimetro DTT, 5% glicerolo, pH 6.5) triplo.
- Equilibrare una colonna di cromatografia di scambio cationico (Vedi Tabella materiali) su un sistema FPLC con Buffer A.
- Caricare il campione della proteina alla colonna di cromatografia di scambio cationico equilibrato ad una portata di 0,5 mL/min.
- Eluire la proteina con una sfumatura di NaCl in 40 mL di miscelazione tampone A e da 0 a 50% B Buffer (25mm HEPES, NaCl di 1m, 1 millimetro DTT, 5% glicerolo, pH 6.5) con una portata di 1 mL/min.
- Controllare la proteina eluita in diverse frazioni usando SDS-PAGE24.
Nota: Il protocollo può essere messo in pausa qui. - Le frazioni di eluato contenente Cul1sortasi•Rbx1 della piscina.
- Concentrare il campione di •Rbx1 disortasiCul1 riunito a 2,5 mL passando il buffer attraverso una membrana di ultrafiltrazione (cut-off 30 kDa).
- Aggiungere AMC al C-terminale di Cul1 attraverso sortasi-mediata transpeptidazione21,22.
- Modificare il buffer del campione di •Rbx1sortasiCul1 nel buffer sortasi (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7.5) utilizzando una colonna di dissalazione (Vedi Tabella materiali).
- Equilibrare una colonna dissalazione con 25 mL di tampone sortasi.
- Caricare 2,5 mL di Cul1sortasi•Rbx1 nella colonna. Scartare il flusso continuo.
- Eluire il campione con 3,5 mL di tampone sortasi. Raccogliere il flusso continuo.
- A 900 µ l della soluzione Cul1sortasi•Rbx1 nel buffer sortasi, aggiungere 100 µ l di soluzione di 600 µM purificato sortasi A e 10 µ l di peptide GGGGAMC 25 mM. Incubare la miscela di reazione a 30 ° C nella notte scura. Nota che questo passaggio genererà Cul1AMC•Rbx1.
Attenzione: Tinture fluorescenti sono sensibili alla luce, quindi evitare di esporli alla luce estranea durante la preparazione della proteina e del campione per quanto possibile.
Nota: Il protocollo può essere messo in pausa qui. - Aggiungere 50 µ l di perle di agarosio Ni-NTA alla miscela di reazione e incubare a temperatura ambiente per 30 min.
- A pellet le perline di agarosio Ni-NTA mediante centrifugazione a 5.000 x g per 2 minuti e raccogliere il surnatante.
- Equilibrare una colonna di cromatografia di esclusione di formato (Vedi Tabella materiali) con buffer (30 mM Tris-HCl, 100 mM NaCl, 1 millimetro DTT, 10% glicerolo) sul sistema FPLC.
- Caricare tutti i Cul1AMC•Rbx1 campione sulla colonna di cromatografia di esclusione di dimensione. Eluire con volume di colonna x 1.5 del buffer.
- Verifica le frazioni di eluato da SDS-PAGE24.
- Piscina le frazioni di eluato contenente Cul1AMC•Rbx1.
- Misurare la concentrazione di proteine utilizzando la sua capacità di assorbimento a 280 nm con uno spettrofotometro.
- Aliquotare la soluzione della proteina e conservare a-80 ° C.
Nota: Il protocollo può essere messo in pausa qui.
- Modificare il buffer del campione di •Rbx1sortasiCul1 nel buffer sortasi (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7.5) utilizzando una colonna di dissalazione (Vedi Tabella materiali).
3. preparazione del FlAsHCand1, la proteina di accettore FRET
Nota: La maggior parte dei passaggi in questa parte sono gli stessi come passaggio 2. Condizioni diverse sono descritti in dettaglio di seguito.
- Costruire il plasmide per esprimere umana TetraCysCand1 in cellule di Escherichia coli .
- Aggiungere la sequenza di DNA codifica "CCPGCCGSG" (tetracysteine/TetraCys tag) prima 15th dell'amminoacido di Cand1 attraverso regolari PCR25 (sequenze dell'iniettore: TGCTGTCCGGGCTGCTGCGGCAGCGGCATGACATCCAGCGACAAGGACTTTAG; CTAACTAGTGTCCATTGATTCCAAG).
- Inserire il prodotto PCR in un vettore pGEX-4T-2. Sequenza del plasmide per confermare l'inserimento del gene è accurata e nel telaio.
- Express TetraCysCand1in Escherichia coli cellule nello stesso modo come passaggio 2.2, tranne che il plasmide si trasforma in cellule competenti di Rosetta.
- Purificazione di TetraCysCand1 dalle cellule e. coli .
- Lisare il pellet di cellule di Escherichia coli ed estrarre TetraCysCand1using perle di glutatione. Questi passaggi sono gli stessi come passi 2.3.1–2.3.12.
- Diluire l'eluato di proteina dalle perle di glutatione con Buffer C (50 mM Tris-HCl, 1 millimetro DTT, 5% glicerolo, pH 7.5) da tre pieghe. Equilibrare una colonna di cromatografia di scambio anionico (Vedi Tabella materiali) sul sistema FPLC con Buffer C e carico il campione diluito della proteina ad una portata di 0,5 mL/min.
- Eluire la proteina con una sfumatura di NaCl in 40 mL di miscelazione Buffer C e da 0 a 50% D Buffer (50 mM Tris-HCl, NaCl di 1m, 1 millimetro DTT, 5% glicerolo, pH 7.5) con una portata di 1 mL/min. Controllare la proteina eluita in diverse frazioni usando SDS-PAGE24e la piscina le frazioni contenenti TetraCysCand1. Si noti che TetraCysCand1 ha un più grande volume di ritenzione rispetto gratis GST.
- Concentrare il campione di Cand1 di TetraCyspassando il buffer attraverso una membrana di ultrafiltrazione (cut-off 30 kDa).
- Equilibrare la colonna di cromatografia di esclusione size con etichettatura buffer (20 mM Tris-HCl, NaCl 100 mM, 2 mM TCEP, 1 mM EDTA, 5% glicerolo) sul sistema FPLC. Carica TetraCysCand1 campione (500 µ l ogni volta) e verificare le frazioni di eluato da SDS-PAGE24.
- Tutte le frazioni contenenti TetraCysCand1 della piscina e concentrare la proteina a ~ 40 µM passando il buffer attraverso una membrana di ultrafiltrazione (cut-off 30 kDa). Stimare la concentrazione di proteine utilizzando la sua capacità di assorbimento a 280 nm. Memorizzare la proteina come 50 aliquote di µ l a-80 ° C.
Nota: Il protocollo può essere messo in pausa qui.
- Preparazione di FlAsHCand1.
- Aggiungere 1 µ l di soluzione FlAsH (Vedi Tabella materiali) a 50 µ l di TetraCysCand1 soluzione.
- Mescolare bene e incubare la miscela a temperatura ambiente al buio per 1-2 h per ottenere FlAsHCand1.
Nota: Il protocollo può essere messo in pausa qui.
4. preparazione del Cand1, la proteina di inseguimento FRET
Nota: Il protocollo di preparazione della proteina è simile al passaggio 3, con le seguenti modifiche.
- Inserire la sequenza di codificazione del full-length Cand1 il vettore pGEX-4T-2.
- Modificare il buffer utilizzato nel passaggio 3.3.5 a un buffer contenente 30 mM Tris-HCl, 100 mM NaCl, 1 millimetro DTT, glicerolo al 10%.
- Eliminare passaggi 3.3.7 e 3.3.8.
5. testare e verificare il dosaggio FRET
- Preparare il tampone FRET contenente 30 mM Tris-HCl, NaCl 100 mM, 0,5 mM DTT, ovoalbumina 1 mg/mL, pH 7,6 e utilizzare a temperatura ambiente.
- Testare il FRET tra Cul1AMC•Rbx1 e Cand1 FlAsHsu un fluorimetro.
- A 300 µ l di tampone FRET, aggiungere Cul1AMC•Rbx1 (FRET donatore) ad una concentrazione finale di 70 nM. Trasferire la soluzione in una cuvetta.
- Disponga la provetta nel supporto del campione di un fluorimetro. Eccitare il campione con luce di eccitazione 350 nm e analizzare i segnali di emissione da 400 nm a 600 nm a incrementi di 1 nm.
- Ripetere il punto 5.2.1 ma cambiamento Cul1AMC•Rbx1 a Cand1 FlAsH(accettore FRET). Esplorare il campione di Cand1 FlAsHutilizzando la stessa procedura descritta al punto 5.2.2.
- (Opzionale) Eccitare il Cand1 FlAsHcon la luce di eccitazione 510 nm e scansione del segnale di emissione da 500 nm a 650 nm.
- A 300 µ l di tampone FRET, aggiungere entrambi Cul1AMC•Rbx1 e Cand1 FlAsHa una concentrazione finale di 70 nM. Analizzare il campione nello stesso modo come descritto al punto 5.2.2.
- Confermare il FRET tra Cul1AMC•Rbx1 e Cand1 FlAsHaggiungendo la proteina chase (adenoide Cand1) (Figura 3).
- A 300 µ l di tampone FRET, aggiungere 70 nM Cul1AMC•Rbx1 e 700 nM Cand1. Analizzare l'emissione di campione come descritto al punto 5.2.2.
- A 300 µ l di tampone FRET, aggiungere 70 nM Cand1 FlAsHe 700 nM Cand1. Analizzare l'emissione di campione come descritto al punto 5.2.2.
- A 300 µ l di tampone FRET, aggiungere 70 nM Cul1AMC•Rbx1 e 70 nM Cand1 FlAsHe mantenere in incubazione il campione a temperatura ambiente per 5 min. Quindi aggiungere 700 nM Cand1 e subito dopo l'aggiunta, la scansione l'emissione del campione come descritto al punto 5.2.2). Si noti che questo passaggio è simile al punto 5.2.5.
- A 300 µ l di tampone FRET, in sequenza aggiungere 70 nM Cul1AMC•Rbx1, 700 nM Cand1 e 70 nM FlAsHCand1. Incubare il campione a temperatura ambiente per 5 minuti e la scansione l'emissione del campione come descritto al punto 5.2.2. Si noti che questo è l'esempio di chase (linea verde in Figura 3).
6. misurare la costante di velocità di associazione (ksu) di Cul1•Cand1
Nota: Dettagli di un fluorimetro fermato-flusso di funzionamento è stato descritto in una precedente relazione26.
- Preparare il fluorimetro fermato-flusso per la misurazione.
- Accendere il fluorimetro flusso interrotto secondo le istruzioni del fabbricante.
- Impostare la luce di eccitazione a 350 nm e uso un filtro passa-banda che consente 450 nm emissione luce passare e blocca 500-650 nm emissione luce.
- Tenere le valvole di campione nella posizione di compilare e collegare una siringa da 3 mL riempita d'acqua. Lavare le due siringhe di campione (A e B) con acqua spostando il campione siringa unità su e giù più volte. Eliminare tutta l'acqua utilizzata in questo passaggio.
- Tenere le valvole di campione nella posizione di compilare e collegare una siringa da 3 mL riempita con buffer di FRET. Lavare le due siringhe di campione con il buffer FRET spostando il campione siringa unità su e giù più volte. Scarta tutti i FRET buffer utilizzato in questo passaggio.
- Prendere una misura di controllo (Figura 4).
- Collegare una siringa da 3 mL e caricare la siringa A con 100 nM Cul1AMC•Rbx1 nel buffer FRET. Girare la valvola del campione la posizione di Guida .
- Collegare una siringa da 3 mL e caricare la siringa B con il buffer FRET. Girare la valvola del campione la posizione di Guida .
- Utilizzare il Pannello di controllo sotto Acquire nel software per prendere cinque colpi (mescolare uguale volume di campioni dalla siringa A e B, siringa) sul fluorimetro flusso interrotto senza registrazione dei risultati.
- Aprire il Pannello di controllo sotto Acquire nel software e programma per registrare l'emissione di Cul1AMC oltre 60 s. Poi prendere un colpo singolo.
- Ripetere il passaggio 6.2.4 2x.
- Girare la valvola del campione la posizione di riempire . Svuotare la siringa B e lavare con il buffer FRET.
- Misura osservato costanti di velocità di associazione (kobs) di Cul1•Cand1 (Figura 4B).
- Mantenere il campione in siringa allo stesso come descritto al punto 6.2.1.
- Collegare una siringa da 3 mL e caricare la siringa B con 100 nM Cand1 FlAsHnel buffer FRET. Girare la valvola del campione la posizione di Guida .
- Utilizzare il Pannello di controllo sotto Acquire nel software per prendere cinque colpi senza registrazione dei risultati.
- Aprire il Pannello di controllo sotto Acquire nel software e programma per registrare l'emissione di Cul1AMC oltre 60 s. Poi prendere un colpo singolo.
- Ripetere il punto 6.3.4 2x.
- Svuotare la siringa B e lavare con il buffer FRET.
- Ripetere i passaggi 6.3.1–6.3.6 parecchie volte con aumento delle concentrazioni di Cand1 FlAsHnel buffer FRET.
- Misura la variazione (diminuzione) in segnali fluorescenti misurati nel tempo da ogni colpo a una singola curva esponenziale. Questo vi darà kobsin ogni misura, e l'unità è s-1. Si noti che la base di questo calcolo è stato discusso anche in un precedente rapporto di27.
- Calcolare la media e la deviazione standard di kobsper ogni concentrazione di Cand1 FlAsHutilizzato. Tracciare il medio kobscontro Cand1 concentrazione (Figura 4), e la pendenza della retta rappresenta la ksu di Cul1•Cand1, con un'unità di M-1 s-1.
7. misurare la costante di dissociazione tasso (koff) di Cul1•Cand1 in presenza di proteina Skp1•F-box.
Nota: Questo passaggio è simile al passaggio 6, con le seguenti modifiche.
- Nella siringa A, sotto la posizione di riempire , caricare una soluzione di 100 nM Cul1AMC•Rbx1 e 100 nM Cand1 FlAsHnel buffer FRET. Girare la valvola del campione nella posizione "Auto".
- In siringa B, sotto la posizione di riempire , caricare una soluzione di Skp1•Skp2 (preparati a seguito di una precedente relazione20). Girare la valvola del campione nella posizione "Auto".
- Aprire il Pannello di controllo sotto Acquire nel software e programma per registrare l'emissione di Cul1AMC oltre 30 s. Poi prendere un colpo singolo. I segnali fluorescenti aumentano nel tempo dopo la miscelazione di soluzioni da A siringa e siringa B (Figura 5).
Risultati
Per testare il FRET tra Cul1AMC e FlAsHCand1, abbiamo determinato in primo luogo l'intensità di emissione di 70 nM Cul1AMC (il donatore) e 70 nM Cand1 FlAsH(il ricettore), rispettivamente (Figura 3A-C, blu linee). In ogni analisi, solo un picco di emissione era presente e l'emissione di Cand1 FlAsH(il ricettore) era basso. Quando 70 nM ogni di Cul1AMC e Cand1 FlAsHs...
Discussione
FRET è un fenomeno fisico che è di grande interesse per lo studio e la comprensione di sistemi biologici19. Qui, presentiamo un protocollo per il testing e usando FRET per studiare la cinetica di legame di due proteine interagenti. Quando si progetta FRET, abbiamo considerato tre fattori principali: la sovrapposizione spettrale tra donatore di emissione e accettore di eccitazione, la distanza tra i due fluorofori e l'orientamento del dipolo dei fluorofori28. Per scegliere...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo Shu-Ou Shan (California Institute of Technology) per penetranti discussione sullo sviluppo del dosaggio FRET. M.G., Y.Z. e X.L. sono stati finanziati dai fondi di avvio dalla Purdue University a Y.Z. e X.L.This lavoro è stato sostenuto in parte da una sovvenzione di seme da Purdue University Center per biologia vegetale.
Materiali
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
Riferimenti
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon