Method Article
Assemblaggio di nanorod oro in Metamolecules plasmoniche chirale utilizzando modelli di Origami del DNA
In questo articolo
Riepilogo
Descriviamo il protocollo dettagliato per l'assemblaggio del DNA origami-basato di nanorod oro in metamolecules chirali plasmoniche con forte chiroottiche risposte. Il protocollo non è limitato alle configurazioni chirali e può essere facilmente adattato per la realizzazione di varie architetture plasmoniche.
Abstract
L'indirizzabilità inerente di strutture di DNA origami li rende modelli ideale per la disposizione delle nanoparticelle metalliche in complesse Nanostrutture plasmoniche. L'alta precisione spaziale di un assembly basato su modelli origami del DNA permette di controllare l'accoppiamento tra plasmoniche risonanze delle singole particelle e sartoria proprietà ottiche di nanostrutture costruito. Recentemente, sistemi chirali plasmoniche attirato molta attenzione a causa della stretta correlazione tra la configurazione spaziale delle assemblee plasmoniche e le loro risposte ottiche (ad es., dicroismo circolare [CD]). In questo protocollo, descriviamo l'intero flusso di lavoro per la generazione di DNA origami base chirali assemblee di nanorod oro (AuNRs). Il protocollo comprende una descrizione dettagliata dei principi di progettazione e procedure sperimentali per la realizzazione di modelli di origami del DNA, la sintesi di AuNRs e il montaggio di origami-AuNR strutture. Inoltre, la caratterizzazione delle strutture mediante microscopia elettronica a trasmissione (TEM) e spettroscopia CD è inclusa. Il protocollo descritto non è limitato alle configurazioni chirali e può essere adattato per la costruzione di varie architetture plasmoniche.
Introduzione
Nanostrutture di DNA, origami di DNA, in particolare, sono stati ampiamente usati per organizzare molecole e altri componenti su scala nanometrica (ad es., proteine e le nanoparticelle [NP]), con precisione nanometrica in geometrie quasi arbitrario1,2 , 3 , 4 , 5. la capacità di organizzare il metallo NPs sui modelli di origami del DNA con un alto rendimento e precisione consente la fabbricazione di strutture plasmoniche con proprietà ottiche romanzo6,7,8, 9 , 10. tecnica di origami di DNA è particolarmente utile per la generazione di strutture plasmoniche chirali, che richiedono architetture realmente tridimensionale11,12,13, 14 , 15 , 16 , 17 , 18 , 19 , 20.
Questo protocollo descrive in dettaglio l'intero processo di fabbricazione delle assemblee chirali basati su modelli origami di DNA di AuNRs. Il software utilizzato per il design21 e struttura Pronostico22,23 di origami di DNA è intuitiva e liberamente disponibile. La fabbricazione di origami e la sintesi di AuNR utilizzare comuni attrezzature di laboratorio di biochimica (ad es., thermocyclers, elettroforesi del gel, piastre, centrifughe). Le strutture sono caratterizzate tramite spettroscopia TEM e CD standard.
La fabbricazione di Nanostrutture plasmoniche simili con metodi top-down (ad es., Litografia a fascio di elettroni) richiederebbe piuttosto complicate e costose attrezzature. Inoltre, modelli di origami del DNA forniscono la possibilità di incorporare riconfigurabilità strutturali in assembly plasmoniche24,25,26,27,28,29 ,30,31,32,33, che è estremamente impegnativo per le strutture fabbricate con tecniche di litografia. Rispetto ad altri approcci molecolari basati34,35,36,37, fabbricazione di base di origami del DNA fornisce un alto livello di precisione spaziale e programmabilità.
Protocollo
1. progettazione del origami del DNA
- Identificare la desiderata relativa disposizione spaziale di AuNRs e la forma adatta del modello origami del DNA (Figura 1A). Stimare i parametri strutturali dell'AuNRs e i modelli di origami. Individuare le posizioni approssimative di staples che hanno bisogno di ulteriore modifica (Figura 1B).
- Scaricare e installare caDNAno18 per progettare un modello di origami del DNA. In caDNAno, instradare i fili dell'impalcatura e fiocco secondo la forma desiderata del modello e generare la sequenza di fili graffetta facendo clic Sullo strumento Seq. Fare clic Sullo strumento vernice e segnare i fili dei punti metallici che richiedono un'ulteriore modifica (Figura 1C).
- Fare clic su Strumento di esportazione per esportare le sequenze di DNA fiocco (Figura 1C) in un file CSV.
- Design double-stranded serrature per fissare l' angolo Θ tra i due fasci di origami. A seconda dell'orientamento relativo dei due fasci, il costrutto di origami possa adattare - o destri (LH/RH) chirali configurazione spaziale (Figura 1B).
- Importare file CSV il staples in un'applicazione di foglio di calcolo. Aggiungere una sequenza di10 di polyA all'estremità delle graffette utilizzati per l'assemblaggio di AuNR (maniglie). Modificare i fili graffetta sui siti serratura progettata con sequenze di blocco.
Nota: Gli assembly nei risultati rappresentativi contengono 36 maniglie sporgenti all'estremità 3' dei fili del fiocco, 18 su ogni pacco di origami di DNA, equamente distribuito sui due eliche in parallelo ogni 21 nt. La distanza tra la prima e l'ultima posizione di impugnatura è 168 nt, circa 57 nm (Vedi il file allegato caDNAno).
2. montaggio dei modelli di origami del DNA
- Preparare un brodo di lavoro di filamenti di poliesteri in fiocco (SM), compresi i fili con maniglie e serrature, mescolando uguali quantità di concentrazione-normalizzata fiocco oligonucleotidi (ad es., 100 µM).
Nota: Origami strutture contengono solitamente parecchie centinaia di filamenti di poliesteri in fiocco. Punti metallici sono in genere acquistati da fornitori specializzata nella sintesi chimica degli oligonucleotidi di DNA in pozzetti (ad es., 96 pozzetti) piastre. - Per 500 µ l di origami di 10 nM, mescolare 50 µ l di Tris-EDTA (TE, 10x), 100 µ l di MgCl2 (100 mM), 25 µ l di NaCl (100 mM), 175 µ l di H2O, 100 µ l di SM (0,5 µM), 5 µ l di fili di blocco (5 µM) e un'impalcatura di 50 µ l (100 nM).
- Tempri la miscela in un termociclatore da 80 ° C a 20 ° C come descritto nella tabella 1.
3. purificazione di origami del DNA
Nota: Questa sezione descrive il protocollo per la purificazione del gel dell'agarosi. Modelli di origami del DNA possono essere purificati anche utilizzando approcci alternativi38,39.
- Per il gel 1%, sciogliere 1 g di agarosio in 100 mL di Tris-Borato-EDTA (TBE, 0.5 x) riscaldando la miscela in un forno a microonde. Aggiungere 10 µ l di 10.000 x macchia di DNA in base alla specifica di macchia. Per ridurre al minimo l'esposizione a luce UV nella fase di estrazione (punto 3.6), utilizzare una macchia di DNA che possa essere visualizzata sotto eccitazione blu.
- Raffreddare la soluzione a circa 40 ° C e aggiungere lentamente 1 mL di MgCl2 (1.3 M) mentre si stringono. Cast gel e incubare per 30 min a temperatura ambiente.
- Impostare il dispositivo per elettroforesi e versare in esecuzione buffer freddo (4 ° C) (0,5 x TBE con 11 mM MgCl2) nella casella di gel. Mettere la vaschetta del gel in un bagno di acqua ghiacciata.
- Aggiungere tampone di caricamento ai campioni origami (buffer di caricamento 6 x contiene 15% polysucrose 400 e 0,25% bromofenolo in acqua). Caricare i campioni nei pozzetti con un volume adeguato secondo il pettine usato (ad es., 50 µ l per un pettine 8 pozzetti di 1,5 mm di spessore).
- Eseguire l'elettroforesi per 2 h a 80 V.
Nota: Per caratterizzare l'origami e separare la struttura aperta e chiusa, utilizzare gel 2% invece di 1% e prolungare il tempo di esecuzione a 4 h. - Immagine del gel con il gel imager (Figura 2). Utilizzare un transilluminatore luce blu per visualizzare le bande, tagliare la banda di origami, smash il gel su un parafilm ed estrarre il liquido. La resa di recupero è di circa il 40%.
- Dispensare il liquido in un'unità filtro centrifugo e spin a 3.000 x g per 5 min, misurare l'assorbimento della soluzione origami a 260 nm con uno spettrometro UV-visibile (UV-VIS). Stimare la concentrazione di origami utilizzando un coefficiente di estinzione di 1,3 x 108 M-1ma-1.
Nota: La concentrazione tipica di origami soluzione dopo purificazione del gel dell'agarosi è 1-2 nM. - Archiviare i modelli di origami purificata a 4 ° C per un uso successivo.
4. sintesi di nanorod oro
Nota: Il protocollo per la sintesi di AuNR è adattato da letteratura precedente40 con modifiche minori.
- Lavare tutti i bicchieri con acqua regia per 5 min, sciacquare con acqua, e trattare con ultrasuoni con acqua ultrapura e asciugare prima dell'uso.
- Preparare 0,2 M esadeciltrimetilammonio bromuro (CTAB), 1 mM HAuCl4, 4mm AgNO3, 64 mM L (+)-acido ascorbico e 6mm NaBH4. Usare acqua fredda (4 ° C) per sciogliere NaBH4 e tenerlo in frigorifero a 4 ° C. Acido ascorbico soluzione deve essere preparata.
Attenzione: CTAB è pericoloso in caso di contatto cutaneo (irritante), contatto con gli occhi (irritante), ingestione e inalazione. Usare indumenti protettivi adatti. In caso di ventilazione insufficiente, usare un apparecchio respiratorio adatto. NaBH4 è estremamente pericoloso in caso di contatto cutaneo (irritante), contatto con gli occhi (irritante), ingestione e inalazione. Indossare occhiali splash, un camice da laboratorio, guanti e un respiratore del vapore e polvere. Assicurarsi di utilizzare un respiratore approvato/certificato o equivalente. - Preparare i semi di Au.
- Aggiungere 500 µ l di CTAB (0,2 M), 250 µ l di acqua ultrapura e 250 µ l di HAuCl4 (1 mM) in un flaconcino di vetro. Mescolare a 450 giri/min a temperatura ambiente per 5 min.
- Aumentare il tasso di agitazione a 1.200 giri/min. Aggiungere 100 µ l di soluzione fredda di NaBH4 (6 mM, 4 ° C). Dopo 2 minuti, fermare l'agitazione e incubare la soluzione in un bagno di acqua a 30 ° C per 30 min prima dell'uso.
- Preparare AuNRs.
- Sciogliere 0,55 g di CTAB e 0,037 g di acido 2,6-dihydroxybenzoic in 15 mL di acqua calda (60-65 ° C) in un pallone. Raffreddare la soluzione a 30 ° C, aggiungere 600 µ l di AgNO3 (4 mM) e mescolare a 450 giri/min per 2 min. Quindi, lasciare la soluzione indisturbato per 15 min a 30 ° C.
- Aggiungere 15 mL di HAuCl4 (1 mM) alla soluzione e mescolare a 450 giri/min per 15 min aggiungere 120 µ l di L (+)-acido ascorbico (64 mM) e poi, immediatamente, mescolare a 1.200 giri/min per 30 µ l di aggiungere 12 s. di semi di Au e continuate a mescolare a 1.200 giri/min per 30 s.
- Incubare la soluzione in un bagno di acqua a 30 ° C per 18 h. Non disturba la soluzione e utilizzare un tappo per chiudere il pallone.
- Trasferire la soluzione risultante in provette da centrifuga e centrifugare a 9.500 x g per 12 min a 20 ° C. Eliminare il supernatante, disperdere il pellet in 20 mL di acqua ultrapura ed eseguire un altro passo di centrifugazione.
- Disperdere la pallina finale in 3 mL di acqua distillata. Stimare la concentrazione di AuNRs da una misurazione di assorbimento UV-VIS utilizzando il coefficiente di estinzione per la risonanza plasmonica longitudinale. Il coefficiente di estinzione può essere previsto utilizzando AuNR forma parametri41. Memorizzare la AuNRs a 4 ° C per un ulteriore uso.
5. funzionalizzazione di nanorod oro con DNA single-stranded
Nota: Questa sezione descrive il protocollo per la funzionalizzazione di AuNR con il DNA a singola elica (ssDNA), seguendo il percorso del cosiddetto basso pH adattato da precedente letteratura42. La AuNRs ricoperta di DNA sono purificati mediante centrifugazione; in alternativa, la purificazione può essere effettuata usando l'elettroforesi del gel dell'agarosi.
- Incubare 20 µ l di filamenti di DNA di tiolo-funzionalizzate polyT (1 mM) con 20 µ l di preparati tris(2-carboxyethyl) cloridrato di fosfina (TCEP, 14mm) per 1 h per ridurre i legami disolfuro.
Nota: Il modulo gruppi tiolo legami con AuNRs, e la sequenza di polyT ibridizza con il manico di10 polyA sul origami, in cui troppi o troppo pochi coppie di basi può causare un malfunzionamento o un assembly instabile.
Attenzione: TCEP può causare gravi ustioni cutanee e gravi lesioni oculari. Indossare guanti protettivi abbigliamento/proteggere gli occhi/viso. - Mix 150 µ l di AuNRs (10 nM) e 40 µ l di tiolo-DNA TCEP-trattato (0,5 mM). Aggiungere alla soluzione di AuNR per raggiungere una concentrazione finale di SDS di 0.05% 1% sodio dodecil solfato (SDS). Regolare il pH a 2,5-3 con ~ 1 µ l di HCl (1 M).
- Incubare per 2 h mentre si stringono alle 70 giri/min.
Nota: Il rapporto di AuNR-a-DNA dovrebbe essere nell'ordine di 1:5, 000-15.000, a seconda delle dimensioni delle aste. Per il AuNRs (70 nm, x 30 nm) preparato seguendo il protocollo descritto nella sezione 4, un eccesso di 13.000 del tiolo-DNA è raccomandato. - Aggiungere NaCl per raggiungere una concertazione di NaCl finale di 0,5 M e incubare per 4 h a temperatura ambiente mentre si stringono alle 70 giri/min.
Nota: Un cambiamento di colore in questa fase potrebbe indicare una funzionalizzazione di DNA non riuscita. - Regolare il pH a ~8.5 con tampone TBE (10x) e incubare per una notte.
- Lavare il DNA-AuNRs 4 x mescolando i campioni con 1 mL di tampone di lavaggio (0,5 x TBE con 0,1% SDS) e centrifugare a 7.000 x g per 30 min. eliminare il surnatante e risospendere il DNA-AuNRs nel liquido restante (~ 40 µ l). Stimare la concentrazione di DNA-AuNRs da una misurazione di assorbimento UV-VIS come descritto nel passaggio 4.4.5.
Nota: La soluzione potrebbe diventare un po' 'nuvolosa' a passi 5.3-5.4 la sostituzione CTAB dalla superficie dell'AuNRs dal tiolo-DNA. La soluzione dovrebbe essere chiara al momento fino a ~ 35 ° c per 5 min.
6. montaggio del nanorod oro sui modelli di origami del DNA
- Aggiungere MgCl2 alla soluzione di DNA purificato-AuNRs, a una concentrazione finale di 10 mM. Mescolare il purificato del DNA-AuNRs e origami per un rapporto 10:1.
Nota: Un rapporto inferiore può ridurre il rendimento di prodotto43. - Tempri la miscela in un mixer con un controllo di temperatura da 40 ° C a 20 ° C mentre si stringono a 400 giri/min, la procedura descritta nella tabella 2.
Nota: Per la caratterizzazione di CD, il campione può essere misurato dopo questo passaggio senza ulteriore purificazione. - Utilizzare l'elettroforesi del gel dell'agarosi per 0,7% (3,5 h a 80 V) per purificare le strutture di origami-AuNR finale.
- Utilizzare un transilluminatore luce bianco per l'imaging. Tagliare la banda di prodotto (dimero origami-AuNR) (Figura 3), smash il gel su un parafilm ed estrarre il liquido. Pipetta il liquido in un'unità filtro centrifugo e spin a 3.000 x g per 5 min. Risospendere il origami-AuNRs nella soluzione. Il rendimento di recupero dal gel è di circa il 50%.
- Stimare la concentrazione delle strutture origami-AuNR da una misurazione di assorbimento UV-VIS come descritto nel passaggio 4.4.5.
7. formazione immagine di microscopia elettronica di trasmissione
Nota: Questa formiato di uranile (UFo) protocollo di colorazione è adattato da precedente letteratura44.
- Mix 200 µ l di soluzione di UFo (0,75%) e 1 µ l di NaOH (5 M) e vortex immediatamente per 2-3 min. Centrifugare la soluzione macchia per 3-4 min a 14.000 x g. Proteggere la macchia da esposizione alla luce (ad es., avvolgendolo nella carta stagnola).
Attenzione: UFo è tossico se inalato o ingerito e può causare irritazione agli occhi. Nel caso di esposizioni brevi e minime, utilizzare un dispositivo di filtro respiratorio. Nel caso di esposizioni più intense e, utilizzare un apparecchio di protezione respiratoria self-contained. Indossare i guanti. Il materiale dei guanti deve essere impermeabile e resistente agli UFo e le sue soluzioni. Indossare occhiali, sigillati ermeticamente. - Effluvio rivestita di carbonio/formvar griglie TEM per 6 s appena prima dell'uso per aumentare l'idrofilia e promuovere l'incollaggio delle strutture. Pipettare 5 µ l del campione gocce sulla griglia TEM, incubare per 5-8 minuti e rimuovere la goccia toccando delicatamente una carta da filtro con il bordo della griglia.
- Pipettare uno grande (~ 20 µ l) e uno piccolo (~ 10 µ l) goccia della soluzione macchia su un parafilm. Mettere la griglia sul drop soluzione piccola macchia e asciugare immediatamente toccando la carta da filtro con il bordo della griglia. Poi, messo sulla goccia di soluzione di grande macchia per 30 s.
- Rimuovere il liquido sulla griglia toccando la carta da filtro con il bordo della griglia. Posizionare la griglia nel supporto griglia. Attendere che la griglia ad asciugare per almeno 10 min.
- Caratterizzare i campioni di origami (Figura 4), AuNRs (Figura 5) e origami-AuNRs (Figura 6) di TEM.
8. misurazione di dicroismo circolare
- Eliminazione dei fogli inceppati dello spettrometro di CD con N2 per 20 min.
Nota: La maggior parte degli spettrometri CD richiedono l'eliminazione con N2 prima accensione della lampada. Consultare il manuale di spettrometro di CD. - Impostare la larghezza di banda, intervallo di scansione e acquisizione step.
Nota: L'intervallo di scansione dipende dalla proprietà ottiche di AuNRs, che dipendono dalle dimensioni dell'AuNRs. - Misura CD vuoto con buffer.
- Misurare gli spettri CD di origami-AuNR campioni (Figura 7).
Nota: Utilizzare quarti o le provette di vetro per la misura di CD. Cuvette in plastica non sono adatti per la spettroscopia CD. Inoltre, la maggior parte CD spettrometri consentono l'acquisizione simultanea di assorbimento e CD dati.
Risultati
Immagini TEM di assembly finale origami-AuNR, AuNRs e modelli di origami del DNA sono indicate in Figura 4, Figura 5e Figura 6A, rispettivamente. A causa della loro preferenza di associazione alle griglie TEM, origami-AuNR assembly sono solitamente visti come fasci paralleli origami e coni retinici (Figura 6A). Ricottura termica è necessaria per il corretto allineamento di AuNRs sui modelli di origami (Figura 6A, B). Il protocollo consente alti rendimenti dell'Assemblea del AuNRs in metamolecules chirali con forti plasmoniche CD risposte (Figura 7).
| Temperatura (° C) | Tempo |
| 80 | 15 min |
| 79 - 71 | 1 ° C/1 min |
| 70 - 66 | 1 ° C/5 min |
| 65 - 60 | 1 ° C/30 min |
| 59 - 37 | 1 ° C/60 min |
| 36 - 30 | 1 ° C/15 min |
| 29 - 20 | 1 ° C/5 min |
| 20 | Tenere premuto |
Tabella 1: Le temperature e le tariffe per la ricottura termica di modelli di origami del DNA.
| Temperatura (° C) | Tempo (min) |
| 40 | 130 |
| 36 | 180 |
| 32 | 180 |
| 22 | Tenere premuto |
Tabella 2: temperature e tempi di permanenza per la ricottura dei modelli di origami del DNA e AuNRs. Il tasso di raffreddamento tra i passaggi è impostato a 0,1 ° C/min. I campioni di DNA origami-AuNR vengono ricotti mentre si stringono a 400 giri/min.

Figura 1 : Design di DNA basato su modelli origami chirali metamolecules. (A) identificare la disposizione spaziale relativa desiderata di nanorod oro (AuNRs) e una forma adatta del modello origami del DNA. (B) stimare i parametri strutturali di AuNRs (DAuNR, LAuNR) e il modello di origami (Worigami, Lorigami, Θ). Individuare le posizioni approssimative delle graffette che hanno bisogno di ulteriore modifica. (C) progettazione di modelli di origami del DNA utilizzando caDNAno. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2 : L'elettroforesi su gel di agarosio di origami. (A) purificazione con elettroforesi in gel d'agarosio 1% per 2 ore a 80 v (B) caratterizzazione con elettroforesi su gel di agarosio al 2% per 4 h a 80 V. per favore clicca qui per visualizzare una versione più grande di questa figura.
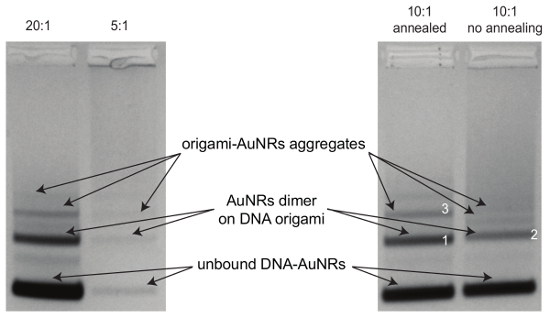
Figura 3 : La purificazione di elettroforesi del gel dell'agarosi di origami-AuNRs. Gel (0,7%) è stato eseguito per 3,5 h a 80 V per i campioni preparati seguendo la procedura di assemblaggio con differenti rapporti di origami di DNA AuNR (20:1, 5:1) e campioni (rapporto 10:1-AuNRs-a-origami di DNA) con o senza procedura di ricottura. Per le immagini TEM dei campioni in bande di 1, 2 e 3, vedere la Figura 6. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4 : Immagine TEM di rappresentante dei modelli di origami del DNA. La struttura di origami è costituito da due fasci 14-elica (80 nm, x 16 nm, x 8 nm) collegati tra loro dal filo dell'impalcatura. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5 : Immagine TEM di rappresentante della AuNRs. Delle dimensioni medie di AuNRs sintetizzato sono 70 x 30 nm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6 : Immagini TEM di origami-AuNR assembly. (A) AuNR dimeri sull'origami dopo ricottura (banda 1 nella Figura 3). (B) AuNR dimeri sull'origami senza ricottura (band 2 in Figura 3). (C) Origami-AuNR aggrega (banda 3 nella Figura 3). Clicca qui per visualizzare una versione più grande di questa figura.
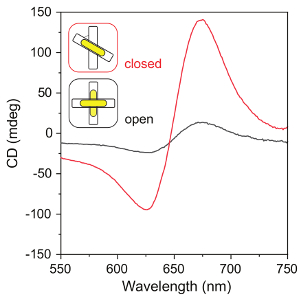
Figura 7 : Spettri CD degli assembly origami-AuNR. Gli spettri CD di strutture chiuse (i modelli di origami fissati dai fili di blocco in una configurazione di mano destra, con il 50° tra due fasci di origami) e la struttura aperta (i modelli di origami senza fili di blocco). Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Il protocollo introduce l'intero flusso di lavoro di progettazione, assemblaggio, purificazione e caratterizzazione di DNA origami base chirali assembly di AuNRs. I modelli di origami di DNA utilizzati nel protocollo sono particolarmente adatti per la fabbricazione degli assembly stimoli-sensible a reagire. Vari tipi di risposte e functionalizes può essere incorporato nei filamenti serratura che definiscono lo stato chirale di origami modello (Figura 1B)24,25,26,31. Per gli assembly statici, modelli a forma di blocco più semplici sono spesso sufficienti14,45,46,47.
L'approccio di base di origami di DNA per la fabbricazione di Nanostrutture plasmoniche eredita limitazioni del DNA origami tecnica48. La dimensione dei modelli origami è in genere limitata dalle dimensioni del filo del patibolo. La stabilità delle strutture di DNA è ridotta in condizioni di legge-sale. Il costo di fiocco sintetico fili rimane piuttosto elevato. Tuttavia, recenti sviluppi nel campo della nanotecnologia del DNA strutturale si prevede di superare queste limitazioni49,50,51,52,53,54 , 55.
Rispetto ad altri approcci molecolari basati per la generazione di assembly chirali di AuNRs34,35,36,37, origami di DNA fornisce un alto livello di precisione spaziale e programmabilità.
Per ottenere risposte ottiche affidabile e riproducibile di assembly chirali, si consiglia di adattare i protocolli per AuNR sintesi40, poiché la qualità e le proprietà ottiche dei prodotti commerciali possono variare tra i batch. Ulteriore ricottura (punto 6.2) sono spesso fondamentale per garantire il fissaggio corretto di AuNRs ai modelli di origami del DNA (Figura 6).
Infine, il protocollo qui descritto non è limitato agli assembly chirali. Origami di DNA fornisce una piattaforma molto flessibile per la fabbricazione di Nanostrutture plasmoniche complesso9,10.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori ringraziano S. Voutilainen per la sua assistenza con spettrometro di CD. Gli autori riconoscono la fornitura di servizi e supporto tecnico di Aalto University presso OtaNano - Nanomicroscopy Center (Aalto-NMC). Questo lavoro è stato supportato dall'Accademia di Finlandia (grant 308992) e programma ricerca e innovazione Orizzonte 2020 dell'Unione europea sotto il Marie Skłodowska-Curie di sovvenzione n. 71364.
Materiali
| Name | Company | Catalog Number | Comments |
| 2,6-Dihydroxybenzoic acid | Sigma-Aldrich | D109606-25 | 98+% |
| AgNO3 | Alfa Aesar | AA1141414 | 99.90% |
| Blue light transilluminator | Nippon Genetics | FG-06 | FastGene LED Transilluminator |
| Bromophenol Blue | Acros Organics | 403160050 | For agarorose gel loading buffer |
| Centrifugal filter units | Merck Millipore | 42600 | DNA extraction from agarose |
| Chirascan CD spectrometer | Applied Photophysics | ||
| Cuvette | Hellma | 105-202-85-40 | Quartz SUPRASIL |
| DNA lobind tubes | Eppendorf | 30108051 | |
| Eppendorf Biospectrometer | Eppendorf | 6135000904 | |
| Eppendorf ThermoMixer C | Eppendorf | 5382000015 | |
| Ficoll 400 | Thermo Fisher Scientific | BP525-10 | Polysucrose 400 (For agarorose gel loading buffer) |
| Gel electrophoresis sets | Thermo Fisher Scientific | ||
| Gel imager | Bio-Rad | Gel Doc XR+ System | |
| HAuCl4•3H2O | Alfa Aesar | AA3640006 | 99.99% |
| HCl | Scharlau | AC07441000 | 1M |
| Hexadecyltrimethylammonium bromide (CTAB) | Sigma-Aldrich | H9151-100 | BioXtra, 98+% |
| L(+)-ascorbic acid | Acros Organics | 401471000 | 99+% |
| M13p7560 scaffold strand | Tilibit nanosystems | ||
| MgCl2•6H2O | Sigma-Aldrich | M2670-500 | BioXtra, 99+% |
| NaBH4 | Acros Organics | 200050250 | 99% |
| NaCl | Sigma-Aldrich | S7653-500 | BioXtra, 99.5+% |
| NaOH | Sigma-Aldrich | S8045-500 | BioXtra, 98+% |
| Parafilm | Sigma-Aldrich | P7668-1EA | PARAFILM M |
| PBS buffer (10X) | Thermo Fisher Scientific | BP3991 | Molecular Biology |
| ProFlex PCR System | Thermo Fisher Scientific | 4484073 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | 74255-250 | 99+% |
| Staple strands | Thermo Fisher Scientific | ||
| Sybr Safe | Invitrogen | S33102 | For DNA stain |
| TBE buffer (10X) | Invitrogen | 15581-044 | Molecular Biology |
| TE buffer (10X) | Thermo Fisher Scientific | BP24771 | Molecular Biology |
| TEM | FEI | FEI Tecnai F12 | |
| Thiol-functionalized ssDNA | Biomers.net | ||
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP-HCl) | Thermo Fisher Scientific | PI20491 | |
| UltraPure Agarose | Invitrogen | 16500-100 | |
| Ultrapure water (Type 1) | Milli-Q Direct 8 system | ||
| Uranyl Formate | Tebu-bio | 24762-1 | |
| White light transilluminator | UVP | TW-26 |
Riferimenti
- Rothemund, P. W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 440, 297-302 (2006).
- Wang, P., Meyer, T. A., Pan, V., Dutta, P. K., Ke, Y. The Beauty and Utility of DNA Origami. Chem. 2, 359-382 (2017).
- Hong, F., Zhang, F., Liu, Y., Yan, H. DNA Origami: Scaffolds for Creating Higher Order Structures. Chemical Reviews. 117, 12584-12640 (2017).
- Roller, E. -M., Argyropoulos, C., Högele, A., Liedl, T., Pilo-Pais, M. Plasmon-Exciton Coupling Using DNA Templates. Nano Letters. 16, 5962-5966 (2016).
- Acuna, G. P., et al. Fluorescence Enhancement at Docking Sites of DNA-Directed Self-Assembled Nanoantennas. Science. 338, 506-510 (2012).
- Roller, E. -M., et al. DNA-Assembled Nanoparticle Rings Exhibit Electric and Magnetic Resonances at Visible Frequencies. Nano Letters. 15, 1368-1373 (2015).
- Liu, Q., Song, C., Wang, Z. -G., Li, N., Ding, B. Precise organization of metal nanoparticles on DNA origami template. Methods. 67, 205-214 (2014).
- Roller, E. -M., et al. Hotspot-mediated non-dissipative and ultrafast plasmon passage. Nature Physics. 13, 761-765 (2017).
- Liu, N., Liedl, T. DNA-Assembled Advanced Plasmonic Architectures. Chemical Reviews. 118, 3032-3053 (2018).
- Kuzyk, A., Jungmann, R., Acuna, G. P., Liu, N. DNA Origami Route for Nanophotonics. ACS Photonics. 5, 1151-1163 (2018).
- Kuzyk, A., et al. DNA-based self-assembly of chiral plasmonic nanostructures with tailored optical response. Nature. 483, 311-314 (2012).
- Shen, X., et al. Three-Dimensional Plasmonic Chiral Tetramers Assembled by DNA Origami. Nano Letters. 13, 2128-2133 (2013).
- Liu, H., Shen, X., Wang, Z. -G., Kuzyk, A., Ding, B. Helical nanostructures based on DNA self-assembly. Nanoscale. 6, 9331-9338 (2014).
- Shen, X., et al. 3D plasmonic chiral colloids. Nanoscale. 6, 2077-2081 (2014).
- Urban, M. J., et al. Plasmonic Toroidal Metamolecules Assembled by DNA Origami. Journal of the American Chemical Society. 138, 5495-5498 (2016).
- Hentschel, M., Schäferling, M., Duan, X., Giessen, H., Liu, N. Chiral plasmonics. Science Advances. 3, 1602735(2017).
- Cecconello, A., Besteiro, L. V., Govorov, A. O., Willner, I. Chiroplasmonic DNA-based nanostructures. Nature Reviews Materials. 2, 17039(2017).
- Lan, X., Wang, Q. Self-Assembly of Chiral Plasmonic Nanostructures. Advanced Materials. 28, 10499-10507 (2016).
- Shen, C., et al. Spiral Patterning of Au Nanoparticles on Au Nanorod Surface to Form Chiral AuNR@AuNP Helical Superstructures Templated by DNA Origami. Advanced Materials. 29, 1606533(2017).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature Communications. 6, 8102(2015).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37, 5001-5006 (2009).
- Kim, D. -N., Kilchherr, F., Dietz, H., Bathe, M. Quantitative prediction of 3D solution shape and flexibility of nucleic acid nanostructures. Nucleic Acids Research. 40, 2862-2868 (2012).
- Maffeo, C., Yoo, J., Aksimentiev, A. De novo reconstruction of DNA origami structures through atomistic molecular dynamics simulation. Nucleic Acids Research. 44, 3013-3019 (2016).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13, 862-866 (2014).
- Kuzyk, A., et al. A light-driven three-dimensional plasmonic nanosystem that translates molecular motion into reversible chiroptical function. Nature Communications. 7, 10591(2016).
- Kuzyk, A., Urban, M. J., Idili, A., Ricci, F., Liu, N. Selective control of reconfigurable chiral plasmonic metamolecules. Science Advances. 3, 1602803(2017).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347, 1446-1452 (2015).
- Zhou, C., Duan, X., Liu, N. DNA-Nanotechnology-Enabled Chiral Plasmonics: From Static to Dynamic. Accounts of Chemical Research. 50, 2906-2914 (2017).
- Ijäs, H., et al. Dynamic DNA Origami Devices: from Strand-Displacement Reactions to External-Stimuli Responsive Systems. International Journal of Molecular Sciences. 19, 2114(2018).
- Lan, X., et al. DNA-Guided Plasmonic Helix with Switchable Chirality. Journal of the American Chemical Society. 140, 11763-11770 (2018).
- Funck, T., Nicoli, F., Kuzyk, A., Liedl, T. Sensing Picomolar Concentrations of RNA Using Switchable Plasmonic Chirality. Angewandte Chemie International Edition. 57, 13495-13498 (2018).
- Zhou, C., Xin, L., Duan, X., Urban, M. J., Liu, N. Dynamic Plasmonic System That Responds to Thermal and Aptamer-Target Regulations. Nano Letters. 18, 7395-7399 (2018).
- Zhan, P., et al. Reconfigurable Three-Dimensional Gold Nanorod Plasmonic Nanostructures Organized on DNA Origami Tripod. ACS Nano. 11, 1172-1179 (2017).
- Ma, W., et al. Attomolar DNA detection with chiral nanorod assemblies. Nature Communications. 4, 2689(2013).
- Ma, W., et al. Chiral plasmonics of self-assembled nanorod dimers. Scientific Reports. 3, 1934(2013).
- Guerrero-Martínez, A., et al. Intense Optical Activity from Three-Dimensional Chiral Ordering of Plasmonic Nanoantennas. Angewandte Chemie International Edition. 50, 5499-5503 (2011).
- Kumar, J., et al. Detection of amyloid fibrils in Parkinson's disease using plasmonic chirality. Proceedings of the National Academy of Sciences. 115, 3225-3230 (2018).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53, 12735-12740 (2014).
- Shaw, A., Benson, E., Högberg, B. Purification of Functionalized DNA Origami Nanostructures. ACS Nano. 9, 4968-4975 (2015).
- Ye, X., et al. Improved Size-Tunable Synthesis of Monodisperse Gold Nanorods through the Use of Aromatic Additives. ACS Nano. 6, 2804-2817 (2012).
- Near, R. D., Hayden, S. C., Hunter, R. E., Thackston, D., El-Sayed, M. A. Rapid and Efficient Prediction of Optical Extinction Coefficients for Gold Nanospheres and Gold Nanorods. The Journal of Physical Chemistry C. 117, 23950-23955 (2013).
- Shi, D., Song, C., Jiang, Q., Wang, Z. -G., Ding, B. A facile and efficient method to modify gold nanorods with thiolated DNA at a low pH value. Chemical Communications. 49, 2533-2535 (2013).
- Gür, F. N., Schwarz, F. W., Ye, J., Diez, S., Schmidt, T. L. Toward Self-Assembled Plasmonic Devices: High-Yield Arrangement of Gold Nanoparticles on DNA Origami Templates. ACS Nano. 10, 5374-5382 (2016).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature Methods. 8, 221-229 (2011).
- Lan, X., et al. Bifacial DNA Origami-Directed Discrete, Three-Dimensional, Anisotropic Plasmonic Nanoarchitectures with Tailored Optical Chirality. Journal of the American Chemical Society. 135, 11441-11444 (2013).
- Lan, X., et al. Au Nanorod Helical Superstructures with Designed Chirality. Journal of the American Chemical Society. 137, 457-462 (2015).
- Zhu, C., Wang, M., Dong, J., Zhou, C., Wang, Q. Modular Assembly of Plasmonic Nanoparticles Assisted by DNA Origami. Langmuir. , (2018).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature Nanotechnology. 6, 763-772 (2011).
- Ducani, C., Kaul, C., Moche, M., Shih, W. M., Högberg, B. Enzymatic production of 'monoclonal stoichiometric' single-stranded DNA oligonucleotides. Nature Methods. 10, 647-652 (2013).
- Praetorius, F., et al. Biotechnological mass production of DNA origami. Nature. 552, 84-87 (2017).
- Wagenbauer, K. F., Sigl, C., Dietz, H. Gigadalton-scale shape-programmable DNA assemblies. Nature. 552, 78-83 (2017).
- Zhang, T., et al. 3D DNA Origami Crystals. Advanced Materials. 30, 1800273(2018).
- Ong, L. L., et al. Programmable self-assembly of three-dimensional nanostructures from 10,000 unique components. Nature. 552, 72-77 (2017).
- Ponnuswamy, N., et al. Oligolysine-based coating protects DNA nanostructures from low-salt denaturation and nuclease degradation. Nature Communications. 8, 15654(2017).
- Agarwal, N. P., Matthies, M., Gür, F. N., Osada, K., Schmidt, T. L. Block Copolymer Micellization as a Protection Strategy for DNA Origami. Angewandte Chemie International Edition. 56, 5460-5464 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon