Method Article
Studi mirati utilizzando la faccia a blocchi seriali e la microscopia elettronica a scansione a fascio ionico focalizzato
In questo articolo
Riepilogo
Qui presentiamo un protocollo per combinare in modo efficiente la faccia a blocchi seriali e la microscopia elettronica a scansione del fascio ionico focalizzato per indirizzare un'area di interesse. Ciò consente una ricerca efficiente, in tre dimensioni e l'individuazione di eventi rari in un ampio campo visivo.
Abstract
Questo protocollo consente l'imaging efficiente ed efficace di campioni di cellule o tessuti in tre dimensioni a livello di risoluzione della microscopia elettronica. Per molti anni la microscopia elettronica (EM) è rimasta una tecnica intrinsecamente bidimensionale. Con l'avvento delle tecniche di imaging al microscopio elettronico a scansione seriale (volume EM), utilizzando un microtoma integrato o un fascio ionico focalizzato per tagliare e poi visualizzare i tessuti incorporati, la terza dimensione diventa facilmente accessibile. La microscopia elettronica a scansione facciaa a blocchi seriali (SBF-SEM) utilizza un ultramicrotoma racchiuso nella camera SEM. Ha la capacità di gestire campioni di grandi dimensioni (1.000 m x 1.000 m) e di visualizzare grandi campi visivi a piccole dimensioni in pixel X,Y, ma è limitato nella dimensione s' dal coltello a diamante. Il fascio di ioni focalizzato SEM (FIB-SEM) non è limitato nella risoluzione 3D (sono raggiungibili i voxel isotropici di 5 nm), ma il campo visivo è molto più limitato. Questo protocollo dimostra un flusso di lavoro per combinare le due tecniche per consentire di trovare singole aree di interesse (ROI) in un grande campo e quindi l'imaging del volume mirato successivo ad alta risoluzione voxel isotropica. La preparazione di celle o tessuti fissi è più impegnativa per le tecniche EM del volume a causa del contrasto supplementare necessario per una generazione efficiente del segnale nell'imaging SEM. Tali protocolli richiedono molto tempo e richiedono molto lavoro. Questo protocollo incorpora anche l'elaborazione dei tessuti assistita da microonde che facilitano la penetrazione dei reagenti, il che riduce il tempo necessario per il protocollo di elaborazione da giorni a ore.
Introduzione
Questo protocollo descrive un flusso di lavoro per il targeting efficiente della microscopia elettronica tridimensionale (EM) ad alta risoluzione in una specifica regione di interesse (ROI). Fin dai suoi inizi negli anni '30, EM è stata una tecnica essenzialmente bidimensionale. Le prime immagini pubblicate furono di interi tessuti o cellule, ma che presto lasciarono il posto a sezioni che venivano tagliate a mano usando un ultramicrotome e immagini utilizzando un microscopio elettronico a trasmissione (TEM). TEM produce micrografie ad altissima risoluzione in cui anche la più piccola delle strutture cellulari è chiaramente distinguibile. Tuttavia, la magrezza della sezione necessaria per l'immagine del tessuto da parte del fascio di elettroni ha reso minima la dimensione della z. Poiché le celle sono strutture tridimensionali, le interazioni tra strutture cellulari e superfici cellulari dovevano essere dedotte da dati limitati. Ciò ha aumentato il potenziale di errata interpretazione, soprattutto nelle strutture complesse. Alcuni microscopisti sono riusciti a ottenere strutture 3D più accurate mediante cellule e tessuti a sezione seriale e poi ricostruirli scrupolosamente da singole immagini TEM1. Questo è stato un processo molto laborioso e prima dell'avvento dell'imaging digitale e del rendering al computer i risultati erano difficili da visualizzare. Negli ultimi anni sono state introdotte due tecniche che sono diventate collettivamente note come microscopia elettronica a volume (volume EM)2 che hanno reso EM in tre dimensioni accessibili a più laboratori.
L'idea di ottenere una pila di immagini da un blocco incorporato all'interno di un microscopio elettronico può essere fatta risalire al 1981, quando Steve Leighton e Alan Kuzirian costruirono un microtoma in miniatura e lo collocarono nella camera di un microscopio elettronico a scansione3 (SBF-SEM) . Questo prototipo fu infine copiato e migliorato 23 anni dopo da Denk e Horstmann4 e successivamente commercializzato. Più o meno nello stesso periodo gli scienziati biologici si sono resi conto di un'altra tecnologia utilizzata principalmente nella scienza dei materiali, il fascio ionico focalizzato. Questa tecnica utilizza un fascio ionico di qualche tipo (Gallium, plasma) per rimuovere una quantità molto piccola di materiale di superficie da un campione (FIB-SEM)5. Entrambe le tecniche impiegano il sezionamento seguito da imaging fornendo una serie di immagini che possono essere combinati in una pila X, Y, z. Entrambe le tecniche forniscono informazioni 3D, ma su scale di risoluzione diverse. SBF-SEM è limitato dalle proprietà fisiche del coltello diamante a fette non più sottili di 50 nm per lunghe corse di imaging seriale; tuttavia la dimensione del blocco campione che può essere sezionato è grande, fino a 1 mm x 1 mm x 1 mm. A causa del grande formato di acquisizione digitale del rilevatore di elettroni sparsi posteriori (32k x 32k pixel) che riceve un segnale dalla faccia del blocco , le dimensioni dei pixel dell'immagine possono essere di dimensioni fino a 1 nm. Questo si traduce in voxel non isotropici in cui la dimensione X,Y è spesso più piccola della . A causa della precisione del fascio ionico, FIB-SEM ha la capacità di raccogliere immagini con voxel isotropici . Tuttavia, l'area totale che può essere immagine è piuttosto piccola. In precedenza è stata pubblicataunatabella riassuntiva di vari campioni e volumi immagine con le due tecniche.
La preparazione dei tessuti per il volume EM è più difficile che per TEM o SEM standard perché i campioni devono essere macchiati per fornire un'adeguata generazione di segnale nel SEM. Spesso, le macchie devono essere ottimizzate non solo per il particolare tipo di tessuto, ma anche per l'aggiunta di in contrasto con alcune strutture cellulari per facilitare l'identificazione e la ricostruzione. Il protocollo qui utilizzato si basa sullo standard NCMIR6. La colorazione aggiuntiva di solito significa ulteriori passaggi del protocollo. Pertanto, per il volume EM, è necessario estendere i protocolli standard per garantire il tempo sufficiente per i reagenti a penetrare nel campione. L'elaborazione assistita a microonde può ridurre il tempo necessario per la colorazione da ore a minuti e rende più efficiente la preparazione del campione EM del volume7. Questo metodo è applicabile a tutti i tipi di cellule e tessuti8 e alle domande di ricerca in cui l'inomogeneità del tessuto rende essenziale il campionamento di un'area specifica9.
Una volta ottenuto, uno stack di dati può essere allineato e le strutture di interesse segmentate dal resto dei dati e modellate in 3D. Anche se l'automazione dell'imaging di molte fette di tessuto ha reso l'acquisizione delle immagini relativamente semplice, il processo di ricostruzione e visualizzazione digitale dei dati è un'attività che richiede molto tempo. Il software a questo scopo non è ancora integrato né completamente automatizzato. Poiché gran parte dei primi lavori di utilizzo del volume EM è stato diretto verso le neuroscienze, le tecniche per colorare e segmentare digitalmente strutture come gli assoni sono abbastanza avanzate rispetto ad altre cellule e organelli. Mentre la letteratura su altri tessuti non neuronali sta crescendo rapidamente, le strutture non lineari o irregolari richiedono un input più manuale.
L'utilizzo di SBF-SEM e FIB-SEM è un approccio utile per il targeting e l'imaging di strutture tissutali specifiche, non omogenee ad alta risoluzione in 3D. Combinandoquesto con l'elaborazione dei tessuti assistita a microonde che riduce notevolmente il tempo necessario per la preparazione del campione. Insieme questo flusso di lavoro renderà la generazione di set di dati di immagini voxel isotropiche ad alta risoluzione di strutture fini un processo efficiente e più rapido.
Protocollo
1. Fissazione e lavorazione del campione per la microscopia elettronica
- Piantide fisso di Arabidopsis thaliana coltivati su piastre di agar in 0.5% paraformaldeide, 2.5% glutaraldeide in 0.1 M fosfato buffer (PB) pH 6.8 per 2 h a temperatura ambiente (RT).
INFORMATIVA: Le aldeidi sono irritanti e corrosive e hanno un potenziale cancerogeno, mutageno e teratogenico. Tutte le soluzioni devono essere maneggiate con attrezzature di protezione adeguate e in una cappa di fumi. - Tagliare le punte delle radici della pianta coltivata al punto 1.1 e mettere 2-3 punte in tubi da 0,5 mL contenenti lo stesso fissativo durante la notte a 4 gradi centigradi.
NOTA: il volume di questa e di tutte le soluzioni nei passaggi rimanenti è determinato dal volume campione; un rapporto minimo tra campione e soluzione è 10:1. I campioni più grandi di 1 mm in qualsiasi dimensione sarà difficile da macchiare, quindi lavorare con blocchi di tessuto più grandi è più difficile. Non tutti i tessuti hanno le stesse caratteristiche; ad esempio, le foglie e gli steli delle piante possono essere difficili da macchiare. Se si desiderano campioni più grandi o tipi di tessuto difficili, è necessario eseguire l'ottimizzazione dell'elaborazione dei campioni prima di passare alla raccolta dei dati. - Preparare la soluzione thiocarbohydrazide (TCH), necessaria fresca e disponibile prima del passaggio 1.7. Aggiungere 0,1 g di tiocarbohydrazide a 10 mL di acqua a doppia distillazione (ddH2O) e sciogliere riscaldando a 60 gradi centigradi in forno per 1 h. Prima dell'uso, filtrare la soluzione TCH utilizzando un filtro di siringa da 0,22 m.
- Rimuovere il fissativo dai tubi e sostituirlo con 0,1 M PB pH 6.8. Mettere i tubi su un tavolo orbitale agitazione a 100 giri e lavare per 10 min. Ripetere il lavaggio utilizzando PBS fresco 5 volte.
- Post fix the root tips placing PB con 2% osmio tetroxide (OsO4) e 0.2% rosso di tonnio in 0.1 M PB pH 6.8. Mettere i tubi nel microonde con i coperchi aperti e avviare il programma 9 (Tabella 1).
AVVISO: l'Osmium è estremamente pericoloso in caso di ingestione, molto pericoloso in caso di inalazione e pericoloso in caso di contatto con la pelle. Maneggiare sempre utilizzando dispositivi di protezione appropriati e in una cappa di fumi.
NOTA: Durante il protocollo, i coperchi dei tubi sono sempre aperti durante i passaggi a microonde. - Lavare le punte della radice due volte con ddH2O per 5 min ciascuno sul banco. Per il terzo e il quarto ddH2O lavaggio utilizzare il programma 15 sul microonde (Tabella 1). Dopo il primo lavaggio ddH2O di 40 secondi, estrarre i campioni dal microonde e sostituire il buffer con ddH2O. Mettere i campioni nel microonde e continuare il programma.
NOTA: Il forno a microonde suonerà un allarme quando è necessario aggiornare il buffer. Assicurarsi che il coperchio della camera a vuoto venga sostituito correttamente ogni volta. - Incubare campioni in una soluzione TCH precedentemente preparata a RT per 2 min in panchina e per ulteriori incubazioni utilizzare il programma a microonde 8 (Tabella 1). Non cambiare soluzione tra panca e microonde.
- Lavare i campioni come descritto al punto 1.6.
- Collocare i campioni nell'1% di OsO4 in ddH2O per il programma microonde 9 (Tabella 1).
- Lavare i campioni come descritto al punto 1.6.
- Incubare campioni in acetato di uranilo dell'1% in ddH2O utilizzando il programma a microonde 16 (Tabella 1).
AVVISO: L'acetato di uranyl è tossico, irritante e ha un potenziale cancerogeno, mutageno e teratogenico. Maneggiare sempre con attrezzature di protezione adeguate. - Lavare i campioni come descritto al punto 1.6.
- Preparare la soluzione principale di Walton per l'uso nel passaggio 1.14. In primo luogo fare una soluzione di stock di acido L-aspartic aggiungendo 0.998 g di acido L-aspartic a 250 mL di ddH2O e regolando il pH a 3.8 con 1 M KOH. Successivamente, sciogliere 0,066 g di nitrato di piombo in 10 mL di soluzione di riserva di acido L-aspartic e regolare il pH a 5,5. Lasciare la soluzione in forno a 60 gradi centigradi per 30 min.
NOTA: non devono formarsi precipitazioni. - Incubare campioni nella soluzione di piombo di Walton per 30 min in forno a 60 gradi centigradi.
- Lavare i campioni come descritto al punto 1.6.
- Campioni disidratati in EtOH in passi graduati del 50%, 70%, 90% in ddH2O, e poi 2x in 100% EtOH. Utilizzare il programma a microonde 10 (Tabella 1) e il forno a microonde richiederà agli utenti ogni 40 s di sostituire la soluzione con il prossimo passo EtOH. Questo è l'ultimo passo fatto nel microonde.
- Ulteriormente disidratare in 100% ossido di propilene 2x per 10 min ciascuno a RT sul banco, sostituendo la soluzione tra i passaggi.
NOTA: l'ossido di propilene può sciogliere alcune materie plastiche come i polistireni; utilizzare fiale di vetro per questo passaggio o pre-test di plastica per la resistenza.
AVVISO: L'ossido di propilene è altamente infiammabile. Maneggiare sempre utilizzando dispositivi di protezione appropriati e in una cappa di fumi. - Iniziare l'infiltrazione delle punte della radice incubando nella resina del 50% di Spurr in ossido di propilene (min 2 h).
AVVISO: I componenti in resina dello Spurr sono irritanti. Maneggiare sempre utilizzando dispositivi di protezione appropriati e in una cappa di fumi. - Sostituire la soluzione con 100% Spurr e lasciare la notte a RT.
- Passare alla resina fresca 100% Spurr 2 volte (incubazione min 2 h).
- Mettete i campioni in uno stampo di incorporamento, contenente nuovamente la resina fresca al 100% di Spurr e polimerizzare in forno a 65 gradi centigradi per 36-48 h.
NOTA: lo stampo di incorporamento utilizzato dipende dal tipo di tessuto e dall'approccio utilizzato per l'imaging. In questo caso, è stato utilizzato uno stampo di incorporamento in silicone piatto (Figura 1A).
2. Preparare i campioni incorporati per l'imaging
- Rimuovere i campioni dal forno, rimuovere la resina dallo stampo di incorporamento (Figura 1B).
- Utilizzando una lama di rasoio, tagliare approssimativamente il campione a un blocco di massimo 0,5 mm x 0,5 mm x 0,5 mm (Figura 1C).
NOTA: Per evitare la ricarica in SBF-SEM, è importante tagliare via quanta più resina nuda possibile e rendere il campione il più piatto / sottile possibile. Idealmente tutti i lati del blocco contengono già tessuto, ma soprattutto il lato che sarà attaccato al perno metallico (vedi passo 2.3) deve contenere tessuto esposto in modo che il tessuto sia in contatto diretto con il metallo conduttivo. - Rimuovere il campione dalla resina estranea e collegarlo a un perno metallico (Figura 1D) con resina epossidica conduttiva, assicurandosi che parte del tessuto tocchi il perno metallico. Lasciare che la resina epossidica curere durante la notte in forno a 65 gradi centigradi.
NOTA: Assicurarsi che il campione sia posizionato al centro del perno, perché il movimento dello stage nell'SB-SEM è limitato. Rimuovere il campione di resina molto piccolo racchiuso dalla resina extra può essere difficile in quanto il piccolo campione avrà la tendenza a volare via quando staccato. Una soluzione semplice ed efficace a questo è quello di coprire il campione con un foglio di pellicola di paraffina come mostrato nel film supplementare di riferimento1. - Fissare il supporto nel supporto per l'ultramicrotoma. Utilizzare una lama di rasoio per rimuovere l'epossidica in eccesso e utilizzare l'ultramicrotoma e un coltello di adiamante per lisciare il viso e i lati del blocco, formando una piramide. Assicurarsi che almeno una parte del tessuto sia già esposta sulla faccia del blocco.
NOTA: Il passo extra dell'utilizzo di un coltello di adiamante è facoltativo, ma rende il blocco risultante più facile da avvicinare nel SBF-SEM perché l'ombra del coltello sulla faccia del blocco è più chiara rendendo così la stima della distanza tra coltello e faccia del blocco più facile da determinare. - Posizionate il blocco campione tagliato nel rivestimento della sferetta e ricoprite il campione con uno strato sottile di platino (Pt) di 2-5 nm.
NOTA: Il platino sulla faccia del blocco sarà tagliato durante l'avvicinamento nel SBF-SEM (vedi sotto), ma il platino sui lati della piramide fornirà ulteriore conduttività. In questo esempio, il campione è stato rivestito con platino, ma anche oro, o oro/palladio è efficace; tuttavia, il rivestimento con oro ha provocato un aumento dei detriti sulla faccia del blocco durante una corsa di imaging.
3. Imaging nell'SBF-SEM
- Inserire il supporto nel microscopio SBF-SEM e avvicinare il coltello alla superficie del campione. Utilizzando il coltello diamante tagliare la parte superiore del campione in modo che lo strato Pt è già stato rimosso e almeno una parte del tessuto è esposto.
NOTA: poiché questo processo è diverso per ogni microscopio SBF-SEM, non tutti i passaggi sono specificati qui. Finché la superficie del campione è libera da Pt e pronta per l'imaging, saranno possibili i prossimi passi. - Iniziare a imaging a bassa risoluzione e tempi di permanazione brevi per ottenere una panoramica del campione e individuare una regione di interesse (Figura 1E).
NOTA: Qui sono stati utilizzati 512 x 512 pixel e tempo di permane di 1s per la scansione rapida e il posizionamento dello stage e sono stati utilizzati 2.000 x 2.000 pixel con un tempo di permane di 1 s per ottimizzare la finestra di imaging e regolare la messa a fuoco. - Utilizzando una tensione accelerata di 1,5-2,0 kV ad una corrente di 80-100 pA, catturare un'immagine del tessuto.
NOTA: L'esempio qui mostrato è stato illustrato su un sistema ad alto vuoto in cui la corrente del fascio deve essere regolata per ridurre al minimo la ricarica, che dipende molto dal campione. Tipicamente, il fascio di elettroni è impostato su 1,5 kV, ma questo dipenderà dal campione. Con un ingrandimento più elevato (solitamente > 10.000x) la resina è troppo influenzata dal fascio per garantire un sezionamento uniforme, quindi in genere la dimensione dei pixel viene impostata a 8-20 nm con una dimensione dell'immagine di 8.000 x 10.000 x 8.000-10.000 pixel con un ingrandimento corrispondente di 430 – 1.400x e dimensioni del campo di 64 X 64 mm e 200 x 200 mm rispettivamente. - Determinare una regione di interesse e decidere quante sezioni sono necessarie per coprire il volume di interesse e avviare la corsa di imaging, utilizzando il rilevatore di elettroni sparsi posteriore.
NOTA: nell'esempio qui presentato, 500 sezioni di 80 nm sono state immagini a 10 pixel e immagini di 10.000 x 10.000 pixel (tempo di dimora 1 s). Il microscopio è stato impostato su 1,6 kV e 100 pA. In generale, il numero di sezioni dipende dal campione e dalle dimensioni del ROI e può variare da 100 s a 1.000 s di sezioni consecutive. Il set di dati risultante è costituito da singole immagini di ogni sezione. Queste immagini devono essere convertite in una pila 3D.
4. Trattamento dei dati SBF-SEM
- Utilizzando Figi, selezionate la sequenza file-import-image e individuate la pila di immagini per caricare le immagini. A seconda delle dimensioni del set di dati, selezionare la casella 'Usa stack virtuale'.
NOTA: se il set di dati è davvero grande, prima convertirlo in 8 bit (se raccolto a 16 bit) e, se necessario, inserire i dati fino a quando non ha una dimensione praticabile. - Uso del pulsante di riproduzione nella parte inferiore dell'immagine scorrere il set di dati per verificare se l'esecuzione dell'immagine ha avuto esito positivo. Verificare la presenza di manufatti di imaging tipici per SBF-SEM, come le sezioni che cadono dal coltello sul tubo, la ricarica in aree di resina nuda, gli artefatti di taglio del coltello (linee orizzontali sull'immagine).
- Utilizzando il comando image-properties regolare le dimensioni in pixel e la profondità voxel (cioè, la sezione-spessore) utilizzate durante l'esecuzione. Se i dati sono già collocati, prenderne in considerazione.
- Utilizzando il comando plugins-registration-linear stack alignment con SIFT per registrare i dati.
NOTA: la registrazione dei dati SBF-SEM è necessaria perché potrebbe esserci un leggero movimento del campione durante l'imaging a causa della ricarica o della deriva del campione. Poiché questo è solo un movimento minimo in XY, è necessaria solo la traduzione. - Controllare l'allineamento scorrendo il dataset e se OK utilizzare il comando Salva nel menu File per salvare il dataset allineato come file 3D-tif.
- Analizzare attentamente il set di dati per verificare se il ROI è incluso e contiene le informazioni necessarie per la domanda biologica. Nell'ultima immagine della pila ( : la faccia a blocchi corrente), selezionare un nuovo ROI per l'imaging FIB-SEM.
NOTA: se non esiste una buona regione per l'imaging FIB-SEM presente sulla faccia del blocco corrente, è possibile tagliare più sezioni dal blocco (che si trova ancora nell'SBF-SEM) fino a quando non viene visualizzato un ROI. Esiste un limite al volume che può essere visualizzato con il FIB. Il ROI sull'immagine SBF-SEM può essere massimo in X,Y di 30-40 m x 15-20 m.
5. Imaging nel FIB-SEM
- Prendere immagini generali dell'esempio nell'SBF-SEM, includendo idealmente uno o più bordi dell'esempio che sono quindi riconoscibili nel FIB-SEM (Figura 2A, C).
NOTA: In questo esempio le immagini di panoramica SBF-SEM sono state scattate con una dimensione in pixel di 10 nm, 8.000 x 8.000 pixel a un tempo di permane. Il microscopio era ancora impostato a 1,6 kV e 100 pA. - Rimuovere il campione dall'SBF-SEM e posizionarlo nel rivestimento sputter. Coprire il campione con 20 nm di platino per l'imaging FIB-SEM.
- Caricare il campione in FIB-SEM e utilizzando il rilevatore di elettroni secondari a 15 kV, 1 nA individuare il ROI identificato nel SBF-SEM sulla faccia a blocchi (Figura 2B, D).
NOTA: L'imaging a 15 kV è necessario per vedere attraverso il rivestimento in platino. - Portare il ROI sul campione nel punto di coincidenza delle travi FIB e SEM, spostando e inclinando lo stage (Figura 3A).
NOTA: la colonna FIB è solitamente montata sotto un angolo (Figura3A). Ciò significa che qualsiasi campione deve essere inclinato in modo tale che la superficie da ingrandire e sezionare sia posizionata parallelamente al fascio FIB. La superficie da immaginare è ora inclinata rispetto al fascio SEM e una trincea di tessuto deve essere rimossa prima che il SEM sia in grado di immaginare il ROI (Figura 3E) - Utilizzando il fascio FIB e il sistema di iniezione del gas, depositare uno strato protettivo di 1 mm di platino sulla superficie sopra il ROI (Figura 3C). Successivamente, utilizzando una corrente di fresatura bassa (50-100 pA), fresare le linee sottili nella deposizione di platino per la messa a fuoco automatica e il tracciamento 3D durante l'esecuzione di immagini (Figura 3D). Utilizzando la colonna FIB e l'iniettore di gas di carbonio, coprire queste linee con deposizione di carbonio.
NOTA: la dimensione del ROI qui corrisponde alla dimensione del ROI sull'immagine SBF-SEM e la dimensione massima può quindi essere 30-40 m x 15-20 m. In questo esempio è stato imageato un ROI di 17 x 8 m. La deposizione di carbonio è necessaria per la protezione delle linee e per creare un contrasto bianco-nero tra il carbonio e il platino che è ideale per la messa a fuoco automatica. Le correnti di fresatura utilizzate per ogni passo sono riportate nella tabella 2. - Utilizzando una corrente di fresatura elevata, minacciare una trincea di 30 m davanti al ROI, creando la superficie di imaging per il fascio SEM (Figura 3E).
NOTA: Il raggio FIB è intrinsecamente distruttivo, ancora di più a correnti elevate. Assicurarsi di mantenere l'imaging ad alte correnti al minimo e l'immagine a bassa ingrandimento e velocità di scansione veloci per evitare di fondere la resina al ROI. - Ammorbidire la superficie di imaging con una corrente di fresatura più vicina alla corrente utilizzata durante la conduzione dell'imaging. Interrompere la lucidatura quando tutti i segni di rilevamento 3D e di messa a fuoco automatica sono chiaramente visibili sulla superficie di imaging (Figura 3F). Il progresso del FIB può essere seguito dall'imaging della superficie con il servizio EM (utilizzando un rilevatore di elettroni sparsi sul retro) durante la lucidatura.
- Determinare l'area da visualizzare sulla superficie appena creata e impostare i parametri di imaging. Assicuratevi che il fascio di elettroni sia focalizzato sulla superficie, impostate la luminosità e il contrasto e impostate la dimensione in pixel e lo spessore della sezione. Mantenere il tempo di imaging al di sotto di 1 minuto regolando il tempo di dimora e la media della linea.
NOTA: È importante utilizzare una bassa tensione per l'imaging con il fascio di elettroni per garantire che solo la superficie del blocco venga immagine (cioè che non vengano immagini elettroni più profondi nel campione). Questo viene fatto mantenendo la tensione al di sotto di 2 kV e utilizzando un rilevatore di elettroni sparsi posteriori con una tensione di rete, permettendo solo elettroni ad alta energia per essere immagine. In questo esempio, il fascio di elettroni è stato impostato a 1,5 kV e 1 nA con una tensione della griglia di 1,2 kV sul rilevatore di elettroni sparsi sul retro. Anche in questo caso, è stata utilizzata una dimensione in pixel di 5 nm con sezioni 5nm per generare un set di dati con voxel isotropici. Un'area di 17 m x 10 m è stata immagine a 6,5 s tempo di permane e una media di linea di 1,0. - Impostare le finestre per la regolazione automatica e il tracciamento 3D, utilizzando le stesse dimensioni in pixel, il tempo di permane e la media della linea utilizzata per l'imaging.
NOTA: questo processo sarà diverso per i diversi sistemi, pertanto viene menzionato solo il passaggio senza specificare le diverse azioni. - Avviare la corsa di imaging e monitorare la stabilità del processo durante le prime 50-100 sezioni. Una volta che il sistema funziona senza intoppi, lasciare la stanza e assicurarsi che ci sia il minor disturbo possibile per la stanza.
NOTA: la durata della conduzione e il numero di sezioni dipendono dalle dimensioni del ROI e dallo spessore della sezione. In FIB-SEM l'asse z è in realtà l'altezza della scatola ROI sull'immagine SBF-SEM (max 15-20 m; Figura 3E). - Registrare i dati FIB-SEM nello stesso modo descritto in precedenza per i dati SBF-SEM.
Risultati
Le immagini dell'SBF-SEM forniscono una panoramica del tessuto, fornendo informazioni sull'orientamento spaziale delle cellule e delle connessioni intercellulari (Figura 4A). La successiva imaging FIB-SEM su una nuova regione, che di solito è una regione di interesse determinata dopo l'ispezione dell'esecuzione sBF-SEM, aggiunge dettagli ad alta risoluzione di celle e/o strutture specifiche (Figura 4B).
Figura 4C ,D mostra la differenza nel rendering dei voxel non isotropici dei dati SBF-SEM (Figura 4C) e dei dati isotropici voxel FIB-SEM (Figura 4D). Lo spessore z utilizzato in SBF-SEM significa che il rendering mostra chiaramente le sezioni, con un effetto 'scala' sulla superficie. Nei dati FIB-SEM le sezioni 5 nm assicurano che il rendering sia molto più uniforme e le singole sezioni si fondono completamente nella superficie.
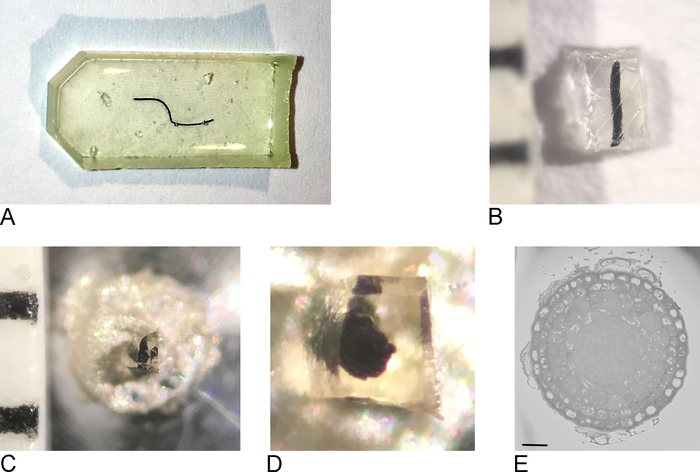
Figura 1: Creazione della faccia di blocco da un campione incorporato in resina. (A) Un root-tip incorporato nella resina. (B) Utilizzando una lama di rasoio la resina in eccesso viene tagliata fino a quando non rimane un blocco di 0,5 mm2. (C,D) Il blocco tagliato è incollato su un perno metallico e dopo una notte in forno, i lati del blocco vengono tagliati e la superficie levigata con un coltello di a diamante utilizzando un ultramicrotoma. (E) All'interno dell'SBF-SEM, il campione è orientato in modo che il blockface e ROI può essere riconosciuto, barra di scala 20 m. Fare clic qui per visualizzare una versione più grande di questa figura.
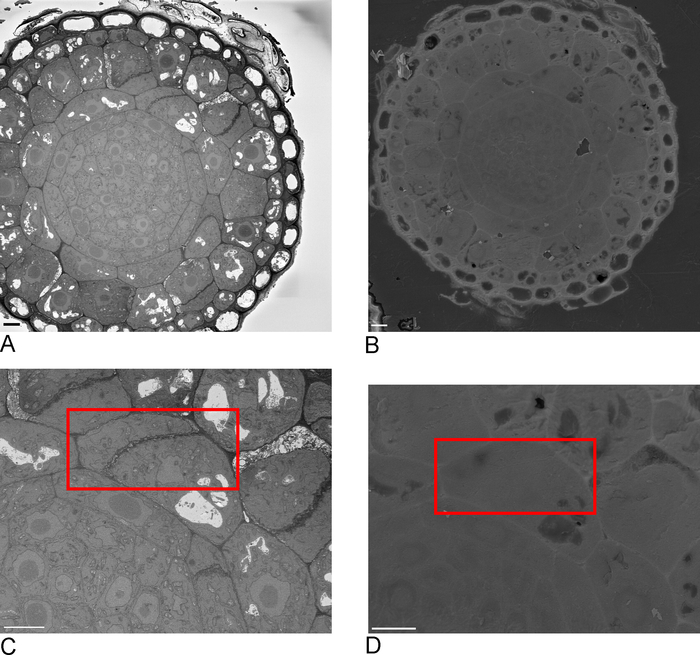
Figura 2: Correlazione tra SBF-SEM e FIB-SEM. Panoramica delle immagini della faccia del blocco utilizzando l'SBF-SEM (A) e il FIB-SEM (B), la barra della scala (C,D) zoom sul ROI. Scatola rossa delinea la regione da immaginare con FIB-SEM, barra della scala - 5 m. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
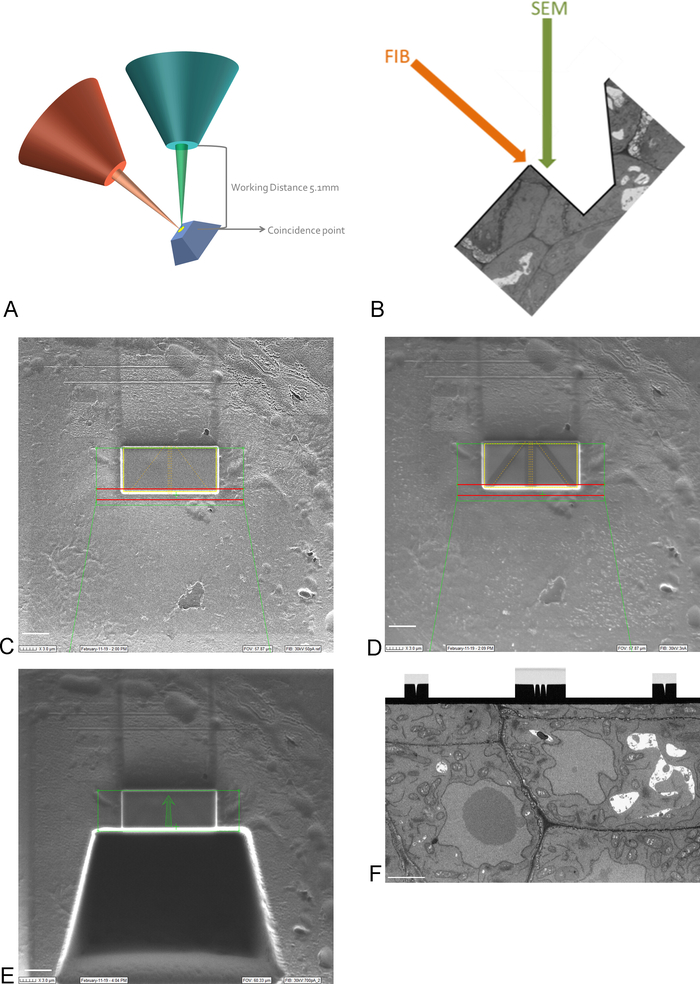
Figura 3: Schema FIB-SEM e passaggi di preparazione. (A) Schema che mostra l'orientamento della trave FIB, del fascio SEM e del campione. Il campione deve essere posizionato al punto di coincidenza delle travi FIB e SEM per poter fresare e immagini sulla stessa regione. (B) Disegno schematico della trincea necessaria per l'imaging SEM delle sezioni rimosse dalla FIB. (C) Immagine scattata con il fascio FIB che mostra la deposizione platinumsul sul ROI, barra in scala di 5 m. (D) Immagine scattata con il fascio FIB che mostra le linee utilizzate per la messa a fuoco automatica e il tracciamento 3D. Le linee al centro vengono utilizzate per la messa a fuoco automatica e le linee esterne forniscono il tracciamento 3D. Deposizione di carbonio in cima alle linee fornisce il contrasto necessario (platino vs carbonio) per eseguire queste attività, scalebar 5 s. (E) Immagine scattata con il fascio FIB dopo la fresatura della trincea, scalebar 5 (F) Immagine scattata con il fascio SEM che mostra la regione di interesse immagine durante l'esecuzione FIB-SEM, barra della scala di 2 m. Fare clic qui per visualizzare una versione più grande di questa figura.
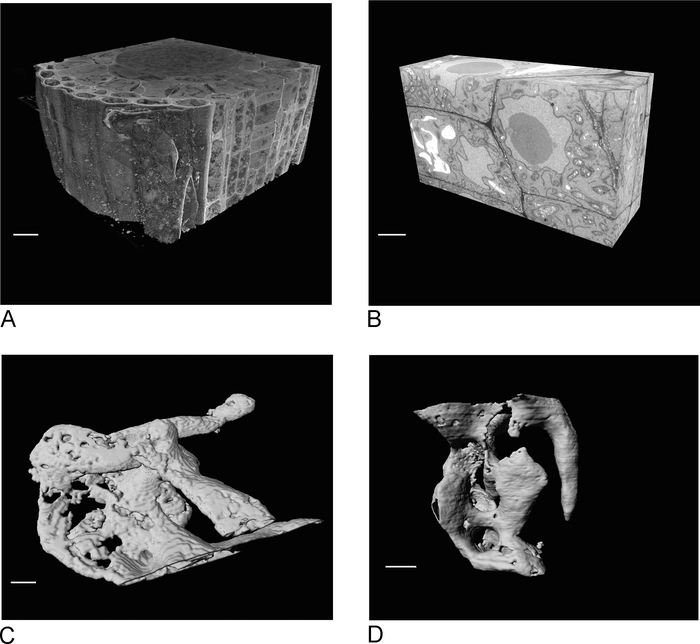
Figura 4: risultati SBF-SEM e FIB-SEM prima e dopo la segmentazione. (A) Vista 3D del set di dati SBF-SEM (100 x 100 x 40 m, barra della scala - 10 m), (B) vista 3D del set di dati FIB-SEM (17 x 10 x 5,4 m, barra della scala x 2 m), (C) Rendering di vacuoles segmentati dai dati SBF-SEM per soglia, scala mented dai dati FIB-SEM per soglia, barra della scala - 2 m. Fare clic qui per visualizzare una versione più grande di questa figura.
| Protocollo per l'elaborazione a microonde | |||||||||
| programma # | descrizione | Prompt utente (attivata/disattivata) | Tempo (hr:min:sec) | Potenza (Watt) | Temp (C) | Carica raffreddatore (spento/auto/on) | Vacuum/Bubbler Pump (ciclo off/bubb/vac/vac on/vap) | Temperatura stabile | |
| Pompa (accesa/disattivata) | Temp (C) | ||||||||
| 8 | Tch | via | 0:01:00 | 150 | 50 | via | CICLO DEL VUOTO | su | 30 |
| via | 0:01:00 | 0 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:01:00 | 150 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| 9 | Osmio | via | 0:02:00 | 100 | 50 | via | CICLO DEL VUOTO | su | 30 |
| via | 0:02:00 | 0 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:02:00 | 100 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:02:00 | 0 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:02:00 | 100 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| 10 | 50% EtOH | su | 0:00:40 | 150 | 50 | via | via | su | 30 |
| 70% EtOH | su | 0:00:40 | 150 | 50 | via | via | su | 30 | |
| 90% EtOH | su | 0:00:40 | 150 | 50 | via | via | su | 30 | |
| 100% EtOH | su | 0:00:40 | 150 | 50 | via | via | su | 30 | |
| 100% EtOH | su | 0:00:40 | 150 | 50 | via | via | su | 30 | |
| 15 | 0,1 M CACODYLATE | su | 0:00:40 | 250 | 50 | via | CICLO DEL VUOTO | su | 30 |
| 0,1 M CACODYLATE | su | 0:00:40 | 250 | 50 | via | CICLO DEL VUOTO | su | 30 | |
| 15 | ddH2O | su | 0:00:40 | 250 | 50 | via | CICLO DEL VUOTO | su | 30 |
| ddH2O | su | 0:00:40 | 250 | 50 | via | CICLO DEL VUOTO | su | 30 | |
| 16 | Acetato di Uranyl | via | 0:01:00 | 150 | 50 | via | CICLO DEL VUOTO | su | 30 |
| via | 0:01:00 | 0 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:01:00 | 150 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:01:00 | 0 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:01:00 | 150 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:01:00 | 0 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
| via | 0:01:00 | 150 | 50 | via | CICLO DEL VUOTO | su | 30 | ||
Tabella 1. Protocollo dettagliato per l'elaborazione a microonde.
| passo | attuale | Tempo stimato |
| Deposizione Platino | 3n A | 10-15 minuti |
| Ottimizzazione della fresatura e contrassegni di tracciamento | 50-100 pA | 4-6 minuti |
| Carbonio deposizione | 3 nA | 5-10 minuti |
| Fresatura Fossa Grossa | 15-30 nA | 30-50 minuti |
| Superficie di lucidatura | 1.5-3 nA | 15-20 minuti |
| Esecuzione di imaging | 700 pA-1,5 nA | Ore-giorni |
Tabella 2. Correnti di fresatura FIB utilizzate per la preparazione del campione e la conduzione dell'imaging
Discussione
La microscopia elettronica di volume è più impegnativa e dispendiosa in termini di tempo rispetto al SEM o TEM convenzionale. A causa della necessità di macchiare tessuti o cellule in blocco, le fasi di lavorazione devono essere abbastanza lunghe da garantire la penetrazione dei reagenti in tutto il campione. L'utilizzo dell'energia a microonde per facilitare la penetrazione consente un'elaborazione più breve ed efficiente e migliora la colorazione. Poiché la preparazione per EM è molto più rigorosa che per la microscopia leggera, tutte le soluzioni e i reagenti devono essere rigorosamente controllati dalla qualità. I cambiamenti nella pH, nella tonicità, nell'uso di reagenti impuri e nell'introduzione di contaminanti dovuti a una tecnica scadente possono tutti avere effetti deleteri sull'immagine finale.
Volume EM richiede anche protocolli personalizzati per ogni tipo di campione diverso. Tessuti mammiferi di diversi tipi: piante, singole cellule come lieviti, trypanosomes, C. elegans, ecc., tutti hanno bisogno delle proprie variazioni per ottenere risultati ottimali. La fissazione e la colorazione devono essere progettate in modo da preservare l'integrità strutturale e mantenere il campione il più vicino possibile alla morfologia in vivo. La fissazione dei tessuti a temperatura fisiologica, pH e tonicità è fondamentale per rendere il campione come vitale come può essere. Il congelamento ad alta pressione (HPF) di campioni può aiutare a preservare una situazione più realistica, (o forse solo produrre artefatti diversi), ma per le cellule diverse da quelle che l'hAV fallirà come ghiaccio vitreo può essere generato solo in piccoli volumi. Pertanto, per molte domande la fissazione chimica è l'unica opzione. Non importa se la fissazione è HPF o chimica, in qualsiasi esperimento EM i risultati strutturali devono essere attentamente confrontati con risultati simili dall'imaging di cellule vive o tessuti per vedere se sono coerenti. La colorazione deve anche essere ottimizzata considerando la domanda specifica che deve essere risolta e il protocollo che verrà utilizzato per la visualizzazione delle immagini digitali.
Avere un sistema SBF-SEM e FIB nelle immediate vicinanze è un grande vantaggio in molti esperimenti. L'ampio campo visivo e l'elevata risoluzione X,Y di SBF-SEM rendono semplice trovare singole strutture/cellule/eventi e forniscono un orientamento spaziale generale delle cellule nei tessuti. Inoltre, la sua capacità di consentire l'imaging attraverso un campione in : è molto potente; tuttavia, le ricostruzioni che richiedono dettagli geometrici fini possono non riuscire o produrre artefatti utilizzando questa tecnica a causa dei voxel non isotropici che genera. Il FIB è limitato dalla fisica del processo a un campo di imaging più piccolo, ma la sua risoluzione 3D è sufficiente per ricostruzioni molto accurate. La combinazione delle due tecniche è semplice in quanto i campioni possono passare da SBF-SEM a FIB senza ulteriori elaborazioni o preparazioni. Riconosciamo che l'utilizzo dell'SBF-SEM per la ricerca in un campione per trovare una particolare area è un uso molto costoso di uno strumento molto più capace. Tuttavia, la possibilità di vedere immediatamente il nuovo blockface e determinare se il ROI è stato raggiunto è un grande vantaggio. Inoltre, le alternative all'uso di sezioni LM semi-sottili seriali (0,5 m) possono rimuovere piccole strutture prima di essere rilevate e ispezionare un blocco utilizzando singole sezioni TEM che devono essere tagliate, indossare una griglia e quindi essere visualizzate in un TEM altrettanto costoso non è efficiente come il metodo presentato.
Poiché esistono molti programmi per segmentare ed eseguire il rendering dei dati e le esigenze di una determinata struttura potrebbero non essere soddisfatte al meglio da una singola applicazione, non è possibile proporre alcun flusso di lavoro standard. Alcune strutture semplici possono essere segmentate con un algoritmo di soglia se rientrano in valori di scala di grigi molto stretti. Le strutture neuronali possono essere segmentate semi-automaticamente utilizzando un programma come Ilastik11, ma sarà meno utile su organelli più casuali o di forma complessa come ER. Microscopy Image Browser è un programma molto flessibile che può allineare, segmentare e renderizzare i dati EM del volume, ma richiede un'interazione significativa da parte dell'utente12. Come regola generale, il tempo necessario per visualizzare digitalmente i risultati supererà notevolmente il tempo necessario per la preparazione del campione e dell'imaging.
Le tecniche Volume EM hanno aperto la terza dimensione all'analisi ultrastrutturale. Altri metodi per ottenere eM 3D hanno limitazioni nel loro volume (tomografia TEM) o nella loro efficienza (sezione seriale TEM). Sebbene per la maggior parte le tecniche di EM in volume siano troppo complesse e costose per essere implementate nei singoli laboratori, il numero di strutture centrali condivise che le offrono è in crescita e il numero di tipi di campione immagine con successo è aumentato rapidamente. Per coloro che hanno una domanda specifica e un particolare tessuto è probabile che qualcuno sarà in grado di offrire consigli e istruzioni sulla sua preparazione e imaging. Le apparecchiature Volume EM possono essere migliorate per includere la capacità di gestire campioni più grandi nell'SBF-SEM e la capacità di fresare calcoli più grandi con il FIB. Il software in grado di segmentare le strutture di interesse in modo più automatizzato semplificherà notevolmente il processo di analisi dei dati e i miglioramenti della velocità di elaborazione ridurranno il tempo necessario per farlo. Nonostante le sue attuali limitazioni, il volume EM è ancora uno strumento utile e la combinazione di SBF-SEM e FIB-SEM fornisce un flusso di lavoro efficiente per identificare eventi rari e imaging ad alta risoluzione.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
L'attrezzatura per il volume EM è stata fornita da una generosa sovvenzione del governo delle Fiandre.
Materiali
| Name | Company | Catalog Number | Comments |
| 3View 2XP | Gatan | NA | In chamber ultramicrotome for SBFI |
| Cacodylate buffer 0.2M solution | EM Sciences | 11652 | |
| Conductive epoxy resin (circuit works) | RS components | 496-265 | |
| Diatome Histo 4.0mm diamond knife | EM Sciences | 40-HIS | |
| Digitizing tablet | Wacom | DTV-1200W | No longer available |
| Eppendorf tubes | Eppendorf | 0030 120.094 | |
| Flat Embedding Mold | EM Sciences | 70900 | |
| Gluteraldehyde 25% solution | EM Sciences | 16220 | |
| High MW Weight Tannic Acid | EM Sciences | 21700 | |
| Lead Citrate | Sigma-Aldrich | 22861 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Osmium Tetroxide 4% solution | EM Sciences | 19170 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Pelco Biowave Pro + | Ted Pella | 36700 | |
| Potassium Ferrocyanide | Sigma-Aldrich | P3289 | |
| Quorum Q150T ES sputter coater | Quorum Technologies | 27645 | |
| Reichert-Jung Ultracut ultramicrotome | NA | NA | No longer available |
| Sodium Cacodylate 0.2M | EM Sciences | 11653 | |
| Spurrs Resin kit | EM Sciences | 14300 | |
| Uranyl Acetate | EM Sciences | 22400 |
Riferimenti
- Linberg, K. A., Fisher, S. K. An ultrastructural study of interplexiform cell synapses in the human retina. Journal of Comparative Neurology. 243, 561-576 (1986).
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Leighton, S. SEM images of block faces, cut by a miniature microtome within the SEM – A technical note. Scanning Electron Microscopy. (pt 2), 71-76 (1981).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three dimensional tissue nanostructure. PLoS Biology. 11, 329 (2004).
- Heymann, J. A., Hayles, M., Gestmann, I., Giannuzzi, L. A., Lich, B., Subramaniam, S. Site specific 3D imaging of cells and tissues with a dual beam microscope. Journal of Structural Biology. 155 (1), 63-73 (2006).
- . NCMIR Methods for 3D EM: A new protocol for preparation of biological specimens for serial block face scanning electron microscopy. V7_01_10 Available from: https://ncmir.ucsd.edu/sbem-protocol (2019)
- Giberson, R. T., Austin, R. L., Charlesworth, J., Adamson, G., Herrera, G. A. Microwave and digital imaging technology reduce turnaround times for diagnostic electron microscopy. Ultrastructural Pathology. 27 (3), 187-196 (2003).
- Kremer, A., et al. Developing 3D EM in a broad biological context. Journal of Microscopy. 259 (2), 80-96 (2015).
- Vanslembrouck, B., Kremer, A., Pavie, B., van Roy, F., Lippens, S., van Hengel, J. Three-dimensional reconstruction of the intercalated disc including the intercellular junctions by applying volume scanning electron microscopy. Journal of Histochemistry and Cell Biology. 149, 479-490 (2018).
- Russel, M. R., et al. 3D correlative light and electron microscopy of cultured cells using serial blockface scanning electron microscopy. Journal of Cell Science. 130 (1), 278-291 (2017).
- Sommer, C., Strähle, C., Köthe, U., Hamprecht, F. A. ilastik: Interactive Learning and Segmentation Toolkit. Eighth IEEE International Symposium on Biomedical Imaging (ISBI). Proceedings. , 230-233 (2011).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy Image Browser: A Platform for Segmentation and Analysis of Multidimensional Datasets. PLoS Biology. 14 (1), e1002340 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon