È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Plasmid di DNA ad alto throughput e trasfezione utilizzando la tecnologia di nanoedispensing acustico
In questo articolo
Riepilogo
Questo protocollo descrive la trasflessazione di plasmide ad alto rendimento delle cellule dei mammiferi in una piastra di 384 pozzetto che utilizza la tecnologia di espulsione acustica delle goccioline. L'erogazione e il multiplexing del DNA, che richiedono molto tempo e richiedono errori, ma anche il reagente di trasfezione, sono basati su software ed eseguiti da un dispositivo nanodispenser. Le cellule vengono poi semiate in questi pozzi precompilati.
Abstract
La trasfezione cellulare, indispensabile per molti studi biologici, richiede il controllo di molti parametri per un risultato accurato e di successo. Il più delle volte eseguita a bassa velocità effettiva, è inoltre dispendiosa in termini di tempo e soggetto a errori, ancora di più quando si multiplano diversi plasmidi. Abbiamo sviluppato un metodo semplice, veloce e preciso per eseguire la trasfezione cellulare in un layout a piastre a 384 pozze utilizzando la tecnologia di espulsione acustica delle goccioline (ADE). Il dispositivo nanodispenser utilizzato in questo studio si basa su questa tecnologia e consente la consegna precisa di nanovolume ad alta velocità da una piastra di pozzo di origine a una di destinazione. Può erogare e multispensare il DNA e il reagente di trasfezione secondo un foglio di calcolo pre-progettato. Qui presentiamo un protocollo ottimale per eseguire la trasfezione ad alta velocità basata su ADE che consente di raggiungere un'efficienza fino al 90% e una cotransfezione quasi al 100% negli esperimenti di cotransfezione. Estendiamo il lavoro iniziale proponendo una macro basata su fogli di calcolo user-friendly, in grado di gestire fino a quattro plasmidi/pozzi da una libreria contenente fino a 1.536 diversi plasmidi e un'applicazione di guida al pipettaggio basata su tablet. La macro progetta i modelli necessari delle lastre di origine e genera i file pronti all'uso per il nanodispenser e l'applicazione basata su tablet. Il protocollo di trasfezione a quattro fasi coinvolge i) un dispense diluente con un gestore di liquidi classico, ii) distribuzione del plasmide e multiplexing, iii) un reagente di trasfezione erosi dal nanodispense, e iv) placcatura cellulare sui pozzi precompilati. Il controllo basato su software descritto di ADE plasmide multiplexing e trasfezione consente anche ai non specialisti del settore di eseguire una trasfezione cellulare affidabile in modo rapido e sicuro. Questo metodo consente di ottenere rapidamente l'identificazione delle impostazioni ottimali per un determinato tipo di cellula e può essere trasposto in approcci manuali e su scala più elevata. Il protocollo facilita le applicazioni, come la proteina UMANA ORFeome (insieme di frame di lettura aperti [ORF] in un genoma) espressione o la convalida della funzione genica basata su CRISPR-Cas9, in strategie di screening non di pool.
Introduzione
Il metodo qui presentato descrive in dettaglio come eseguire il plasmide del DNA e la trasfezione nelle cellule dei mammiferi ad alta produttività utilizzando un nanodispenser liquido a base acustica in una piastra di 384 pozze, anche per i non specialisti del settore. Questo metodopubblicato di recente 1 permette di eseguire fino a 384 condizioni indipendenti di multiplexing e trasfezione del DNA plasmide in un esperimento, in meno di 1 h. Gli esperimenti di cotrafezione o singolo hanno avuto successo, raggiungendo un quasi 100% cotrafezione all'interno della popolazione di cellule trasfette. Questo protocollo semplifica la trasfezione perché la maggior parte dei passaggi noiosi, dispendiosi in termini di tempo e soggetti a errori sono ora guidati dal software (vedere Figura 1 per una panoramica generale). Ulteriori sforzi sono stati fatti per sviluppare strumenti dedicati per migliorare la facilità d'uso evitando gli errori umani durante il processo complessivo e per promuovere la trasfezione di successo anche per i non specialisti nel campo. Il protocollo descritto include un foglio di calcolo macro "user-friendly" che abbiamo sviluppato al fine di gestire 384 condizioni di trasfezione indipendenti con possibilità di multiplexing fino a quattro plasmidi in ogni pozzo. La macro genera automaticamente modelli delle lastre di origine per caricare il volume di plasmid di DNA previsto dall'avvio di soluzioni di stock e i file necessari per guidare il software nanodispenser sul progetto sperimentale che è stato inserito. Poiché l'erogazione manuale del DNA in una piastra sorgente 384-well è noiosa e soggetta a errori, abbiamo anche sviluppato un'applicazione dedicata basata su tablet per guidare l'utente durante l'erogazione della soluzione di DNA secondo il modello.
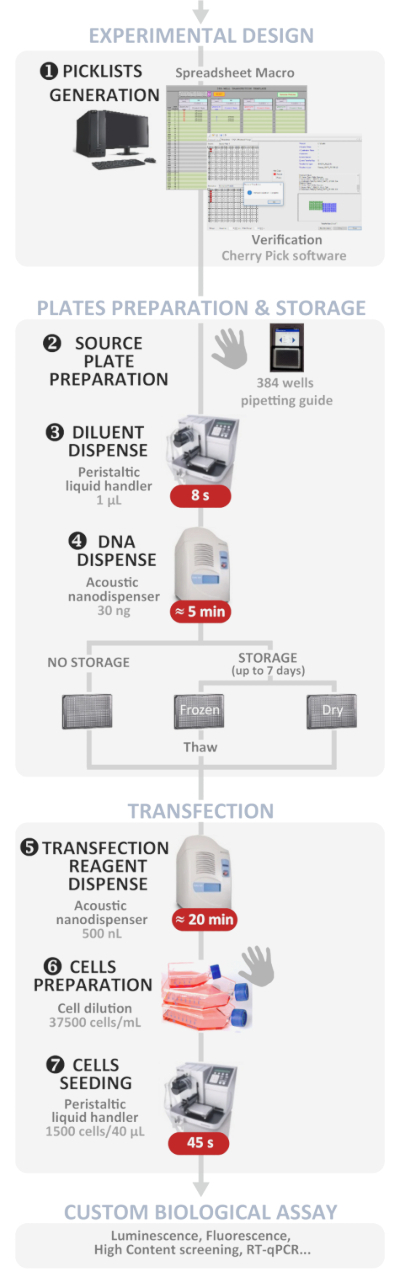
Figura 1: flusso di lavoro sperimentale. Rappresentazione schematica del protocollo di trasfezione inversa automatizzato ottimale ad alta velocità effettiva (dalla progettazione sperimentale al test biologico personalizzato). I passaggi manuali sono indicati dal simbolo della mano e il tempo approssimativo per ogni passo è scritto in una casella rossa. Fare clic qui per visualizzare una versione più grande di questa figura.
Molti esperimenti basati sulle cellule iniziano con la trasfezione del DNA plasmida, e anche se molti reagenti dedicati sono stati e sono ancora in fase di sviluppo per migliorare l'efficienza della trasfezione e/o facilitare la procedura, molto resta da fare2,3 , 4. La trasfezione cellulare plasmida di DNA prevede diversi passaggi per raggiungere un'elevata efficienza, come un assorbimento complesso iniziale, fuga endosomica e trasporto citoplasmatico al nucleo5,6. Oltre alla precipitazione di calcio o tecniche fisiche come elettroporazione o microiniezione utilizzando dispositivi dedicati7, i moderni metodi chimici si sono concentrati sul miglioramento della somministrazione di cellule del DNA, abbassando la citossicità cellulare8, 9. L'uso di lipidi o polimeri cationici che formano complessi simili a liposomi e, più recentemente, sistemi chimici polimerici non liposomici ha reso la trasfezione più facile e più efficiente10. Nonostante questi sviluppi, la trasfezione cellulare richiede ancora competenze specifiche per essere eseguita con precisione poiché la maggior parte di questi protocolli di trasfezione fisica o chimica richiedono agli scienziati di preparare manualmente ogni condizione di reazione alla trasfezione del DNA, compromissione della velocità effettiva. Per aggirare questo problema, sono stati sviluppati protocolli di trasfezione inversa utilizzando reagenti di trasfezione chimica11,12,13, consentendo all'utente di testare o combinare diversi plasmidi in modo più veloce. In questi protocolli, si formano complessi di acido nucleico con reagenti di trasfezione prima di seminare le cellule sui complessi. Tuttavia, questi protocolli inversi sono ancora limitati dalla gestione manuale delle soluzioni del DNA e dalla combinazione di ciascuna delle condizioni indipendenti. Anche se è possibile eseguirli in un formato di piastra 96-well, la preparazione del DNA e dispensa sarà noioso, e probabilmente ci saranno errori. Quando sono necessarie e multiplexed con l'altro diverse quantità di diversi plasmidi di DNA, la trasfezione cellulare diventa ancora più difficile da raggiungere e gli errori umani diventano abbastanza inevitabili. Scalare fino al formato a 384 pozze in un approccio di trasfezione inverso, nonostante poche condizioni di trasfezione del DNA multiplexed, diventa una sfida impossibile a causa dei seguenti motivi. i) I volumi di DNA, reagente di trasfezione o miscela di reazione da gestire sono inferiori a 1 -L per ogni pozzo. ii) Il multiplexing di plasmidi per 384 condizioni indipendenti diventa estremamente complicato. La consegna in ciascuno dei 384 pozzi è anche iii) molto dispendioso in termini di tempo e iv) soggetto a errori. Infatti, erogare la soluzione giusta nei pozzi attesi è difficile da gestire perché i bassi volumi già erogati non consentono il monitoraggio visivo tra i pozzi vuoti e quelli già riempiti. v) Infine, c'è un alto rischio di essiccazione della miscela per evaporazione prima che le cellule vengano aggiunte a causa del tempo necessario per eseguire le fasi di erogazione necessarie. In sintesi, il fattore limitante per impostare analisi di trasfezione del DNA ad alto consumo sembra essere la miniaturizzazione dell'analisi, che implica multiplexing e gestione a basso volume che non possono più essere gestiti manualmente, ma sono anche difficilmente raggiungibili in un affidabile dai classici gestori di liquidi peristatici.
Come prova di difficoltà per automatizzare tali saggi e ottenere un alto rendimento, solo pochi tentativi di automatizzare la trasfezione sono stati pubblicati finora: un formato di piastra di 96 pozzetti che utilizza un dispositivo di movimentazione dei liquidi commerciali e precipitazioni di fosfato di calcio14 e, più recentemente, un reagente lipoplex e un chip microfluidico che consentono 280 trasfettazioni indipendenti15 ma richiedono competenze specializzate in questo campo. Un altro metodo, l'acoustoforesi, che consente la levitazione liquida e porta alla manipolazione e miscelazione dei fluidi, è stato utilizzato per eseguire la trasfezione del DNA nei formati da 24 a 96 pozzi16. Anche se fattibile, questo approccio soffre di una produttività estremamente bassa in quanto la miscela di cellule con miscela di trasfezione del DNA richiede un'incubazione di 60 s per ogni singolo punto prima della semina. Ciò implica una durata di almeno 96 min per una piastra completa di 96 pozze. Inoltre, questo protocollo è ben lungi dall'essere suscettibile al pubblico generale dei biologi in quanto questo lavoro è stato fatto con un dispositivo progettato e fabbricato in-house che attualmente non è disponibile sul mercato. Al contrario, negli ultimi anni, è emersa una tecnologia di erogazione basata sull'acustica basata su software di facile utilizzo con dispositivi di dispenser nanovolume. Utilizzando energia acustica focalizzata, questi dispositivi consentono l'espulsione strettamente controllata di piccoli volumi di liquidi da 2,5 nL a 500 nL da una piastra di origine a una destinazione17. Questa tecnologia, chiamata espulsione acustica delle goccioline (ADE), presenta numerosi vantaggi: è completamente automatizzata, senza contatto, senza punta, precisa e altamente riproducibile, e ha un alto throughput18. In primo luogo dedicato alla fornitura di soluzioni di zolfo dimetilico (DMSO), le impostazioni sono state migliorate per erogare tamponi acquosi19. I nanodispenser acustici, quindi, sembrano adatti per i protocolli di trasfezione inversa delle cellule e potrebbero aggirare la maggior parte delle limitazioni manuali di cui sopra. Poiché in precedenza non sono stati descritti tentativi di trasfezione plasmida utilizzando questa tecnologia, abbiamo recentemente valutato l'idoneità di un sistema di erogazione basato su acustica per eseguire la trasfezione inversa delle celle.
Approfittando della velocità effettiva dei nanodispenser e della facilità d'uso, abbiamo ottimizzato un protocollo di trasfezione inversa per le cellule HeLa verificando diversi parametri che possono influenzare la trafezione del DNA su una piastra singola di 384 pozzetto, vale a dire la quantità totale di DNA e concentrazione iniziale del DNA sorgente, volume diluente, reagente di trasfezione e numero di cellule di diffusione. Il protocollo sviluppato aggira i limiti manuali sopra descritti della trasfezione cellulare e presenta diversi vantaggi rispetto ad altri tentativi di trasfezione automatizzati. In primo luogo, è miniaturizzato, consentendo così un reagente di trasfezione conveniente salvando i preparati del plasmide del DNA e il reagente di trasfezione. In secondo luogo, è molto più ad alto rendimento e riproducibile rispetto al protocollo manuale (anche per i principianti), in quanto la trasfezione di un'intera piastra di 384 pozzetto può essere ottenuta in meno di 1 h. Infine, è basato sul software, consentendo il controllo della quantità di DNA erogato e il multiplexing di diversi plasmidi. Infatti, grazie al software nanodispenser (Tabella dei materiali), l'utente può elaborare un piano di studio per controllare i volumi da erogare da una piastra di origine definita a una di destinazione.
Il protocollo qui presentato è destinato principalmente a coloro che hanno accesso a un nanodispenser e vorrebbero impostare esperimenti di trasfezione ad alta produttività, ma anche per coloro che vogliono ottimizzare rapidamente i loro parametri di trasfezione per un determinato tipo di cellula l'applicazione di questo protocollo per eseguire il cross-test di diversi parametri a velocità effettiva elevata. In effetti, abbiamo dimostrato che i parametri ottimizzati identificati con questo protocollo su nanoscala possono essere trasposti in esperimenti di trasfezione su larga scala e manuali. Infine, poiché il reagente di trasfezione utilizzato nel presente protocollo consente la trasfezione del DNA o del siRNA secondo il produttore, il protocollo è anche di interesse per coloro che mirano a eseguire approcci di array per la sovraespressione genica o il knockdown. Le piastre di destinazione precompilate con DNA possono essere conservate fino a 7 giorni prima dell'uso in un saggio di trasfezione senza perdita di efficacia, che è un altro vantaggio del seguente protocollo per questo tipo di applicazione.
Protocollo
1. Preparazione anticipata
- Preparazione dei programmi di gestione dei liquidi peristaltici
NOTA: Per le fasi di diluente e di erogazione cellulare del protocollo, è necessario preparare un programma dedicato, tenendo conto dell'altezza della testa di erogazione alla piastra utilizzata e dell'intento di passo.- Per il passo di diluente 1 L, montare una cassetta da 1 ll e preparare un programma con le impostazioni come descritto nei passaggi 1.1.1.1 e 1.1.1.2.
- Regolare il parametro della portata su Alta per ottenere la migliore velocità effettiva, in quanto non sono previsti danni materiali biologici in questo passaggio. Regolare l'altezza di erogatore a 9,6 mm (secondo la piastra di coltura cellulare utilizzata, Figura supplementare 1) per consentire alla caduta di 1 -L di toccare il fondo dei pozzi durante la dispensazione.
NOTA: Questo passaggio è fondamentale per evitare la ritenzione delle goccioline sulla testa di erogazione fino a raggiungere un volume sufficiente per cadere. - Regolare l'altezza di compensazione della piastra a 14,4 mm per consentire uno spostamento libero della testa di erogazione sopra la piastra dopo l'erogazione di ogni riga. Controlla visivamente le impostazioni corrette dell'altezza della testa del gestore liquido peristale: assicurati che non vengano mantenute gocce sulle punte di erogazione durante l'erogazione e verifica che la testa sia sufficientemente alta da consentire lo spostamento della testa dopo l'erogazione di ogni riga.
NOTA: Evitare la ritenzione di caduta è un parametro cruciale in quanto comprometterà l'accuratezza del volume della dispensazione.
- Regolare il parametro della portata su Alta per ottenere la migliore velocità effettiva, in quanto non sono previsti danni materiali biologici in questo passaggio. Regolare l'altezza di erogatore a 9,6 mm (secondo la piastra di coltura cellulare utilizzata, Figura supplementare 1) per consentire alla caduta di 1 -L di toccare il fondo dei pozzi durante la dispensazione.
- Per l'erogazione delle sospensioni a celle da 40,l, montare una cassetta da 10 l e preparare un programma con le impostazioni descritte nei passaggi 1.1.2.1-1.1.2.2.
- Regolare il parametro della portata su Bassa per erogare le cellule a bassa velocità per evitare di promuovere potenziali danni alle cellule da stress di taglio e un alto impatto sul fondo dei pozzi. Regolare l'altezza di dispensa a 11,43 mm (secondo la piastra di coltura cellulare utilizzata, Figura supplementare 1), abbastanza alta da ridurre l'impatto cellulare sul fondo dei pozzetti durante il processo di erogazione, ma abbastanza bassa da evitare la ritenzione delle goccioline sul testa di erogazione. Regolare l'altezza di cancellazione della piastra a 16 mm per consentire lo spostamento libero della testa di erogazione sopra la piastra dopo l'erogazione di ogni riga.
- Controlla visivamente le impostazioni corrette dell'altezza della testa del gestore liquido peristale: assicurati che non vengano mantenute gocce sulle punte di erogazione durante l'erogazione e verifica che la testa sia sufficientemente alta da consentire lo spostamento della testa dopo l'erogazione di ogni riga.
NOTA: Evitare la ritenzione di goccia è un parametro cruciale in quanto porterà all'erogazione di un numero di cella inaffidabile.
- Per il passo di diluente 1 L, montare una cassetta da 1 ll e preparare un programma con le impostazioni come descritto nei passaggi 1.1.1.1 e 1.1.1.2.
- Preparazione del plasmide del DNA (protocollo di estrazione miniprep classico)
- Coltivare un ceppo di batteri Trasformati dH5 in mezzo LB integrato con 125 g/mL ampicillin selection antibiotic (Tabella dei materiali) durante la notte a 37 gradi centigradi e sotto agitazione delicata (200 giri/) su uno shaker orbitale ( Tabella deimateriali).
- Raccogliere 2 mL della coltura, pellet le cellule da centrifugando per 5 min a 6.000 x g, e scartare il supernatante.
- Risospendere il pellet cellulare con 250 litri di buffer di sospensione contenente RNase A (Tabella dei materiali). Aggiungere 250 l di tampone di lisi e incubare per 5 min a temperatura ambiente, secondo le istruzioni del produttore.
- Interrompere la reazione di lisi aggiungendo 300 l di buffer di neutralizzazione (Tabella dei materiali) e vorriccio a breve. Centrifugare i tubi per 5 min a 11.000 x g.
- Posizionare una nuova minicolonna plasmide (Tabella dei materiali) in un tubo di raccolta da 2 mL e decantare il supernatale nella colonna centrifugando per 1 min a 11.000 x g.
- Eliminare il flusso attraverso e riporre la minicolonna nel tubo di raccolta.
- Lavare la minicolonna plasmide con 500 l di tampone di lavaggio opzionale (Table of Materials) e centrifugare per 1 min a 11.000 x g, secondo le istruzioni del produttore.
- Eliminare il flusso attraverso e posizionare la minicolonna plasmide indietro nel tubo di raccolta.
- Aggiungere 700 l di washing buffer (Table of Materials) integrati con etanolo e centrifuga per 1 min a 11.000 x g, secondo le istruzioni del produttore.
- Scartare il flusso attraverso e centrifugare la minicolonna plasmide e il suo tubo di raccolta 1x più per 2 min a 11.000 x g per asciugare la membrana di silice.
- Mettere la minicolonna di plasmide essiccata in un nuovo tubo da 1,5 mL e aggiungere 30 -L di acqua distillata preriscaldata a 60 gradi centigradi, incubarla per 2 min a temperatura ambiente, quindi centrificarla per 1 min a 11.000 x g.
- Scartare la minicolonna del plasmide e mantenere l'eluato contenente il plasmide di DNA purificato.
- Misurare la concentrazione di DNA del DNA eluito utilizzando uno spettrofotometro a microvolume (Tabella dei materiali).
- Accendere lo spettrofotometro e scegliere le impostazioni di misurazione del DNA.
- Sollevare il braccio di campionamento dello spettrofotometro e della pipetta 1 -L di acqua sul piedistallo di misurazione per eseguire una calibrazione in bianco.
- Abbassare il braccio di campionamento, avviare la misurazione in bianco e attendere il completamento.
- Sollevare il braccio di campionamento e pulire il campione dai piedistalli superiore e inferiore.
- Pipetta 1 - L della soluzione del DNA sul piedistallo inferiore per misurarlo.
- Abbassare il braccio di campionamento, avviare la misurazione della concentrazione del DNA e attendere il completamento.
- Sollevare il braccio di campionamento e pulire il campione dai piedistalli superiore e inferiore.
- Per ulteriori misurazioni della concentrazione del DNA, ripetere i passaggi 1.2.13.5-1.2.13.7.
- Una volta che le misurazioni sono finite, conservare le soluzioni di DNA a 4 gradi centigradi fino all'uso.
2. Progettazione sperimentale e generazione dei picklist per guidare le erogate adE
NOTA: è stata sviluppata una macro di foglio di calcolo "user-friendly" dedicata per gestire le quantità di DNA e mescolare fino a quattro plasmidi in un formato a 384 pozze. Sulla base della progettazione sperimentale inserita, questa macro genera i file necessari per guidare il protocollo di trasfezione del DNA basato su ADE da nanodispenser. Per generare questi file, è necessario compilare diversi campi nel foglio modello, come illustrato nella Figura 2.
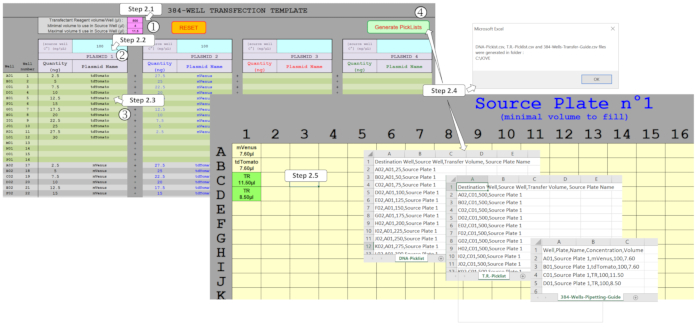
Figura 2 : generazione degli elenchi a discesa per guidare la dispensazione ADE utilizzando la macro del foglio di calcolo. Diversi parametri devono essere riempiti, vale a dire (1) il reagente di refezione di trasfezione (TR) e i volumi minimi/massimi da utilizzare nella piastra di origine, (2) le concentrazioni di plasmide iniziali da erogare nella piastra di origine e (3) il design intero, comprese le quantità di plasmide previste e multiplexing in ciascuno dei 384-pozzi. (4) L'attivazione di Generate Picklists consente di verificare i diversi campi e, una volta compilati correttamente, vengono generati automaticamente elenchi a discesa per la snidatura del DNA e del TR e il necessario modello di piastra sorgente. Fare clic qui per visualizzare una versione più grande di questa figura.
- Immettere i parametri del protocollo nanodispenser nei campi rosa. Impostare il valore della miscela TR (Transfection reagent) su 500 nL. Impostare il valore minimo del volume nei pozzi della piastra di origine su 4. Impostare il volume massimo nei pozzi della piastra di origine a 11,25.
NOTA: il nanodispenser qui utilizzato può trasferire solo un massimo di 500 nL in una corsa di ADE. Questi campi rosa sono precompilati con i valori consigliati, ma possono essere modificati in base alle esigenze dell'utente. - Nei campi blu corrispondenti al DNA sottostante, inserite 100 concentrazioni di inizio DNA ng/L.
NOTA: questo valore è la concentrazione ottimale definita in precedenza, ma può tuttavia essere modificato per esigenze utente diverse. - Immettere la quantità di DNA desiderata nei campi grigio/verde. Inserire gli importi e i nomi dei plasmi per i 384 pozzi, garantendo la stessa ortografia se lo stesso plasmide viene utilizzato in diversi pozzi.
- Generare il progetto della piastra di origine, i file degli elenchi a discesa e il file guida di pipettaggio. Fare clic su Genera elenchi di selezione per consentire alla macro di generare il FILE DNA-Picklist.csv, T.R.-Picklist.csv e 384-Wells-Pipetting-Guide.csv dai dati raccolti nel foglio corrispondente. Se richiesto, correggere i valori delle celle piene di arancia in quanto indica errori o volumi che non possono essere gestiti dal nanodispenser.
- Stampare i modelli dal foglio Piastra di origine. Sono indicati i nomi di plasmide e il volume minimo per riempire i pozzi. Allo stesso modo, i volumi di miscela reagente di trasfezione che dovranno essere riempiti nei seguenti pozzi sono indicati come TR ed evidenziati in verde.
3. Preparazione della piastra di origine del DNA mediante l'applicazione guida di tubazione 384 pozzetti
- Diluire il plasmide di DNA immagazzinato dal punto 1.2.14 a 100 ng/L utilizzando acqua distillata.
- Calibrare la griglia 384-po per le dimensioni piastra: aprire l'applicazione guida tubo 384-po 'su un tablet (Figura 3). Posizionare la piastra di origine sulla griglia sullo schermo inferiore, e nel menu di calibrazione in alto a sinistra, fare clic su o - (o utilizzare il cursore rosso) per migliorare o ridurre le dimensioni della griglia e dei pozzi al fine di regolare i pozzetti verdi ai quattro pozzetti d'angolo della piastra .

Figura 3 : Utilizzo dell'applicazione di guida di pipettaggio a 384 pozzetti. (1) Calibrazione della griglia 384-well alla dimensione della piastra; (2) Montaggio di un adattatore universale di lamiere stampata in 3D sulla tavoletta utilizzando nastro adesivo a doppio lato; (3) Posizionamento della piastra sull'adattatore; (4) Spostamento della griglia per centrarla alla piastra montata. (5) Blocco della fase di calibrazione. (6) Apertura del file 384 wells pipetting guide.csv. (7) Dato l'elenco dei file, l'applicazione indicherà il nome della piastra di origine previsto, il reagente (DNA o reagente di trasfezione), la concentrazione e il volume da erogare nei pozzi bersaglio, che saranno illuminati uno per uno. (8) I pulsanti freccia sinistra e destra consentono all'utente di seguire la guida alla cattura per erogare facilmente i reagenti in base al modello di piastra di origine macro del foglio di calcolo. Fare clic qui per visualizzare una versione più grande di questa figura.
- Utilizzando il nastro fronte/retro, montare l'adattatore di lamiera stampato in 3D sullo schermo per evitare movimenti della lastra di origine durante l'erogazione. Se necessario, spostare la griglia calibrata utilizzando le frecce di rotazione e i pulsanti Su/Giù/Destra/Sinistra per regolare la griglia sullo schermo alla posizione della lamiera. Una volta che le dimensioni della griglia e dei pozzi sono correttamente calibrate e posizionate, spuntare la casella Blocca calibrazione.
- Fare clic su FILE e aprire il file 384 wells pipetting guide.csv. Seguire le istruzioni dello schermo per erogare manualmente il volume indicato del plasmide indicato alla concentrazione indicata nel pozzo bianco evidenziato corrispondente alla destinazione di destinazione appropriata della piastra prevista. Utilizzare le frecce - o per tornare indietro o oltre nel processo di erogazione del DNA. Interrompere l'erogazione quando si raggiunge la prima soluzione reagente trasfusione da caricare.
- Una volta completati i deportanti del DNA, rimuovere la piastra sorgente dall'adattatore. Se devono essere riempite più piastre di origine, posizionare una nuova piastra di origine sull'adattatore e seguire le istruzioni di erogazione. Una volta terminata la dispensazione del DNA, centrificare le lastre di sorgente riempite di DNA (a 1.500 x g per 2 min) per garantire un adeguato livellamento del liquido e rimuovere le bolle che portano all'imprecisione nei trasferimenti basati su ADE.
4. Dispensazione diluente del gestore di liquidi peristaltica 1 svalutazione diluente nella piastra di destinazione
NOTA: Eseguire i passaggi 4.1-4.5 in un armadio di sicurezza biologico.
- Disinfettare la testa della cassetta 1 L spruzzandola con un disinfettante spray (Tabella dei materiali) e consentire a questa soluzione di entrare nel supporto della punta. Assorbire il disinfettante residuo sull'assorbimento della carta. Montare la cassetta 1 L sul dispositivo di gestione del liquido peristaltico. Accendere il dispositivo e assicurarsi che l'impostazione del tipo di cassetta sia corretta (1 o L), così come il formato della piastra (384 pozzi).
- Disinfettare l'intero lume del tubo: inserire l'organizzatore del tubo (tenendo insieme gli otto tubi) in un recipiente sterile e riempirlo con 5 mL di alcool del 70%. Utilizzando la funzione di innesco del gestore liquido peristale, prima lavare l'alcol nel tubo e poi risciacquarlo passando 5 mL di acqua distillata e 5 mL di mezzo senza sieri (il mezzo di Eagle modificato di Dulbecco [DMEM] integrato con 100 U/mL penicillina-streptomycin; Tabella del materiale), riempiendo successivamente lo stesso recipiente. Assicurarsi che nessuna punta sia intasata ispezionando visivamente il flusso di liquido da tutti.
- Prime il tubo con mezzo senza sieri riempiendo un nuovo recipiente sterile con 10 mL di mezzo preriscaldato senza siero e immergendosi l'organizzatore del tubo in esso. Premere il pulsante primo del gestore liquido perificato per circa 10 s. Ancora una volta, assicurarsi che nessuna punta sia intasata ispezionando visivamente il flusso di liquido da tutti.
- Riempire la piastra con 1 -L di diluente. Posizionare una piastra di coltura sterile 384-well (destinazione) sul supporto peristaltico della piastra di gestione liquida e rimuovere il coperchio.
- Eseguire il programma precalibrato per erogare 1 ll in ogni pozzetto della piastra 384-po. Il tempo di erogazione è di circa 8 s. Quindi sostituire il coperchio della piastra 384-pozzetto.
NOTA: In alternativa, questo passaggio può essere gestito manualmente, in un armadio di sicurezza, utilizzando una micropipetta multicanale.
5. Esecuzione di un'indagine per controllare i volumi erogati manualmente
NOTA: per informazioni dettagliate, vedere la figura 4.
- Eseguire il programma nanodispenser, andare alla scheda diagnostica, selezionare la piastra di origine Fuori scatola, caricare la piastra di origine sul supporto piastra, e spuntare In per inserire la piastra. Quando richiesto, selezionare 384LDV_AQ_B2 per impostare il nanodispenser sulla modalità di erogazione del buffer acquosa, quindi premere OK.
- Selezionare Sondaggio nel menu Varie e fare clic su Avvia. Selezionare i pozzi precompilati da analizzare e fare clic sul pulsante Vai. Verificare che i volumi misurati corrispondano a quelli previsti e assicurarsi che non siano stati caricati pozzi con volumi superiori a 12 gradi, in quanto ciò eviterà trasferimenti.
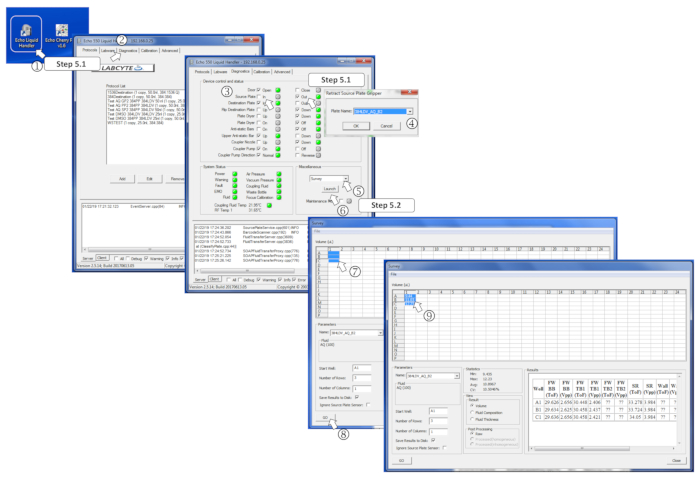
Figura 4 : definizione dei parametri software del sondaggio. (1) Avviare il programma di nanodispenser. (2) Aprire la scheda Diagnostica. (3) Inserire la piastra di origine spuntando fuori per la piastra di origine e quindi, In. (4) Quando richiesto, definire il tipo di piastra di origine nel menu. (5) Nella casella Varie, selezionare Sondaggio nel menu a discesa. (6) Avviare il programma di rilevamento facendo clic su Avvia. (7) Selezionare i pozzi precompilati da misurare. (8) Avviare l'analisi facendo clic su Vai. (9) Una volta eseguita l'indagine, i volumi misurati sono scritti nei pozzi corrispondenti selezionati. Fare clic qui per visualizzare una versione più grande di questa figura.
6. Smilata dal DNA ADE nella piastra di destinazione
- Eseguire il software picklist, impostare i tipi di piastra di origine e di destinazione a 384_well su 384_LDV e Greiner 384PS_781096, rispettivamente (Figura 5). Impostare il dispositivo su una modalità di erogazione del buffer acquosa selezionando 384LDV_AQ_B2e deselezionare "ottimizza la velocità effettiva di trasferimento".

Figura 5 : esecuzione delle dispensazioni basate su elenchi a discesa. (1) Avviare il software nanodispenser. Nella scheda Protocollo selezionare (2) il formato della piastra di esempio, (3) il tipo di piastra di destinazione e (4) viene deselezionata "Ottimizza velocità effettiva trasferimento". (5) Selezionare la scheda Distinta di prelievo. (6) Fare clic su Importa e selezionare il file .csv appropriato (DNA-PickList o T.R.-Picklist). (7) Una volta selezionato, fare clic su Importa. (8) Fare clic su Riproduci e salvare il protocollo. (9) Eseguire una simulazione di srogazione facendo clic su Simula, o (10) Avviare la dispensazione programmata facendo clic su Esegui. Fare clic qui per visualizzare una versione più grande di questa figura.
- Selezionare la scheda "Lista di prelievo", fare clic su Importa, selezionare il file DNA-Picklist.csv. Fare clic su Riproduci e salvare il protocollo. Fare clic su Simula per eseguire una simulazione delle dispensazioni programmate per assicurarsi che il elenco a discesa corrisponda al progetto sperimentale previsto. Una volta completato, fare clic su Chiudi.
- Fare clic su Riproduci, quindi Esegui, per avviare il programma di erogazione: quando richiesto, inserire la piastra sorgente richiesta (soluzioni DNA riempite manualmente) e la piastra di destinazione (riempita con diluente) nel nanodispenser.
NOTA: Il tempo di erogazione è di circa 5-20 minuti per una piastra completa di 384 pozze, a seconda dei volumi selezionati e del numero totale di dispensazioni nel progetto sperimentale. - In alternativa, mettere in pausa il protocollo qui come le piastre diluenti e piene di DNA in grado di gestire lo stoccaggio asciutto o congelato per un massimo di 7 giorni. Per lo stoccaggio a secco, lasciare asciugare i piatti sul banco a temperatura ambiente e poi conservarli allo stesso modo. Scongelare e centrifugare (a 1.500 x g per 2 min) piastre conservate congelate prima dell'uso in una fase di trasfezione (sezione 7).
7. Dispensazione reagente di trasfusione guidata da ADE
- In un armadietto di biosicurezza, reagente di trasfezione lipopoliplex estemporaneamente diluito nel mezzo senza siero a una concentrazione finale di 1x. Vortice e immediatamente erogare questa miscela reagente trasfezione secondo le piastre di origine predefinite progettate dalla macro e utilizzando l'applicazione guida di pipettaggio 384 ben descritta come descritto nel passaggio 3.4.
NOTA: Non centrifugare la piastra di origine una volta caricata con il reagente in quanto non si nota alcuna trasfezione dopo la centrifugazione. - Eseguire il programma nanodispenser per eseguire un "sondaggio" come descritto nella sezione 5, al fine di controllare i volumi di tutti i pozzi TR riempiti manualmente delle piastre di origine per evitare errori di erogazione dovuti a volumi superiori a 12 .
- Fare clic su Reimposta per cancellare l'elenco di selezione del DNA nel software di selezione e verificare che i parametri del dispositivo siano ancora impostati su buffer acquosi e sui tipi di piastra di origine e di destinazione utilizzati, come nel passaggio 6.1.
- Fare clic su Importa e scegliere il file TR-Picklist.csv. Fare clic su Riproduci e salvare il protocollo, se richiesto, e (questo è opzionalmente ma fortemente raccomandato) eseguire una simulazione delle dispensazioni miscela reagente di trasfusione programmata per garantire la corretta progettazione dei dispensazioni facendo clic sul Pulsante Simula. Una volta completato, fare clic su Chiudi.
- Fare clic su Riproduci, quindi sul pulsante Esegui per avviare il programma di erogazione: come richiesto, posizionare le lastre di origine (riempite con MISCELA TR) e la piastra di destinazione (diluente e riempita di DNA) nel nanodispenser.
NOTA: Il tempo di erogazione è inferiore a 20 min per una piastra completa di 384 pozze durante l'erogazione di 500 nL di miscela TR. - Incubare 15-30 min a temperatura ambiente dopo aver aggiunto il TR al DNA come indicato dal protocollo del produttore.
8. Dispensazione cellulare basata su gestore di liquidi peristaltici
- Preparare il gestore di liquidi peristaltico per l'erogazione delle cellule. Disinfettare la testa di una cassetta da 10 l spruzzandola con Aniospray Surf 29 Disinfectant e assorbendo il resto sulla carta. Montare la cassetta sul dispositivo di gestione dei liquidi peristalitici, modificare l'impostazione del tipo di cassetta su 10 , e assicurarsi che il formato della piastra sia impostato su 384 pozzi.
- Disinfettare il tubo di cassette da 10 L come descritto in precedenza al punto 4.2. Immergi l'organizzatore del tubo in un recipiente sterile e sciacqua il tubo con 5 mL di alcool del 70%, poi con 5 mL di acqua distillata, e infine, con 5 mL di mezzo senza siero, successivamente riempito nello stesso recipiente e fino a quando ogni tubo è vuoto.
- Preparare la sospensione cellulare a erogazione. Da un piatto confluente di cellule HeLa B10, lavare le cellule 1x con 1x soluzione salina con tamponamenti fosfati (PBS), quindi dissociare le cellule con la metapsina / EDTA per 5 min a 37 .
- Verificare la dissociazione cellulare al microscopio e arrestare l'azione trypsin/EDTA aggiungendo 10 mL di mezzo completo (DMEM integrato con 10% siero bovino fetale e 100 U/mL penicillina-streptomica; vedere la Tabella del Materiale) nella parabola di coltura. Raccogliere le cellule in un tubo da 50 mL e contare le cellule al microscopio, utilizzando una cellula di Malassez o un contatore cellulare automatico.
- Preparare almeno 25 mL di sospensione cellulare HeLa ad una concentrazione di 37.500 cellule/mL nel mezzo completo (ad es. 1.500 cellule/40 l) per una piastra completa di 384 pozzetti, per garantire l'innesco del tubo e la dispensazione di 40 l/po.
- Per erogare le cellule, riempire un nuovo vaso sterile con la sospensione cellulare preparata e mescolarlo per evitare sedimentazioni che portano a imprecisione nella densità cellulare della dispensazione. Inserire l'organizzatore del tubo in questa soluzione e premere il pulsante Prime fino a quando la sospensione cellulare inizia a lavare dalla testa di erogazione. Assicurarsi che nessuna punta sia intasata ispezionando visivamente il flusso di liquido da tutti e che ogni tubo sia caricato con sospensioni cellulari.
- Caricare il DNA e la piastra di destinazione riempita da TR da 384 pozzetti sul supporto peristaltico peristaltico per la piastra di gestione del liquido e rimuovere il coperchio. Eseguire il programma precalibrato per erogare 40 -L della sospensione cellulare sulla piastra completa 384 pozze (cioè 1.500 cellule/pozzo). Il tempo di erogazione è di circa 8 s. Sostituire il coperchio della piastra 384-bene.
NOTA: In alternativa, la sospensione a celle da 40 once può essere erogata manualmente utilizzando una micropipetta multicanale.
9. Analisi biologica personalizzata (monitoraggio dell'efficienza della trasfezione cellulare)
NOTA: seguendo le impostazioni sperimentali e l'intento dell'esperimento, utilizzare i metodi necessari per la luminescenza, la fluorescenza, lo screening ad alto contenuto e la reazione a catena quantitativa di trascrizione inversa (RT-qPCR). In questa sezione del protocollo, l'efficienza della trasfezione cellulare viene valutata mediante microscopia a fluorescenza automatizzata e analisi delle immagini.
- Incubare la piastra a 37 gradi centigradi con il 5% di CO2 in un'atmosfera satura d'acqua e fino a una corretta espressione proteica.
NOTA: Qui, un tempo di incubazione di 48 h viene utilizzato per le cellule HeLa per monitorare l'efficienza della trasfezione, utilizzando plasmidi che esprimono tdTomato e mVenus. - Rimuovere il mezzo di coltura 48 h post-trasfezione invertendo la piastra, aggiungere 30 l/pozzetto di 10% di formalina utilizzando il gestore liquido peristaltico (10 - cassetta l') e incubare per 15 min a temperatura ambiente.
- Rimuovere la formalina invertendo la piastra; quindi incubare le cellule per 15 min a temperatura ambiente con 0,1 ng/mL Hoechst diluito in soluzione 1x PBS.
- Lavare le cellule 3x per 15 min con 80 luna di 1x PBS regolate a pH - 8 al fine di recuperare il segnale ad alta fluorescenza perso dal 6,9 pH della fase di incubazione della soluzione di formalina.
- Utilizzando un microscopio fluorescente automatizzato, acquisire le immagini di due o tre canali fluorescenti (Hoechst, tdTomato e mVenus) in sequenza con obiettivi 10x e un set di filtri di emissione adeguato (4,6-diamidino-2-fenilitine [DAPI], dsRed e fluoresceina isothiocyanate [FITC], rispettivamente).
- Per valutare l'efficienza della trafezione, utilizzare il software di analisi delle immagini per determinare l'efficienza della trafezione utilizzando l'analisi dello script basata sulla colorazione dei nuclei.
Risultati
Al fine di determinare se la tecnologia ADE potrebbe essere utilizzata per un protocollo di trasfezione inversa automatizzato, abbiamo monitorato l'efficienza della trasfezione cellulare mediante microscopia a fluorescenza, utilizzando un plasmide fluorescente rosso fluorescente. In primo luogo puntando a determinare i migliori parametri di trasfezione, diversi volumi diluenti e quantità totali di DNA sono stati testati in modo incrociato. Il volume diluente è stato utilizzato per conse...
Discussione
L'istituzione e l'ottimizzazione di un accurato metodo di trasflessazione ad alto throughput per una determinata linea cellulare richiedono agli scienziati di seguire alcuni parametri chiave descritti in questa sezione. Incoraggiamo vivamente a partire dai valori raccomandati in tutto il protocollo poiché queste impostazioni ottimizzate per le cellule HeLa si sono dimostrate efficienti anche per le cellule HEK. Tuttavia, poiché i parametri migliori possono dipendere dalle linee cellulari e dai reagenti di trasfezione, ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori hanno rivelato una ricevuta del seguente sostegno finanziario per la ricerca, la paternità e/o la pubblicazione di questo articolo: Inserm, Lille University, Lille Pasteur Institute, Conseil Régional du Nord, e PRIM-HCV1 e 2 (P Interdisciplinaire sur le Médicament), Agence Nationale de la Recherche (ANR-10-EQPX-04-01), la Feder (12001407 (D-AL) Equipex Imaginex BioMed) e la Comunità Europea (ERC-STG INTRACELL nTB 260901). Gli autori desiderano ringraziare il Dr. S. Moureu, il Dr. B. Villemagne, il Dr. R. Ferru-Clément e il Dr. H. Groult per la loro revisione critica e le correzioni del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| 384LDV Microplate | Labcyte | LP-0200 | |
| 384-well Microplate μClear Black | Greiner | 781906 | |
| Ampicilin | Sigma | A9393-5G | Selection antibiotic for bacteria transformed with ampicilin expressing vector |
| Android Tablet | Samsung | Galaxy Note 8 | used to guide the user while the source plate manual dispense |
| Aniospray Surf 29 | Anios | 2421073 | disinfectant to clean the MicroFlo head |
| Columbus software | Perkin Elmer | image analysis software | |
| Dulbecco's Modified Eagle Medium (DMEM), high glucose, GlutaMAX Supplement, pyruvate | Thermo Fisher Scientific | 10566032 | cell culture medium |
| Echo Cherry Pick 1.5.3 software | Labcyte | Software enabling ADE-based dispenses by the Echo550 device from a *.csv file; nanodispenser software | |
| Echo550 | Labcyte | ADE-based dispenser | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | to add in cell culture medium |
| Formalin solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | to fix cell |
| HeLa cells | ATCC | HeLa (ATCC® CCL-2™) | |
| Hoechst 33342, Trihydrochloride, Trihydrate | Thermo Fisher Scientific | H3570 | 10 mg/mL Solution in Water |
| INCell Analyzer 6000 | GE Healthcare | 29043323 | automated laser-based confocal imaging platform |
| LB medium | Thermoischer Scientific LB Broth Base (Lennox L Broth Base)®, powder | 12780052 | culture medium for bacteria growth |
| Lysis Buffer (A2) | Macherey-Nagel | 740912.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| MicroFlo 10µL cassette | Biotek Instruments Inc | 7170013 | to use with the Microflo Dispenser |
| MicroFlo 1μL cassette | Biotek Instruments Inc | 7170012 | to use with the Microflo Dispenser |
| MicroFlo Dispenser | Biotek Instruments Inc | 7171000 | peristaltic pump-based liquid handler device |
| Microvolume spectrophotometer | Denovix | DS-11 Spectrophotometer | Measure the DNA concentration of samples |
| mVenus plasmid | mVenus cDNA was cloned by enzymatic restriction digestion and ligation in Age1/BsrG1 sites of the tdTomato-N1 plasmid | Vector type: Mammalian Expression, Fusion Protein: mVenus | |
| Neutralization Buffer (A3) | Macherey-Nagel | 740913.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| NucleoSpin Plasmid kit | Macherey-Nagel | 740588.50 | used to prepare plasmid from bacterial culture |
| Optimal-Modified Eagle Medium (Opti-MEM) Medium | Thermo Fisher Scientific | 31985070 | |
| optional Wash bufferWash Buffer (A4) | Macherey-Nagel | 740914.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| orbital shaker | incubated large capacity shaker | 444-7084 | Used to grow bacteria under gentle agitation and 37°C |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | 10,000 U/mL |
| Phosphate-Buffered Saline | Thermo Fisher Scientific | 10010001 | |
| Plasmid mini-columns | Macherey-Nagel | 740499.250 | Silica membrane mini-column to prepare plasmid from bacterial culture |
| Resuspension Buffer (A1) | Macherey-Nagel | 740911.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| RNAse A | Macherey-Nagel | 740505 | Enzyme from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| tdTomato-N1 plasmid | Addgene | Plasmid #54642 | Vector type: Mammalian Expression, Fusion Protein: tdTomato |
| TransIT-X2 Dynamic Delivery System | Mirus Bio | MIR 6000 | |
| Wash Buffer (AW) | Macherey-Nagel | 740916.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| 3D printer | Creality | CR10S | used to print the plate adapter |
| Blender Software | https://www.blender.org/ Free software under GNU General Public License (GPL). | version 2.79b | used to design the plate adapter |
Riferimenti
- Colin, B., Deprez, B., Couturier, C. High-Throughput DNA Plasmid Transfection Using Acoustic Droplet Ejection Technology. SLAS Discovery: Advancing Life Sciences R & D. , 2472555218803064 (2018).
- Mirus Bio. . Optimising Transfection Performance. , (2019).
- Thermo Fisher Scientific. . Factors Influencing Transfection Efficiency | Thermo Fisher Scientific - FR. , (2019).
- Boussif, O., et al. A versatile vector for gene and oligonucleotide transfer into cells in culture and in vivo: polyethylenimine. Proceedings of the National Academy of Sciences of the United States of America. 92 (16), 7297-7301 (1995).
- Figueroa, E., et al. A mechanistic investigation exploring the differential transfection efficiencies between the easy-to-transfect SK-BR3 and difficult-to-transfect CT26 cell lines. Journal of Nanobiotechnology. 15 (1), 36 (2017).
- Kirchenbuechler, I., Kirchenbuechler, D., Elbaum, M. Correlation between cationic lipid-based transfection and cell division. Experimental Cell Research. 345 (1), 1-5 (2016).
- Zhang, Z., Qiu, S., Zhang, X., Chen, W. Optimized DNA electroporation for primary human T cell engineering. BMC Biotechnology. 18 (1), 4 (2018).
- Cao, D., et al. Transfection activity and the mechanism of pDNA-complexes based on the hybrid of low-generation PAMAM and branched PEI-1.8k. Molecular bioSystems. 9 (12), 3175-3186 (2013).
- Bos, A. B., et al. Development of a semi-automated high throughput transient transfection system. Journal of Biotechnology. 180, 10-16 (2014).
- Colosimo, A., et al. Transfer and expression of foreign genes in mammalian cells. BioTechniques. 29 (2), 314-318 (2000).
- Villa-Diaz, L. G., Garcia-Perez, J. L., Krebsbach, P. H. Enhanced transfection efficiency of human embryonic stem cells by the incorporation of DNA liposomes in extracellular matrix. Stem Cells and Development. 19 (12), 1949-1957 (2010).
- Sabatini, D. M. . Reverse transfection method. , WO2001020015A1 (2001).
- Raymond, C., et al. A simplified polyethylenimine-mediated transfection process for large-scale and high-throughput applications. Methods (San Diego, CA). 55 (1), 44-51 (2011).
- Junquera, E., Aicart, E. Recent progress in gene therapy to deliver nucleic acids with multivalent cationic vectors. Advances in Colloid and Interface Science. 233, 161-175 (2016).
- Woodruff, K., Maerkl, S. J. A High-Throughput Microfluidic Platform for Mammalian Cell Transfection and Culturing. Scientific Reports. 6, 23937 (2016).
- Vasileiou, T., Foresti, D., Bayram, A., Poulikakos, D., Ferrari, A. Toward Contactless Biology: Acoustophoretic DNA Transfection. Scientific Reports. 6, 20023 (2016).
- Hadimioglu, B., Stearns, R., Ellson, R. Moving Liquids with Sound: The Physics of Acoustic Droplet Ejection for Robust Laboratory Automation in Life Sciences. Journal of Laboratory Automation. 21 (1), 4-18 (2016).
- Grant, R. J., et al. Achieving accurate compound concentration in cell-based screening: validation of acoustic droplet ejection technology. Journal of Biomolecular Screening. 14 (5), 452-459 (2009).
- Sackmann, E. K., et al. Technologies That Enable Accurate and Precise Nano- to Milliliter-Scale Liquid Dispensing of Aqueous Reagents Using Acoustic Droplet Ejection. Journal of Laboratory Automation. 21 (1), 166-177 (2016).
- Day, R. N., Davidson, M. W. The fluorescent protein palette: tools for cellular imaging. Chemical Society Reviews. 38 (10), 2887-2921 (2009).
- Zielinski, D., Gordon, A., Zaks, B. L., Erlich, Y. iPipet: sample handling using a tablet. Nature Methods. 11 (8), 784-785 (2014).
- Brunner, S., et al. Cell cycle dependence of gene transfer by lipoplex, polyplex and recombinant adenovirus. Gene Therapy. 7 (5), 401-407 (2000).
- Nii, T., et al. Single-Cell-State Culture of Human Pluripotent Stem Cells Increases Transfection Efficiency. BioResearch Open Access. 5 (1), 127-136 (2016).
- Noonan, D. J., Henry, K., Twaroski, M. L. A High-Throughput Mammalian Cell-Based Transient Transfection Assay. Signal Transduction Protocols. 284, 051-066 (2004).
- . . Transfection | TransIT Transfection Reagents | Mirus Bio. , (2015).
- American Type Culture Collection. . General protocol for transfection of stem cells, primary cells, and continuous cell lines with ATCC TransfeX Transfection Reagent. , (2017).
- American Type Culture Collection. . Transfection Reagents for Nucleic Acid Transfer into ATCC Cells. , (2017).
- de Los Milagros Bassani Molinas, M., Beer, C., Hesse, F., Wirth, M., Wagner, R. Optimizing the transient transfection process of HEK-293 suspension cells for protein production by nucleotide ratio monitoring. Cytotechnology. 66 (3), 493-514 (2014).
- Promega. . FuGENE® 6 Transfection Reagent. , (2019).
- Olden, B. R., Cheng, Y., Yu, J. L., Pun, S. H. Cationic polymers for non-viral gene delivery to human T cells. Journal of Controlled Release: Official Journal of the Controlled Release Society. 282, 140-147 (2018).
- Park, E., Cho, H. B., Takimoto, K. Effective gene delivery into adipose-derived stem cells: transfection of cells in suspension with the use of a nuclear localization signal peptide-conjugated polyethylenimine. Cytotherapy. 17 (5), 536-542 (2015).
- Wood, R. W., Loomis, A. L. The physical and biological effects of high-frequency sound-waves of great intensity. The London, Edinburgh, and Dublin Philosophical Magazine and Journal of Science. 4 (22), 417-436 (1927).
- Mamat, U., et al. Eliminating Endotoxin at the Source - A Novel Competent Cell Line with Modified Lipopolysaccharide for Low-Endotoxin Plasmid Production. , (2014).
- Ivanova, N. V., Kuzmina, M. L. Protocols for dry DNA storage and shipment at room temperature. Molecular Ecology Resources. 13 (5), 890-898 (2013).
- Lesnick, J., Lejeune-Dodge, A., Ruppert, N., Jarman, C. . High-Precision Cell Dispensing with the Labcyte Echo® Liquid Handler. , (2017).
- Yang, X., et al. A public genome-scale lentiviral expression library of human ORFs. Nature Methods. 8 (8), 659-661 (2011).
- Peng, J., Zhou, Y., Zhu, S., Wei, W. High-throughput screens in mammalian cells using the CRISPR-Cas9 system. The FEBS journal. 282 (11), 2089-2096 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon