Method Article
Mappatura dell'Emergente Organizzazione Spaziale delle Cellule Mammarie utilizzando Micropatterns e Quantitative Imaging
In questo articolo
Riepilogo
Il metodo qui presentato utilizza il micromodellamento insieme all'imaging quantitativo per rivelare l'organizzazione spaziale all'interno delle culture dei mammiferi. La tecnica è facile da stabilire in un laboratorio di biologia cellulare standard e offre un sistema trattabile per studiare patterning in vitro.
Abstract
Un obiettivo fondamentale in biologia è capire come emergono i modelli durante lo sviluppo. Diversi gruppi hanno dimostrato che la creazione di modelli può essere realizzata in vitro quando le cellule staminali sono spazialmente confinate su micromodelli, creando così modelli sperimentali che offrono opportunità uniche per identificare, in vitro, i principi fondamentali della Organizzazione.
Qui descriviamo la nostra implementazione della metodologia. Abbiamo adattato una tecnica di fotomodellamento per ridurre la necessità di attrezzature specializzate per rendere più facile stabilire il metodo in un laboratorio di biologia cellulare standard. Abbiamo anche sviluppato un framework di analisi delle immagini gratuito, open-source e facile da installare per misurare con precisione il posizionamento preferenziale delle sottopopolazioni di cellule all'interno di colonie di forme e dimensioni standard. Questo metodo permette di rivelare l'esistenza di eventi di patterning anche in popolazioni di cellule apparentemente disorganizzate. La tecnica fornisce informazioni quantitative e può essere utilizzata per disaccoppiare le influenze dell'ambiente (ad esempio, segnali fisici o segnali endogeni), su un determinato processo di modellazione.
Introduzione
Nei sistemi dei mammiferi, il patterning è una proprietà emergente del comportamento collettivo delle cellule e quindi, i modelli possono formarsi in vitro se vengono forniti segnali appropriati alle celle1,2,3,4, 5 Del numero 3( , 6. Un modo per rivelare la capacità intrinseca delle cellule di auto-organizzarsi in vitro è quello di costringere le cellule a formare gruppi/colonie di forme e dimensioni definite7,8,9,10 . Una tecnica che consente questo è micropatterning11. La micropatterning consente di definire con precisione la posizione in cui le molecole di matrice extracellulare (ECM) vengono depositate su una superficie. Questo, a sua volta determina dove le cellule possono aderire e quindi controlla come le cellule si organizzano spazialmente.
Il micropatterning è una tecnica con numerose applicazioni, ad esempio il micropatterning consente la standardizzazione delle condizioni iniziali prima della differenziazione12. È importante sottolineare che il micropatterning consente di controllare facilmente le dimensioni, la forma e la spaziatura delle colonie cellulari e questa proprietà può essere utilizzata per elaborare esperimenti volti a interrogare la risposta collettiva delle cellule al morfogeno o ai segnali fisici7 , 8 (IN vio , 10 del sistema , 13 del sistema , 14 Del sistema , 15 Mi lasa del sistema , 16 , 17.
Sono stati sviluppati diversi metodi di micromodellazione11. Le tecniche di fotomodellazione sono forse i metodi più semplici per stabilire18. Questi approcci hanno anche il vantaggio di precisione in quanto possono essere utilizzati per controllare la forma delle singole celle18,19,20. Tuttavia, richiedono anche costose attrezzature specializzate, tra cui un rivestimento di spin, una camera al plasma e un detergente UVO (UV-Ozone) che generalmente non sono prontamente disponibili nei laboratori di biologia standard. Per facilitare l'adozione della tecnica, abbiamo adattato il protocollo per richiedere solo la lampada UVO. Iniziamo da vetrini di plastica disponibili in commercio che possono essere tagliati con forbici o con un foro per il formato desiderato.
Un'importante utilità dei micromodelli è la capacità di standardizzare le colonie al fine di confrontare le singole colonie tra più repliche. Questo permette di chiedere in che misura la formazione del modello all'interno di queste colonie è riproducibile, ed esplorare i fattori che influenzano la robustezza del processo di modellazione. È importante sottolineare che la quantificazione dei modelli "mediati" in più colonie standardizzate può anche rivelare processi di modellazione che altrimenti non sarebbero evidenti. Il vantaggio di essere in grado di quantificare il patterning sulle colonie standardizzate dipende dalla possibilità di misurare con precisione l'espressione proteica, idealmente a livello di singola cellula. Tuttavia, le cellule su micromodelli sono spesso strettamente imballate, rendendole difficili da segmentare con alta precisione. Le cellule spesso si organizzano in tre dimensioni anziché in due dimensioni e può essere difficile rilevare e conservare informazioni tridimensionali (3D) durante la segmentazione. Una volta che le celle sono state segmentate con successo, sono necessari metodi di calcolo per estrarre le informazioni di patterning dai set di dati risultanti.
Abbiamo sviluppato strumenti di segmentazione e analisi delle immagini per aiutare a superare questi problemi. Questo metodo di analisi utilizza solo software libero e open-source e non richiede la conoscenza della riga di comando o della programmazione per implementare. Per illustrare il metodo qui, usiamo cellule staminali embrionali di topo (mES) che esprimono spontaneamente un marcatore di brachiury di differenziazione precoce (Tbra)21,22. Anche se nessuna disposizione spaziale apparente è visivamente rilevabile, il metodo consente la creazione di una mappa del posizionamento preferenziale delle cellule di T . Mostriamo anche che il patterning Tbra contrasta con l'assenza di una localizzazione preferenziale delle cellule che esprimono Id1, una lettura diretta del percorso della proteina morfogenetica ossea (BMP)23. Discutiamo anche le attuali limitazioni del metodo e come questa tecnica può essere adattata ad altri sistemi sperimentali.
Protocollo
NOTA: una panoramica del metodo è disponibile nella Figura 1.
1. Design della maschera
- Progettare la maschera fotografica secondo le linee guida descritte in Azioune et al. Consultare la Tabella dei materiali per un riferimento al produttore del software e della maschera utilizzato per questo studio.
NOTA: è possibile creare più geometrie, dimensioni o spaziatura tra le forme su una maschera. La lampada UV può contenere una fotomaschera da 15 cm che può contenere fino a 49 disegni diversi (presupponendo chip 2 cm x 2 cm).
2. Procedura di produzione di micropattern
- Preparare i materiali necessari.
- Preparare una soluzione di 0,1% poloxamer 407 (10 mg per 10 mL) in salina tampone di fosfato (PBS) e lasciare su uno shaker a temperatura ambiente. Il poloxamer 407 richiederà circa 20 min per sciogliersi.
- Laici pellicole da laboratorio (vedi la Tabella deiMateriali) sul fondo di un piatto Petri di 10 cm quadrati. Questo sarà utilizzato come camera per la deposizione di matrice.
- Pulire la superficie della fotomaschera, prima con 100% acetone, poi con 100% isopropanolo e infine con ddH2O. Se possibile, asciugare l'aria la fotomaschera o altrimenti asciugare la maschera con un tovagliolo di carta pulito.
- Preparare un pezzo di plastica quadrato, rigido e opaco con le stesse dimensioni della fotomaschera (in seguito indicato come 'titolare').
NOTA: Questo verrà utilizzato per mantenere coperture in plasticaalips a contatto con la fotomaschera durante la fase di illuminazione. - Accendere la lampada UVO ed eseguire un'illuminazione di riscaldamento per 10 min.
- Crea chip fotomodellati.
NOTA: È possibile adattare la procedura per creare chip di qualsiasi dimensione desiderata. Per semplicità, descriviamo qui la procedura per generare un chip micropattern rotondo da 12 mm.- Utilizzando un punzone da 12 mm, tagliare i vetrini di plastica idrofobica per creare 12 mm di copricapi rotondi e metterli in una nuova parabola Petri pulita.
AVVISO: Utilizzare i guanti in ogni momento per evitare il contatto della pelle con la superficie della plastica in quanto ciò potrebbe danneggiare il trattamento superficiale. - Rimuovere con attenzione la pellicola protettiva dalle coperture con una pinzetta.
NOTA: Evitare di danneggiare la superficie di plastica in quanto ciò potrebbe influenzare il posizionamento delle cellule sul chip durante la procedura di semina (sezione 3). - Posizionare la fotomaschera su una superficie pulita e stabile (ad esempio, scatola fotomaschera), lato cromato rivolto verso l'alto e aggiungere una goccia di 2 ddH2O nella posizione del disegno del chip desiderato.
- Posare un coperchio sulla goccia di ddH2O e premere delicatamente.
NOTA: Assicurarsi che il lato di plastica che si affaccia sulla fotomaschera sia il lato che è stato protetto dalla pellicola che è stata rimossa nel passaggio precedente. - Posizionare il supporto sopra i vetrini di plastica e fissare con attenzione questo panino con morsetti al fine di mantenere i pezzi di plastica a contatto con la fotomaschera.
NOTA: Posizionare i morsetti il più vicino possibile alla posizione dei vetrini di plastica per garantire che i vetrini di plastica siano perfettamente mantenuti a contatto con la superficie della fotomaschera. - Posizionare l'assieme nella lampada UVO a circa 2 cm dalla sorgente luminosa e illuminare per 10 min.
NOTA: La potenza della luce è stimata a 6 mW/cm2 a 254 nm di lunghezza d'onda quando il chip è posizionato ad una distanza di 2 cm dalla sorgente. - Tenere il panino con la fotomaschera nella parte inferiore e rimuovere con attenzione i morsetti mantenendo la pressione con una mano per evitare che i vetrini si muovano durante lo smontaggio del panino. Rimuovere il supporto, assicurandosi che tutti i pezzi di plastica siano ancora sulla maschera e non attaccati al supporto.
- Aggiungere ddH2O sopra i chip e staccare delicatamente i trucioli dalla fotomaschera.
NOTA: Se il chip di plastica è attaccato alla fotomaschera, staccare il chip utilizzando una punta di pipetta di plastica per spingere il chip tenendo le pinzette leggermente sopra il chip nel caso in cui il chip si stacchi improvvisamente. - Infine, posizionare i chip fotomodellati all'interno della camera di deposizione a matrice.
NOTA: Assicurarsi che il lato illuminato del chip sia rivolto verso l'alto.
- Utilizzando un punzone da 12 mm, tagliare i vetrini di plastica idrofobica per creare 12 mm di copricapi rotondi e metterli in una nuova parabola Petri pulita.
- Depositare la matrice.
NOTA: Tutte le procedure descritte in questa sezione devono essere eseguite in una cappa di coltura tissutale.- Filtrare la soluzione poloxamer 407 con un filtro in poliethersulfone (PES) da 0,22 m.
- Preparare la soluzione di rivestimento ECM mescolando 500 g/mL di poloxamer filtrato sterile 407 e 1 mg/mL di gelatina.
NOTA: Vedere anche la Tabella 1 per ulteriori informazioni su altre possibili molecole ECM. - Aggiungere 200 l della soluzione di rivestimento su ogni chip illuminato. La pellicola di laboratorio impedirà che la caduta cada al di fuori del chip.
- Aggiungere un piatto di Petri di 3 cm confortato con ddH2O per limitare l'evaporazione e posizionarlo con il chip a 4 gradi centigradi durante la notte.
3. Procedura di seeding
NOTA: i passaggi descritti di seguito sono stati ottimizzati per le cellule staminali embrionali del tono CGR8 (mESC)24 utilizzando il supporto mESC standard (vedi anche la Tabella dei Materiali). Tuttavia, in linea di principio è possibile adattare la procedura per qualsiasi tipo di cellula. Si noti inoltre che la coltura cellulare convenzionale delle cellule staminali embrionali del topo non è descritta qui come una documentazione approfondita può essere trovata altrove25.
- Aspirare la soluzione di rivestimento e incubare i trucioli due volte per almeno 5 min con PBS sterile.
- Nel frattempo, preparare una sospensione cellulare di 5,5 x 105 celle / mL nel mezzo caldo.
- Pipet 200 - L di sospensione cellulare su ogni chip (100.000 celle/cm2).
- Chiudere la camera di semina e lasciare che le cellule aderiscano per 1 h nell'incubatrice.
- Dopo 1 h, riempire i pozzetti di una piastra multiwell (4-bene o 24-bene a seconda del numero di trucioli) con 500 l/pozzedi di mezzo caldo e trasferire i trucioli nella piastra con una pinzetta sterile.
- Agitare vigorosamente la piastra per staccare le cellule non aderenti. Aspirare il mezzo e sostituire immediatamente con un mezzo caldo fresco. Controllare al microscopio per verificare se la modellistica è visibile (Figura 2a).
NOTA: potrebbe essere necessario ottimizzare il tempo di adesione quando si utilizzano linee cellulari diverse da mESC o altre proteine a matrice (vedere anche tabella 2). - Ripetere questo passaggio fino a quando i modelli diventano chiaramente visibili come illustrato nella Figura 2a.
NOTA: questo passaggio è fondamentale e determina l'esito positivo della procedura. Una procedura di lavaggio troppo intensa può staccare le cellule, al contrario un lavaggio insufficiente può causare la permansi di cellule tra i modelli (Figura 2c,d).
4. Fissazione
NOTA: Dopo 48 h in coltura le cellule dovrebbero formare colonie dense che seguono rigorosamente la forma dei modelli (come mostrato nella Figura 2b).
- Lasciando i trucioli nella piastra, rimuovere il 90% del mezzo, lasciando abbastanza mezzo per evitare che i trucioli si asciughino.
NOTA: È importante che il chip non si asciughi mai per evitare artefatti colorazione e per evitare il distacco cellulare dalla superficie. A causa dell'idrofobicità della superficie del chip tra modelli adesivi, il chip può avere una tendenza a de-umido. In questa fase, il fissativo può causare il distacco di grandi colonie a cupola dal chip. I fusti dovrebbero essere molto delicati, eseguiti idealmente pipetting liquid sul lato del pozzo e non direttamente sul chip. - Aggiungere almeno 500 l di paraformaldeide (PFA)-soluzione di fissazione a base di bene e incubare per 10 min.
NOTA: Se le colonie appaiono particolarmente spesse (più di 5 strati cellulari), potrebbe essere necessario regolare il tempo di fissaggio a 20 min. - Dopo la fissazione, lavare 3 volte con la soluzione di lavaggio (PBS con 0.01% poloxamer 407). Un lavaggio supplementare con 50 mM NH4Cl diluito in soluzione di lavaggio può essere intercalato per disinnescare l'attività residua di collegamento incrociato PFA.
- Incubare i campioni per almeno 30 min nella soluzione di blocco.
NOTA: In questa fase, i campioni possono essere conservati a 4 gradi centigradi per circa una settimana prima della colorazione. In tal caso, sigillare la piastra con pellicola di laboratorio per evitare l'evaporazione.
5. Immunostaining
- Preparare una camera di colorazione posizionando un foglio di pellicola da laboratorio nella parte inferiore di un piatto Petri di 10 cm quadrati.
- Preparare le soluzioni anticorpali (vedere la tabella 3 per un elenco di anticorpi e diluizioni utilizzate in questo articolo).
- Posizionare il chip nella camera di colorazione con il lato che sostiene le cellule rivolte verso l'alto e aggiungere immediatamente 100 l di soluzione anticorpale primaria sul chip.
AVVISO: In questa fase, i chip non dovrebbero essere facilmente de-umidi come nel passaggio 4.1. Tuttavia, la cura dovrebbe ancora essere presa perché è importante che i chip non si asciughino. Se è necessario elaborare più chip, applicare il passaggio 5.3 a ogni chip in sequenza. - Incubare per 1 h su una piattaforma rotante a temperatura ambiente.
NOTA: Se le cellule hanno formato grandi strutture 3D, può essere necessario un tempo di incubazione più lungo per consentire una colorazione uniforme del campione. Il tempo di incubazione può essere aumentato fino a 24 h. Tuttavia, la camera di colorazione deve contenere un piatto di 3 cm riempito con acqua e la camera di colorazione deve essere sigillata con pellicola di laboratorio per evitare l'evaporazione. - Trasferire le patatine in un piatto multiwell fresco e lavare 3 volte con la soluzione di lavaggio.
- Eseguire l'incubazione con anticorpi secondari come descritto nei passaggi 5.3 e 5.4.
- Montare il chip su una diapositiva di microscopia utilizzando 20 -L di qualsiasi supporto di montaggio standard (ad esempio Mowiol).
6. Imaging
NOTA: l'imaging può essere eseguito su un microscopio confocale standard. In questo caso forniamo solo raccomandazioni per garantire una qualità dell'immagine che sarà sufficiente per la successiva analisi quantitativa.
AGGIORNAMENTO: Per evitare qualsiasi distorsione dell'operatore, le colonie di immagine devono essere scelte solo utilizzando il segnale di involucro nucleare (per vedere se una colonia segue correttamente la forma del modello). Evitare di controllare il segnale dei marcatori di interesse, tranne quando si regolano le impostazioni del microscopio.
- Assicurarsi che la profondità del bit di acquisizione sia 12 o 16 bit.
- Identificare le impostazioni appropriate per massimizzare l'intervallo dinamico per ogni canale di immagine. In particolare, evitare il ritaglio dell'immagine.
- Includere un canale per l'immagine dell'autofluorescenza del micropattern (vedere la figura3).
- Regolare le dimensioni dell'immagine e il fattore di zoom per ottenere dimensioni voxel comprese tra 0,1 e 0,6 m negli assi x e y e comprese tra 0,2 e 2 m nell'asse z.
NOTA: Ad esempio, in questo studio, abbiamo utilizzato un microscopio confocale a scansione invertita con un obiettivo 40x (apertura numerica uguale a 1,3), una dimensione dell'immagine di 1024 x 1024 pixel senza zoom digitale e una dimensione a z di 0,5 m. Ciò ha portato ad una dimensione voxel di 0,38 m x 0,38 m x 0,5 m. - Per ogni colonia, definire la posizione minima e massima lungo l'asse z per garantire che l'intera colonia venga acquisita. Almeno un aereo con segnale basso o nessun dovrebbe essere incluso sotto e sopra la colonia.
- Assicurarsi che gli orientamenti z-stack siano acquisiti in modo coerente (sempre dall'alto verso il basso o sempre dal basso verso l'alto)
- Regola la velocità di scansione, la risoluzione dell'immagine, la media dei fotogrammi e i vantaggi del rivelatore per identificare un valore ottimale tra la qualità dell'immagine e il tempo di imaging. Come indicazione, il tempo di imaging per una colonia come mostrato nella Figura 3 è stato di circa 2-3 min. Eseguire l'acquisizione dell'immagine.
AVVISO: Tutte le immagini, per essere comparabili, devono essere acquisite sullo stesso microscopio con le stesse impostazioni oggettive e di acquisizione. - Al termine dell'acquisizione, salvare tutte le immagini e assegnare una convenzione di denominazione univoca per identificare la condizione sperimentale che ogni immagine rappresenta.
NOTA: Vedere Figura 4 come esempio, questa convenzione verrà utilizzata in un secondo momento durante la procedura di analisi. Si noti che le immagini possono essere salvate in qualsiasi formato supportato da BioFormats 26. Se le colonie sono più grandi del campo visivo, il plugin di cucitura di ImageJ può essere utilizzato27. Si noti inoltre che in caso di cucitura, potrebbe essere necessaria una correzione di roll off dell'illuminazione.
7. Analisi dell'immagine
NOTA: le specifiche del computer consigliate per questa procedura sono: 16 GB di RAM, una CPU multi-core da 3,33 GHz e almeno 50 GB di spazio su disco (o più a seconda del numero di chip che sono stati imageizzati). Il software è stato testato su Linux, Windows e MacOS. PickCells è un'applicazione di analisi delle immagini multipiattaforma con un'interfaccia utente grafica dedicata all'analisi dell'organizzazione collettiva delle cellule in complesse immagini multidimensionali (Blin et al, in preparazione). Si noti che ulteriori informazioni su PickCells così come la documentazione per i moduli specifici menzionati qui possono essere trovati online: https://pickcellslab.frama.io/docs/. Si noti inoltre che l'interfaccia è soggetta a modifiche mentre continuiamo a migliorare il software. Se l'interfaccia differisce da ciò che è mostrato nella figura o video, si prega di fare riferimento al manuale online.
- Installare ed eseguire PickCells seguendo la documentazione disponibile online.
- Importare immagini e verificare l'accuratezza delle informazioni fornite (Figura 4-1).
- Documentare il nome di ogni canale.
- Nuclei di segmento basati sul segnale di inviluppo nucleare utilizzando il modulo Nessys28 (Figura4-2) e fornire un prefisso ("nuclei" per esempio) che verrà utilizzato per denominare le immagini segmentate generate.
NOTA: la documentazione sull'utilizzo e le regolazioni dei parametri è reperibile in https://framagit.org/pickcellslab/nessys. - Esaminare e modificare le segmentazioni, se necessario, utilizzando il modulo dell'editor di segmentazione (Figura 4-3)
NOTA: se per qualsiasi motivo il processo di segmentazione non ha fornito risultati soddisfacenti, eliminare manualmente le immagini nella cartella del database ed eliminare anche il nodo 'risultato di segmentazione' nella vista MetaModel. Ripetere quindi i passaggi 7.4 e 7.5. Se la segmentazione di un solo sottoinsieme di immagini non ha fornito risultati soddisfacenti, utilizzare l'applicazione autonoma Nessys (vedere il collegamento nella 7.4), tentare la segmentazione sulle "immagini difettose" e sostituire il file corrispondente nella cartella del database). - Segmentare il segnale di autofluorescenza del modello utilizzando il modulo di segmentazione di base (Figura 4-4)
- Fornire un prefisso ("modello", ad esempio) che verrà utilizzato per denominare le immagini segmentate generate.
- Selezionare il canale contenente il segnale di autofluorescenza.
- Applicare la riduzione del rumore; generalmente utilizzando un filtro gaussiano con una dimensione del kernel di 10 x 10 x 0,5 voxel dà risultati soddisfacenti.
- Impostare la soglia inferiore in modo che lo sfondo venga visualizzato in blu mentre il primo piano appare bianco. Impostare anche la soglia superiore sul valore massimo per evitare che intensità elevate vengano escluse dal risultato finale (aree rosse).
- Selezionare Salta per l'ultimo passaggio.
- Fare clic su Fine e attendere l'elaborazione di tutte le immagini.
- Per quanto riguarda i nuclei, i risultati della segmentazione possono ora essere ispezionati visivamente e corretti se necessario utilizzando il modulo dell'editor di segmentazione (Figura 4-5).
- Creare oggetti nuclei e calcolare le funzioni di base degli oggetti.
- Avviare il modulo delle funzionalità intrinseche dalla barra delle applicazioni a sinistra dell'interfaccia principale (Figura 4-6).
- Chiudete i pannelli Sistema ellissoide ed Estrattore superficie per mantenere aperto solo il pannello Funzioni di base.
- Scegliere Nucleus come tipo di oggetto e scegliere il prefisso indicato nel passaggio 7.4 per le "immagini segmentate".
- Premere Calcolo e attendere che tutte le immagini siano state elaborate.
NOTA: Dopo questo passaggio, non sarà possibile modificare nuovamente le segmentazioni dei nuclei.
- Creare oggetti pattern e calcolare le funzioni oggetto di base. Ripetere i passaggi da 7.8.1 a 7.8.4, solo questa volta scegliere Tipo personalizzato come tipo di oggetto e il prefisso specificato nel passaggio 7.6.1 per le immagini segmentate.
NOTA: dopo questo passaggio, non sarà possibile modificare nuovamente le segmentazioni del modello. - Memorizzare il nome dell'immagine a cui appartiene ogni nucleo come attributo del nucleo.
- Fare clic su Dati > Nuovo attributo e selezionare Nucleus nella finestra di dialogo popup e fare clic su OK.
- Selezionare Raccogli dati da altri oggetti connessi al nodo e fare clic su Avanti.
- Nel pannello a sinistra, selezionare Immagine, quindi fare doppio clic sul segno di interrogatorio sotto il flag Fine nel pannello Definizione percorso per impostare il nodo dell'immagine come destinazione del percorso.
- Espandere il riquadro Attributi disponibili nel riquadro sinistro e selezionare l'attributo name.
- Espandere il riquadro Operazione di riduzione e selezionare Ottieni uno, quindi fare clic sul pulsante di modifica e fare clic su Avanti.
- Digitare "Nome immagine", premere TAB e fare clic su OK.
- Creare un attributo "coordinata normalizzata" negli oggetti nuclei (Figura 4-7).
- Adattare i passaggi da 7.10.1 a 7.10.6 per memorizzare le coordinate del modello centroide come attributo del nucleo. Denominare questo nuovo attributo "Coordinata modello".
- Quindi, fare clic su Dati > Nuovo attributo, selezionare Nucleus e fare clic su Ok.
- Selezionare Definisci una funzione tra vettori spaziali o direzionali del nodo e fare clic su Avanti.
- Per Tipo di funzione selezionare Operazione di matrice, per V1 selezionare Vettore elemento, quindi Centroide, quindi V2 selezionare Vettore elemento, quindi Coordinato motivoe.
- Fare clic su Avanti, digitare "Coordinata normalizzata" nel campo Nome e fare clic su Fine.
- Esportare i dati in un file con valori separati da tabulazioni.
8. Analisi R
- Scaricare e installare Rstudio.
NOTA: le informazioni sul software e i link per il download sono disponibili https://www.rstudio.com/. - Scaricare gli script R necessari per questa analisi.
NOTA: gli script possono essere scaricati dal repository GitLab: https://framagit.org/pickcellslab/hexmapr. - Aprire Rstudio.
NOTA: se si eseguono gli script per la prima volta, installare i pacchetti R necessari (ggplot2 e scale). - Da Rstudio, aprire il modello di mappaina. Script R.
- Impostare la directory di lavoro sul percorso del file di origine.
- Seguire le istruzioni fornite nello script per adattare lo script a qualsiasi set di dati specificato per ottenere mappe spaziali, come illustrato nella Figura 5.
- Eseguire lo script per generare mappe di densità.
Risultati
Il metodo di fotomodellamento descritto qui consente di organizzare con precisione le cellule coltivate in colonie di forme e dimensioni definite. Il successo di questa procedura dovrebbe essere chiaramente evidente immediatamente dopo la procedura di semina cellulare (passaggio 3.7) in quanto le celle aderenti si raggruppano in base al design della fotomaschera come mostrato nella Figura 2a. A 1 h dopo la semina delle cellule, i singoli modelli potrebbero non essere pienamente confluenti (solo poche cellule per modello), tuttavia, poiché le cellule proliferano nel tempo, i modelli verranno colonizzati completamente con solo pochissime celle al di fuori delle superfici adesive (Figura 2b). L'aspetto esatto delle impostazioni cultura dipenderà dalla cella. Ad esempio, mESC forma colonie a forma di cupola10. Un chip in cui la modellazione non è chiara da 1 a 2 h dopo il seeding delle celle indica il fallimento della procedura (Figura 2c, d).
Le colonie grandi e spesse a volte possono essere difficili da macchiare in modo omogeneo. Suggeriamo di fissare e permeabilizzare le cellule in un unico passaggio (sezione 4) in quanto ciò può migliorare la penetrazione degli anticorpi29. Questo è il motivo per cui la soluzione fissativa scelta contiene un detergente. La figura 3 mostra il segnale di fluorescenza previsto dopo l'immunostaining. Si noti che le celle positive Luminose Id1 si trovano all'interno di regioni dense (regioni NPC luminose) delle colonie (Figura 3A). Suggerimenti come questo sono utili per valutare la qualità della procedura di colorazione degli anticorpi. Si noti inoltre che i micromodelli creati con la tecnica attuale sono autofluorescenti. Questo segnale (Figura 3A,B ha lasciato la maggior parte delle immagini) è utile durante la fase di analisi per registrare spazialmente le colonie tra loro e creare i risultati illustrati nella Figura 5. Il segnale di autofluorescenza è generalmente il più luminoso quando il campione è eccitato con un laser da 405 nm e questo canale deve essere lasciato senza colorazione per questo scopo. Figura 3 mostra anche come le celle sono vincolate con precisione su modelli di forme diverse.
L'analisi dei dati di imaging viene eseguita in PickCells, un software libero e open source sviluppato nel nostro laboratorio (Blin et al., in preparazione). Questo software include i moduli di analisi delle immagini per leggere e ordinare le immagini confocal (Figura 4-1), al segmento (Figura 4-2,4-4) e oggetti segmentati curati (Figura 4-3,4-5), per calcolare l'oggetto caratteristiche quali le coordinate o l'intensità media (Figura 4-6) e per esportare i dati (Figura 4-7,4-8). È importante sottolineare che abbiamo sviluppato un robusto metodo di segmentazione nucleare chiamato Nessys28 che è particolarmente adatto per popolazioni dense ed eterogenee di cellule come le cellule coltivate su micromodelli (Figura 3). Figura 4 -2 mostra un output rappresentativo del modulo Nessys in cui a ogni singola cella viene data con precisione un'identità di colore univoca. Dovrebbe essere necessaria solo una modifica minima, tuttavia la modifica è fattibile se l'utente decide così (Figura 4-3). Infine PickCells fornisce una serie di moduli di visualizzazione per visualizzare i dati. Un esempio è dato in Figura 4-6: una colonia ad anello viene resa in 3D dove i nuclei sono codificati a colori in base alla loro posizione lungo l'asse z. Una volta convalidata l'analisi in PickCells, i dati possono essere esportati per creare le mappe spaziali in R utilizzando gli script disponibili in (https://framagit.org/pickcellslab/hexmapr) come illustrato nella Figura 530.
Recentemente abbiamo dimostrato che il confinamento spaziale di mESC supiccoli (30.000 m 2) di micropatterns su disco o ellissi guida il patterning di una sottopopolazione di cellule che esprimono il marcatore mesodermico Tbra10. Così, per illustrare il nostro metodo qui, ci chiediamo se il patterning di Tbra può essere influenzato dalla segnalazione BMP in colonie più grandi (90,000 sm2). La figura 5A mostra che quando mESC viene coltivato su micromodelli a disco di grandi dimensioni, le cellule Tbra sono preferibilmente limitate alla periferia del modello (mappa di densità Tbra), dove la densità delle cellule locali è la più bassa (vedere la mappa blu a sinistra della figura 5A ). Questo patterning di Tbra è confermato dalla mappa dell'intensità media di Tbra.
Questi dati dimostrano che il metodo può rivelare informazioni sottovisive. Infatti, dalla Figura 3, l'ispezione visiva di una colonia non è sufficiente per identificare qualsiasi forma di organizzazione spaziale in espressione Tbra. Ciò è particolarmente spiegato dall'importante variabilità da colonia a colonia quantificata e mostrata nel pannello più a destra della figura 5A.
La tecnica mostra anche che non esiste alcun patterning rilevabile per Id1 (un obiettivo di segnalazione BMP) che può indicare che il patterning T non è guidato dalla segnalazione BMP in questo contesto.
Il micropatterning consente di forzare le colonie ad adottare quasi tutte le geometrie desiderate. Ciò è particolarmente utile per interrogare il modo in cui il sistema risponde a varie geometrie. Per esempio, potremmo ragionare che se un gradiente morfogeno si accumula al centro della colonia, la creazione di un buco nella colonia interromperebbe questo gradiente. È interessante notare che osserviamo ancora patterning su un micropattern anello anche se in modo meno robusto (Figura 5B).
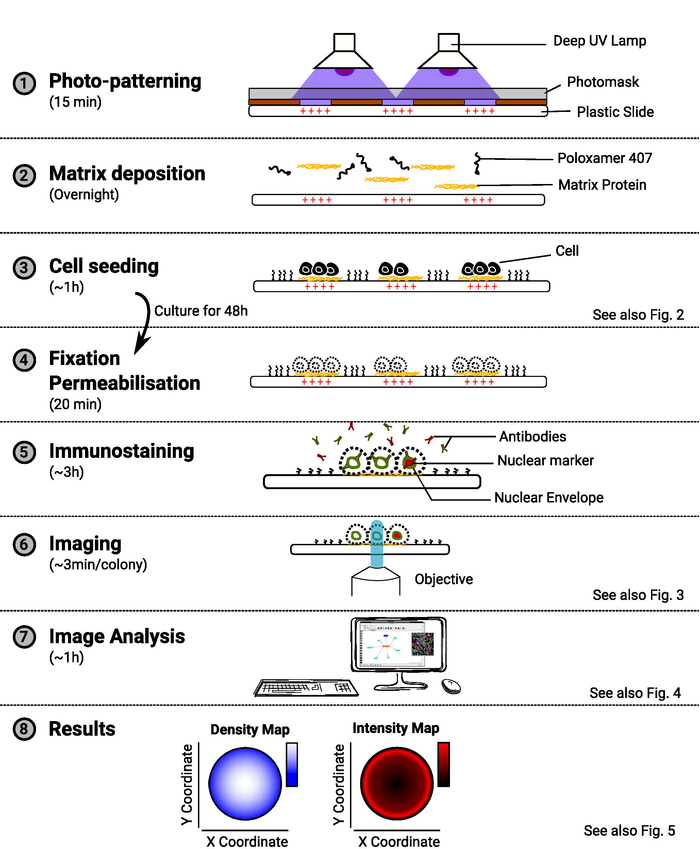
Figura 1: Panoramica del metodo. Il diagramma che mostra i passaggi principali del metodo. Per ogni fase, la quantità di tempo stimata è indicata sotto il nome dell'attività e uno schema illustra lo scopo della procedura. Un riferimento a una figura rilevante è anche fornito quando disponibile. Fare clic qui per visualizzare una versione più grande di questa figura.
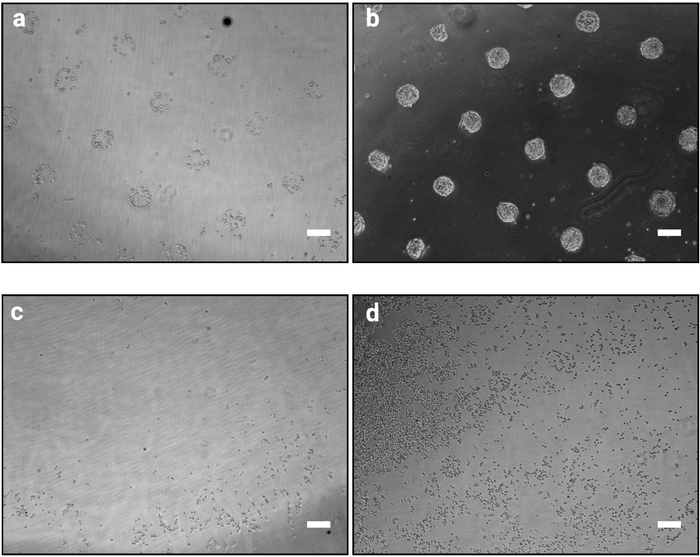
Figura 2: Aspetto della coltura 1 h e 48 h dopo il seeding delle cellule su micromodelli. Immagini luminose di semi mESC su micromodelli. (a) Organizzazione cellulare prevista 1 h dopo la semina, i modelli dovrebbero essere chiaramente identificabili. (b) Risultato atteso dopo 48 h di colture. mESC hanno proliferato e sono ancora strettamente confinati alle forme del modello. (c-d) Possibili risultati non ottimali, o pochissime cellule aderiscono alla plastica tranne che alla periferia del vetrino (c) o le cellule aderiscono tra i modelli (d). Vedere la tabella 2 per una guida alla risoluzione dei problemi. Fare clic qui per visualizzare una versione più grande di questa figura.
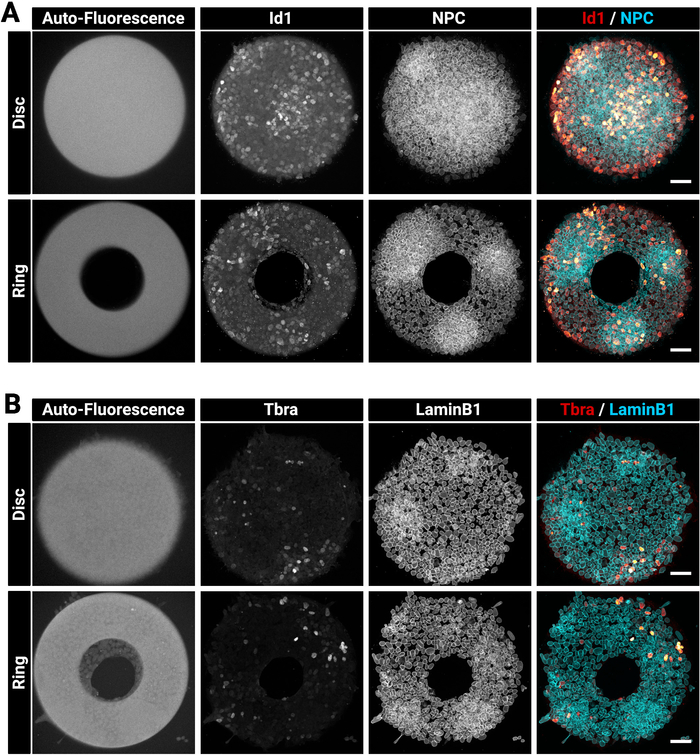
Figura 3: Immagini confocali rappresentative di colonie immunostainse coltivate su micromodelli.
Rappresentante colonie mESC dopo immunofluorescenza per (A) Id1 e Nuclear Pore Complex o (B) Tbra e LaminB1. Per ogni colorazione, viene mostrata una colonia coltivata su un micromodello a disco e una colonia coltivata su un micromodello ad anello. I singoli canali vengono forniti come immagini in scala di grigi. Si noti il chiaro segnale di auto-fluorescenza del micropattern (405 nm eccitazione laser). La barra della scala rappresenta 50 m. Fare clic qui per visualizzare una versione più grande di questa figura.
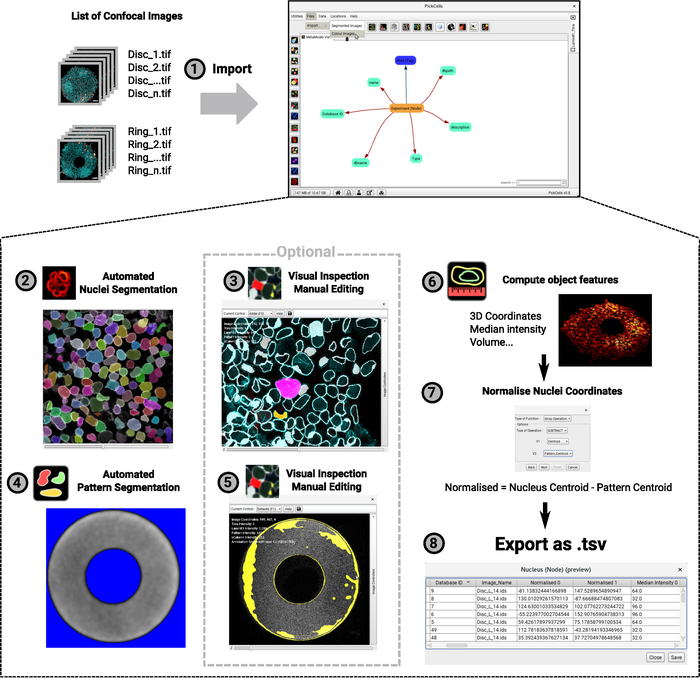
Figura 4: Diagramma di flusso della procedura di analisi delle immagini. Un elenco di immagini confocali 3D viene importato in PickCells per l'analisi (1). In questo esempio viene illustrato un esperimento con due forme distinte (dischi e anelli come nella Figura 2). La convenzione di denominazione delle immagini viene visualizzata a sinistra e l'interfaccia PickCells a destra. Quindi, il modulo Nessys viene utilizzato per segmentare automaticamente i nuclei (2). Nella schermata, a ogni singolo nucleo viene assegnato un colore univoco che indica una segmentazione accurata. Anche l'autofluorescenza del pattern viene segmentata, questa volta, utilizzando il modulo di "segmentazione di base" (4). Lo sfondo appare nel segnale blu e bianco sarà definito come la forma del modello. Le forme segmentate vengono quindi ispezionate visivamente per garantire un'accurata segmentazione e modificate se necessario utilizzando il modulo dell'editor di segmentazione (3-5). Gli screenshot mostrano il contorno delle forme rilevate. Le forme rosa e gialle sono state modificate. Infine, le entità geografiche vengono calcolate ed esportate in file per essere successivamente elaborate in R (6–7). Viene fornita una schermata di una colonia resa come vista 3D (6). Per i passaggi da 2 a 6 le icone trovate nell'interfaccia PickCells al momento della scrittura di questo articolo vengono fornite accanto all'indice del passaggio. Fare clic qui per visualizzare una versione più grande di questa figura.
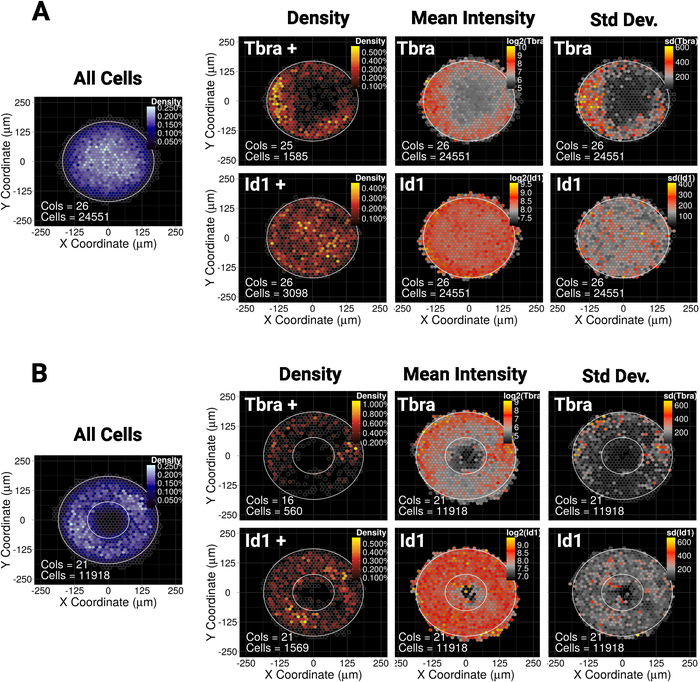
Figura 5: Risultati rappresentativi per due diversi fattori di trascrizione e forme di micropattern
Mappa spaziale binned per mESC coltivata per 48 h su (A) micropatterns a forma di disco o (B) micropatterns a forma di anello. Per ogni forma di micromodello, la mappa della densità delle cellule, indipendentemente dal fenotipo cellulare, viene visualizzata a sinistra con una scala di colori blu. Quindi per ogni marcatore (Tbra nella riga superiore e Id1 nella riga inferiore), vengono fornite tre mappe distinte, da sinistra a destra: mappa della densità delle celle del marcatore che esprime solo le celle (analisi basata sulla soglia), la mappa dell'intensità media del marcatore (log2) e la mappa del deviazione standard dell'intensità del marcatore. Le intensità sono date come unità di fluorescenza arbitrarie. Per ogni mappa, la forma del micromodello è data come contorno bianco. Fare clic qui per visualizzare una versione più grande di questa figura.
| Tipo ECM | Gelatina | Fibronectin | Matrice membrana del seminterrato | |
| concentrazione | Concentrazione ECM | 1 mg/mL | 20 g/mL | 200 g/mL |
| Poloxamer 407 concentrazione | 500 g/mL | 400 g/mL | 1 mg/mL | |
| Testato con | MESC (in quadra in più) | sì | sì | sì |
| mEpiSC (in nome com/mEpiSC) | No | sì | sì | |
| Mezzo senza siero | No | sì | sì |
Tabella 1: concentrazioni testate di poloxamer 407 e ECM. Questa tabella fornisce una panoramica delle concentrazioni di ECM e poloxamer 407 che abbiamo testato nel nostro laboratorio. Per ogni combinazione ECM/poloxamer 407, viene mostrato il tipo di cellula per il quale è stato raggiunto con successo il patterning, nonché se la coltura conteneva o meno il siero. mESC - cellule staminali embrionali del topo, mEpiSC - cellula staminale del topo.
| procedura | osservazione | Possibile problema | soluzione |
| Micropatterning | Attacco a celle basse | inappropriato rapporto di concentrazione ECM/poloxamer 307 | Aumentare il rapporto di concentrazione ECM/poloxamer 307 |
| Tempo di attacco cellulare troppo breve | Aumentare il tempo di incubazione per dare abbastanza tempo alle cellule per aderire correttamente ai modelli (passaggio 3.4). Per ottimizzare questo passaggio, il controllo delle cellule al microscopio può aiutare a rilevare un cambiamento nella morfologia cellulare che indica che le cellule hanno iniziato ad aderire. | ||
| Lavamenti troppo intensi (passaggio 3.6) | Quando si sostituisce il supporto, evitare di pipettare direttamente sulle chips. Invece, pipet delicatamente il mezzo sulle pareti del pozzo invece | ||
| Le cellule aderiscono tra i modelli | inappropriato rapporto di concentrazione ECM/poloxamer 307 | Diminuire il rapporto di concentrazione ECM/poloxamer 407 | |
| Tempo di attacco cellulare troppo lungo | Diminuire il tempo di incubazione (passaggio 3.4). | ||
| I fusti inefficaci (passaggio 3.6) | Scuotere vigorosamente la piastra è di solito sufficiente per staccare le cellule in eccesso. Per i tipi di cellule che tendono ad aderire fortemente al chip, pipetting direttamente sul chip può migliorare il risultato. Aumentare il numero di lavamenti può anche aiutare, in particolare per garantire che nessuna cellula rimanga galleggiante nel mezzo dopo questo passaggio. | ||
| Le celle non seguono rigorosamente la forma del modello | Tipo di cella 'Incompatibile' e geometria del motivo | Pianificare/progettare più geometrie/dimensioni da aggiungere alla fotomaschera per poter testare e identificare le dimensioni ottimali del modello per una determinata forma di pattern e tipo di cella. Si prega di consultare la sezione "Limitazioni" nella discussione | |
| Foto-patterning non ottimale, questo può essere diagnosticato osservando la nitidezza del segnale di autofluorescenza. I contorni del motivo devono essere nitidi come in Fig. Se i bordi dei motivi appaiono sfocati, il passaggio di fotopattern deve essere migliorato. | I bordi del motivo sfocato indicano che il vetrino di plastica non era abbastanza vicino alla superficie della maschera durante la fase di illuminazione. Assicurarsi che i pezzi che contengono le diapositive alla fotomaschera siano uniformi e che all'assieme venga applicata una pressione costante e sufficiente durante la procedura di illuminazione. | ||
| Colorazione | Colorazione non omogenea | tempo di incubazione anticorpale troppo breve | Aumentare il tempo di incubazione degli anticorpi (fino a 24h a temperatura ambiente) |
| colonie appiattite durante la procedura di montaggio | Montare i vetrini micropattern in una camera come chamlide o camere citoo-camera per eseguire sia l'immunostaining che l'imaging senza la necessità di montare le cellule. Questo conserverà meglio la struttura 3D delle colonie. | ||
| Distacco delle colonie durante la procedura di colorazione | Deumidizione del chip | Lasciare abbastanza mezzo o utilizzare 2 pipette, uno per rimuovere il mezzo e l'altro per aggiungere la soluzione fresca | |
| Le colonie appaiono tosate al microscopio | colonie sono state tosate durante il montaggio del chip sul vetrino di microscopia | Essere molto delicato quando si montano i chip. In alternativa, montare i micromodelli in una camera come chamlide o citoocamera per eseguire sia l'immunostaining che l'imaging senza la necessità di montare le cellule. Questo conserva anche l'ultrastruttura della colonia. |
Tabella 2: Guida alla risoluzione dei problemi. Questa tabella fornisce una panoramica dei possibili risultati non ottimali. Le potenziali fonti dei problemi sono anche elencate insieme alle soluzioni consigliate.
Discussione
Qui descriviamo un metodo per analizzare i modelli emergenti nelle colture di cellule. Un approccio di micropatterning semplificato viene utilizzato per standardizzare la forma e le dimensioni delle colonie cellulari, e presentiamo strumenti di analisi delle immagini e script R che consentono il rilevamento e la quantificazione dei modelli all'interno di queste colonie.
La pipeline che proponiamo è simile in una certa misura con un metodo precedentemente pubblicato31 in cui gli autori si concentrano sulle condizioni di coltura, utilizzando micromodelli disponibili in commercio, per ottenere la formazione di strato germinale riproducibile nelle colonie ESC per il studio dei primi eventi di gastrulazione in vitro. Il nostro obiettivo è più mirato a fornire una pipeline generalizzabile per la scoperta della formazione di modelli in vitro dove l'organizzazione collettiva delle cellule può diventare evidente solo dopo l'analisi statistica. Per questo motivo, forniamo un robusto flusso di lavoro di analisi delle immagini che consente l'identificazione e l'analisi accurata della posizione nucleare nello spazio 3D su più colonie (vedi anche la sezione "Vantaggio e limitazioni del metodo di rilevamento dei pattern" di questo discussione). Abbiamo anche deciso di sviluppare un semplice approccio di micropatterning interno che offra un'alternativa più flessibile ed economica alle soluzioni disponibili in commercio a lungo termine che speriamo siano utili alla comunità.
Infine, notiamo che durante la revisione di questo manoscritto, è stato rilasciato un nuovo pacchetto per l'analisi del patterning in vitro simile ai nostri script R32. Questo nuovo pacchetto accetta tabelle di funzionalità delle celle come input che possono essere ottenute da piattaforme di imaging ad alta velocità effettiva. Crediamo che la tabella delle caratteristiche dei nuclei generata al punto 7 del nostro protocollo potrebbe in linea di principio servire come input a questo nuovo pacchetto anche se non abbiamo testato questa possibilità noi stessi.
Adattabilità del metodo ad altri tipi di cellule e geometrie di colonia
Vi presentiamo questo approccio nel contesto dello studio dell'emergere di fattori di trascrizione mesodermici nelle colture di cellule pluripotenti in presenza di siero. Tuttavia, il metodo è facilmente adattabile ad altri tipi di cellule e alle colture senza siero, anche se potrebbe essere necessario ottimizzare le concentrazioni di ECM/poloxamer 407 (cfr. tabella 1 per le concentrazioni testate e Tabella 2 per una guida alla risoluzione dei problemi). Il metodo può anche essere adattato a dimensioni più grandi o più piccole di micromodelli e ad una vasta gamma di forme in base alle esigenze dell'utente. Tuttavia, mentre si stabilisce il metodo, è importante essere consapevoli del fatto che non tutte le combinazioni di forma/tipo di cella sono ottimali. Ad esempio, mESC esprime alti livelli di E-cadherin33,34 consentendo a queste cellule di formare strutture collettive che si estendono su aree prive di ECM. Queste celle non seguono rigorosamente le geometrie con angoli acuti o che includono piccoli fori nel modello. Si noti, ad esempio, che sull'anello della figura 3B, le celle sono in fase di colonizzazione dell'area centrale. Nelle nostre mani una zona centrale più piccola non ha costretto mESC a formare colonie ad anello. Si consiglia quindi di includere una varietà di geometrie durante la progettazione della fotomaschera per poter testare e identificare le dimensioni e le curve ottimali che saranno adatte al tipo di cella scelta.
Un altro fattore importante da prendere in considerazione è la lunghezza dell'esperimento e il tasso di proliferazione delle cellule. Per alcuni tipi di cellule che proliferano rapidamente (comprese le cellule pluripotenti) può essere difficile mantenere le cellule su micromodelli per molti giorni (Per mESC tre giorni è un massimo). Inoltre, la semina di cellule su micromodelli non sempre avviene in modo ottimale per ogni colonia, quindi è consigliabile seminare un eccesso di colonie per avere pezzi di ricambio.
Vantaggio e limitazioni del metodo di rilevamento dei pattern
Un particolare vantaggio del metodo è la capacità di rilevare modelli "medi" combinando i risultati dell'analisi delle immagini da più colonie di replica (Figura 5). Questo può rivelare eventi di patterning che non sono evidenti dall'ispezione di singole colonie. Uno svantaggio di questo approccio di "media" è che può mancare alcuni tipi di modelli ripetitivi, ad esempio piccole macchie o strisce strette. Tuttavia, questi tipi di pattern possono invece essere rivelati con una combinazione di dimensioni del modello scelte con cura8. Inoltre, la pipeline di analisi delle immagini qui descritta fornisce dati quantitativi sia a singola cellula che a risoluzione della colonia che offrono la possibilità di studiare il livello di variabilità tra le composizioni (Figura5) o di eseguire l'analisi dei vicini a più bilance10.
Un altro importante vantaggio del metodo di media è che offre l'opportunità di mappare la posizione preferenziale di molti marcatori senza essere limitato da fluorofori disponibili dei canali di rilevamento. Infatti, anche se ci avvalemo solo di due marcatori di differenziazione nel lavoro qui presentato, la capacità di standardizzare le colonie ed estrarre modelli "mediati" consente di confrontare insieme le mappe di distribuzione di diverse serie di colonie per per rivelare le relazioni spaziali generalizzate dei marcatori l'uno all'altro.
Inoltre, anche se il nostro obiettivo è stato quello di studiare i marcatori di differenziazione, il metodo di analisi può essere esteso per studiare altri processi biologici per i quali sono disponibili marcatori nucleari. Ad esempio, la microdoratura di una linea cellulare contenente un indicatore del ciclo cellulare dell'ubiquitazione a fluorescenza a fluorescenza35 (FUCCI) consentirebbe di studiare in che modo la geometria a livello di colonia può influenzare gli eventi del ciclo cellulare nel gruppo.
Indicazioni future
Il metodo è suscettibile di analisi delle immagini a media velocità effettiva, tuttavia, l'acquisizione di immagini non è attualmente completamente automatizzata e può diventare limitante per esperimenti di grandi dimensioni. Le disposizioni regolari delle colonie dovrebbero rendere possibile creare routine di acquisizione completamente automatizzate simili a quelle sviluppate per una singola cellula mediadi 20. Tuttavia, poiché le dimensioni del campo necessario per immaginare una colonia sono grandi, probabilmente richiedono mosaici, e poiché le colonie sono tridimensionali, è altamente auspicabile ridurre sia le dimensioni del set di dati che il tempo di acquisizione mediante l'imaging solo di colonie pertinenti. Pertanto, gli sforzi futuri potrebbero essere dedicati a sviluppare un microscopio "intelligente" in grado di identificare le colonie rilevanti e adattare le coordinate di imaging per ogni campione. Ciò non solo ridurrà i tempi e gli sforzi, ma previene anche potenziali distorsioni dell'operatore.
Le pipeline di analisi possono anche essere rese più efficienti riducendo il numero di passaggi che l'utente deve eseguire. Abbiamo in programma di costruire un meccanismo di costruzione di tubazioni e di integrare R direttamente nel nostro software (vedi anche problemi pickcells-api-3 e pickcells-rjava1 nel tracker di problemi dei nostri repository di codice [https://framagit.org/groups/pickcellslab/-/issues]). La completa automazione della procedura di analisi ridurrà i tempi e gli sforzi e limiterà i potenziali errori degli utenti.
Infine, notiamo che il nostro metodo di analisi non cattura ancora completamente la natura dinamica del patterning cellulare. Alcune informazioni dinamiche limitate possono essere estratte esaminando una serie temporale di immagini istantanee8,10,36. Tuttavia, essere in grado di registrare la storia della popolazione cellulare è altamente auspicabile se vogliamo capire meglio come emerge il modello. Una limitazione è che il tracciamento accurato delle singole cellule in una popolazione di cellule dense 3D rimane un compito molto impegnativo37. Il nostro metodo di rilevamento cellulare utilizza l'involucro nucleare e si comporta particolarmente bene su popolazioni cellulari dense e sovrapposte28. I giornalisti vivi dell'involucro nucleare sono prontamente disponibili28,38 e uno dei vantaggi della tecnica di micropatterning è che può essere utilizzato per impedire alle cellule di muoversi al di fuori del campo visivo durante l'imaging a lungo termine. Nel complesso, siamo fiduciosi che il monitoraggio automatizzato delle celle sarà realizzabile utilizzando una combinazione di strumenti di recente creazione28,39,40 e che questo dovrebbe portare nuove intuizioni nel fondamentale principi di auto-organizzazione.
Divulgazioni
Gli autori non hanno nulla da rivelare
Riconoscimenti
Questo lavoro è stato finanziato da una borsa di studio post-dottorato di Sir Henry Wellcome (da WT100133 a G.B.), da una Wellcome Trust Senior Fellowship (da WT103789AIA a S.L.), e da una borsa di dottorato del Wellcome Trust (108906 / 15 / s . Siamo anche grati al dottor Manuel Thery per i suoi consigli sull'adattamento della tecnica di fotomodellazione.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Gibco | 31350-010 | handle in fume hood |
| 1× Master quartz anti-reflective chromium photomask | Toppan Photomask | Custom design | |
| 24-well plates | Corning | 3526 | |
| 4-well plates | Nunclon Delta | 176740 | |
| 5% Donkey Serum | Sigma | D9663 | |
| Anti Nuclear pore complex | Abcam | ab24609 | Mouse monoclonal, use 1:1000 |
| Anti-Id1 | Biocheck | 37-2 | Rabbit polyclonal, use 1:200 |
| Anti-Lamin B1 | Abcam | ab16048 | Rabbit polyclonal, use 1:1000 |
| Anti-Tbra | R&D | AF2085 | Goat ployclonal, use 1:400 |
| Blocking Solution | NA | NA | 0.1% TritonX-100 0.01% Pluronic F-127 5% Donkey Serum 0.003% Sodium Azide In PBS |
| CGR8 mESC | NA | NA | Reference: Mountford et al. PNAS, 1994 |
| Fixation solution | NA | NA | 4% PFA diluted in washing solution |
| Foetal bovine serum | Gibco | 10270-106 | Serum batches must be tested to ensure compatibility with ESC maintenance |
| Gelatin | Sigma | G1890 | |
| Glasgow Minimum Essential Medium | Sigma | G5154 | |
| Laboratory Film | VWR | 291-1212 | |
| Layout Editor Software | LayoutEditor | NA | https://layouteditor.com/ |
| Layout Editor Software | Klayout | NA | https://www.klayout.de/ |
| L-glutamine | Gibco | 25030-024 | |
| LIF | Millipore | ESG1107 | |
| MEM NEAA | Gibco | 11140-035 | |
| mESC Culture medium | NA | NA | GMEM 10% FCS 100 U/ml LIF 100 nM 2-mercaptoethanol 1× non-essential amino acids, 2 mM L- glutamine 1 mM sodium pyruvate |
| NH4Cl 50mM | Sigma | 9718 | |
| Paraformaldehyde | Sigma | 158127 | CAUTION: Toxic, handle undiluted stocks in fume hood and wear protective equipment |
| PBS (immunostaining) | Sigma | P4417 | Dilute in 200ml of ddH2O |
| PBS (tissue culture) | Gibco | 11140-035 | |
| Plastic slides | Ibidi | IB-10813 | |
| Poloxamer 407 | Sigma | P2443 | |
| ProLong Gold Antifade Mountant | Molecular Probes | P36930 | |
| Sodium Azide | Sigma | 8591 | CAUTION: Toxic, handle in fume hood and wear protective equipment |
| Sodium pyruvate | Gibco | 11360070 | |
| TritonX-100 | Sigma | T8532 | |
| Trypsin | Gibco | 25200056 | |
| UVO cleaner | Jetlight, USA | 42-220 | CAUTION: Follow manufacturer’s safety recommendations |
| Washing Solution | NA | NA | 0.1% TritonX-100 0.01% Pluronic F-127 In PBS |
Riferimenti
- Sasai, Y. Cytosystems dynamics in self-organization of tissue architecture. Nature. 493 (7432), 318-326 (2013).
- Turner, D. A., Baillie-Johnson, P., Martinez Arias, A. Organoids and the genetically encoded self-assembly of embryonic stem cells. BioEssays. 38 (2), 181-191 (2016).
- Simunovic, M., Brivanlou, A. H. Embryoids, organoids and gastruloids: new approaches to understanding embryogenesis. Development. 144 (6), 976-985 (2017).
- Tewary, M., Shakiba, N., Zandstra, P. W. Stem cell bioengineering: building from stem cell biology. Nature Reviews. Genetics. 19 (10), 595-614 (2018).
- Shahbazi, M. N., Zernicka-Goetz, M. Deconstructing and reconstructing the mouse and human early embryo. Nature Cell Biology. 20 (8), 878 (2018).
- Laurent, J., et al. Convergence of microengineering and cellular self-organization towards functional tissue manufacturing. Nature Biomedical Engineering. 1 (12), 939 (2017).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Developmental Cell. 6 (4), 483-495 (2004).
- Tewary, M., et al. A stepwise model of Reaction-Diffusion and Positional-Information governs self-organized human peri-gastrulation-like patterning. Development. , (2017).
- Warmflash, A., Sorre, B., Etoc, F., Siggia, E. D., Brivanlou, A. H. A method to recapitulate early embryonic spatial patterning in human embryonic stem cells. Nature Methods. 11 (8), 847-854 (2014).
- Blin, G., Wisniewski, D., Picart, C., Thery, M., Puceat, M., Lowell, S. Geometrical confinement controls the asymmetric patterning of brachyury in cultures of pluripotent cells. Development. 145 (18), (2018).
- . . Micropatterning in cell biology. Pt. A. , (2014).
- Bauwens, C. L., et al. Control of human embryonic stem cell colony and aggregate size heterogeneity influences differentiation trajectories. Stem Cells. 26 (9), 2300-2310 (2008).
- Heemskerk, I., Burt, K., Miller, M., Chabra, S., Guerra, M. C., Warmflash, A. Morphogen dynamics control patterning in a stem cell model of the human embryo. bioRxiv. , 202366 (2017).
- Nemashkalo, A., Ruzo, A., Heemskerk, I., Warmflash, A. Morphogen and community effects determine cell fates in response to BMP4 signaling in human embryonic stem cells. Development. 144 (17), 3042-3053 (2017).
- Peerani, R., Onishi, K., Mahdavi, A., Kumacheva, E., Zandstra, P. W. Manipulation of signaling thresholds in “engineered stem cell niches” identifies design criteria for pluripotent stem cell screens. PloS One. 4 (7), e6438 (2009).
- Peerani, R., et al. Niche-mediated control of human embryonic stem cell self-renewal and differentiation. The EMBO journal. 26 (22), 4744-4755 (2007).
- Etoc, F., et al. A Balance between Secreted Inhibitors and Edge Sensing Controls Gastruloid Self-Organization. Developmental Cell. 39 (3), 302-315 (2016).
- Azioune, A., Carpi, N., Tseng, Q., Théry, M., Piel, M. Protein micropatterns: A direct printing protocol using deep UVs. Methods in Cell Biology. 97, 133-146 (2010).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. Journal of Cell Science. 123 (Pt 24), 4201-4213 (2010).
- Degot, S., et al. Improved Visualization and Quantitative Analysis of Drug Effects Using Micropatterned Cells. JoVE (Journal of Visualized Experiments). (46), e2514 (2010).
- Beddington, R. S. P., Rashbass, P., Wilson, V. Brachyury - a gene affecting mouse gastrulation and early organogenesis. Development. 116 (Supplement), 157-165 (1992).
- Wilkinson, D. G., Bhatt, S., Herrmann, B. G. Expression pattern of the mouse T gene and its role in mesoderm formation. Nature. 343 (6259), 657-659 (1990).
- Hollnagel, A., Oehlmann, V., Heymer, J., Rüther, U., Nordheim, A. Id Genes Are Direct Targets of Bone Morphogenetic Protein Induction in Embryonic Stem Cells. Journal of Biological Chemistry. 274 (28), 19838-19845 (1999).
- Mountford, P., et al. Dicistronic targeting constructs: reporters and modifiers of mammalian gene expression. Proceedings of the National Academy of Sciences of the United States of America. 91 (10), 4303-4307 (1994).
- Smith, A. G. Culture and differentiation of embryonic stem cells. Journal of tissue culture methods. 13 (2), 89-94 (1991).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Blin, G., Sadurska, D., Migueles, R. P., Chen, N., Watson, J. A., Lowell, S. Nessys: a novel method for accurate nuclear segmentation in 3D. bioRxiv. , 502872 (2018).
- Weiswald, L. -. B., Guinebretière, J. -. M., Richon, S., Bellet, D., Saubaméa, B., Dangles-Marie, V. In situ protein expression in tumour spheres: development of an immunostaining protocol for confocal microscopy. BMC cancer. 10, 106 (2010).
- Deglincerti, A., et al. Self-organization of human embryonic stem cells on micropatterns. Nature Protocols. 11 (11), 2223-2232 (2016).
- Ostblom, J., Nazareth, E. J. P., Tewary, M., Zandstra, P. W. Context-explorer: Analysis of spatially organized protein expression in high-throughput screens. PLOS Computational Biology. 15 (1), e1006384 (2019).
- Larue, L., Ohsugi, M., Hirchenhain, J., Kemler, R. E-cadherin null mutant embryos fail to form a trophectoderm epithelium. Proceedings of the National Academy of Sciences. 91 (17), 8263-8267 (1994).
- Pieters, T., van Roy, F. Role of cell-cell adhesion complexes in embryonic stem cell biology. J Cell Sci. 127 (12), 2603-2613 (2014).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Morgani, S. M., Metzger, J. J., Nichols, J., Siggia, E. D., Hadjantonakis, A. -. K. Micropattern differentiation of mouse pluripotent stem cells recapitulates embryo regionalized cell fate patterning. eLife. 7, e32839 (2018).
- Ulman, V., et al. An objective comparison of cell-tracking algorithms. Nature Methods. 14 (12), 1141 (2017).
- Moir, R. D., Yoon, M., Khuon, S., Goldman, R. D. Nuclear Lamins a and B1. The Journal of Cell Biology. 151 (6), 1155-1168 (2000).
- McDole, K., et al. In Toto Imaging and Reconstruction of Post-Implantation Mouse Development at the Single-Cell Level. Cell. 175 (3), 859-876 (2018).
- Stegmaier, J., et al. Real-Time Three-Dimensional Cell Segmentation in Large-Scale Microscopy Data of Developing Embryos. Developmental Cell. 36 (2), 225-240 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon