È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Una pipeline di bioinformatica per analizzare in modo accurato ed efficiente i trascrittomi di microRNA nelle piante
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Una pipeline bioinformatica, vale a dire miRDeep-P2 (miRDP2 in breve), con criteri di miRNA vegetale aggiornati e un algoritmo revisionato, potrebbe analizzare in modo accurato ed efficiente i trascrittimi di microRNA nelle piante, in particolare per le specie con genomi complessi e di grandi dimensioni.
Abstract
I microRNA (miRNA) sono da 20 a 24 nucleotidi (nt) piccoli RNA (sRNA) ampiamente esistenti nelle piante e negli animali che svolgono ruoli potenti nella regolazione dell'espressione genica a livello post-trascrizione. Le librerie di sRNA di sequenziamento con metodi di sequenziamento di nuova generazione (NGS) sono state ampiamente impiegate per identificare e analizzare i trascrittomi di miRNA nell'ultimo decennio, con conseguente rapido aumento della scoperta di miRNA. Tuttavia, due grandi sfide sorgono nell'annotazione del miRNA vegetale a causa dell'aumento della profondità delle librerie di sRNA sequenziati, nonché delle dimensioni e della complessità dei genomi delle piante. In primo luogo, molti altri tipi di sRNA, in particolare, brevi RNA interferenti (siRNA) provenienti dalle librerie di sRNA, sono erroneamente annotati come miRNA da molti strumenti computazionali. In secondo luogo, diventa un processo che richiede molto tempo per l'analisi dei trascrittomi di miRNA in specie vegetali con genomi grandi e complessi. Per superare queste sfide, abbiamo recentemente aggiornato miRDeep-P (uno strumento popolare per le analisi del transcriptome di miRNA) a miRDeep-P2 (miRDP2 in breve) impiegando una nuova strategia di filtraggio, riportando l'algoritmo di punteggio e incorporando miRNA vegetale appena aggiornato criteri di annotazione. Abbiamo testato miRDP2 su popolazioni di sRNA sequenziate in cinque piante rappresentative con crescente complessità genomica, tra cui Arabidopsis, riso, pomodoro, mais e grano. I risultati indicano che miRDP2 ha elaborato questi compiti con un'efficienza molto elevata. Inoltre, miRDP2 ha sovraperformato altri strumenti di previsione per quanto riguarda la sensibilità e l'accuratezza. Nel loro insieme, i nostri risultati dimostrano il miRDP2 come uno strumento veloce e preciso per analizzare i trascriptomi di miRNA vegetale, quindi uno strumento utile per aiutare la comunità ad annotare meglio i miRNA nelle piante.
Introduzione
Una delle scoperte più interessanti degli ultimi due decenni in biologia è il ruolo proliferatinte delle specie di sRNA nella regolazione delle diverse funzioni del genoma1. In particolare, i miRNA costituiscono un'importante classe di sRNA da 20 a 24 nt negli eucarioti, e funzionano principalmente a livello post-trascrizione come regolatori genici di primo piano durante le fasi di sviluppo del ciclo di vita, nonché nelle risposte di stimolo e stress2,3. Nelle piante, i miRNA derivano da trascrizioni primarie chiamate pri-miRNA, che sono generalmente trascritte dalla polimerasi RNA II come singole unità di trascrizione4,5. Elaborati da macchinari cellulari evolutivamente conservati (Drosha RNase III negli animali, simili a DICER nelle piante), i pri-miRNA vengono ascontenuti nei precursori immediati del miRNA, pre-miRNA, che contengono sequenze che formano strutture intra-molecolari di ciclo stelo6,7. I pre-miRNA vengono poi trasformati in intermedi a doppio filamento, vale a dire duplex di miRNA, costituiti dal filamento funzionale, dal miRNA maturo e dal partner meno frequentemente funzionale, miRNA2,8. Dopo aver caricato nel complesso di silenziamento indotto dall'RNA (RISC), i miRNA maturi potrebbero riconoscere i loro obiettivi di mRNA in base alla complementarità della sequenza, ottenendo una funzione regolatoria negativa2,8. I miRNA potrebbero destabilizzare le trascrizioni di destinazione o impedire la traduzione di destinazione, ma il modo precedente è dominato negli impianti8,9.
Dalla scoperta fortuita del primo miRNA nel nematode Caenorhabditis elegans10,11, molta ricerca è stata impegnata per l'identificazione del miRNA e la sua analisi funzionale, soprattutto dopo la disponibilità del metodo NGS. L'ampia applicazione del metodo NGS ha notevolmente promosso l'utilizzo di strumenti computazionali progettati per catturare la caratteristica unica dei miRNA, come la struttura del ciclo stelo dei precursori e il loro accumulo preferenziale di letture di sequenza su miRNA e miRNA maturi. Di conseguenza, i ricercatori hanno ottenuto un notevole successo nell'identificazione dei miRNA in specie diverse. Sulla base di un modello di probabilità descritto in precedenza12, abbiamo sviluppato miRDeep-P13, che è stato il primo strumento computazionale per scoprire i miRNA vegetali dai dati NGS. miRDeep-P era specificamente finalizzato a vincere le sfide di decodifica dei miRNA vegetali con una lunghezza precursore più variabile e grandi famiglie paralologiche13,14,15. Dopo il suo rilascio, questo programma è stato scaricato migliaia di volte e utilizzato per annotare trascrittomi di miRNA in più di 40 specie di piante16. Spinti da strumenti basati su NGS come miRDeep-P, c'è stato un drammatico aumento del numero di miRNA registrati nel repository miRNA pubblico miRBase17, dove sono attualmente ospitati oltre 38.000 elementi di miRNA (rilascio 22.1) rispetto ai soli 500 elementi di miRNA (rilascio 2.0) nel 200818.
Tuttavia, due nuove sfide sono sorte dall'annotazione del miRNA vegetale. In primo luogo, alti rapporti di falsi positivi hanno avuto un forte impatto sulla qualità delle annotazioni del miRNA vegetale16,19 per i seguenti motivi: 1) un diluvio di RNA interferenti brevi endogeni (siRNA) dalle librerie di sRNA NGS sono stati erroneamente annotati come miRNA a causa della mancanza di un rigoroso criterio di annotazione miRNA; 2) per le specie senza informazioni a priori miRNA, i falsi positivi previsti sulla base dei dati NGS sono difficili da eliminare. Usando miRBase come esempio, Taylor et al.20 trovarono che un terzo delle voci di miRNA vegetale nel repository pubblico21 (release 21) mancava di prove di supporto convincenti e anche tre quarti delle famiglie di miRNA vegetali erano discutibili. In secondo luogo, diventa un processo estremamente dispendioso in termini di tempo per prevedere i miRNA vegetali con genomi grandi e complessi16. Per superare queste sfide, abbiamo aggiornato miRDeep-P aggiungendo una nuova strategia di filtraggio, riportando l'algoritmo di punteggio e integrando nuovi criteri per l'annotazione miRNA dell'impianto e rilasciato la nuova versione miRDP2. Inoltre, abbiamo testato il miRDP2 utilizzando set di dati sRNA NGS con dimensioni del genoma in graduale aumento: Arabidopsis, riso, pomodoro, mais e grano. Rispetto ad altri cinque strumenti ampiamente utilizzati e alla sua vecchia versione, miRDP2 ha analizzato questi dati sRNA e ha analizzato più velocemente i trascriptomi di miRNA con una maggiore precisione e sensibilità.
Contenuto del pacchetto miRDP2
Il pacchetto miRDP2 è costituito da sei script Perl documentati che devono essere eseguiti in sequenza dallo script bash preparato. Dei sei script, tre (convert_bowtie_to_blast.pl, filter_alignments.ple excise_candidate.pl) vengono ereditati da miRDeep-P. Gli altri script vengono modificati rispetto alla versione originale. Le funzioni dei sei script sono descritte di seguito:
preprocess_reads.pl filtra le letture di input, incluse quelle troppo lunghe o troppo corte (<19 nt o >25 nt) e le letture correlate alle sequenze ncRNA Rfam, nonché le letture con RPM (Leggi per milione) inferiori a 5. Lo script recupera quindi le letture correlate a sequenze mature di miRNA note. I file di input sono letture originali in formato FASTA/FASTQ e output bowtie2 della mappatura delle letture alle sequenze di miRNA e ncRNA.
La formula per il calcolo di RPM è la seguente:
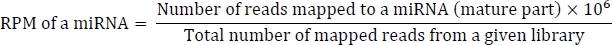
convert_bowtie_to_blast.pl modifica il formato bowtie in formato analizzato BLAST. Il formato parsed BLAST è un formato separato tabulare personalizzato derivato dal formato BLASToutput NCBI standard.
filter_alignments.pl filtra l'allineamento delle letture di sequenziamento profondo in un genoma. Filtra gli allineamenti parziali e le letture multiallineate (taglio di frequenza specificato dall'utente). L'input di base è un file in formato analizzato BLAST.
excise_candidate.pl taglia le potenziali sequenze precursori da una sequenza di riferimento utilizzando le letture allineate come linee guida. L'input di base è un file in formato parsed BLAST e un file FASTA. L'output è tutte le potenziali sequenze precursori in formato FASTA.
mod-miRDP.pl richiede due file di input, il file di firma e il file di struttura, che viene modificato dall'algoritmo miRDeep-P di base modificando il sistema di punteggio con parametri specifici dell'impianto. I file di input sono file di struttura precursore delle parentesi punti e leggono il file delle firme di distribuzione.
mod-rm_redundant_meet_plant.pl richiede tre file di input: chromosome_length, precursori e original_prediction generati da mod-miRDP.pl. Genera due file di output, il file previsto non ridondante e il file previsto filtrato in base ai nuovi criteri di miRNA dell'impianto aggiornati. I dettagli sul formato del file di output sono descritti nella sezione 1.4.
Protocollo
1. Installazione e collaudo
- Scaricare le dipendenze richieste: Bowtie222 e RNAfold23. Si consigliano pacchetti compilati.
- Scarica Bowtie2, uno strumento di mappatura di lettura, dal suo sito di casa (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml).
- Scarica RNAfold, uno strumento del pacchetto di Vienna utilizzato per prevedere la struttura secondaria dell'RNA, da http://www.tbi.univie.ac.at/~ivo/RNA/.
- Prima di installare miRDP2, assicurarsi che queste due dipendenze siano installate correttamente e personalizzare il file di ambiente bash (ad esempio, .bashrc) per impostare un percorso corretto per queste due dipendenze.
NOTA: Altri strumenti di mappatura come Bowtie24 sono adatti anche a miRDP2; può essere utilizzato Bowtie o Bowtie2 dopo la versione 1.1.3.
- Per scaricare il pacchetto miRDP2, passare a https://sourceforge.net/projects/mirdp2/files/latest_version/ e recuperare i file tarball.
- Prima di installare miRDP2, assicurarsi che Perl si trova nel percorso. Per installare miRDP2, estrarre tutto il contenuto del file tarball scaricato in una cartella (righe di comando come in 1.4.2), quindi impostare il percorso della cartella nel percorso.
NOTA: si consiglia di utilizzare un computer o un nodo di elaborazione con almeno 8 GB di RAM e 100 GB di spazio di archiviazione per l'esecuzione di miRDP2. - Testare la pipeline MiRDP2.
- Per verificare se miRDP2 è stato installato correttamente, utilizzare i dati di test e l'output previsto trovato in https://sourceforge.net/projects/mirdp2/files/TestData/. I dati dei test contengono un file di sequenziamento GSM formattato e un file del genoma di Arabidopsis thaliana.
- Spostare tutti i file scaricati nella directory di lavoro corrente:
mv miRDP2-v.tar.gz TestData.tar.gz ncRNA_rfam.tar.gz
cd - Estrarre i file di tarball compressi:
tar –xvzf miRDP2-v.tar.gz
tar –xvzf TestData.tar.gz
tar –xvzf ncRNA_rfam.tar.gz - Costruire l'indice di riferimento del genoma dell'Arabidopsis:
bowtie2-build -f ./TestData/TAIR10_genome.fa ./TestData/TAIR10_genome - Costruire l'indice di riferimento ncRNA:
bowtie2-build -f ./ncRNA_rfam.fa ./1.1.3/script/index/rfam_index - Eseguire la pipeline miRDP2:
bash ./1.1.3/miRDP2-v1.1.3_pipeline.bash –g ./TestData/TAIR10_genome.fa -i ./ TestData/TAIR 10_genome –f ./TestData/GSM2094927.fa –o .
NOTA: i comandi Linux utilizzati sono in grassetto e corsivo, con opzioni della riga di comando in corsivo. : indica la versione di miRDP2 (la versione corrente è 1.1.3). Il comando bowtie2-build dovrebbe richiedere circa 10 minuti e la pipeline miRDP2 dovrebbe terminare in pochi minuti
- Controllare gli output dei test.
- Si noti che in viene generata automaticamente una cartella denominata 'GSM2094927-15-0-10', contenente tutti i file intermedi e i risultati.
- Verificare che il file di output delimitato da tabulazioni GSM2094927-15-0-10_filter_P_prediction, l'output finale dei miRNA previsti, contenga colonne che indicano id cromosoma, direzione del filamento, lettura rappresentativa id, id precursore, posizione di miRNA maturi, precursore posizione, sequenza matura e sequenza precursore. Si noti il file letto aggiuntivo derivato da questo file per facilitare un'ulteriore analisi.
- Controllare il file "progress_log", che fornisce informazioni sui passaggi completati, e i file "script_log" e "script_err", che contengono output e avvisi del programma.
NOTA: Attualmente, abbiamo testato miRDP2 su due piattaforme Linux, tra cui CentOS versione 6.5 su un server cluster, e Cygwin 2.6.0 su PC Windows system, e miRDP2 dovrebbe funzionare su sistemi simili che supportano Perl.
2. Identificazione di nuovi miRNA
- Prima di eseguire la pipeline, assicurarsi che le letture di input siano pre-elaborate in un formato corretto.
NOTA: la nuova versione 1.1.3 di miRDP2 può accettare i file di formato FASTQ originali come input, anche se il processo di formattazione delle letture viene eseguito come nelle versioni precedenti.- In primo luogo, rimuovere gli adattatori dalle estremità 5' e 3 ' del sequenziamento profondo letti (se presente).
- In secondo luogo, analizzare le letture di sequenza completa in formato FASTA.
- In terzo luogo, rimuovere la ridondanza in modo che le letture con sequenza identica siano rappresentate con una voce FASTA singola e univoca.
- Infine, assicurarsi che tutti gli identificatori FASTA siano univoci. Ogni identificatore di sequenza deve terminare con un '_x' e un numero intero, che indica il numero di copia della sequenza esatta recuperata nei set di dati di sequenza completa. Un modo per garantire un identificatore FASTA univoco consiste nell'includere un numero in esecuzione nell'ID. Per riferimento, vedere il file GSM2094927.fa nei dati di test (https://sourceforge.net/projects/mirdp2/files/TestData/).
- Per esempi di letture formattate correttamente, vedere quanto segue:
>read0_x29909
TTTGGATTGAAGGGACTCTA
>read1_x36974
TTCCACAGCTTCTTGAACTG
>read2_x32635
TTCCACAGCTTCTTGAACTT
- Creare indici di riferimento.
- Per quanto riguarda il riferimento del genoma, per risparmiare tempo, scaricare i file di indice Bowtie2 dal sito web di iGenomes (https://support.illumina.com/sequencing/sequencing_software/igenome.html) se le sequenze genomiche delle specie di interesse sono state indicizzate. In caso contrario, gli utenti indicizzano le sequenze di riferimento e mantengono il file di indice per un po' fino al termine del progetto, poiché potrebbe essere necessario reindicizzare la sequenza genomica. I dettagli su come indicizzare un riferimento al genoma sono inclusi nel manuale bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml).
- Un altro indice ncRNA non-miRNA è necessario anche per filtrare le sequenze rumorose da altri frammenti di RNA non codificanti. Il file è una raccolta di principali sequenze ncRNA di Rfam, tra cui rRNA, tRNA, snRNA e snoRNA. Per creare questo indice, fare riferimento alla parte 1.4, in quanto l'indice deve essere posizionato e denominato correttamente, vale a dire /script/index/rfam_index.
- Eseguire miRDP2.
- Per utilizzare miRDP2 per rilevare nuovi miRNA da dati di sequenza completa, eseguire lo script bash nel pacchetto per avviare la pipeline di analisi (un esempio è disponibile nel passaggio 1.4):
/miRDP2-v._pipeline.bash –g -i -f -o
in cui : indica la versione dello script bash della pipeline. Ci sono tre parametri che possono essere modificati: 1) il numero di posizioni diverse a cui una lettura potrebbe essere mappata, 2) il numero di mancata corrispondenza per l'esecuzione bowtie2 e 3) la soglia di RPM (Letture per milione). Modificarli utilizzando rispettivamente le opzioni –L, -M e –R. Una spiegazione dettagliata è riportata nella sezione 3.1.
- Per utilizzare miRDP2 per rilevare nuovi miRNA da dati di sequenza completa, eseguire lo script bash nel pacchetto per avviare la pipeline di analisi (un esempio è disponibile nel passaggio 1.4):
- Controllare le uscite miRDP2.
- Si noti che la cartella di output verrà generata automaticamente in e denominata '-15-0-10'; gli ultimi 3 numeri indicano i valori (in questo caso di default) per i parametri 1, 2 e 3, rispettivamente. Il file _filter_P_prediction contiene informazioni sui miRNA previsti finali che soddisfano i nuovi criteri di annotazione del miRNA vegetale appena aggiornati. I dettagli sul formato del file di output sono descritti nella parte 1.4.
3. Modifiche e cautela con miRDP2
- Parametri che possono essere modificati
- Utilizzare l'opzione '-L' per impostare il limite di posizioni a cui è possibile eseguire il mapping di una lettura (parametro 1). Il mapping di lettura a troppi siti è probabilmente associato a sequenze ripetute e non è probabile che si ripercano in miRNA. L'impostazione predefinita è 15. Per specie specifiche, se ci sono famiglie di miRNA con molti membri, il primo parametro può essere aumentato manualmente per adattarsi al paesaggio del genoma.
- Utilizzare l'opzione '-M' per impostare le mancate corrispondenze consentite per bowtie (parametro 2). L'impostazione predefinita è 0.
- Utilizzare l'opzione '-R' per impostare la soglia per le letture potenzialmente corrispondenti ai miRNA maturi (parametro 3). Per ridurre il consumo di tempo e i falsi positivi, filtrare le letture per RPM. Solo le letture che superano una determinata soglia RPM possono rappresentare sequenze mature di miRNA anziché rumore di fondo e verrebbero mantenute per un'ulteriore analisi. L'impostazione predefinita è 10 RPM.
- Si noti che la modifica di questi parametri può influire sul consumo di tempo e sulle prestazioni. In generale, un aumento dei parametri 1 e 2 e una diminuzione del parametro 3 genererebbero un risultato meno rigoroso e tempi di esecuzione più lunghi e viceversa.
- Ridondanza e miRNA
- Si noti che i miRNA di output da miRDP2 possono differire dai miRNA noti. Abbiamo scoperto che questo è dovuto principalmente a uno dei due motivi: eterogeneità dei miRNA maturi o relativa abbondanza di miRNA e miRNA. Abbiamo scoperto che questo non influisce sulla selezione ottimale della lunghezza dei precursori e sulla profilazione dei geni miRNA noti.
Risultati
La pipeline di annotazione miRNA, miRDP2, qui descritta viene applicata a 10 librerie pubbliche di sRNA-seq di 5 specie vegetali con gradualmente aumentata lunghezza del genoma, tra cui l'Arabidopsis thaliana, Oryza sativa (riso), Solanum lycopersicum (pomodoro), zelo mays (mais) e Triticum aestivum (grano) (Figura 1A). Complessivamente, per ogni specie, 2 biblioteche sRNA rappresentative di tessuti diversi (collassati in letture ...
Discussione
Con l'avvento di NGS, un gran numero di loci miRNA sono stati identificati da una quantità sempre crescente di dati di sequenziamento dello sRNA in diverse specie29,30. Nel database della comunità centralizzata miRBase21, gli elementi di miRNA depositati sono aumentati di quasi 100 volte nell'ultimo decennio. Tuttavia, rispetto ai miRNA negli animali, i miRNA vegetali hanno molte caratteristiche uniche che rendono l'identificazione/annot...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da Beijing Academy of Agriculture and Forestry Sciences (KJCX201917, KJCX20180425 e KJCX20180204) a XY e National Natural Science Foundation of China (31621001) a LL.
Materiali
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
Riferimenti
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon