È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Ottimizzazione dei processi mediante microreattori automatizzati ad alta velocità effettiva nella coltivazione delle cellule ovaio dell'ammasso cinese
In questo articolo
Riepilogo
Qui, presentiamo una procedura dettagliata per eseguire un progetto di esperimento in un micro-bioreattore automatizzato seguito dal raccolto cellulare e dalla quantificazione delle proteine utilizzando una colonna di proteine A.
Abstract
L'ottimizzazione dei bioprocessi per aumentare la resa dei prodotti desiderati è importante nell'industria biofarmaceutica. Ciò può essere ottenuto mediante la selezione della deformazione e lo sviluppo di parametri di bioprocesso. A questo scopo sono stati utilizzati flaconi Shake. Essi, tuttavia, non sono in grado di controllare i parametri di processo come il pH e l'ossigeno disciolto (DO). Questa limitazione può essere superata con l'aiuto di un microbioreattore automatico. Questi bioreattori imitano la coltivazione su scala più ampia. Uno dei principali vantaggi di questo sistema è l'integrazione del Design of Experiment (DOE) nel software. Questa integrazione consente di stabilire un progetto in cui più parametri di processo possono essere variati contemporaneamente. I parametri di processo critici e le condizioni ottimali del bioprocesso possono essere analizzati all'interno del software. L'obiettivo del lavoro qui presentato è quello di introdurre l'utente alle fasi coinvolte nella progettazione del processo nel software e nell'incorporazione del DOE all'interno della corsa di coltivazione.
Introduzione
Il mercato biofarmaceutico globale ha varato più di 250 miliardi di dollari nel 2018 ed è in continua espansione1. Le aziende farmaceutiche si stanno allontanando dalla produzione di piccoli farmaci molecolari a terapie biotecnologicamente prodotte come le proteine ricombinanti. Questi da soli sono responsabili di un fatturato di oltre 150 miliardi di dollari1. Le cellule dei mammiferi sono ora ampiamente utilizzate per la produzione di queste proteine ricombinanti farmaceutiche. Nel periodo attuale, tra i 68 prodotti approvati prodotti dalle cellule dei mammiferi, 57 sono prodotti dalle cellule ovariche di Hamster cinesi (CHO)2. Le cellule CHO sono specificamente utilizzate per la produzione di proteine ricombinanti che richiedono modifiche post-traduzionali. Queste cellule sono preferite in quanto crescono in una sospensione e quindi consentono risultati riproducibili in unsierolibero chimicamente 3,4. L'altro vantaggio dell'utilizzo di cellule CHO è che la struttura glicana del prodotto assomiglia a quella dell'anticorpo monoclonale umano (mAb) e si traduce in una maggiore resa di proteine ricombinanti e produttività specifica a causa dell'amplificazione della genetion5.
La resa della coltura cellulare ricombinante CHO (rCHO) è aumentata di cento volte negli ultimi due decenni. Questo miglioramento è attribuito all'ottimizzazione dei parametri di processo, alla strategia di alimentazione e allo sviluppo di un mezzo chimico definito chimicamente privo di siero6. Con l'aumento del fabbisogno dei prodotti farmaceutici, la pressione aumenta sull'efficienza dei costi e dei tempi per lo sviluppo del processo di produzione7. Per ridurre la pressione assicurando la qualità del prodotto ha reindirizzato l'attenzione dell'industria farmaceutica sulla qualità di progettazione (QbD). QbD viene utilizzato per comprendere la produzione del prodotto e il processo. Uno strumento vitale utilizzato nell'ObD è il Design of Experiment (DOE). Aiuta ad aumentare la comprensione del processo rivelando la relazione tra varie variabili di input e i dati di output risultanti. L'applicazione dell'approccio DOE per ottimizzare il bioprocesso è vantaggiosa durante le prime fasi del progetto nell'assimilare le condizioni di processo e nell'aumento della quantità e della qualità del titolo. Questo approccio è utile rispetto alla strategia antiquata: OFAT (one-factor-at-a-time). Gli approcci statistici al DOE utilizzando Classico, Shainin o Taguchi sono di gran lunga superiori all'OFAT8.
Il processo e l'ottimizzazione dei supporti possono essere eseguiti in flaconi shake. I flaconi sono relativamente poco costosi. Tuttavia, non è possibile controllare parametri quali temperatura, pH e ossigeno disciolto (DO). Per superare questi inconvenienti, possono essere utilizzati bioreattori multiuso che vanno dal volume di lavoro da 0,5 L a 5 L. I reattori forniscono un ampio monitoraggio on-line e controllo dei processi. Tuttavia, l'uso del bioreattore multiuso è un tempo e un lavoro intensivo. Per superare questi svantaggi, viene utilizzato un nuovo bioreattore monouso che combina il processo completo di monitoraggio del bioreattore bench-top e di facile gestione del pallone di agitazione. Il sistema di screening ad alta produttività e la tecnologia monouso hanno contribuito a migliorare l'efficienza delle prestazioni di processo e dello sviluppo9.
In questo articolo sono elencate le linee guida per caricare la ricetta nel software automatizzato di microbioreattore (AMBR). Nel corso di questo esperimento viene studiata l'influenza delle diverse velocità e del pH sulla concentrazione cellulare vitale (VCC) e sul timore. Il risultato sperimentale e l'analisi vengono effettuati con la progettazione di software sperimentale MODDE 12. L'analisi dei prodotti viene effettuata in un sistema di cromatografia liquida ad alta pressione (HPLC) con una colonna Protein A. Si basa sul principio che la regione Fc del mAb si lega alla proteina A con alta affinità10,11. Con questo metodo, è possibile identificare e quantificare il mAb. La quantificazione viene effettuata sulle aree di picco di eluizione misurata a 280 nm.
Protocollo
1. Procedura di precultura
NOT: Per questo protocollo vengono utilizzate cellule RICO DG44 ricombinanti con una concentrazione cellulare vitale di 1 x 107 celle/mL.
- Scongelare la fiala contenente 1,2 mL di cellule a temperatura ambiente e trasferire immediatamente la sospensione cellulare in un tubo di centrifuga conica da 15 mL contenente 10 mL di mezzo di semi freddi.
- Centrifugare il tubo centrifuga conico per 5 minuti a 190 x g e temperatura ambiente e scartare il supernatante.
- Preriscaldare 150 mL del mezzo seme in un flacone di scossone da 500 mL a 36,8 gradi centigradi.
- Risospendere delicatamente il pellet cellulare in 10 mL di mezzo semi preriscaldato e trasferire le cellule nel pallone di agitazione.
- Utilizzare 1 mL del campione dal pallone per misurare il VCC iniziale e la vitalità utilizzando un contatore cellulare.
NOT: La redditività dovrebbe essere superiore al 70% dopo lo scongelamento per una coltivazione di successo. - Incubare il pallone di scossa in uno shaker orbitale (diametro dello shaker di 19 mm) a 36,8 e 7,5% di CO2 con una velocità di agitazione di 120 giri/m.
NOT: Queste condizioni variano a seconda del ceppo cellulare e del mezzo. - Tre giorni dopo aver passato le cellule, rimuovere il pallone di agitazione dallo shaker e posizionarlo sotto l'armadio di flusso laminare. Prendere 1 mL di campione per misurare la concentrazione finale delle cellule. Calcolare il volume da trasferire al nuovo mezzo di semi preriscaldato in modo che la concentrazione iniziale delle cellule nel nuovo passaggio sia 2 x 105 celle/mL.
- Passare le cellule 5 volte in totale prima di trasferirle al bioreattore per la coltivazione principale.
2. Coltivazione principale
- Misurare la concentrazione cellulare finale della precoltura. Calcolare il volume da trasferire al bioreattore in modo che la concentrazione cellulare iniziale nel reattore sia 3 x 105 cellule/mL.
- Riempire il reattore con un mezzo di produzione un giorno prima dell'inoculazione per abcillare il reattore e impostare i parametri di processo come temperatura, pH e DO.
NOT: Le condizioni di coltivazione sono 36,8 gradi centigradi e il 60% di concentrazione di ossigeno disciolto (DO). Abbiamo testato velocità di stirrer di 1050 rpm e 1300 rpm insieme a pH di 6.9, 7.1 e 7.3. La durata totale della coltivazione è di 12 giorni prima che le cellule siano raccolte. Il processo batch viene eseguito per 72 ore dopo le quali il supporto di alimentazione viene aggiunto ogni 24 ore. Il protocollo da utilizzare per la coltivazione è elencato in dettaglio nel segmento successivo.
3. Scrivere la ricetta nel software automatizzato di microbioreattore
NOT: Ci sono due modi per scrivere una ricetta nel software di coltura cellulare AMBR: viene creata utilizzando una procedura guidata o aggiungendo ogni passaggio manualmente. Ai fini di questo protocollo, vengono visualizzati i passaggi che utilizzano la procedura guidata.
- Creazione di un nuovo esperimento
- Aprire il software di coltura delle celle AMBR e nella scheda Introduzione fare clic su Crea nuovo esperimento.
- Caricamento della ricetta
- Nella scheda Nuovo esperimento, inserisci il nome dell'esperimento insieme alla data in cui deve essere condotto.
- Attivare il check point per la stazione di coltura e i vasi da utilizzare durante la coltivazione. I tag Auto Add DOE saranno attivati anche per una transizione facile durante la programmazione dell'esperimento DOE. Fare clic su Avanti per passare alla scheda successiva.
- Impostare le informazioni sull'aggiunta di supporti nel recipiente insieme a antifoam, inoculum, mangimi e glucosio.
- Attivare il punto di controllo Aggiungi piastra multimediale. Definire il tipo di piastra, il nome e la posizione della piastra contenente il supporto.
AVVISO: A seconda del tipo di piastra e se la piastra contiene un coperchio, attivare il controllo su È coperchio per garantire il corretto funzionamento del gestore liquido - Fare clic su Aggiungi supporto alle navi. Immettere il volume dei supporti da aggiungere alle navi. Definire la mappatura del trasferimento del supporto dalla piastra ai recipienti. Fare clic su Avanti per passare alla scheda successiva.
- Attivare il punto di controllo Aggiungi piastra multimediale. Definire il tipo di piastra, il nome e la posizione della piastra contenente il supporto.
- Impostare le condizioni di coltivazione nel reattore.
- Dopo che le informazioni multimediali sono state inserite nel software, assegnare le condizioni di coltivazione. Fare clic su Condizione Supporto e compilare la temperatura, il DO di destinazione, il limite di pH superiore e l'agitazione rpM (Up stirring o Down stirring).
- Impostare l'aggiunta di inoculos nei vasi.
- Attivare Aggiungi piastra di celle. Definire il tipo, il nome e la posizione della piastra contenente il supporto.
- Fare clic su Aggiungi celle alle navi. Immettere l'ora di inoculazione e il volume dei supporti da aggiungere alle navi.
- Definire il percorso percorso dal gestore liquido per il trasferimento della cella dalla piastra ai vasi. Fare clic su Avanti per passare alla scheda successiva.
NOT: Assicurarsi che le punte Reuse Pipette siano disattivate per evitare la contaminazione incrociata e una concentrazione iniziale errata delle cellule vitali.
- Impostare l'aggiunta di mangime, glucosio e antifoam.
NOT: La procedura per l'aggiunta di mangime, glucosio e antifoam è simile tra loro. Ai fini di questo protocollo la procedura è elencata per "Feed". Questo può essere replicato per glucosio e antifoam.- Attivare Aggiungi piastra di alimentazione e definire il tipo, il nome e la posizione della piastra. Fare clic su Aggiungi alimentazione alle navi e immettere il volume dell'avanzamento da aggiungere alle navi. Definire la mappatura del trasferimento del mangime dalla piastra ai recipienti.
- A seconda della coltivazione, aggiungere il numero di addizione del mangime. Per questa coltivazione, il reattore viene alimentato dopo 72 ore per ogni 24 ore.
- Aggiungere manualmente il ritardo tra l'alimentazione immettendo i dati in Ritardo dalle celle aggiunte. Il primo giorno di alimentazione è dopo 72 ore di inoculazione e il successivo è dopo 96 ore e così via.
NOT: L'aggiunta di anti-foam è programmata per essere aggiunta ogni giorno per evitare la schiuma durante la coltivazione.
- Set campionamento durante la coltivazione.
- Attivare Aggiungi piastra di prova e definire il tipo, il nome e la posizione della piastra.
- Controllare Take Sample from Vessels e inserire il volume del campione da rimuovere dalle navi. Definire la mappatura del trasferimento del campione dai vasi alla piastra. Assicurarsi che il volume non diminuisca al di sotto di 10 mL durante l'intero corso di coltivazione.
- Aggiungere il numero di campioni da prelevare durante la coltivazione. Simile all'alimentazione, aggiungere il tempo del campione rimosso dalla nave per ogni punto campione di ingresso.
- Salvare il processo. Ora è pronto per l'esecuzione.
NOT: Per garantire il corretto funzionamento del protocollo, passare alla scheda Passaggi di processo nel software di coltura delle celle AMBR e selezionare la vista Passo processo per visualizzare il flusso della ricetta.
- Nella scheda Nuovo esperimento, inserisci il nome dell'esperimento insieme alla data in cui deve essere condotto.
- Progettazione di esperimenti nel microbiore automatizzato
- Per eseguire il software DOE del bioreattore, assicurarsi che la ricetta nel software principale sia salvata e pronta all'uso.
- Aprire il software AMBR 15 DOE e fare clic su Investigazione e selezionare Nuovo.
- Immettere il nome della nuova indagine DOE nella finestra di dialogo Crea indagine.
- Per assegnare un esperimento all'indagine DOE, aprire la ricetta creata per studiare i diversi parametri. Fare clic su Sfoglia e selezionare il rispettivo esperimento.
- Definire il fattore DOE.
- Le etichette delle navi sono già elencate nella colonna. Per definire il fattore DOE desiderato, selezionare il parametro e fare clic sulla colonna denominata Fattore DOE. Selezionare Nuovo e aggiungere le unità, l'abbreviazione, il limite inferiore e superiore dei fattori (ad esempio, temperatura, DO, pH).
- Definire il fattore di risposta.
- Una volta definiti i fattori DOE, definire la risposta in base alla quale sarebbe stata strutturata l'analisi sperimentale.
- Nella scheda Risposte definire i valori da considerare per l'analisi dei dati.
- Fare clic su Modifica risposte DOE e definire il nome della risposta, l'abbreviazione, le unità, i limiti minimi e massimi (ad esempio, tibore, concentrazione cellulare praticabile).
- Una volta definite le risposte, selezionare la variabile AMBR per ogni risposta e definire la variabile. Una risposta può essere associata automaticamente a una variabile micro-bioreattore, Scegliere la variabile richiesta dall'elenco a discesa.
- Modificare l'equazione per ogni risposta in base al requisito. La scelta è tra i dati minimo, massimo, primo, ultimo e medio.
- Creare un progetto.
- Utilizzare la procedura guidata Avvia progetto per selezionare il tipo di progetto sperimentale, per aggiungere o rimuovere il numero di repliche e punti centrali.
- Selezionare l'obiettivo che determina la scelta dei progetti e dei modelli:
Screening: utilizza modelli lineari e di interazione per trovare i fattori importanti
Ottimizzazione (RSM), Utilizza modelli quadratici e cubici per la modellazione e l'ottimizzazione dettagliate
Obiettivo diviso: i modelli per la formulazione e i fattori di processo possono essere scelti separatamente - Una volta deciso l'obiettivo, selezionare il modello e il progetto insieme al numero di punti centrali e repliche.
- Fare clic su Fine e passare alla scheda successiva.
- Definire l'esperimento.
NOT: I fattori DOE sono elencati nella colonna di destra del software. Selezionando i fattori desiderati, verrebbero evidenziate le navi in esecuzione che sperimentano con il parametro desiderato. I vasi all'interno della stazione di coltura possono essere spostati facendo clic con il pulsante destro del mouse sulla nave e spostandola nella posizione desiderata.- Creare pacchetti di lavoro che possono essere importati nel software di impostazioni cultura delle celle AMBR. A seconda del numero di esperimenti, i diversi pacchetti di lavoro vengono creati e memorizzati per un'ulteriore implementazione
- Esecuzione dell'esperimento nei pacchetti di lavoro creati sul computer portatile di controllo AMBR
- Nella scheda Esperimento, fare clic su Crea esperimento DOE e cercare il pacchetto di lavoro creato utilizzando il software DOE.
- Inizializzare il processo facendo clic su Avvia.
- Analisi dei risultati sperimentali
- Una volta eseguito l'esperimento, esportare i dati utilizzando Esporta risultati DOE. Viene visualizzata la finestra Esporta risultati DOE e le righe che indicano la nave di coltura e la stazione sono elencate nella tabella.
- Selezionare le righe desiderate e fare clic su Righe selezionate esportate o Esporta dati sperimentali per memorizzare tutti i risultati e salvare il file per un'ulteriore analisi.
- Importare i dati nel modulo AMBR DOE passando alla scheda Risultati e selezionando Importa risultati.
- Cercare il file di dati desiderato e fare clic su Risultati analisi.
- Analizzare ulteriormente i risultati in MODDE.
4. Esecuzione della coltivazione nel microbiore automatizzato
NOT: I seguenti passaggi vengono eseguiti dall'utente con l'aiuto del protocollo scritto nel software di cui sopra. I passaggi vengono eseguiti dall'utente, salvo diversa.
- Caricamento delle navi
- Aprire i vasi di coltura gamma sterilizzati sotto un armadio di flusso laminare e orientarsi nella stazione di coltura come raffigurato nella Figura 2.
- Pulire la piastra di morsetto con 70% di etanolo e acqua a doppia distillazione. Quindi, autoclave la piastra e posto sulla parte superiore della nave.
- Montare la piastra di morsetto con una piastra di stirrer, assicurando si assicura che ogni perno sia fissato saldamente.
- Stringere la piastra di agitazione e la piastra di morsetto sull'assieme di agitazione.
- Esecuzione del software di microbioreattore
- Utilizzare il programma scritto nella Sezione 3 per eseguire la coltivazione.
- Visualizzare i passaggi del processo programmati o completati nella scheda Processo. Alterare i passaggi di coltivazione durante l'esecuzione del processo in base alle esigenze mettendo in pausa il gestore liquido e quindi modificando la ricetta del processo.
- Aggiungere antifoam ai vasi prima che lo stirrer venga avviato per garantire che non vi sia schiuma eccessiva durante la coltivazione. L'antifoam verrà aggiunto regolarmente e la schiuma rilevata visivamente.
- Aggiunta di supporti nella nave
- Riempire manualmente la 24 piastra fornita con i microreattori con supporti sterili e posizionarla nel ponte designato del sistema. Assicurarsi che la piastra sia posizionata nel ponte designato dal programma scritto (sezione 3). Il riempimento della nave avverrà come previsto nella sezione 3.2.2.
NOT: La temperatura e l'agitatore iniziano subito dopo l'aggiunta del supporto e dell'antifoam. Il lettore di sensori viene attivato 1 ora dopo il riempimento dell'imbarcazione (fase Start Monitor). La gassazione per ogni nave inizia una volta che il lettore è stato attivato. I supporti vengono lasciati a cedere per un minimo di 6 ore prima della ricalibrazione e inoculazione del pH. I parametri di processo possono essere modificati nel software come indicato nella sezione 3.2.3.
- Riempire manualmente la 24 piastra fornita con i microreattori con supporti sterili e posizionarla nel ponte designato del sistema. Assicurarsi che la piastra sia posizionata nel ponte designato dal programma scritto (sezione 3). Il riempimento della nave avverrà come previsto nella sezione 3.2.2.
- Inoculazione
- Misurare la concentrazione cellulare vitale dopo il passaggio di 5th. Calcolare il numero di cellule da trasferire ai vasi per garantire che la concentrazione iniziale delle cellule in tutti i vasi sia 3 x 105 cellule/mL.
- Trasferire le celle in una piastra di pozzo profondo 24 in modo che il volume delle sospensioni sia almeno 1,6 volte il volume richiesto. Per un volume richiesto di 2 mL dell'inoculo, trasferire 3,2 mL di sospensione cellulare in ogni pozzo della piastra.
- Posizionare la piastra 24 pozzo nel ponte designato. Le navi saranno inoculate come nel paragrafo 3.2.4.
- Campionamento giornaliero e analisi
- Eliminare ogni giorno un campione di 460 l dai vasi utilizzando il gestore liquido. Diluire 200 l del campione con 800 l di buffer 1x PBS filtrato (diluizione 5x), quindi inserirlo nel contatore cellulare.
- Centrifugare il campione rimanente per 5 min a 190 x g e temperatura ambiente e conservare il supernatante per ulteriori analisi (glucosio, lattato, glutammina e glutammato).
- Congelare 100 gradi l del supernatante a -20 gradi centigradi fino alla fine della coltivazione per la quantificazione delle proteine.
- Fine della coltivazione
- Quando il controllo dei parametri di processo (ad esempio, temperatura, agitazione, pH e DO) è terminato, interrompere il monitoraggio del processo.
- Svitare la piastra stirrer e la piastra di morsetto.
- Rimuovere i vasi di coltura e pulire le stazioni di coltura. Posizionare le piastre di essiccazione sulle stazioni di coltura e avvitarle.
- Nel frattempo, pulire accuratamente le piastre di morsetto con il 70% di etanolo e l'acqua doppia.
- Fare clic su Stop nel software del bioreattore una volta completato il ciclo di essiccazione.
- Raccolto di coltura cellulare
- Raccogliere le cellule il giorno 12 della coltivazione rimuovendo manualmente il contenuto dei vasi in tubi di centrifuga 50 mL. Centrifugare il brodo cellulare a 190 x g per 30 min.
- Scartare il pellet cellulare e conservare il supernatante a -20 gradi centigradi.
5. Misurazione della concentrazione di mAb
- Utilizzare una colonna A di 1,7 ml per la quantificazione della proteina durante la corsa di coltivazione.
-
Preparare l'equilibratione e il tampone di eguagliazione prima di scongelare i campioni.
- Utilizzare una soluzione di 0,5 M Na2HPO4 contenente 0,5 M NaCl con un pH di 7,9 come buffer di equilibratione e una soluzione di 100 mM glicina contenente 0,5 M NaCl con un pH di 2 come buffer di elusione.
- Filtrare entrambi i buffer attraverso una membrana di 0,2 m e degas prima di essere posizionati per l'analisi.
- Svuotare il sistema di cromatografia liquida ad alte prestazioni (HPLC) con buffer di equilibration appena preparato.
- Caricare la colonna di proteina A sul sistema HPLC.
- Eseguire la cromatografia con una portata di 1 mL/min. Impostare la temperatura del forno della colonna a 30 gradi centigradi e la temperatura del campionista automatico a 10 gradi centigradi
- Scongelare i campioni congelati a temperatura ambiente e filtrare 225 l di ciascun campione attraverso una membrana PVDF di 0,22 m. Diluire i campioni con maggiore concentrazione della proteina desiderata sono in un rapporto 1:20 con il tampone di equilibratimento e filtrano attraverso la membrana prima di essere inseriti nell'autocampionatore.
- Collocare i campioni nell'autocampionatore. Caricare il metodo e la sequenza nel software e avviare la sequenza.
NOT: Il metodo è costituito da tre fasi (vedere Figura 1): iniezione del campione nella colonna per i primi due minuti; seguito da buffer di eluizione per 8 min e rigenerazione a colonne con buffer equilibration per 10 min.
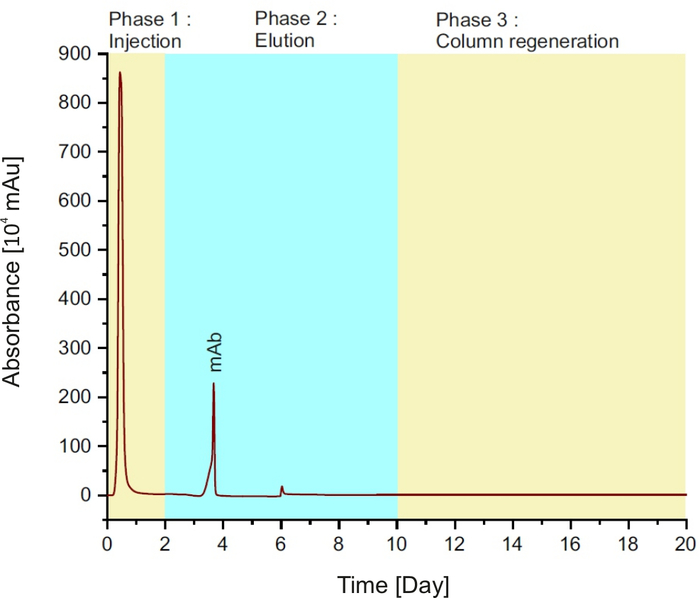
Figura 1: Proteina Un cromatogramma, che rappresenta le diverse fasi durante una singola corsa. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati
Una panoramica della coltivazione effettuata in questo studio è presentata nella Figura 2.
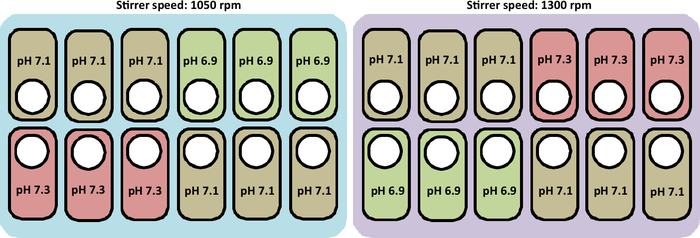
Figura 2: Rappresentazione schematica delle condizioni sperimentali per testare i profili di pH e di velocità dell'agitazione nelle stazioni di coltura. La figura rappresenta anche il layout corretto per posizionare i vasi.
Discussione
L'ottimizzazione del processo per aumentare la resa è di fondamentale importanza nell'industria biofarmaceutica. I flaconi di agitazione potrebbero essere potenzialmente utilizzati per lo screening del ceppo; tuttavia, il monitoraggio dei parametri di processo come pH e DO non sono disponibili nei flaconi. I microreattori hanno un vantaggio in quanto consentono un monitoraggio e un controllo continui del processo. Questi cicli di controllo nel microreattore forniscono anche una condizione simile a quelli su larga scala ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare il Bundesministerium f'r Bildung und Forschung (BMBF), il Ministero federale dell'istruzione e della ricerca, in Germania, e il team di BioProcessing di Sartorius Stedim Biotech GmbH, Germania, per il loro sostegno.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 mL disposable pipette tips, sterilized | Sartorius Stedim Biotech GmbH | A-0040 | |
| 200 mM L-glutamine | Corning, Merck | 25-005-CV | |
| 24 Well deep well plates | Sartorius Stedim Biotech GmbH | A-0038 | |
| 5 mL disposable pipette tips, sterilized | Sartorius Stedim Biotech GmbH | A-0039 | |
| ambr 15 automated microbioreactor system | Sartorius Stedim Biotech GmbH | 001-2804 | |
| ambr 15 Cell Culture 24 Disposable Bioreactors - Sparged | Sartorius Stedim Biotech GmbH | 001-1B86 | |
| Antifoam C Emulsion | Sigma-Aldrich, Merck | A8011 | |
| Bottle Top Sterile filter | Corning, Merck | CLS431474 | 0.1 μm pore size |
| CEDEX Detergent (3% Mucosol) | Roche Innovatis AG | 05-650-658-001 | |
| Cell counter | Roche Innovatis AG | 05-650-216-001 | CEDEX HiRes |
| CHO DG44 cell line | Cellca, Sartorius Stedim Biotech GmbH | ||
| CHOKO Feed Media A (FMA) | Sigma-Aldrich, Merck | CR80025 | |
| CHOKO Feed Media B (FMB) | Sigma-Aldrich, Merck | CR80026 | |
| CHOKO Production Medium | Sigma-Aldrich, Merck | CR80027 | |
| CHOKO Stock Culture Meium | Sigma-Aldrich, Merck | CR80028 | |
| Chromaster high pressure liquid chromatography system | VWR International | ||
| Conical Centrifuge tube | Corning, Merck | SIAL0790 | |
| Ethanol | Merck | 1070179026 | |
| Glycine | Carl Roth | 56-40-6 | |
| HPLC Vials | VWR International | SUPLSU860181 | |
| PBS | Sigma-Aldrich,Merck | P4417 | |
| Protein A Column | Thermo Fisher Scientific | 1502226 | POROS™ A 1.7 mL |
| Sodium chloride | Sigma-Aldrich,Merck | 7647-14-5 | |
| Sodium phosphate dibasic anhydrous | Sigma-Aldrich,Merck | 7558-79-4 | |
| Trypan Blue | VWR International | VWRVK940 | |
| YSI | YSI Inc | 2900D | YSI 2900 Select |
Riferimenti
- Walsh, G. Biopharmaceutical benchmarks 2018. Nature Biotechnology. 36, 1136 (2018).
- Kim, J. Y., Kim, Y., Lee, G. M. CHO cells in biotechnology for production of recombinant proteins: current state and further potential. Applied Microbiology and Biotechnology. 93 (3), 917-930 (2012).
- Lai, T., Yang, Y., Ng, S. K. Advances in Mammalian cell line development technologies for recombinant protein production. Pharmaceuticals (Basel). 6 (5), 579-603 (2013).
- Carlage, T., et al. Analysis of dynamic changes in the proteome of a Bcl-XL overexpressing Chinese hamster ovary cell culture during exponential and stationary phases. Biotechnology Progress. 28 (3), 814-823 (2012).
- Hacker, D. L., de Jesus, M., Wurm, F. M. 25 years of recombinant proteins from reactor-grown cells - where do we go from here. Biotechnology Advances. 27 (6), 1023-1027 (2009).
- Shukla, A. A., Gottschalk, U. Single-use disposable technologies for biopharmaceutical manufacturing. Trends in Biotechnology. 31 (3), 147-154 (2013).
- Ao, S., Gelman, L. . Advances in electrical engineering and computational science. Lecture notes in electrical engineering. 39, (2009).
- Bareither, R., et al. Automated disposable small scale reactor for high throughput bioprocess development: a proof of concept study. Biotechnology and Bioengineering. 110 (12), 3126-3138 (2013).
- Kang, Y., Ludwig, D. L., Balderes, P. What can cell culture flocculation offer for antibody purification processes. Pharmaceutical Bioprocessing. 2 (6), 483-485 (2014).
- Choe, W., Durgannavar, T. A., Chung, S. J. Fc-Binding Ligands of Immunoglobulin G: An Overview of High Affinity Proteins and Peptides. Materials (Basel). 9 (12), (2016).
- Schäpper, D., et al. Application of microbioreactors in fermentation process development: a review. Analytical and Bioanalytical Chemistry. 395 (3), 679-695 (2009).
- Zhang, Z., et al. Microbioreactors for Bioprocess Development. Journal of the Association for Laboratory Automation. 12 (3), 143-151 (2007).
- Claßen, J., et al. Spectroscopic sensors for in-line bioprocess monitoring in research and pharmaceutical industrial application. Analytical and Bioanalytical Chemistry. 409 (3), 651-666 (2017).
- Janoschek, S., et al. A protocol to transfer a fed-batch platform process into semi-perfusion mode: The benefit of automated small-scale bioreactors compared to shake flasks as scale-down model. Biotechnology Progress. 35 (2), 2757 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon