Method Article
IR-TEx: uno strumento open source per l'integrazione dei dati per la trascrittomica dei Big Data progettato per il raggio del malaria Anopheles gambiae
In questo articolo
Riepilogo
IR-TEx esplora i profili trascrizionali legati alla resistenza agli insetticidi nella specie Anopheles gambiae. Qui sono disponibili istruzioni complete per l'utilizzo dell'applicazione, modifiche per esplorare più set di dati trascrittomici e usare il framework per creare un database interattivo per raccolte di dati trascrittomici da qualsiasi organismo, generati in qualsiasi piattaforma.
Abstract
IR-TEx è un'applicazione scritta in Shiny (un pacchetto R) che permette l'esplorazione dell'espressione di (oltre ad assegnare funzioni) trascrizioni la cui espressione è associata a fenotipi di resistenza agli insetticidi nelle zanzare gambiae di Anopheles. L'applicazione può essere utilizzata online o scaricata e utilizzata localmente da chiunque. L'applicazione locale può essere modificata per aggiungere nuovi set di dati di resistenza agli insetticidi generati da più piattaforme -omiche. In questa guida viene illustrato come aggiungere nuovi set di dati e gestire i dati mancanti. Inoltre, IR-TEx può essere ricodificato completamente e facilmente per utilizzare set di dati da qualsiasi dato sperimentale, rendendolo una risorsa preziosa per molti ricercatori. Il protocollo illustra l'utilità dell'IR-TEx nell'identificare nuovi candidati alla resistenza agli insetticidi utilizzando come esempio il transferte microsomico glutathione, GSTMS1. Questa trascrizione è regolata in più popolazioni resistenti ai piretroidi provenienti dalla Costa D'Avorio e dal Burkina Faso. L'identificazione delle trascrizioni co-correlate fornisce ulteriori informazioni sui ruoli putativi di questo gene.
Introduzione
La capacità di misurare l'espressione di un gran numero di trascrizioni simultaneamente attraverso piattaforme di microarray e tecnologia RNAseq ha portato alla generazione di vasti set di dati che associano l'espressione trascrizione a un particolare fenotipo sia negli organismi modello che in quelli non modello. Questi set di dati sono una risorsa estremamente ricca per i ricercatori, il cui potere può essere aumentato combinando set pertinenti in un approccio di integrazione dei Big Data. Tuttavia, questa metodologia è limitata a quelle con particolari competenze bioinformatiche. Descritto qui è un programma, IR-TEx (precedentemente pubblicato da Ingham et al.1) che è scritto in un pacchetto R chiamato Shiny2 e consente agli utenti con poca formazione bioinformatica di integrare e interrogare questi set di dati con relativa facilità.
IR-TEx, trovato a http://www.lstmed.ac.uk/projects/IR-TEx, è stato scritto per esplorare le trascrizioni associate alla resistenza agli insetticidi in Anopheles gambiae, il principale vettore di malaria africano1. La malaria è una malattia parassitaria causata dalle specie di Plasmodium, trasmessa tra gli esseri umani attraverso i morsi di zanzare femminili di Anopheles. Puntare sul vettore della zanzara con insetticidi si è dimostrato il mezzo più efficace per prevenire la morbilità e la mortalità correlate alla malaria in Africa. Anche l'aumento degli strumenti (cioè reti insetticide di lunga durata) è stato fondamentale nelle drastiche riduzioni dei casi di malaria dal 20003. Con un numero molto limitato di insetticidi disponibili, c'è una forte pressione evolutiva sulle zanzare, e la resistenza è ormai diffusa nei vettori della malaria africana4 .
Inoltre, le mutazioni del sito bersaglio5 e la clearance metabolica degli insetticidi6,7 rimangono i principali meccanismi studiati di resistenza, ma altri potenti meccanismi resistenti stanno emergendo1. Molti di questi nuovi meccanismi non sono stati precedentemente associati alla resistenza agli insetticidi, ma sono stati rilevati cercando modelli comuni di espressione genica in più popolazioni resistenti utilizzando l'app IR-TEx e successivamente convalidati funzionalmente dagli approcci genomici1.
Descritto di seguito è un approccio passo-passo all'utilizzo di IR-TEx, sia sul Web che quando installato localmente. Il protocollo descrive come i nuovi set di dati di resistenza agli insetticidi possono essere integrati nel pacchetto esistente e spiega come operare con i dati mancanti. Infine, descrive come utilizzare questo software con altri set di dati -omici che non sono correlati alla resistenza agli insetticidi, combinando così i dati provenienti da diversi approcci -omici mentre opera con valori mancanti e normalizzazione in modo che i dati siano comparabili.
Protocollo
1. Utilizzo dell'applicazione Web IR-TEx
- Esecuzione dell'applicazione in un Web browser
- Aprire l'applicazione Web IR-TEx seguendo il collegamento nella parte inferiore della pagina disponibile in http://www.lstmed.ac.uk/projects/IR-TEx.
- Una volta inizializzata la pagina Web, fare clic sul pulsante Applicazione nella parte superiore della pagina, che visualizzerà l'applicazione e gli output associati.
- Leggere ogni output relativo alla voce predefinita di AGAP008212-RA (CYP6M2) nella casella ID trascrizione con le seguenti condizioni: An. coluzzii dataset che sono (i) esposti a insetticidi piretroidi o (ii) non esposti ad alcuna classe di insetticidi, e trascrizioni associate con una correlazione di >0.98.
- Esplorare l'espressione di una trascrizione di interesse
- Per selezionare una trascrizione di interesse, inserisci l'ID della trascrizione nella casella ID trascrizione, ricordando che le trascrizioni terminano in -RX a seconda dell'isoformazione di interesse.
- Selezionare i set di dati da interrogare selezionando le caselle pertinenti per (i) Paesi; (ii) Stato di esposizione, (iii) Specie di interesse; e (iv) Classe di insecticidi di interesse, il tutto garantendo che questi criteri si traducano in >1 incluso il set di dati (vedi tabella supplementare 1 in Ingham et al.1).
NOTA: (iii) si riferisce al membro del complesso di specie An. gambiae che l'utente interessato. Attualmente sono disponibili dati per An. coluzzii e An. arabiensis. - Fate clic su Aggiorna vista nella parte inferiore del menu di selezione o premete Invio, ignorando valore di correlazione assoluto (per ora).
- Concedere all'applicazione il tempo necessario per l'aggiornamento.
- Leggere il primo grafico come: log2 volte cambio tra una popolazione resistente e una popolazione di zanzare in laboratorio della trascrizione di interesse in ogni set di dati che soddisfa i criteri selezionati nel passaggio 1.2 (Figura 1). I dettagli di tutti i set di dati sono disponibili in Ingham etal.
- Leggere le informazioni sotto il grafico come: i cambiamenti di piegatura tra le zanzare resistenti e suscettibili per ogni set di dati rilevante, oltre ai valori p corretti (Q). Ogni riga rappresenta singoli probe nel microarray. La metodologia per la visualizzazione grafica è stata riportata in precedenza1.
- Leggere la tabella aggiuntiva riportata di seguito come il numero di esperimenti in cui la trascrizione degli interessi è significativa e il numero totale di esperimenti corrispondenti ai criteri selezionati nel passaggio 1.2.
- Per scaricare i dati in formato separato da tabulazioni, fare clic sul pulsante Download sotto le due tabelle. Ciò consente all'utente di esplorare i dati in modo più semplice utilizzando un programma come Excel.This allows the user to explore data in an easier manner using a program such as Excel.
- Interpretare la mappa come segue: ogni punto rappresenta i siti di raccolta approssimativi di zanzare resistenti in ogni set di dati in cui la trascrizione di interesse è espressa in modo differenziale. I colori seguono un sistema a semaforo che viene spiegato nell'app (Figura 2).
- Per i passaggi 1.2.5 e 1.2.8, salvare gli output grafici facendo clic con il pulsante destro del mouse, scegliendo Salva immagine con nome... e scegliendo una cartella appropriata.
NOTA: nell'istanza di un errore di output da parte dell'applicazione, è probabile che nessun set di dati corrisponda ai criteri immessi. Se ciò si verifica, consultare la Tabella supplementare 1 in Ingham etal.
- Identificazione delle funzioni/percorsi putativi di trascrizione di interesse
- Le correlazioni (valore minimo r2 immesso) dei modelli di espressione delle trascrizioni in più set di dati possono essere utilizzate per prevedere la funzione di trascrizione e potenzialmente chiarire le trascrizioni coregolate dallo stesso percorso. Utilizzando l'esempio di Ingham et al.1 (AGAP001076-RA; CYP4G16), seguire i passaggi 1.2.1–1.2.2 nella sezione precedente, selezionando tutti i set di dati per la massima potenza.
- Prima di fare clic su Aggiorna vista, spostare il dispositivo di scorrimento Valore di correlazione assoluto su 0,85 e fare clic su Aggiorna visualizzazione o premere Invio.
- Esaminare la tabella di correlazione (tabella più in basso) per trovare le trascrizioni multiple che sono ora visualizzate e correlate ('r'85) con la trascrizione immessa.
- Manipolare il dispositivo di scorrimento Valore di correlazione assoluto e osservare eventuali modifiche nel grafico e nella tabella più in basso; le uscite del passaggio 1.3.2 rimarranno invariate. Come illustrato nella Figura 3 (z r > 0,9, sr & > 0,8), abbassando la severità del valore di correlazione verranno visualizzate più trascrizioni, ma verrà introdotto più rumore.
- Leggere la tabella sotto l'output grafico, che (oltre ai parametri descritti nel passaggio 1.2.6) contiene il valore di correlazione per ogni trascrizione.
- Per scaricare i dati in un formato separato da tabulazioni, fare clic sul pulsante Download.
- L'analisi dell'arricchimento funzionale può essere eseguita nell'elenco dei documenti di trascrizione scaricati utilizzando DAVID analysis8. Una volta sul sito web DAVID (disponibile in https://david.ncifcrf.gov/),selezionare Analisi funzionale. Incollare l'elenco completo dei geni, utilizzando gli ID genetico [identificatore senza -RX, che può essere fatto in Excel inserendo una colonna a destra dell'ID sistematico e digitando LEFT(X1,10), dove X1 è la cella ID sistematico]. Selezionare l'identificatore come elenco di VectorBase_ID e geni e fare clic su Invia elenco.
- Fare clic sul pulsante Clustering annotazioni funzionali per ottenere una panoramica degli arricchimenti presenti in questa rete di correlazione, consentendo l'assegnazione di una funzione potenziale alla trascrizione. È possibile esplorare gli arricchimenti in profondità esaminando le diverse categorie e facendo clic sui pulsanti , per ognuna e successivamente facendo clic su Grafico.
2. Download e implementazione locale di IR-TEx
- Download ed esecuzione di IR-TEx
- Vai al link trovato allhttp://github.com/LSTMScientificComputing/IR-TEx; e fare clic su Clona o scarica Scarica zip. Indirizzare a una cartella desiderata e decomprimere il file in tale cartella.
- Scaricare l'ultima versione del software R per il sistema operativo appropriato dal collegamento disponibile allhttp://cran.r-project.org/mirrors.html. Installare il programma.
- Scaricare e installare il software R Studio più recente, sempre per il sistema operativo appropriato dal collegamento disponibile allhttp://www.rstudio.com/products/rstudio/download/.
- Una volta installato, aprire R Studio File di codifica supplementare 1 ed eseguire ogni riga per configurare il sistema per IR-TEx.
- Una volta che tutti i pacchetti sono stati installati e aggiornati correttamente come richiesto, andare a File Aprire, individuare IR-TEx.R, evidenziare E aprire. Questo dovrebbe essere visibile nella finestra superiore di R Studio.
- Per eseguire l'app, premi il pulsante Esegui app in alto a destra nella finestra e verrà visualizzata una seconda finestra in cui verrà caricata l'app. Una volta completato il caricamento, per la piena funzionalità fare clic su Apri nel browser situato in alto a destra nella finestra caricata.
- Aggiunta di set di dati di resistenza a IR-TEx (generati utilizzando Anopheles gambiae 15k Agilent array)
- Per aggiungere un nuovo set di dati analizzato generato sulla stessa piattaforma di microarray (A-MEXP-2196) al set di dati disponibile, scaricare l'app e individuare la cartella decompressa scaricata nella sezione 2.1.
- Apri file aggiuntivo 1, che rappresenta un output di un'analisi limma su A-MEXP-2196 1. Utilizzando Excel, nella colonna H1, scrivere Fold_Change, e in H2, scrivere il valore di 2 B2, in cui B2 è la modifica della piega del registro. Applicarlo in tutta la colonna H per produrre modifiche di piegatura grezza.
- Disponi file aggiuntivo 1 in modo che la colonna A sia l'ID, la colonna B è la modifica della piegatura rispetto alla colonna H (copia colonna H, evidenzia la colonna B, quindi fai clic con il pulsante destro del mouse e incolla i valori) e la colonna C è il valore p regolato. Eliminare tutte le altre colonne e salvarle come file delimitato da tabulazioni.
- Aprire il file di codifica supplementare 2 ed eseguirlo utilizzando il foglio delimitato da tabulazioni prodotto nel passaggio 2.2.3.
NEWFILE_FC c('PAESE','STATO DI ESPOSIZIONE','SPECIES','INSECTICIDE')
NEWFILE_Q c('PAESE','STATO DI ESPOSIZIONE','SPECIES','INSECTICIDE')
NOTA: i campi racchiusi tra virgolette singole devono essere modificati per riflettere le informazioni del nuovo set di dati. Lo stato di esposizione si riferisce al fatto che i campioni siano stati raccolti in seguito all'esposizione agli insetticidi (esposti/non esposti). Insetticida: se 'non esposto', utilizzare 'none'. Vedere Fold_Changes.txt. per i metadati di altri campioni. Assicurarsi che l'ortografia sia coerente. - Aprire geography.txt, scorrere fino all'ultima riga occupata e selezionare di seguito. Digitare il nome del set di dati, seguito da Q e NEWFILE_Q nella colonna 1, la latitudine del sito della raccolta di campioni nella colonna 2 e la longitudine nella colonna 3. Salvare le modifiche.
- Se vengono utilizzate voci novelle (ad esempio Gambia), che non sono disponibili per la selezione nel set di dati (vedere Ingham et al. Supplementary Table 11), queste dovranno essere aggiunte al codice. A tale scopo, aprire IR-TEx.R in RStudio e individuare la riga 26 come indicato da RStudio, a quel punto dovrebbe iniziare quanto segue:
'sidebarPanel(....'.
NOTA: ognuna delle righe del procedimento si riferisce a un elemento di metadati immessi nelle righe sotto il nome del set di dati in Fold_Changes.txt nel passaggio 2.2.5. - Per aggiungere i nuovi metadati, scorrere fino alla fine della riga dei metadati prescelta e individuare il termine 'selezionato'. Immediatamente dopo questo dovrebbe essere una virgola e una parentesi chiusa; a questo punto, fare clic sul cursore all'interno della parentesi chiusa. Dopo l'apostrofo finale, digita una virgola, seguita da un apostrofo, seguito dai nuovi metadati (ad esempio, 'Gambia') e salva le modifiche. Per un esempio, vedere di seguito.
checkboxGroupInput('CountryInput','Select Relevant Countries',c('Burkina Faso','Cote D'Ivoire','Camerun','Equatorial Guinea',''''''''''''''''''''Strada',''''''''','Uganda',', 'Togo', 'Gambia'), selezionato:c('Burkina Faso','Cote D'Ivoire','Cameroon','Guinea Equatoriale',''''''''''''''''''''''''''''''''''''''''Uganda','Togo')) - Eseguire l'app. La nuova voce di metadati dovrebbe essere visualizzata come una casella di controllo non selezionata sotto l'intestazione pertinente. Se l'utente desidera che venga selezionato, deve essere aggiunto dopo il selezionato: c(..., come mostrato di seguito:
checkboxGroupInput('CountryInput','Select Relevant Countries',c('Burkina Faso','Cote D'Ivoire','Camerun','Equatorial Guinea',''''''''''''''''''''Strada',''''''''','Uganda',', 'Togo', 'Gambia'), selezionato:c('Burkina Faso','Cote D'Ivoire','Cameroon','Guinea Equatoriale',''''''''''''''''''''''''''''''''''''''''Uganda','Togo', 'Gambia')) - Per aggiungere set di dati di resistenza non eseguiti su A-MEXP-2196, vedere la sezione 3.
3. Modifica di IR-TEx per l'utilizzo con set di dati diversi
- Utilizzare su più piattaforme -omics e procedere con i dati mancanti
- Per procedere con "0" nei set di dati: consultare l'origine del set di dati per il significato specifico di "0". Si consiglia di sostituire "0" con "NA". Come per le variazioni di piegatura grezza (B/A), "0" indica un segnale non rilevato nelle condizioni sperimentali B. Nel caso in cui la condizione sperimentale A presenti un'espressione sostanziale, l'utente può applicare un valore di modifica di piegatura ridotto.
- Apri file aggiuntivo 2.txt, un file RNAseq adattato da Uyhelji et al.9. Questo file rappresenta il modello in cui devono essere basati i nuovi dati: colonna A - identificatore, colonna B - modifica della piega non elaborata e colonna C - valore p modificato. Utilizzare questo file per eseguire i passaggi seguenti.
- Eseguire il codice R per trovare la corrispondenza degli identificatori in un singolo file delimitato da tabulazioni tra piattaforme, quindi organizzare e normalizzare i dati (Supplemental Coding File 2). Le istruzioni sono contenute all'interno del file. Qualsiasi FILEPATH sarà separato da "/" per MacOS o "//" per Windows (cambiarli da """ come appariranno).
- Eseguire l'output del file prodotto alla fine del file di codifica supplementare 2 in un percorso di scelta da utilizzare nel passaggio 3.1.5. Supplemental Coding File 2 emetterà un nuovo file Fold_Changes.txt. Eseguire il backup del file originale.
- Eseguire il codice contenuto nel file di codifica supplementare 3. Individuare il file di output denominato FC_distribPlot.png nella cartella specificata come FILEPATH. Controllare le distribuzioni della modifica di log2 pieghe per verificare che le distribuzioni delle modifiche di ripiegata del log2 siano quasi identiche tra i set di dati.
- Seguire le istruzioni del passaggio 2.2.6 per modificare altri file e garantire la compatibilità del nuovo Fold_Changes.txt.
- Modifica di IR-TEx per l'utilizzo con set di dati completamente nuoviModifying IR-TEx for use with completely new datasets
- Aprire IR-TEx.R in RStudio e individuare le righe (23-34) che iniziano con:
'tabPanel('
e termina in:
submitButton("Visualizzazione aggiornamento", icona("refresh"))
), - Modificare l'AGAP008212-RA trovato nelle righe seguenti in una trascrizione di interesse per i nuovi dati.
textInput('textInput','ID trascrizione','AGAP008212-RA'), - Individuare le quattro opzioni che iniziano con:
checkboxGroupInput(
Queste opzioni possono essere modificate per rappresentare i metadati importanti in base ai quali l'utente desidera filtrare i nuovi dati. In ogni istanza, l'utente deve modificare il Seleziona paesi rilevanti; Selezionare Stato esposizione; Seleziona Specie rilevanti; e Selezionare Classe Insetticida per essere rappresentativi dei dati (ad esempio, Selezionare il tipo di tessuto; Selezionare Sesso; Selezionare Staffa Età; Selezionare Stato malattia). - Identificare i metadati associati al set di dati e all'input per sostituire le opzioni esistenti immediatamente dopo la prima c('. In ogni caso, le opzioni saranno contenute all'interno di segni vocali e separate dalla selezione successiva da una virgola. Dopo la selezione finale, la staffa deve essere chiusa. Un esempio per Selezione stato della malattia è:An example for Select Disease Status is:
c('Infetto', 'Non infettato', 'Sconosciuto') - Scegli quale di questi metadati sarà selezionato all'apertura dell'app. Questi possono essere modificati modificando le opzioni dopo selezionato : c('. Un esempio per Selezione stato della malattia è:An example for Select Disease Status is:
selected:c('Infetto', 'Non infetto')
In questo modo l'app selezionerà solo i set di dati corrispondenti a questi criteri al caricamento iniziale. - Per creare una nuova tabella dati, seguire il layout disponibile in Fold_Changes.txt e le istruzioni nella sezione 2. Modificare i metadati in ogni modifica descritta nel passaggio 3.2.4, esattamente come scritto nel codice (R fa distinzione tra maiuscole e minuscole). Nella colonna di disintossicazione, i nomi dei geni di input e nella colonna del tipo di trascrizione, iminputno le descrizioni dei geni per ogni trascrizione. Seguire la sezione 3.2 quando si aggiungono nuovi set di dati.
- Se il mapping non è rilevante per i requisiti sperimentali, individuare le seguenti righe di codice e inserire il simbolo ' ' in primo piano:
Linee 49-51:
br(),br(),
withSpinner(plotOutput("Geography")),
textOutput('Geography_legend'),
Linee 493 a partire:
output: geografia <- renderPlot(
Alla riga 602 finale:
output-Geography_legend <- renderingText(
paste("Solo trascrizioni significative (p", as.expression("<'"),"0.05): FC > 5 : Rosso, FC > 1 - Ambra, FC < 1 : Verde",sep "")
})
- Aprire IR-TEx.R in RStudio e individuare le righe (23-34) che iniziano con:
Risultati
Utilizzando il file Fold_Changes.txt incluso in IR-TEx, abbiamo confrontato le trascrizioni che erano significativamente espresse in datadimi gambiai Anopheles coluzzii e Anopheles resistenti ai controlli suscettibili della Costa D'Avorio e del Burkina Faso. Ciò ha prodotto 18 trascrizioni di interesse (Tabella 1; questa ricerca può essere eseguita utilizzando Excel, R o altri programmi). Due di questi, un ATPase (AGAP006879) e la z-crystallin (AGAP007160), sono stati precedentemente segnalati, con il primo che ha un effetto significativo sulla resistenza ai piretroidi1. Oltre a queste due trascrizioni, erano presenti due trascrizioni di disintossicazione, GSTMS1 (FC- 1,95 e 1,85) e UGT306A2 (FC- 2,29 e 2,28).
La convalida qPCR di due di queste trascrizioni (GSTMS1, una trascrizione di disintossicazione; e AGAP009110-RA, una trascrizione sconosciuta, specifica per zanzara che contiene un dominio di legame di z-1,3-glucan) sono state eseguite come descritto in precedenza1. L'analisi è stata eseguita utilizzando i gruppi di primer descritti nel file aggiuntivo 3 e ha mostrato che queste trascrizioni sono state significativamente upregolate in una popolazione multiresistente proveniente dalla Costa d'Avorio (Tiassalé) e un'altra dal Burkina Faso (Banfora), rispetto al N'Gousso ( Figura4A)suscettibile al laboratorio .
Poiché entrambe le trascrizioni hanno mostrato una significativa upregulation in ciascuna delle popolazioni resistenti, è stato eseguito un knockdown indotto dall'RNAi sulle zanzare della colonia DistM Tiassalé. Questa colonia proviene dalla Costa D'Avorio ed è resistente a tutte le principali classi di insetticida utilizzate nella salute pubblica, come descritto in precedenza1,10. L'attenuazione dell'espressione di GSTMS1 ha provocato un aumento significativo (p - 0,021) della mortalità dopo l'esposizione alla deltametria rispetto ai controlli iniettati da GFP, dimostrando l'importanza di questa trascrizione nella resistenza ai piretroidi (Figura 4B). Al contrario, il knockdown di AGAP009110-RA non ha provocato variazioni significative (p - 0,082) nella mortalità dopo l'esposizione(Figura 4B).
GSTMS1 è un GST microsomico ed è uno dei tre trovati nelle zanzare A. gambiae 11. Anche se i membri delle classi epsilon e delta delle STO sono stati precedentemente implicati nella disintossicazione insetticida12,13,14, questa è la prima prova della nostra conoscenza per un ruolo di GST microsomici nella resistenza ai piretroidi15. Per esplorare la funzione putativa di questa trascrizione nelle zanzare sl gambiae di Anopheles, sono state identificate l'espressione e la correlazione in IR-TEx. GSTMS1 è stato significativamente sovraespresso in 20 dei 21 set di dati disponibili per queste specie, ad eccezione dell'isola di Bioko. In ogni posizione, la sovraespressione era inferiore a cinque volte rispetto alle popolazioni suscettibili (Figura 5).
Poiché i GG microsomici sono stati in gran parte ignorati come potenziali disintossicazioni insetticidi, si sa poco del loro ruolo nella resistenza agli insetticidi15. Esplorando la correlazione di altre trascrizioni, le funzioni putative possono essere chiarite attraverso l'assunzione di coregolamentazione o coinvolgimento negli stessi percorsi. Per massimizzare la potenza nella rete di correlazione, sono stati selezionati tutti i set di dati microarray presenti in IR-TEx, di >0.75 è stato selezionato. La tabella 2 mostra l'output di IR-TEx.
Queste trascrizioni sono arricchite nell'attività dell'osoreductae e nel metabolismo del glucosio/carboidrati nello strumento di annotazione funzionale di DAVID8. Sia la deidroasi glucosio-6-fosfato che la gamma-lyase di citathione mantengono il livello di glutathione nelle cellule dei mammiferi16,17 e quindi collegano direttamente con GSTMS1, un glutathione-S-transferase. La catalasi è un risponditore di stress ossidativo ad azione rapida che protegge le cellule dai danni reattivi delle specie di ossigeno, un sottoprodotto dell'esposizione ai piretroidi. L'idrolasi valaciclovir è un idrolasi che può svolgere un ruolo nella disintossicazione nelle cellule dei mammiferi18. CYP4H17 è presente anche nella rete di correlazione. I p450 citocromatici sono metabolizzanti diretti di insetticidi piretroidi, e questi prodotti di ripartizione possono essere ulteriormente metabolizzati dalle SGT. Infine, CYP4H17 è stato implicato nella resistenza ai piretroidi in A. funestus19. Nel loro insieme, questi dati sostengono fortemente un ruolo per GSTMS1 nella disintossicazione xenobiotica.
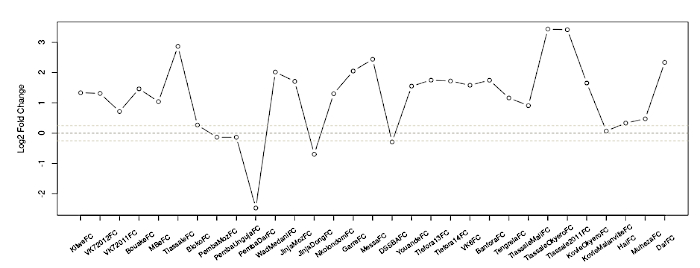
Figura 1: Modifica del registro2 di AGAP002865-RA in tutti i set di dati. L'asse x descrive in dettaglio i diversi set di dati, le cui informazioni sono disponibili nella Tabella supplementari 1 di una pubblicazione precedente1e l'asse y mostra la modifica di piegatura del log2 nella trascrizione degli interessi. Le linee tratteggiate grigio chiaro indicano soglie approssimative per significato, prese qui come un cambio di piegatura di <0.8 o un cambio di piegatura di >1.2. La linea nera tratteggiata indica un cambio di piegatura di 1 (cioè nessuna differenza di espressione tra le popolazioni resistenti e le popolazioni suscettibili). Fare clic qui per visualizzare una versione più grande di questa figura.
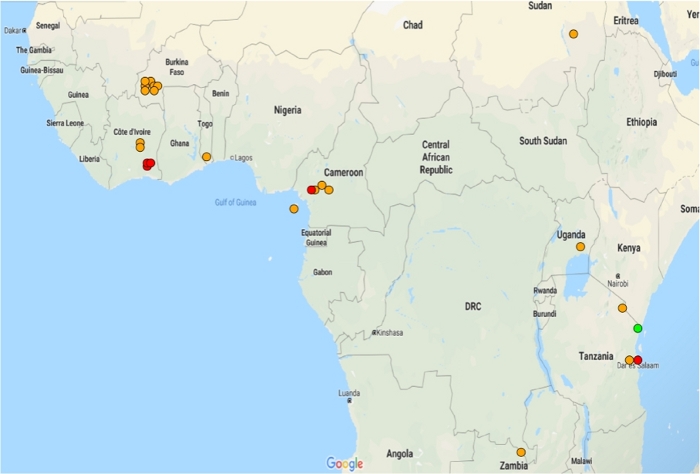
Figura 2: Distribuzione di microarray che mostra una significativa espressione differenziale di AGAP002865-RA nelle popolazioni resistenti. Le modifiche di piegatura sono rappresentate in un sistema a semaforo: cambio piegatura verde di <1, cambio piega arancione di >1 e cambio piega rossa di >5. Vengono visualizzati solo i set di dati con un'espressione differenziale significativa (p - 0,05). Fare clic qui per visualizzare una versione più grande di questa figura.
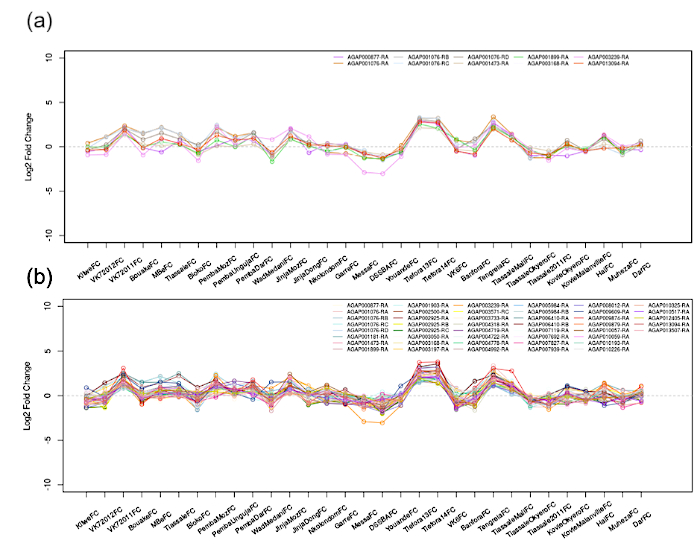
Figura 3: Reti di correlazione di AGAP001076-RA (CYP4G16). Le correlazioni pairwise vengono calcolate in tutte le trascrizioni nei 31 set di dati microarray, con un cut-off definito dall'utente applicato. Mostrato qui è (A) > 0,9 e (B) > 0,8. Tutte le trascrizioni visualizzate nel grafico soddisfano questo criterio e seguono le modifiche di espressione di AGAP001076-RA. Fare clic qui per visualizzare una versione più grande di questa figura.
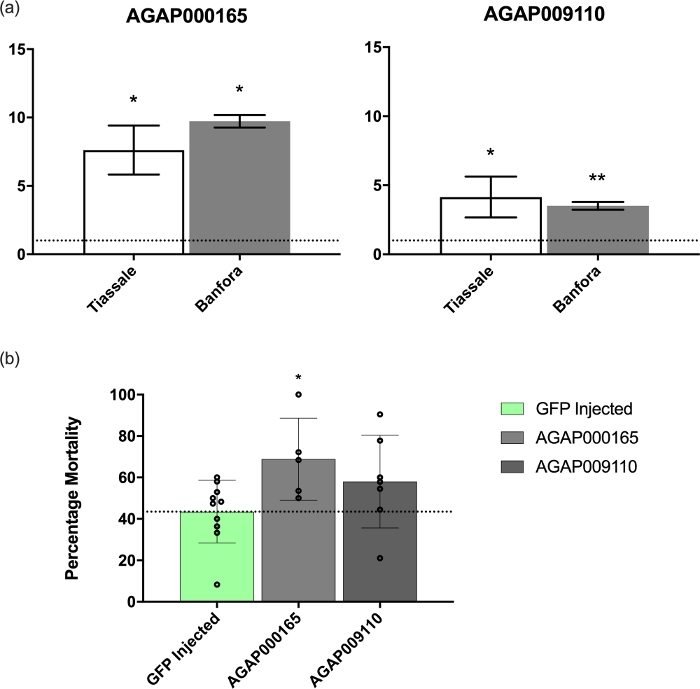
Figura 4: espressione mRNA e fenotipo all'attenuazione di GSTMS1 e AGAP009110-RA. (A) espressione mRNA di GSTMS1 e AGAP009110-RA in due popolazioni multiresistenti An. coluzzii provenienti rispettivamente dalla Costa D'Avorio e dal Burkina Faso. I livelli sono stati confrontati con il laboratorio-suscettibile An. coluzzii N'Gousso. Livelli di significatività calcolati da ANOVA con un test post-hoc di Dunnett. (B) Attenuazione indotta dall'RNAi di entrambe le trascrizioni rispetto ai controlli iniettati dalla GFP. L'attenuazione del GSTMS1 mostra un aumento significativo della mortalità dopo l'esposizione alla deltametrina (calcolata da ANOVA con un test post-hoc per i tukey; Fare clic qui per visualizzare una versione più grande di questa figura.
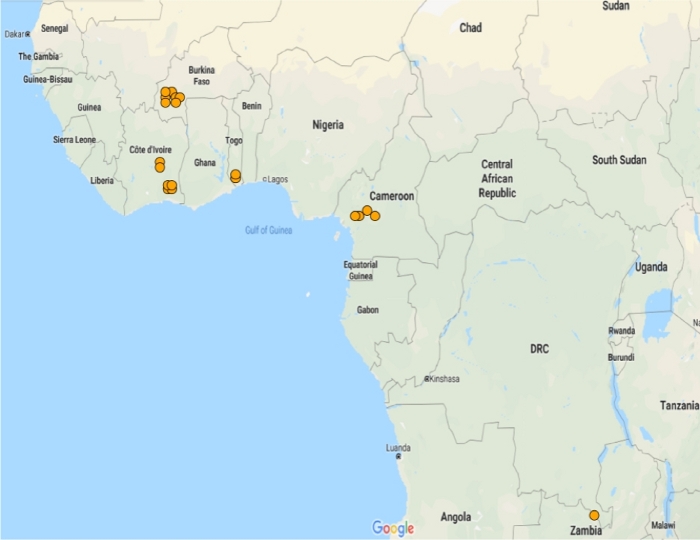
Figura 5: Espressione di GSTMS1 nelle popolazioni di Anopheles gambiae e Anopheles coluzzii. Mappa che mostra l'espressione significativamente differenziale di GSTMS1 nei set di dati di microarray disponibili. GSTMS1 è risultato essere significativamente differenziale in 20 dei 21 set di dati microarray. Fare clic qui per visualizzare una versione più grande di questa figura.
| ID trascrizione | Descrizione | Burkina Faso | Costa d'Avorio |
| AGAP006879-RA | Atpasi | 27.94 | 43.05 |
| AGAP007160-RB | a-crystallin | 11.49 | 10.58 |
| AGAP007160-RC | a-crystallin | 11.14 | 10.38 |
| AGAP007160-RA | a-crystallin | 9.78 | 9.84 |
| AGAP009110-RA | Sconosciuto | 9.26 | 5.96 |
| AGAP007780-RA | Dihydrogenasi NADH | 10.49 | 3.77 |
| AGAP006383-RA | oligosaccharyltransferasis complesso subunit beta | 3.69 | 5.57 |
| AGAP007249-RB | Flightin | 4.61 | 3.86 |
| AGAP003357-RA | Proteina 1-attivazione RAG1 | 4.31 | 4.05 |
| AGAP007249-RA | Flightin | 4.48 | 3.46 |
| AGAP001998-RA | mRpS10 (in quadra mRpS10) | 3.46 | 2.85 |
| AGAP007589-RA | UGT306A2 | 2.29 | 2.28 |
| AGAP000165-RA | GSTMS1 | 1.95 | 1.85 |
| AGAP002101-RA | isoleucyl-tRNA sinotasi | 0.57 | 0.59 |
| AGAP002969-RA | sintetasi asparaginyl-tRNA | 0.45 | 0.45 |
| AGAP004199-RA | famiglia di vettori solute 5 (trasportatore monocarboxylato accoppiato al sodio), membro 8 | 0.35 | 0.48 |
| AGAP004684-RA | proteina di elaborazione del rRNA CGR1 | 0.36 | 0.22 |
| AGAP006414-RA | Cht8 | 0.024 | 0.36 |
Tabella 1: Trascrizioni significativamente differenziali nella stessa piega cambiano direzione attraverso le popolazioni del Burkina Faso e della Costa D'Avorio. ID trascrizione, descrizione del gene e modifica media della piega per ogni set di dati dei due paesi che rappresentano le popolazioni An. coluzzii e An. gambiae.
| Correlazione | Nome sistematico | Tipo di trascrizione |
| 1 | AGAP000165-RA | GSTMS1 |
| 0.82 | AGAP004904-RA | Catalasi |
| 0.76 | AGAP007243-RA | 26S protease regolatoria sottounità 8 |
| 0.79 | AGAP008358-RA | CYP4H17 |
| 0.76 | AGAP009436-RA | Idrolasi Valacyclovir |
| 0.75 | AGAP010739-RA | Glucosio-6-fosfato 1-dehydrogenasi |
| 0.85 | AGAP011172-RA | cistathionine gamma-lyase |
| 0.76 | AGAP012678-RA | Glucosio-6-fosfato 1-dehydrogenasi |
Tabella 2: Trascrizioni co-correlate con GSTMS1. La tabella mostra l'output della rete di correlazione per GSTMS1 su IR-TEx con di >0.75. La tabella mostra la correlazione di Spearman, l'ID della trascrizione e la descrizione del gene per ogni trascrizione co-correlata.
File aggiuntivo 1: file di output dall'array A-MEXP-2196 analizzato su limma. Il file ha origine da un knockdown Met rispetto a una matrice di controlli GFP, descritta in modo più dettagliato in ArrayExpress (E-MTAB-4043) e da un'altra precedente pubblicazione1. Le colonne rappresentano l'identificatore AGAP (SystematicName), la modifica della piega del log (logFC), i valori dell'espressione del log (AveExpr), la statistica t (t), il valore p non corretto (P.Value), il valore p modificato (adj. statistica P.Val) e B (B)20. Ai fini di questo file, le zanzare sono Anopheles coluzzi della Costa D'Avorio e non sono esposte agli insetticidi, con una latitudine di raccolta e longitudine di -5.4 e 6.0, rispettivamente. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
File aggiuntivo 2: file di output dall'esperimento RNAseq. Analisi di RNAseq ricavata da Uyhelji et al.9 che descrive i cambiamenti nel trascrittoma delle zanzare Anopheles quando sono esposte al 50% di salinità. Questo file è adattato dalla tabella S2 della pubblicazione e include l'identificatore AGAP (SystematicID), la modifica della piega non elaborata (Fold_Change) e il valore p regolato (q_value). Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
File aggiuntivo 3: elenco di primer per risultati rappresentativi. L'identificatore AGAP, il nome del gene, il dsRNA in avanti, l'inversione del dsRNA, il forward e il primer inverso qPCR vengono impostati per ogni trascrizione. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
File di codifica supplementare 1. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
File di codifica supplementare 2. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
File di codifica supplementare 3. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
Discussione
La trascrittomica dei Big Data produce elenchi di migliaia di trascrizioni espresse in modo differenziale per ogni condizione sperimentale. Molti di questi esperimenti sono eseguiti su organismi e fenotipi correlati e sono quasi esclusivamente analizzati come esperimenti indipendenti. L'utilizzo di queste ricche fonti di dati esaminando i dati in modo olistico e senza ipotesi teoriche 1) porterà all'identificazione di nuove trascrizioni candidate e 2) impedire lo scarto di dati preziosi semplicemente perché ci sono troppe informazioni da convalidare in vivo1.
IR-TEx offre agli utenti uno sfondo di bioinformatica limitato con la possibilità di esaminare facilmente più set di dati, visualizzare le modifiche nei set di dati e scaricare le informazioni associate1. Sebbene IR-TEx non supporti la ricerca di più di una trascrizione in ogni ricerca, gli utenti possono esaminare i file Fold_Changes.txt associati semplicemente utilizzando Excel, R o altri programmi appropriati. Un'ulteriore utilità di IR-TEx deriva dall'uso di reti di correlazione per prevedere la funzione di trascrizione, l'input di ipotetiche proteine o trascrizioni con funzioni sconosciute e l'uso di software a valle per la ricerca di arricchimenti1.
Nell'esempio dimostrato in questo protocollo, IR-TEx viene utilizzato in base alla funzione originale. Qui, permette l'esplorazione delle trascrizioni associate alla resistenza agli insetticidi e alla visualizzazione della distribuzione di sovra e sottoespressione attraverso la mappatura grafica. Le trascrizioni di interesse sono convalidate in vivo per determinare se l'eccessiva o sottoespressione di determinate trascrizioni contribuisce a un fenotipo osservato1 (ad esempio, resistenza agli insetticidi). È stato dimostrato qui, come precedentemente riportato1, che un set di dati può essere utilizzato in un approccio basato su ipotesi per identificare le trascrizioni di interesse su base nazionale. IR-TEx può quindi essere utilizzato per esplorare l'espressione della trascrizione e 2) contestualizzare la funzione della trascrizione applicando una rete di correlazione a coppie tra tutte le trascrizioni contenute in ogni set di dati -omics. In questo caso, GSTMS1 ha dimostrato di essere co-correlato con una serie di altre trascrizioni implicate nella disintossicazione. Questi dati (insieme all'abbattimento della trascrizione che ha provocato un significativo aumento della mortalità dopo l'esposizione agli insetticidi) dimostrano l'importanza di questa trascrizione nella clearance xenbiotica.
IR-TEx rappresenta una risorsa preziosa per esplorare le trascrizioni relative alla resistenza agli insetticidi sul web o utilizzando applicazioni locali. Questo protocollo dimostra come modificare IR-TEx per diverse piattaforme -omics e dati completamente nuovi. La guida illustra come utilizzare IR-TEx per integrare dati da più piattaforme -omiche e set di dati con dati mancanti, nonché come ricodificare IR-TEx semplicemente in modo che sia utile per chiunque cerchi set di dati tracriomici.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato finanziato da una MRC Skills Development Fellowship a V.I. (MR/R024839/1) e dalla Royal Society Challenge Grant (CH160059) a H.R.
Materiali
| Name | Company | Catalog Number | Comments |
| Laptop with browser | Any | - | - |
| R Program | The R Project for Statistical Computing | - | https://www.r-project.org/ |
| R Studio | R Studio | - | https://www.rstudio.com/ |
Riferimenti
- Ingham, V. A., Wagstaff, S., Ranson, H. Transcriptomic meta-signatures identified in Anopheles gambiae populations reveal previously undetected insecticide resistance mechanisms. Nature Communications. 9 (1), 5282 (2018).
- Chang, W., Cheng, J., Allaire, J., Xie, Y., McPherson, J. . shiny: Web Application Framework for R. , (2017).
- Bhatt, S., et al. The effect of malaria control on Plasmodium falciparum in Africa between 2000 and 2015. Nature. 526 (7572), 207-211 (2015).
- Ranson, H., Lissenden, N. Insecticide Resistance in African Anopheles Mosquitoes: A Worsening Situation that Needs Urgent Action to Maintain Malaria Control. Trends in Parasitology. 32 (3), 187-196 (2016).
- Donnelly, M. J., et al. Does kdr genotype predict insecticide-resistance phenotype in mosquitoes. Trends in Parasitology. 25 (5), 213-219 (2009).
- Stevenson, B. J., et al. Cytochrome P450 6M2 from the malaria vector Anopheles gambiae metabolizes pyrethroids: Sequential metabolism of deltamethrin revealed. Insect Biochemistry and Molecular Biology. 41 (7), 492-502 (2011).
- Müller, P., et al. Field-Caught Permethrin-Resistant Anopheles gambiae Overexpress CYP6P3, a P450 That Metabolises Pyrethroids. PLoS Genetics. 4 (11), 1000286 (2008).
- Huang, D., et al. The DAVID Gene Functional Classification Tool: a novel biological module-centric algorithm to functionally analyze large gene lists. Genome Biology. 8 (9), 183 (2007).
- Uyhelji, H. A., Cheng, C., Besansky, N. J. Transcriptomic differences between euryhaline and stenohaline malaria vector sibling species in response to salinity stress. Molecular Ecology. 25 (10), 2210-2225 (2016).
- Edi, C. V., Benjamin, K. G., Jones, C. M., Weetman, D., Ranson, H. Multiple-Insecticide Resistance in Anopheles gambiae Mosquitoes, Southern Côte d’Ivoire. Emerging Infectious Diseases. 18 (9), 1508-1511 (2012).
- Ding, Y., Ortelli, F., Rossiter, L., Hemingway, J., Ranson, H. The Anopheles gambiae glutathione transferase supergene family: annotation, phylogeny and expression profiles. BMC Genomics. 4 (1), 1-16 (2003).
- Enayati, A. A., Ranson, H., Hemingway, J. Insect glutathione transferases and insecticide resistance. Insect Molecular Biology. 14 (1), 3-8 (2005).
- Ranson, H., et al. Identification of a novel class of insect glutathione S-transferases involved in resistance to DDT in the malaria vector Anopheles gambiae. The Biochemical Journal. 359, 295-304 (2001).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), 27 (2014).
- Pavlidi, N., Vontas, J., Van Leeuwen, T. The role of glutathione S-transferases (GSTs) in insecticide resistance in crop pests and disease vectors. Current Opinion in Insect Science. 27, 97-102 (2018).
- Salvemini, F., et al. Enhanced glutathione levels and oxidoresistance mediated by increased glucose-6-phosphate dehydrogenase expression. Journal of Biological Chemistry. 274 (5), 2750-2757 (1999).
- Deplancke, B., Gaskins, H. R. Redox control of the transsulfuration and glutathione biosynthesis pathways. Current Opinion in Clinical Nutrition & Metabolic Care. 5 (1), (2002).
- Puente, X. S., López-Otn, C. Cloning and expression analysis of a novel human serine hydrolase with sequence similarity to prokaryotic enzymes involved in the degradation of aromatic compounds. Journal of Biological Chemistry. 270 (21), 12926-12932 (1995).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. G3: Genes, Genomes, Genetics. 7 (6), 1819-1832 (2017).
- Smyth, G. K. Linear models and empirical bayes methods for assessing differential expression in microarray experiments. Statistical Applications in Genetics and Molecular Biology. 3 (1), 3 (2004).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon