È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Determinazione dell'efficienza dell'inibitore chimico contro la crescita intracellulare del Toxoplasma Gondii utilizzando un saggio di crescita basato su Luciferase
In questo articolo
Riepilogo
Presentato qui è un protocollo per valutare l'efficacia dell'inibizione dei composti chimici contro la crescita intracellulare in vitro di Toxoplasma gondii utilizzando un analisi della crescita basata sulla luciferasi. La tecnica viene utilizzata per confermare la specificità dell'inibizione mediante la delezione genetica del gene bersaglio corrispondente. L'inibizione di LHVS contro la proteasi TgCPL viene valutata come esempio.
Abstract
Il toxoplasma gondii è un patogeno protozoo che colpisce ampiamente la popolazione umana. Gli attuali antibiotici utilizzati per il trattamento della toxoplasmosi clinica sono limitati. Inoltre, essi esibiscono effetti collaterali negativi in alcuni gruppi di persone. Pertanto, la scoperta di nuove terapie per la toxoplasmosi clinica è imperativa. Il primo passo dello sviluppo di nuovi antibiotici è quello di identificare composti chimici che mostrano un'elevata efficacia nell'inibizione della crescita dei parassiti utilizzando una strategia di screening ad alto throughput. Come agente patogeno intracellulare obbligato, Toxoplasma può replicarsi solo all'interno delle cellule ospiti, il che vieta l'uso di misurazioni ottiche di assorbimento come indicatore rapido della crescita. Presentato qui è un protocollo dettagliato per un saggio di crescita di luciferasi. Ad esempio, questo metodo viene utilizzato per calcolare il tempo di raddoppio dei parassiti Toxoplasma di tipo selvatico e misurare l'efficacia del morfnurea-leucyl-omofenicolo-vicinquel-vinile sulfone fenil (LHVS, un composto che mira la proteasi cisteina) per quanto riguarda l'inibizione della crescita intracellulare. È stato descritto anche un protocollo di eliminazione genica basato su CRISPR-Cas9 nel Toxoplasma che utilizza regioni omologhe da 50 bp per la ricombinazione dipendente dall'omologia (HDR). Quantificando i efficacidi di inibizione di LHVS in wild-type e TgCPL (Toxoplasma cathepsin L-like protease)-deficiente-deficiente parassiti, è dimostrato che LHVS inibisce la crescita del parassita di tipo selvatico più efficiente rispetto alla crescita zcpl, suggerendo che TgCPL è un obiettivo che LHVS si lega a Toxo. L'elevata sensibilità e il facile funzionamento di questo analisi della crescita di luciferasi lo rendono adatto per monitorare la proliferazione di Toxoplasma e valutare l'efficacia dei farmaci in modo ad alto consumo.
Introduzione
Toxoplasma gondii è un parassita intracellulare di grande successo che infetta circa un terzo della popolazione umana. Il suo alto tasso di trasmissione è principalmente dovuto alle sue diverse vie di trasmissione, tra cui il consumo di carne poco cotta, l'esposizione ai serbatoi di mammiferi e la trasmissione congenita durante la nascita. T. gondii provoca principalmente infezioni opportunistiche che possono portare a una grave morbilità e mortalità in individui immunocompromessi1,2,3,4,5,6. Gli antibiotici attualmente utilizzati per il trattamento della toxoplasmosi acuta sono particolarmente inefficienti nel trattamento delle infezioni congenite e latenti e causano gravi reazioni in alcuni individui3,7,8. Pertanto, esiste un'urgente necessità di identificare nuove terapie. Comprendere le differenze nei processi subcellulari all'interno di Toxoplasma e del suo ospite aiuterà a identificare potenziali bersagli farmacologici. Pertanto, sono necessarie tecniche di manipolazione del genoma efficienti e convenienti per studiare i ruoli dei singoli geni all'interno di Toxoplasma. Inoltre, Toxoplasma appartiene al phylum Apicomplexa, che include diversi altri patogeni umani significativi, come Plasmodium spp. e Cryptosporidium spp. Quindi, Toxoplasma può essere utilizzato come organismo modello per aiutare a studiare la biologia di base in altri parassiti apicomplessi.
Per identificare nuovi antibiotici contro gli agenti patogeni microbici, viene inizialmente eseguito lo screening ad alto throughput di una biblioteca di composti chimici per determinarne l'efficacia nella repressione della crescita microbica. Finora, sono stati sviluppati diversi saggi di crescita basati su microplaciper per misurare la crescita intracellulare di T. gondii (cioè, quantificazione radioattiva 3H-uracil basata sull'incorporazione9, rilevamento quantitativa dei parassiti a base di ELISA utilizzando anticorpi specifici T. gondii10,11, misurazione basata sulle proteine dei reporter utilizzando ceppi Toxoplasma che esprimono YFP12,13e un'imaging ad alto contenuto recentemente sviluppata comesay14).
Queste strategie individuali hanno tutti vantaggi unici; tuttavia, alcune limitazioni limitano anche le loro applicazioni. Ad esempio, poiché il toxoplasma può replicarsi solo all'interno di cellule animali nucleate, l'autofluorescenza e il legame non specifico di anticorpi anti-T.gondii per ospitare le cellule causano interferenze nelle misurazioni basate sulla fluorescenza. Inoltre, l'uso di isotopi radioattivi richiede una particolare conformità alla sicurezza e potenziali problemi di sicurezza. Alcuni di questi saggi sono più adatti per valutare la crescita in un unico momento piuttosto che un monitoraggio continuo della crescita.
Presentato qui è un protocollo basato su luciferasi per la quantificazione della crescita intracellulare del Toxoplasma. In uno studio precedente, il gene della luciferasi di NanoLuc è stato clonato sotto il promotore di tubulina Toxoplasma, e questo costrutto di espressione luciferasi è stato transincato in parassiti di tipo selvaggio (RHku80) per creare un RHku80zhxg::ceppo NLuc (chiamato RHku80::NLuc qui dopo)15. Questo ceppo è servito come ceppo parentale per la determinazione della crescita intracellulare e la delezione genica in questo studio. Utilizzando il ceppoRH-ku80::NLuc, la crescita dei parassiti nei fibroblasti prepuzio (HFF) è stata monitorata in un periodo di 96 ore dopo l'infezione per calcolare il tempo di raddoppio del parassita.
Inoltre, l'efficacia dell'inibizione di LHVS contro la crescita dei parassiti può essere determinata tracciando i tassi di crescita di Toxoplasma contro le concentrazioni seriali di LHVS per identificare il valore di IC50. Letteratura precedente ha riferito che TgCPL è un obiettivo principale di LHVS nei parassiti e che il trattamento con LHVS diminuisce lo sviluppo di infezioni toxoplasma acute e croniche16,17,18,19. Inoltre, ilku80ceppo parentale per la modifica del genoma èstato utilizzato come ceppo parentale per generare un ceppo TgCPL-deficiente (RHku80::NLuc), e l'inibizione di LHVS è stata misurata rispetto a questo mutante. Osservando un upshift di valori DiC50 per LHVS nei parassiti carenti di TgCPLrispetto al ceppo WT, è stato convalidato che TgCPL è preso di mira da LHVS in vivo.
In questo protocollo, RHku80::NLuc viene utilizzato come ceppo parentale, che manca di un efficiente percorso di giunzione finale non omologa (NHEJ), facilitando così la ricombinazione dipendente da omologia crossover (HDR)20,21. Inoltre, le regioni omologhe di 50 bp sono affiancate ad entrambe le estremità di una cassetta di resistenza ai farmaci da PCR. Il prodotto PCR funge da modello di riparazione per rimuovere l'intero locus genico tramite HDR utilizzando strumenti di editing del genoma basati su CRISPR-Cas9. Tali regioni omologhe brevi possono essere facilmente incorporate in primer, fornendo una comoda strategia per la produzione del modello di riparazione. Questo protocollo può essere modificato per eseguire la delezione genica universale e l'etichettatura genica endogena.
Ad esempio, nella nostra pubblicazione più recente, tre geni proteasi, TgCPL, TgCPB ( Proteasi a cathepsina B-comeToxoplasma) e TgSUB1 ( proteasi simile allatossoplasma 1), sono stati geneticamente ablati in TgCRT (Toxoplasma cloroleccanter-resistanceer) parassiti carenti utilizzando questo metodo15. Inoltre, TgAMN (aminopeptidase putativo N [TgAMN, TGGT1_221310]) è stato etichettato endogenamente15. Il laboratorio di Lourido ha anche riferito di aver usato brevi regioni omologhe nell'intervallo di 40-43 bp per l'introduzione della mutazione genica diretta dal sito e dell'etichettatura genica endogena nel genoma del Toxoplasma utilizzando un metodo simile22. Queste modifiche del genoma di successo suggeriscono che una regione omologa di 40-50 bp è sufficiente per una ricombinazione efficiente del DNA nel ceppo tgKU80-carente,che semplifica notevolmente la manipolazione del genoma in Toxoplasma gondii.
Protocollo
Toxoplasma gondii è classificato nel Gruppo di Rischio 2 e deve essere gestito a livello di biosicurezza 2 (BSL-2). Il protocollo è stato rivisto e approvato dal Comitato di Biosicurezza Istituzionale dell'Università di Clemson.
1. Saggio sulla crescita di Toxoplasma basato su Luciferate
- Fibroblasti di prepuzio umano di semi (HFF) 1 settimana prima dell'inoculazione dei parassiti per garantire che le cellule ospiti siano completamente confluenti. Eseguire un saggio fittizio in una piastra trasparente per garantire che i parassiti rimangano intracellulari per tutto il periodo di valutazione.
NOTA: Qui, il saggio è condotto in 96 microplacche ben. Secondo le esigenze sperimentali, può essere scalato fino a 384 o 1536 microplacche ben. - Passare i parassiti toxoplasma in HFF confluenti 2 giorni prima dell'uso trasferendo 0,3-0,4 mL di parassiti completamente lisci in una flacone T25. Incubare le cellule ospiti infette a 37 gradi centigradi con il 5% di CO2 per 2 giorni.
- Siringa 5 mL di parassiti appena lised attraverso un ago di sicurezza 21 G 5x per liberare i parassiti intracellulari, quindi passare attraverso un filtro di 3 m per rimuovere i detriti delle cellule ospiti. Risciacquare i parassiti residui dal flacone usando 7 mL di fenolo medio-libero D10 medio, quindi passare attraverso il filtro di nuovo.
- Centrifuga parassiti a 1000 x g per 10 min a temperatura ambiente (RT). Versare il supernatante e risospendere il pellet in 10 mL di fenolo senza rosse D10.
- Contare i parassiti usando un emocitometro per determinare la concentrazione.
- Diluire i parassiti a 1 x 104 parassiti/mL per il ceppo di tipo selvaggio (WT). Per i ceppi di parassiti carenti di crescita, aumentare la concentrazione di conseguenza per osservare un aumento significativo nei segnali luciferasi.
- Aspirati i media con attenzione da 96 microplacche ben pre-semied con HFF e inoculato 150 -L di sospensione parassita in pozzi in un formato di tre colonne e cinque file, che rappresenta tre repliche tecniche e cinque momenti.
- Incubare la micropiastra a 37 e il 5% di CO2 per 4 h.
- Aspirati i media con attenzione dai pozzi per rimuovere i parassiti non invasi, quindi riempiono i pozzetti con supporti senza rosso fenolo RT in ogni riga (ad eccezione della prima riga).
- Mescolare volumi uguali di PBS e 2x luciferase assaggio tampone e diluire il substrato luciferasi a 12,5 M.
- Aggiungere 100 l di substrato luciferasi diluito in ogni pozzetto della fila superiore. Incubare le microplacche a RT per 10 min per consentire alle cellule di eseguire completamente la liscivia.
- Misurare l'attività della luciferasi utilizzando un lettore di microplacino. Le impostazioni del lettore di lastre sono elencate nella Tabella 1. Ogni lettura rappresenta il numero iniziale di parassiti invasi a 4 h dopo l'infezione.
- Ripetere i passaggi da 1,9 a 1,12 per ogni riga ogni 24 ore per 4 giorni senza modificare il supporto. Queste letture riflettono il numero totale di parassiti replicati a 24 h, 48 h, 72 h e 96 h dopo l'infezione.
- Calcolare le letture medie ad ogni punto temporale e dividerle per le letture medie a 4 h per determinare i cambiamenti di piega nella crescita dei parassiti nel tempo.
- Tracciare i dati utilizzando un software di grafica. Una tabella di lettura di crescita rappresentativa e le trame diRH-ku80:: i parassitiNLuc sono mostrati in Figura 1A, B.
- Per calcolare il tempo di raddoppio, tracciare i valori log2 delle modifiche di piegatura nei singoli punti temporali nel tempo di incubazione. Utilizzare una funzione di regressione lineare per calcolare la pendenza, che rappresenta il tempo di raddoppio di ogni deformazione (Figura 1A,C).
2. Valutazione dell'efficacia dell'inibizione del composto chimico rispetto alla crescita del toxoplasma
NOTA: Qui, la valutazione dell'inibizione dell'LHVS nella crescita di Toxoplasma è presentata come esempio. Vengono testate otto diverse concentrazioni di LHVS, e vengono eseguite tre repliche tecniche per ciascuna delle tre repliche biologiche per entrambi i ceppi RHku80::NLuc eRH-ku8 zcpl::NLuc.
- Prima dell'infezione da parassiti, gli HFF di semi a 96 microplacche ben nel formato di tre righe e nove colonne per una replica biologica per ogni ceppo. Le cellule ospiti potranno crescere per almeno 7 giorni prima dell'uso.
- Passare i parassitiRH:ku80::NLuc eRH-ku8-cpl::NLuc per 2 giorni prima dell'uso. Seguire i passaggi da 1.2-1.6 per la purificazione e la quantificazione dei parassiti. Risospendere i parassiti nei supporti fenolici senza rosso a 1 x 104 parassiti/mL.
- Aspirare i supporti da una piastra contenente HFF confluenti e inoculare ogni pozzo con 150 l di sospensione parassita. Incubare la micropiastra a 37 e il 5% di CO2 per 4 h.
- Preparare LHVS a otto diverse concentrazioni in un serbatoio di 12 pozzi per diluizione seriale. In generale, le concentrazioni sono ridotte di tre volte in modo diluizione seriale.
NOTA: La concentrazione più bassa è ridotta di 6.561 volte rispetto alla più alta concentrazione. Il cambiamento di piegatura della diluizione può essere regolato di conseguenza in base alle diverse proprietà dei singoli composti. - Alle 4 h dopo l'infezione, aspirare i media per rimuovere i parassiti non invasi e riempire ogni pozzo dalle colonne 2-9 con 150 - L di supporti integrati con LHVS a diverse concentrazioni. Lasciare la prima colonna riempita con un supporto regolare per fungere da controllo non trattato.
- Incubare la micropiastra a 37 e il 5% di CO2 per altri 96 h.
- Eseguire i passaggi 1.9-1.11 e misurare l'attività luciferasi dei singoli pozzi.
- Media delle attività luciferasi di tre repliche tecniche da pozzi di ogni singola concentrazione di LHVS.
- Dividere l'attività media di luciferasi per ogni concentrazione di LHVS per l'attività di luciferasi media derivata da parassiti non trattati per calcolare l'attività di luciferasi normalizzata come percentuale.
- Tracciare le attività di luciferasi normalizzate rispetto alle singole concentrazioni di LHVS utilizzando un software grafico (Figura 2). L'inibizione della pirimethamina contro la crescita parassitaria è misurata anche come controllo. Pyrimethamine è un antibiotico clinico usato per trattare la toxoplasmosi acuta inibendo il metabolismo degli acidi folichi nel Toxoplasma.
- Calcolare i valori IC50 per i singoli composti utilizzando il metodo incorporato nel software di grafica, risposta normalizzata vs. L'IC50 viene calcolato utilizzando la seguente formula:
Y : 100/(1 X/IC50)
Dove: Y rappresenta le attività di luciferasi normalizzate di cellule infette in diverse concentrazioni di inibitori, e X rappresenta concentrazioni individuali di inibitore.
3. Delezione genica a base di CRISPR-Cas9 nei parassiti di Toxoplasma
- Generazione di un costrutto plasmide che esprime l'RNA (sgRNA) e il Cas9 per l'eliminazione di un gene di interesse
- Vai a www.ToxoDB.org e recupera l'intera sequenza di codifica genica, inclusi introni ed esoni, insieme a 1,5 kb 5'-UTR e 3'-UTR (regioni non tradotte).
NOTA: In questo caso, TgCPL (TGGT1_321530) è destinato come esempio rappresentativo. - Copiare la sequenza TgCPL recuperata nel software di analisi della sequenza (fare riferimento alla Tabella dei materiali per il nome e la versione) ed etichettare le regioni 5' e 3'-UTR.
- Seleziona l'icona Strumenti nella barra dei menu in alto, quindi seleziona Clonazione Trovare Siti CRISPR.
- Scegliere 3'(Cas9)' per la posizione del sito PAM e selezionare la cartella contenente la sequenza genomica Toxoplasma nella sezione del punteggio di specificità. Lasciare le altre impostazioni come predefinite.
- Scegliere uno sgRNA con i seguenti due criteri: 1) mostrando un punteggio di specificità elevato, generalmente >98%, e 2) privo di una G dopo il NGG, una sequenza di motivi adiacenti (PAM) protospaziale. Lo sgRNA selezionato si trova di solito in siti vicini all'inizio e arresto dei codoni del gene di interesse.
- Copiate la sequenza dello sgRNA selezionato e incollatela nel seguente modello di primer.

La porzione in rosso rappresenta la sequenza di sgRNA TgCPL selezionata. Può essere sostituito con diversi sgRNA per vari geni di interesse.
NOTA: Se lo sgRNA selezionato non inizia con G, aggiungere G all'inizio dello sgRNA per contribuire a migliorarne l'espressione. - Eseguire una reazione PCR per modificare il plasmide preesistente esprimendo sgRNA (Figura 3A) che si rivolge toxoplasma uracil phosphoribosyltransferase (TgUPRT) gene23 utilizzando un premix PCR con le impostazioni fornite nella tabella 2.
- Eseguire il prodotto PCR su un gel di agarose per confermare l'amplificazione di successo. Un prodotto PCR da 10 kb dovrebbe essere amplificato (Figura 3B).
- Estrarre il prodotto PCR utilizzando un kit di estrazione gel di DNA e circolarlo utilizzando un kit di mutagenesi diretto al sito. Fare riferimento alla tabella 3 per la ricetta. Incubare la reazione per 10-20 min a RT.
- Trasforma il prodotto PCR circolare in E. coli e scegli 10 cloni per un'ulteriore verifica dell'incorporazione dello sgRNA progettato.
- Crescere due cloni ed estrarre plasmidi. Tagliare le plasmidi purificate con BamHI ed EcoRV. I plasmidi candidati produrranno due bande a 2,4 kb e 7,2 kb (Figura 3C).
- Inviare i plasmidi per il sequenziamento di Sanger utilizzando i primer inversi M13 per confermare la sostituzione corretta dello sgRNA TgUPRT con lo sgRNA progettato (Figura 3D).
- Vai a www.ToxoDB.org e recupera l'intera sequenza di codifica genica, inclusi introni ed esoni, insieme a 1,5 kb 5'-UTR e 3'-UTR (regioni non tradotte).
- Generazione di un modello di riparazione per la cancellazione genica tramite il meccanismo HDR
- Secondo i siti di targeting dello sgRNA selezionato, individuare 50 bp di 5'-UTR o 3'-UTR del gene bersaglio per la ricombinazione dipendente dall'omologia (HDR, vedi sezione discussione). La selezione delle regioni segue i criteri elencati di seguito, a seconda della posizione dei bersagli sgRNA.
- Se il sito di scissione di Cas9 si trova a monte del codone iniziale, selezionare quanto segue: una sequenza di DNA di 50 bp a monte del sito di scissione come regione HDR sinistra e una sequenza di DNA di 50 bp a valle del codone di arresto come regione HDR destra.
- Se il sito di scissione di Cas9 si trova tra i codoni di avvio e arresto, selezionare le seguenti opzioni: una sequenza di DNA di 50 bp a monte del codone di partenza come regione HDR sinistra e una sequenza di DNA di 50 bp a valle del codone di arresto come regione HDR destra.
- Se il sito di scissione di Cas9 si trova a valle del codone di arresto, selezionare quanto segue: una sequenza di DNA di 50 bp a monte del codone iniziale come regione HDR sinistra e una sequenza di DNA di 50 bp a valle del sito di scissione come regione HDR destra.
NOTA: Per il gene TgCPL, il sito di scissione si trova tra i codoni di partenza e arresto. Così, i seguenti primer sono progettati per amplificare il modello di riparazione utilizzando pMDC64 come modello, che codifica una cassetta di resistenza alla pirimethamina. Le sequenze in nero anneal al plasmide pMDC64 per amplificazione PCR. Le regioni etichettate in rosso sono sequenze specifiche di TgCPLper la ricombinazione omologa.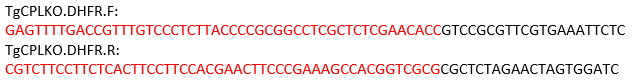
- Eseguire PCR utilizzando un premix PCR nelle condizioni PCR descritte nella tabella 4.
- Eseguire il prodotto PCR su un gel di agarose (Figura 3E), seguito dall'estrazione del gel e dalle procedure standard di quantificazione dell'acido nucleico.
NOTA: se la banda prevista non può essere amplificata correttamente, ottimizzare le condizioni PCR e/o cambiare le coppie di primer.
- Secondo i siti di targeting dello sgRNA selezionato, individuare 50 bp di 5'-UTR o 3'-UTR del gene bersaglio per la ricombinazione dipendente dall'omologia (HDR, vedi sezione discussione). La selezione delle regioni segue i criteri elencati di seguito, a seconda della posizione dei bersagli sgRNA.
- Trasfezione toxoplasma
- PassareRHku80::NLuc parassiti per 2 giorni in una fiaschetta T25 contenente HFF confluenti. Una fiaschetta T25 di parassiti completamente lisci è sufficiente per due o tre trasfettazioni.
- Siringa e parassiti purificanti dal filtro come descritto al punto 1.2. Risospendere i parassiti nel tampone di citomix e girare verso il basso a 1.000 x g per 10 min a RT.
- Lavare i parassiti pelleta con 10 mL di tampone di citomix e far girare verso il basso i parassiti a 1.000 x g per 10 min a RT.
- Versare con attenzione il supernatante e risospendere i parassiti nello stesso buffer ad una concentrazione di 1 x 108 parassiti/mL.
- Mescolare 2 g di DNA modello di riparazione con 20 g di plasmidi espressione sgRNA/Cas9 (rapporto di massa : 1:5, equivalente a un rapporto molare 1:3). Se la resa dell'amplificazione del modello di riparazione è bassa, ridurre di conseguenza l'input di entrambi i pezzi di DNA. È possibile utilizzare un minimo di 0,5 g di modello di riparazione.
- Mescolare 400 l di resospensione parassita, DNA, e 5 . Portare il volume totale a 500 o L con tampone di citomix, se necessario.
- Trasferire la miscela di parassiti e DNA in una cuvette di elettroporazione (larghezza 4 mm di gap) ed eseguire l'elettroporazione (2 kV tensione, resistenza 50 ) utilizzando un apparato di elettroporazione.
- Trasferire i parassiti elettroporati in un flacone T25 contenente HFF confluenti nel mezzo D10 fresco. Applicare antibiotico appropriato per la selezione di farmaci dopo 24 h.
- Mantenere la pressione selettiva del farmaco fino a quando la crescita dei parassiti transgenici è stabile.
- Purifica il DNA genomico dalla popolazione knockout e verifica l'integrazione della cassetta di resistenza alla pirimethamina nel locus TgCPL da PCR. Dopo la verifica, passare alla sezione 3.4. In caso contrario, eseguire un altro giro di trasfezione parassita e selezione di farmaci. L'incapacità di rilevare la corretta integrazione della cassetta di resistenza ai farmaci di solito suggerisce che il gene bersaglio è essenziale o che il locus genico non è accessibile.
- Clonazione di parassiti da urlo
- Semina due 96 microplacche ben con cellule HFF e incuba a 37 e 5% di CO2 per 1 settimana prima di clonare i parassiti.
- Passare 0,3-0,4 mL della popolazione di parassiti transgenici in una fiaschetta T25 contenente HFF confluenti e farli crescere per 2 giorni. Considerare il passaggio di più parassiti se il mutante mostra difetti di crescita.
NOTA: Per ottenere la migliore resa e vitalità, le cellule ospiti sono fortemente infettate dai parassiti e la maggior parte dei parassiti sono mantenuti nello stadio intracellulare. - Siringa infettato cellule ospiti e filtranti parassiti appena lised come menzionato nel passo 1.3. Risospendere i parassiti nel mezzo D10 e farli scendere a 1.000 x g per 10 min a RT.
- Risospendere i parassiti pelleta in 10 mL di D10 medio.
- Contare i parassiti usando un emocitometro per determinare la concentrazione di parassiti.
- Condurre una diluizione in due fasi per portare la concentrazione a 10 parassiti/mL nel mezzo D10 integrato con l'antibiotico appropriato. Di solito, la resospensione iniziale del parassita viene diluita di 1.000 volte, seguita da una seconda diluizione a 10 parassiti/mL.
- Aspirati supporti da 96 microplacche contenenti HFF confluenti e inoculati 150 -L di parassiti diluiti in ogni pozzo.
- Incubare piastre a 37 gradi centigradi con 5% di CO2 per 7 giorni senza disturbi per consentire la formazione di placca. Il periodo di incubazione può essere più lungo se i parassiti transgenici presentano difetti di crescita.
- Schermare le piastre utilizzando un microscopio a contrasto di fase e contrassegnare solo i pozze contenenti una singola placca.
- Eseguire colonia PCR per identificare i cloni corretti.
- Utilizzare punte pipette per raschiare il fondo di ogni pozzo per sollevare monostrati HFF infetti.
- Pipet 75 -L della sospensione cellulare da ciascuno marcato bene in tubi di microcentrifuga da 1,5 mL.
- I tubi di centrifuga per 10 min alla massima velocità a RT. Aspirano con attenzione il superatante e risospendono nuovamente il pellet in 10,25 l di buffer di lisi contenente buffer di diluizione e additivo per il rilascio del DNA fornito nel kit (Tabella dei Materiali).
- Incubare i campioni per 4 min a RT, quindi 2 min a 98 gradi centigradi. Successivamente, i campioni possono essere utilizzati per la PCR o conservati a -20 gradi centigradi fino all'uso. Tre serie di reazioni PCR sono utilizzate per testare l'integrazione della cassetta di resistenza ai farmaci e la perdita del gene di interesse (Figura 4A). Fare riferimento alla tabella 5 per l'impostazione della reazione PCR e alla tabella 6 per le impostazioni del termociclore.
- Identificare i cloni corretti e trasferire quattro cloni in flaconi T25 contenenti HFF confluenti.
- Dopo che singoli cloni lisizzano le cellule ospiti, purifica il DNA genomico per un'ulteriore verifica PCR.
- Se è disponibile un anticorpo che riconosce la proteina di interesse, seguire una procedura di immunobloment standard per verificare la perdita della proteina bersaglio nei correggere knockout Toxoplasma. Immagini rappresentative per lo screening di un mutante TgCPL-deletion sono mostrate nella figura 4B,C.
Risultati
La figura 1 rappresenta un esempio di una curva di crescita per la deformazioneRH-ku80::NLuc e il calcolo derivato per il tempo di raddoppio. Generalmente, il saggio viene eseguito in tre repliche tecniche per ciascuna delle tre repliche biologiche per tenere conto delle variazioni delle letture di attività luciferasi. Al fine di calcolare il cambiamento di piega normalizzata della crescita del parassita, ogni lettura a 24-96 h post-infezio...
Discussione
Questo protocollo descrive un protocollo basato su luciferasi per valutare la crescita intracellulare del Toxoplasma e valutare l'efficacia dell'inibizione dei composti chimici contro la crescita dei parassiti. Rispetto alle strategie esistenti per misurare la crescita intracellulare del Toxoplasma, questo metodo presenta un'elevata sensibilità e specificità. Durante il monitoraggio della crescita del parassita, si raccomanda un saggio fittizio in una micropiastra chiara 96 bene per confermare che il ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare i dottori Sibley e Carruthers per aver condiviso pSAG1-Cas9-sgRNA-TgUPRT plasmid e anticorpi anti-TgCPL e TgActin. Questo lavoro è stato sostenuto dal fondo Clemson Startup (a . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . I finanziatori non hanno avuto alcun ruolo nella progettazione dello studio, nella raccolta e nell'analisi dei dati, nella decisione di pubblicazione o nella preparazione del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| Agarose gel extraction kit | New England BioLabs | T1020L | |
| BamHI | New England BioLabs | R0316S | |
| Biotek Synergy H1 Hybrid Multi-Mode Microplate Reader | BioTek Instuments | ||
| BTX Gemini Twin Waveform Electroporation System | Harvard Apparatus | ||
| Chemically competent E. coli cells | New England BioLabs | C29871 | |
| CloneAmp HiFi PCR premix | Takara Bio | 639298 | |
| Coelenterazine h | Prolume | 301-10 hCTZ | |
| EcoRV | New England BioLabs | R3195S | |
| Phire Tissue Direct PCR Master Mix | Thermo Scientific | F170L | |
| Plasmid miniprep kit | Zymo Research | D4054 | |
| Q5 Site-Directed Mutagenesis kit | New England BioLabs | E0554S | |
| Software | |||
| Geneious software for sgRNA design (version: R11) | |||
| GraphPad Prism software (8th version) | |||
| SnapGene for molecular cloning (version: 4.2.11) |
Riferimenti
- Blader, I. J., Coleman, B. I., Chen, C. T., Gubbels, M. J. Lytic Cycle of Toxoplasma gondii: 15 Years Later. Annual Review of Microbiology. 69 (1), 1-23 (2014).
- Jones, J. L., Kruszon-Moran, D., Rivera, H., Price, C., Wilkins, P. P. Toxoplasma gondii Seroprevalence in the United States 2009-2010 and Comparison with the Past Two Decades. The American Journal of Tropical Medicine and Hygiene. 90 (6), (2014).
- Kieffer, F., Wallon, M. Congenital toxoplasmosis. Handbook of Clinical Neurology. 112, 1099-1101 (2013).
- Hoffmann, S., Batz, M. B., Morris, G. J. Annual cost of illness and quality-adjusted life year losses in the United States due to 14 foodborne pathogens. Journal of Food Protection. 75 (7), 1292-1302 (2012).
- Dubey, J. Toxoplasmosis. Journal of the American Veterinary Medical Association. 205 (11), 1593-1598 (1994).
- Lindsay, D., Dubey, J. Toxoplasma gondii: the changing paradigm of congenital toxoplasmosis. Parasitology. 138 (14), 1-3 (2011).
- Deng, Y., Wu, T., Zhai, S., Li, C. Recent progress on anti-Toxoplasma drugs discovery: Design, synthesis and screening. European Journal of Medicinal Chemistry. 183, 111711 (2019).
- Butler, N. J., Furtado, J. M., Winthrop, K. L., Smith, J. R. Ocular toxoplasmosis II: clinical features, pathology and management. Clinical & Experimental Ophthalmology. 41 (1), 95-108 (2013).
- Pfefferko, E., Pfefferko, L. C. Specific Labeling of Intracellular Toxoplasma gondii with Uracil. Journal of Eukaryotic Microbiology. 24 (3), 449-453 (1977).
- Merli, A., Canessa, A., Melioli, G. Enzyme immunoassay for evaluation of Toxoplasma gondii growth in tissue culture. Journal of Clinical Microbiology. 21 (1), 88-91 (1985).
- Derouin, F., Chastang, C. Enzyme immunoassay to assess effect of antimicrobial agents on Toxoplasma gondii in tissue culture. Antimicrobial Agents and Chemotherapy. 32 (3), 303-307 (1988).
- McFadden, D., Seeber, F., Boothroyd, J. Use of Toxoplasma gondii expressing beta-galactosidase for colorimetric assessment of drug activity in vitro. Antimicrobial Agents and Chemotherapy. 41 (9), 1849-1853 (1997).
- Gubbels, M. J., Li, C., Striepen, B. High-Throughput Growth Assay for Toxoplasma gondii Using Yellow Fluorescent Protein. Antimicrobial Agents and Chemotherapy. 47 (1), 309-316 (2003).
- Touquet, B., et al. High-content imaging assay to evaluate Toxoplasma gondii infection and proliferation: A multiparametric assay to screen new compounds. PLoS ONE. 13 (8), e0201678 (2018).
- Thornton, L. B., et al. An ortholog of Plasmodium falciparum chloroquine resistance transporter (PfCRT) plays a key role in maintaining the integrity of the endolysosomal system in Toxoplasma gondii to facilitate host invasion. PLOS Pathogens. 15 (6), e1007775 (2019).
- Larson, E. T., et al. Toxoplasma gondii cathepsin L is the primary target of the invasion-inhibitory compound morpholinurea-leucyl-homophenyl-vinyl sulfone phenyl. The Journal of Biological Chemistry. 284 (39), 26839-26850 (2009).
- Dou, Z., McGovern, O. L., Cristina, M., Carruthers, V. B. Toxoplasma gondii Ingests and Digests Host Cytosolic Proteins. mBio. 5 (4), e01188-14 (2014).
- Cristina, M., et al. Toxoplasma depends on lysosomal consumption of autophagosomes for persistent infection. Nature Microbiology. 2, 17096 (2017).
- Parussini, F., Coppens, I., Shah, P. P., Diamond, S. L., Carruthers, V. B. Cathepsin L occupies a vacuolar compartment and is a protein maturase within the endo/exocytic system of Toxoplasma gondii. Molecular Microbiology. 76 (6), 1340-1357 (2010).
- Huynh, M. H., Carruthers, V. B. Tagging of endogenous genes in a Toxoplasma gondii strain lacking Ku80. Eukaryotic cell. 8 (4), 530-539 (2009).
- Fox, B. A., Ristuccia, J. G., Gigley, J. P., Bzik, D. J. Efficient gene replacements in Toxoplasma gondii strains deficient for nonhomologous end joining. Eukaryotic Cell. 8 (4), 520-529 (2009).
- Sidik, S. M., Hackett, C. G., Tran, F., Westwood, N. J., Lourido, S. Efficient Genome Engineering of Toxoplasma gondii Using CRISPR/Cas9. PLoS ONE. 9 (6), e100450 (2014).
- Shen, B., Brown, K. M., Lee, T. D., Sibley, D. L. Efficient Gene Disruption in Diverse Strains of Toxoplasma gondii Using CRISPR/CAS9. mBio. 5 (3), e01114-14 (2014).
- Radke, J. R., et al. Defining the cell cycle for the tachyzoite stage of Toxoplasma gondii. Molecular and Biochemical Parasitology. 115 (2), 165-175 (2001).
- Ran, A. F., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B., Valen, E. CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Research. 44 (W1), W272-W276 (2016).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 2812 (2014).
- Peng, D., Tarleton, R. EuPaGDT: a web tool tailored to design CRISPR guide RNAs for eukaryotic pathogens. Microbial Genomics. 1 (4), e000033 (2015).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature Biotechnology. 32 (12), 1262-1267 (2014).
- Sidik, S. M., et al. A Genome-wide CRISPR Screen in Toxoplasma Identifies Essential Apicomplexan Genes. Cell. 166 (6), 1423-1435 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon