È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging live ad alta produttività di microcolonie per misurare l'eterogeneità nella crescita e nell'espressione genica
In questo articolo
Riepilogo
I fenotipi di crescita del lievito sono misurati con precisione attraverso l'imaging time-lapse altamente parallelo di cellule immobilizzate che crescono in microcolonie. Allo stesso tempo, la tolleranza allo stress, l'espressione proteica e la localizzazione delle proteine possono essere monitorate, generando set di dati integrati per studiare come le differenze ambientali e genetiche, così come l'eterogeneità gene-espressione tra le cellule isogeniche, modulano la crescita.
Abstract
Misurazioni precise dell'eterogeneità tra e all'interno dello sforzo nei tassi di crescita microbica sono essenziali per comprendere gli input genetici e ambientali nella tolleranza allo stress, nella patogenicità e in altri componenti chiave dell'idoneità. Questo manoscritto descrive un saggio basato sul microscopio che tiene traccia di circa 105 microcoloni Saccharomyces cerevisiae per esperimento. Dopo l'imaging automatizzato time-lapse del lievito immobilizzato in una piastra multiwell, i tassi di crescita dei microcoloni vengono facilmente analizzati con un software di analisi delle immagini personalizzato. Per ogni microcolonia, è anche possibile monitorare l'espressione e la localizzazione di proteine fluorescenti e la sopravvivenza dello stress acuto. Questo saggio consente una stima precisa dei tassi di crescita medi dei ceppi, nonché una misurazione completa dell'eterogeneità nella crescita, nell'espressione genica e nella tolleranza allo stress all'interno delle popolazioni clonali.
Introduzione
I fenotipi di crescita contribuiscono in modo critico alla forma fisica del lievito. La selezione naturale può distinguere in modo efficiente tra lignaggi con tassi di crescita diversi dall'inverso della dimensione effettiva della popolazione, che può superare i 108 individui1. Inoltre, la variabilità dei tassi di crescita tra gli individui all'interno di una popolazione è un parametro evolutivamente rilevante, in quanto può servire come base per strategie di sopravvivenza come la copertura dellepuntate 2,3,4,5,6. Pertanto, i test che consentono misurazioni altamente accurate dei fenotipi di crescita e delle loro distribuzioni sono fondamentali per lo studio dei microrganismi. Il saggio di crescita del microcolonio qui descritto può generare misurazioni individuali del tasso di crescita per ~10 5 microcolonie per esperimento. Questo saggio fornisce quindi un potente protocollo per studiare la genetica evolutiva e la genomica del lievito. Si presta particolarmente bene a testare come la variabilità all'interno delle popolazioni di singole cellule geneticamente identiche viene generata, mantenuta e contribuisce alla forma fisicadella popolazione 7,8,9,10.
Il metodo qui descritto (Figura 1) utilizza immagini a campo luminoso a basso ingrandimento catturate periodicamente di cellule che crescono in mezzi liquidi su una piastra inferiore in vetro da 96 o 384 pozze di vetro per tenere traccia della crescita in microcolonie. Le cellule aderiscono alla lettalina concanavalina A, che rivestise il fondo della piastra del microscopio e forma colonie bidimensionali. Poiché le microcolonie crescono in un monostrato, l'area della microcolonia è altamente correlata con la cella numero7. Pertanto, è possibile generare stime accurate del tasso di crescita del microcolony e del tempo di ritardo con un software di analisi delle immagini personalizzato che tiene traccia del tasso di cambiamento dell'area di ogni microcolony. Inoltre, la configurazione sperimentale può monitorare l'abbondanza e persino le localizzazioni subcellulari di proteine etichettate fluorescentmente espresse in queste microcolonie. L'elaborazione a valle dei dati di questo test di crescita della microcolonia può essere ottenuta mediante analisi personalizzate o con il software di analisi delle immagini esistente, come Processing Images Easily (PIE)11, un algoritmo per il riconoscimento robusto dell'area della colonia e l'analisi della crescita ad alta produttività da immagini a basso ingrandimento e luminose, disponibile tramite GitHub12.
Poiché le stime del tasso di crescita derivate dal saggio di crescita della microcolonia sono generate da un gran numero di misurazioni a colonia singola, sono estremamente accurate, con errori standard di diversi ordini di grandezza più piccoli delle stime stesse per un esperimento di dimensioni ragionevoli. Pertanto, il potere del saggio di rilevare differenze di tasso di crescita tra diversi genotipi, trattamenti o condizioni ambientali è elevato. Il formato multiwell-plate consente di confrontare numerose combinazioni di ambiente e genotipo diverse in un unico esperimento. Se i ceppi esprimono in modo costitutivo marcatori fluorescenti diversi, possono essere mescolati nello stesso pozzo e distinti dalla successiva analisi dell'immagine, che potrebbe aumentare ulteriormente la potenza consentendo la normalizzazione dei dati pozzo per pozzo.
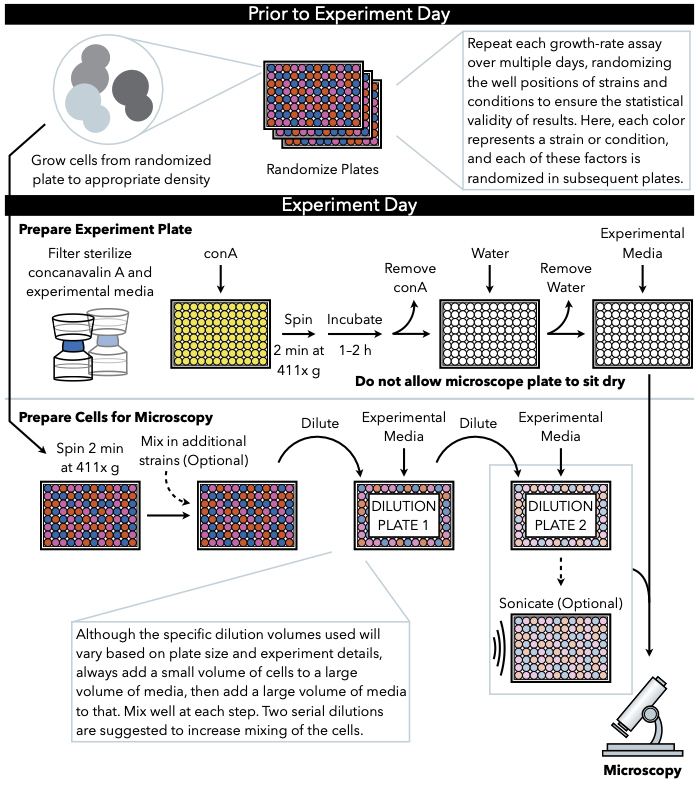
Figura 1: Rappresentazione schematica del protocollo. Questo protocollo segue due fasi principali, che sono la preparazione della piastra sperimentale e la preparazione delle cellule all'immagine. La randomizzazione delle piastre e la crescita delle cellule devono essere condotte prima e prima del giorno dell'esperimento. La miscelazione ripetuta delle cellule in ogni fase durante la diluizione è imperativa nei passaggi fino alla placcatura, e quindi si consiglia di preparare prima la piastra sperimentale in modo che sia pronta per la placcatura immediatamente dopo il completamento della diluizione cellulare. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Preparazione di piastre randomizzate (prima del giorno dell'esperimento)
- Pianificare i ceppi e le condizioni da testare con il test di crescita. A questo punto, assegnare casualmente ceppi e condizioni a qualsiasi pozzo.
NOTA: Quando si considera la configurazione della piastra, è consigliabile includere più di una replica per sforzo e condizione di crescita su una singola piastra per tenere conto del rumore ben correlato nelle misurazioni. Per ulteriori dettagli, vedere Discussione. - Randomizzare computazalmente la posizione di ogni deformazione e condizione ambientale per le repliche delle piastre che verranno eseguite in giorni diversi.
- Far crescere tutte le cellule che verranno utilizzate nell'esperimento fino alla saturazione nel mezzo estratto di lievito-peptone-destrosio (YEPD; 2% glucosio) in uno shaker a 30 °C (o qualsiasi altra temperatura appropriata).
- Creare le piastre di serie randomizzate manualmente o con un robot di movimentazione dei liquidi. Aggiungere 10 μL delle cellule sature designate a ciascun pozzo di una piastra sterile di coltura del tessuto a U.bottom. Se più ceppi verranno testati in un unico pozzo, non combinarli a questo punto; questa combinazione sarà fatta poco prima delle diluizioni cellulari il giorno dell'esperimento per garantire che tutti i ceppi siano alle concentrazioni corrette quando placcati come cellule fondatrici della microcolonia.
- Aggiungere 10 μL di glicerolo al 30% ad ogni pozzo di ogni piastra. Pipetta su e giù in modo che le cellule e il glicerolo diventino ben mescolati.
- Sigillare ogni piastra con un coperchio di lamina e congelare immediatamente a -70 °C fino a quando non è pronto per l'uso.
NOTA: È importante creare tutte le piastre randomizzate nello stesso giorno e congelarle, in modo che le condizioni di pre-crescita delle cellule in ogni piastra siano state identiche e non generino variazioni tecniche nel saggio del tasso di crescita.
2. Pre-crescita del lievito
NOTA: In genere, questo inizia prima della giornata dell'esperimento ed è altamente dipendente dalla domanda sperimentale. Per informazioni dettagliate, vedere Discussione.
- Rimuovere una piastra di serie (lievito da 10 μL, 10 μL di glicerolo per pozzo) dal congelatore a -70 °C e aggiungere 180 μL del supporto da utilizzare per l'esperimento. Se l'esperimento verrà condotto utilizzando mezzi limitanti per i nutrienti, non pre-coltivare il lievito fino alla saturazione nel mezzo che limita i nutrienti in quanto può verificarsi la sporulazione del lievito. Invece, pre-crescere in media non limitanti.
- Coltivare il lievito tremando a 30 °C. Valutare se eseguire il saggio a partire dalle cellule in fase di log o in fase stazionaria per determinare se sarà necessario diluire le cellule più volte prima dell'esperimento. Se si prevede che i ceppi o le condizioni del lievito nel saggio abbiano tassi di crescita significativamente diversi, sarà necessario un periodo di pre-crescita di due giorni affinché tutte le diverse condizioni raggiungano la fase stazionaria.
3. Configurazione microscopio
- Preparazione della piastra del microscopio
- Assicurarsi che l'incubatore di microscopi sia su e riscaldare la camera del microscopio alla temperatura di crescita desiderata per le condizioni sperimentali. Per gli esperimenti standard che utilizzano cellule Saccharomyces cerevisiae, l'incubatore dovrebbe riscaldare la camera del microscopio a 30 °C per garantire che le condizioni di crescita per le cellule siano corrette durante il test del tasso di crescita.
- Sanificare il workbench, le pipette e altri utensili con il 70% di etanolo. Recupera una piastra al microscopio e posizionala sulla panca sopra una salvietta priva di pelucchi e statici.
NOTA: Non toccare mai la parte inferiore della piastra del microscopio, anche con i guanti ad acqua, e impostare sempre la piastra del microscopio sopra una salvietta priva di pelucchi e statici ogni volta che tocca qualsiasi superficie. In questo modo si evita che sbavature o graffi ostacolino le misurazioni del tasso di crescita una volta che l'esperimento è stato immaginato. - Scongelare 5 ml di 5x concanavalina Una soluzione, diluire a 1x con acqua e filtrare sterilizzare attraverso una siringa dotata di un filtro da 0,2 μm.
- Filtrare sterilizzare tutti gli altri liquidi che verranno utilizzati nel saggio con un filtro da 0,2 μm, compresi i mezzi sperimentali, per rimuovere eventuali cristalli o detriti che potrebbero essere stati materializzati nelle soluzioni. La presenza di cristalli ridurrebbe la qualità delle immagini al microscopio.
- Pipetta 200 μL di concanavalina Una soluzione in ogni pozzo della piastra del microscopio.
- Centrifugare la piastra per 2 min a 411 x gravità (g) con una salvietta priva di pelucchi e statici sotto la piastra, per garantire che la soluzione di concanavalina A copra uniformemente il fondo di ogni pozzo e che non ci siano bolle d'aria.
- Coprire la piastra con il coperchio e lasciarla riposare per 1-2 ore. Il tempo preciso in cui si trova la piastra è flessibile, ma è importante essere coerenti tra le diverse corse dell'esperimento.
- Rimuovere tutta la soluzione di concanavalina Una dalla piastra per aspirazione o scaricarla con forza nel lavandino o in un recipiente. Fare attenzione a non toccare la parte di vetro del piatto. È accettabile se alcune gocce di concanavalina Una soluzione rimangono nei pozzi.
- Lavare i pozzi della piastra del microscopio aggiungendo 400 μL di acqua sterile. Rimuovere l'acqua come fatto con la concanavalina A nel passaggio precedente. Non lasciare che il piatto si sieda asciutto.
- Aggiungere immediatamente 185 μL di mezzi di crescita sperimentali nella piastra. 15 μL di cellule correttamente diluite verranno aggiunti a questa piastra.
- Diluizione cellulare del lievito
NOTA: I passaggi seguenti descrivono una diluizione del lievito da una coltura satura (circa10 8 cellule/mL) 400 volte per raggiungere una concentrazione di 250.000 cellule/mL, di cui 15 μL saranno diluiti in 400 μL nella piastra inferiore del vetro, dando un numero finale di circa 4000 cellule per pozzo in una piastra da 96 porcile. Se si utilizza una piastra da 384 pozza, il numero finale di cellule per pozzo dovrebbe essere di circa 700 e le diluizioni devono essere regolate di conseguenza. Questo rapporto dovrebbe essere regolato per le cellule raccolte in fase di log, che crescono in mezzi pre-crescita più ricchi o più poveri o da ceppi diversi. La densità finale delle cellule per pozzo dovrebbe essere ridotta quando si esegue il saggio del tasso di crescita per periodi di tempo superiori a 10 h.- Impostare due piastre di coltura da 96 porri per diluizioni seriali: etichettare come la piastra 1 e 2 e aggiungere 90 μL di mezzi di crescita sperimentali (cioè il supporto in cui il lievito crescerà al microscopio) a ogni piastra di diluizione seriale.
NOTA: Indipendentemente dalla diluizione finale utilizzata, si raccomandano almeno due diluizioni seriali di cellule, in ognuna delle quali un piccolo volume di lievito viene pipettato in un volume maggiore di mezzi sperimentali e quindi un grande volume di mezzi sperimentali viene miscelato vigorosamente con una pipetta (come nei passaggi 3.2.5 e 3.2.6). - Recuperare la piastra delle cellule dalla pre-crescita e centrifugare la piastra per 2 minuti a 411 x g.
NOTA: È molto importante non contaminare diversi pozzi nella piastra. Lo scopo di questa fase di centrifugazione prima di rimuovere il rivestimento del foglio dalle piastre è quello di garantire che le goccioline riempite di lievito da un pozzo non volino fuori dal foglio e finiscano in altri pozzi. Fare attenzione a non inclinare o agitare mai le piastre per evitare che il lievito venga a contatto con il rivestimento del foglio dopo la centrifugazione. - Sbucciare con cura il foglio e rimescolare le cellule mediante pipettazione vigorosa con una pipetta impostata su circa la metà del volume totale nella piastra mentre si sposta la pipetta intorno al pozzo per mescolare. Verificare che tutte le celle siano state rimorsi dal fondo dei pozzi.
- Se verranno utilizzati più ceppi all'interno di singoli pozzi, i ceppi devono essere mescolati in questo momento al rapporto necessario per l'esperimento. Se per generare misurazioni del tasso di crescita verrà utilizzato un ceppo di riferimento, il rapporto tra riferimento e deformazione di prova dovrebbe essere 1:1.
- Pipetta 10 μL di lievito da mezzi di crescita in piastra di diluizione 1. Aggiungere 100 μL di mezzi sperimentali di crescita ad ogni pozzo ad un volume finale di 200 μL per pozzo. Pipetta su e giù vigorosamente.
- Pipetta 10 μL di lievito dalla piastra 1 nel piatto 2. Aggiungere 100 μL di mezzi di crescita sperimentali ad ogni pozzo e pipettare su e giù vigorosamente per mescolare.
NOTA: Questi passaggi di diluizione sono fondamentali per aiutare a separare i cluster di lievito che sono bloccati insieme alla fine della fase di pre-crescita e garantire che un numero approssimativamente uguale di cellule di lievito finisca in ogni pozzo. Avere un numero costante di lieviti in ogni pozzo aiuta a rimuovere il rumore sperimentale e le distorsioni nelle misurazioni del tasso di crescita (vedi Risultati rappresentativi).
- Impostare due piastre di coltura da 96 porri per diluizioni seriali: etichettare come la piastra 1 e 2 e aggiungere 90 μL di mezzi di crescita sperimentali (cioè il supporto in cui il lievito crescerà al microscopio) a ogni piastra di diluizione seriale.
- Sonicazione
NOTA: La sonicazione è facoltativae deve essere eseguita solo per i ceppi di lievito che hanno un'alta propensione ad aderire l'uno all'altro (ad esempio, alcuni ceppi selvatici). Per i ceppi di laboratorio, la sonicazione non è generalmente necessaria e può essere saltata procedendo al passaggio 3.4.- Sanificare una testa del sonicatore a 96 pin con il 70% di etanolo posizionandolo in una piastra da 96 pozzetta riempita con etanolo al 70% e asciutta con una salvietta priva di pelucchi e statici.
- Impostare un programma di sonicazione sufficientemente forte da spezzare le cellule di lievito flocculate, ma non uccide le cellule o causa elevate risposte allo stress. Alcuni test potrebbero essere necessari per identificare il miglior programma di sonicazione per un dato esperimento. Il programma di sonicazione utilizzato in questo esperimento è: ampiezza = 10, tempo di processo = 10 s, pulse-on = 1 s, pulse-off = 1 s. Questo programma esatto probabilmente non è applicabile a tutti i sonicatori, quindi il test è suggerito prima del giorno dell'esperimento.
- Mescolare il lievito in lastra di diluizione seriale 2 ancora una volta tubazione su e giù vigorosamente cinque volte.
- Posizionare la piastra di diluizione 2 sulla piattaforma e fissarla con i perni del sonicatore nella sospensione cellulare ma non toccare il fondo della piastra. Eseguire il programma di sonicazione utilizzando un'adeguata protezione dell'orecchio.
- Dopo l'esecuzione del programma, pulire la testa del sonicatore con il 70% di etanolo e quindi con acqua, quindi procedere immediatamente alla preparazione della piastra del microscopio in modo che le cellule non si clulino di nuovo.
- Preparare la piastra per microscopio:
- Pipetta 15 μL di lievito dalla piastra di diluizione seriale 2 nella piastra del microscopio a un volume di 200 μL. Aggiungere 200 μL di mezzi di crescita sperimentali ad ogni pozzo ad un volume finale di 400 μL per pozzo e pipettare su e giù vigorosamente per mescolare.
- Coprire la piastra con una membrana traspirante. È importante sigillare bene la piastra con questa membrana, ad esempio utilizzando un rullo di gomma.
- Per aderire le cellule di lievito alla concanavalina A sulla superficie del vetro, centrifugare la piastra con una salvietta priva di pelucchi e statici sotto di essa per 2 minuti a 411 x g.
- Al microscopio, pulire la parte superiore e inferiore della piastra con una salvietta priva di pelucchi e statici e soffiare aria compressa sulla piastra per sbarazzarsi dei detriti.
- Posizionare la piastra sul microscopio, assicurandosi che sia livellata e che il pozzo A1 si trova nell'angolo in alto a sinistra.
4. Misurazioni del tasso di crescita della microscopia time-lapse
NOTA: Durante la microscopia time-lapse vengono controllate al computer le seguenti caratteristiche: posizione x, y e z, persiane e filtri a fluorescenza. Un sistema di messa a fuoco automatica basato su hardware è ottimale per prevenire la deriva del piano focale durante l'imaging time-lapse. In alternativa, è possibile utilizzare un ciclo di messa a fuoco automatica basato su software. Per mantenere l'umidità nella camera del microscopio, si consiglia di tenere un becher con acqua purificata nella camera per tutta la durata dell'esperimento.
- Creare un elenco di posizioni (x,y) da immaginere, in modo che ogni pozzo della piastra del microscopio sia completamente visualizzato. Evitare di sovrapporre le immagini in modo che nessuna cella sia analizzata più volte.
- Immagine in campo luminoso con illuminazione diascopica (DIA) ad un ingrandimento di 15x. Impostare l'esposizione su ~5 ms.
- Ingrandire l'immagine digitalmente in modo che le celle siano chiaramente visibili. Utilizzare le manopole di messa a fuoco per identificare la messa a fuoco ideale per l'esperimento nei quattro pozzi all'angolo della piastra e in un pozzo al centro della piastra. Mettere a fuoco in modo da ottenere il massimo contrasto delle celle.
- Impostate la posizione z (o posizione di messa a fuoco automatica) affinché l'esperimento sia una media delle posizioni z/autofocus identificate per ciascuno di questi pozzi. Se la piastra del microscopio è ben realizzata e il fondo in vetro non ha difetti, le posizioni di messa a fuoco ideali dovrebbero essere simili per ogni pozzo.
NOTA: Quando si analizzano le immagini con la pipeline di analisi delle immagini PIE (Processing Images Easily)11,12, è utile che le celle siano leggermente fuori fuoco al microscopio in modo che ci sia un bordo scuro all'esterno della cella e un interno di colore chiaro, che aiuta a migliorare accurate stime delle colonie e delle dimensioni. - Se si utilizzano ceppi fluorescenti, identificare i canali e le esposizioni con cui l'immagine, assicurandosi che nessun pixel sia sovraesposto. Quando si imposta il tempo di esposizione per i canali fluorescenti, disattivare la modalità "acquisizione dal vivo" al microscopio per evitare di esporre le cellule all'eccitazione della fluorescenza per lunghi periodi di tempo, in quanto ciò può sia fotolicenza delle cellule che causare stress.
- Impostare l'acquisizione della sequenza temporale per acquisire immagini all'intervallo di tempo desiderato per il periodo di tempo desiderato.
- Esegui l'esperimento.
Risultati
La novità di questo protocollo è che il tasso di crescita può essere calcolato per le singole cellule all'interno di una popolazione monitorando la loro crescita in microcolonie attraverso l'imaging time-lapse (Figura 2A). Poiché le microcolonie crescono per molte ore in modo planare a causa della presenza di concanavalina A, le loro aree possono essere tracciate durante l'esperimento e una vestibilità lineare al cambiamento nel log naturale dell'area nel tempo può essere utilizzata pe...
Discussione
Il protocollo qui descritto è un saggio versatile che consente di monitorare simultaneamente la crescita cellulare e l'espressione genica a livello delle singole microcolonie. La combinazione di queste due modalità produce intuizioni biologiche uniche. Ad esempio, lavori precedenti hanno usato questo saggio per mostrare una correlazione negativa tra l'espressione del gene TSL1 e il tasso di crescita della microcolonia nelle cellule di tipo selvatico isogenico misurandoentrambi contemporaneame...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Naomi Ziv, Sasha Levy e Shuang Li per i loro contributi allo sviluppo di questo protocollo, David Gresham per le attrezzature condivise e Marissa Knoll per l'aiuto nella produzione video. Questo lavoro è stato sostenuto dalla sovvenzione dei National Institutes of Health R35GM118170.
Materiali
| Name | Company | Catalog Number | Comments |
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are recommended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
Riferimenti
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization--a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon