È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un modello proinflammatorio di coltura degenerativa degli organi per simulare la malattia del disco intervertebrale in fase iniziale.
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo presenta un nuovo modello sperimentale di coltura proinfiammatoria e degenerativa di organi bovini per simulare la degenerazione del disco intervertebrale in fase iniziale.
Abstract
La degenerazione del disco intervertebrale sintomatico (IVD) (IDD) è un importante fardello socioeconomico ed è caratterizzata da infiammazione e degradazione dei tessuti. A causa della mancanza di terapie causali, c'è un'urgente necessità di modelli sperimentali innovativi di coltura degli organi per studiare i meccanismi coinvolti nella progressione della malattia, trovare obiettivi terapeutici e ridurre la necessità di modelli animali. Qui presentiamo un nuovo protocollo di modello tridimensionale di coltura di organi che imita il microambiente proinfiammatorio e catabolico, presente durante l'IDD.
Inizialmente, gli IVD caudali bovini venivano sezionati, puliti e coltivati nel mezzo di coltura tissutale. Il carico fisiologico dinamico o patologico è stato applicato in un bioreattore su misura per 2 ore al giorno. Gli IVD sono stati assegnati a un gruppo di controllo (mezzo di glucosio elevato, carico fisiologico, iniezione salina tamponata da fosfati) e a un gruppo patologico (mezzo a basso glucosio, carico patologico, iniezione di fattore di necrosi tumorale-alfa) per quattro giorni. È stata eseguita l'analisi dell'espressione genica da cellule pulpose del nucleo raccolte degli IVD e il saggio immunoassorbente legato all'enzima del supporto di coltura degli organi condizionati.
I nostri dati hanno rivelato una maggiore espressione di marcatori infiammatori e una riduzione dell'altezza del disco dopo il caricamento nel gruppo patologico rispetto al gruppo di controllo. Questo protocollo è affidabile per simulare l'infiammazione e la degenerazione dell'IVD e può essere ulteriormente ampliato per ampliare il suo ambito di applicazione.
Introduzione
Lo loto antisoccito (LBP) può colpire individui di tutte le età ed è una delle principali cause di disabilità intutto il mondo 1,2,3. Il costo totale associato a LBP supera $ 100 miliardi all'anno4,5. La degenerazione del disco intervertebrale sintomatico (IVD), una condizione caratterizzata da infiammazione e degradazione dei tessuti, è una delle principali cause di LBP6,7. In particolare, l'IDD è caratterizzato da una rottura in graduale evoluzione della matrice extracellulare (ECM) dell'IVD, indotta e innescata da molteplici fattori che portano a una patologia accelerata, disturbi neurologici ed eventualmente disabilità. Inoltre, l'IDD è associato al rilascio di citochine proinfiammatorie, biomeccanica della colonna vertebrale alterata, angiogenesi e crescita nervosa, che aumenta la sensazione di dolore, causando del tutto LBP cronico (discopatia attiva)6,8. Ad oggi, le opzioni di trattamento includono discectomia e successiva fusione delle vertebre adiacenti, impianto di una protesi IVD o approcci non chirurgici, come farmaci antinfiammatori non steroidei, oppioidi e rilassanti muscolari per pazienti con IDD9. Entrambe le attuali opzioni terapeutiche standard, chirurgiche e non chirurgiche, sono solo parzialmente efficaci e non riescono ad affrontare il problema biologicosottostante 9,10. La malattia degenerativa del disco in fase iniziale è caratterizzata da una risposta iniziale del tessuto infiammatorio, in particolare un aumento dell'espressione fattore-alfa della necrosi tumorale (TNF-alfa)11. Questi primi cambiamenti del disco si verificano principalmente a livello cellulare senza interrompere l'architettura del disco e potrebbero in precedenza essere mimicked da carenza nutrizionale in condizioni pro-infiammatorie12. Pertanto, è fondamentale una simulazione precisa della situazione in vivo per indagare questi meccanismi di degenerazione e trovare obiettivi terapeutici adeguati. Inoltre, a queste simulazioni di proprietà molecolari, l'ambiente di carico meccanico dei dischi gioca un ruolo chiave nei cambiamenti patologici e fisiologici dell'IVD. Di conseguenza, combinare questi approcci ci farebbe fare un passo avanti per imitare il complesso microambiente dei D IVD in vivo. Attualmente non ci sono studi che considerino al meglio l'aspetto del carico dinamico insieme all'ambiente pro-infiammatorio e nutrizionale.
Sebbene i modelli animali di grandi dimensioni consentano di investigre potenziali interazioni in vivo pertinenti, sono costosi e richiede molto lavoro. Inoltre, poiché l'uso di modelli animali nella ricerca è stato a lungo oggetto di controversie, la riduzione del numero di animali necessari per rispondere a importanti domande di ricerca è di grande interesse. Infine, attualmente non esiste un modello animale ideale per imitare l'IDD nella ricerca IVD13,14. Pertanto, è necessario stabilire una sostituzione economica e affidabile, come un modello di coltura di organi per simulare l'IDD e i processi infiammatori e degenerativi associati. Recentemente, l'applicazione del presente protocollo sull'istituzione di un modello di coltura proinfiammatoria e degenerativa degli organi per simulare la malattia del disco intervertebrale in fase iniziale ci ha permesso di indagare l'effetto dei farmaci antinfiammatori nella coltura di organi IDD15.
Qui descriviamo come ottenere dischi intervertebrali bovini e induciamo lo stato di IDD in fase iniziale attraverso un microambiente catabolico e proinfiammatorio causato dall'iniezione intradiscale diretta di fattore alfa della necrosi tumorale (TNF-α) e dal carico degenerativo in un bioreattore in condizioni medie nutritive basse. La figura 1 illustra il modello sperimentale e mostra il bioreattore utilizzato per simulare le condizioni di carico degenerative e fisiologiche.
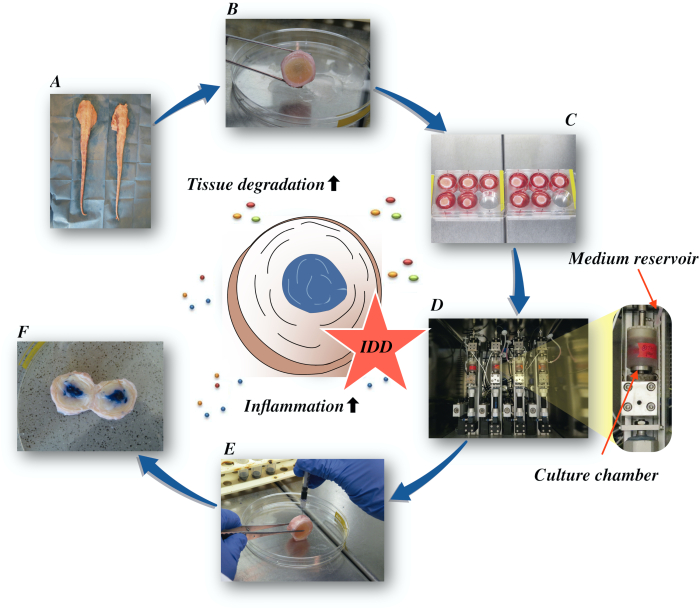
Figura 1: Illustrazione dell'impostazione sperimentale. A: coda bovina; B: dischi intervertebrali bovini sezionati; C: trasferimento del disco su una piastra di pozzo con mezzo di coltura; D: caricamento della simulazione in un bioreattore; E: tecnica di iniezione intradiscale; F: IVD dopo iniezione di pbs/tripano colorante blu per rivelare la distribuzione. IDD: degenerazione del disco intervertebrale. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
Gli esperimenti sono stati eseguiti utilizzando code bovine ottenute da mattatoi locali. I materiali biologici utilizzati nell'attuale studio sono prelevati dalla catena alimentare e non richiedono alcuna approvazione etica nel diritto svizzero ed europeo.
1. Dissezione del disco intervertebrale bovino
- Risciacquare accuratamente l'intera coda con acqua del rubinetto per rimuovere sporco e capelli sulla superficie.
NOTA: Con estremità distali intatte, è possibile utilizzare un massimo di 9 IVD (coccigeo 1-9) per coda per gli esperimenti a seconda della dimensione desiderata degli IVD. Considerando il diametro desiderato tra 15-20 mm, abbiamo usato 12 code bovine con 5 IVD per coda per gli esperimenti. - Immergere l'intera coda in una scatola contenente l'1% di soluzione di betadina per 10 minuti. Asciugare brevemente la coda con garza sterile e posizionarla su un drappo sterile.
NOTA: Durante la dissezione del disco, umidificare le code con la garza bagnata della soluzione di Ringer per prevenire la disidratazione. Conservare le code (o segmenti a sinistra) avvolte in garza bagnata fino al completamento dell'intera procedura di dissezione. - Utilizzare un bisturi (n. 20) per rimuovere il tessuto molle nel modo più completo possibile dalla colonna vertebrale caudale per facilitare l'identificazione degli IVD. Rimuovere i processi spinosi e trasversali delle vertebre con pinze di rimozione ossea.
NOTA: selezionare gli IVD con il diametro desiderato. Nello studio attuale sono stati utilizzati IVD con un diametro di 15-20 mm. - Tagliare trasversalmente con pinze ossee attraverso il centro di ogni corpo vertebrale per ottenere singoli segmenti di movimento. Metti i segmenti di movimento in una piastra di Petri con garza bagnata con la soluzione di Ringer.
- Individuare l'IVD e la vertebra palpando e spostando delicatamente i segmenti di movimento. Effettuare due tagli paralleli con la sega a banda nella piastra di crescita degli IVD, uno su ciascun lato dell'IVD. Identificare la posizione della piastra di crescita toccando e trovando il sito convesso della parte ossea della piastra finale (dura) adiacente al disco (morbida) con una distanza di sicurezza di circa 0,5-1 mm dall'IVD verso la vertebra. Assicurarsi che la lama della sega a nastro sia raffreddata con la soluzione di Ringer durante il taglio delle vertebre.
- Trasferire gli IVD in una piastra di Petri pulita con garza pulita bagnata con la soluzione di Ringer.
NOTA: La garza deve essere inumidita e non troppo bagnata per evitare il gonfiore degli IVD, - Utilizzare la lama bisturi per raschiare il corpo vertebrale (osso rosso / rosa), piastra di crescita (cartilagine bianca), lasciare intatta la piastra finale (giallo-rosa). Rendere le due superfici piatte e parallele per la procedura di caricamento. Trasferire gli IVD raschiati in una piastra di Petri fresca con garza bagnata con la soluzione di Ringer.
NOTA: Indossare un guanto a catena per proteggere la mano tenendo premuto l'IVD e raschiando. - Misurare l'altezza e il diametro del disco con una pinza. Pulire i coaguli di sangue nell'osso delle vertebre con la soluzione di Ringer utilizzando un sistema di lavanda a getto.
- Trasferire gli IVD su tubi di plastica da 50 ml, un IVD per tubo. Aggiungere 25 mL di salina tamponata da fosfati (PBS) + 10% penicillina/streptomicina (P/S) per IVD e lasciarla tremare per 15 minuti su uno shaker orbitale a temperatura ambiente.
- Aspirare il supernatante e aggiungere 10 mL di PBS + 1% P/S per IVD per 2 minuti per risciacquare gli IVD.
2. Cultura e carico IVD
- Trasferire dischi in camere IVD e aggiungere il mezzo di coltura IVD (Modified Eagle Medium (DMEM di Dulbecco, DMEM ad alto glucosio 4,5 g/L per il gruppo fisiologico e 2 g/L di DMEM a basso glucosio per il gruppo patologico) + 1% P/S + 2% siero fetale di vitello + 1% ITS (contiene 5 μg/mL di insulina, 6 μg/mL transferrina e 5 ng/mL acido selenioso) + 50 μg/mL ascorbato-2-fosfato + 1% amminoacido non essenziale + 50 μg/mL agente antimicrobico per le cellule primarie) e posto in un'incubatrice a 37 °C, umidità dell'85% e 5% CO2.
- Coltura dei dischi per 4 giorni all'interno di un sistema bioreattore secondo i gruppisperimentali 16. Nel gruppo patologico, mantenere condizioni di carico degenerative a 0,32-0,5 MPa, 5 Hz per 2 h /giorno. Nel gruppo di controllo fisiologico utilizzare un protocollo di carico di 0,02-0,2 MPa, 0,2 Hz per 2 h/giorno.
NOTA: Posizionare gli IVD in camere contenenti 5 mL di supporto IVD durante le procedure di carico. Il volume dipende dalle dimensioni delle camere di carico del bioreattore. Tra le procedure di carico, posizionare gli IVD in piastre a sei potte con 7 mL di mezzo di coltura IVD per il recupero a gonfiore libero. - Per analizzare le variazioni dell'altezza del disco durante il periodo sperimentale, misurare l'altezza del disco con una pinza dopo la dissezione IVD (linea di base) e quindi giornalmente dopo il periodo di gonfiore libero e dopo il caricamento dinamico per la durata sperimentale.
3. Iniezione del fattore alfa della necrosi tumorale intradiscale (TNF-α)
- Subito dopo il primo ciclo di caricamento dinamico il primo giorno, posizionare gli IVD in una piastra di Petri in posizione verticale e stabilizzare gli IVD con una pinzetta.
- Iniettare TNF-α ricombinante (100 ng in 70 μL di PBS per IVD) con un ago di insulina calibro 30 nel tessuto pulposo del nucleo del gruppo patologico17. Iniettare lentamente ad una velocità di circa 70 μL in 1 min.
- Dopo l'iniezione, tirare la siringa a metà strada all'interno dell'IVD e tirare lo stantuffo della siringa per creare un vuoto che impedisce alla soluzione iniettata di fuoriuscire, prima di rimuovere completamente l'ago e la siringa dall'IVD.
NOTA: Eseguire un esperimento pilota iniettando PBS contenente colorante blu trypan per valutare la distribuzione della soluzione iniettata dopo il carico e la coltura notturna.
4. Espressione genica
- Raccogli gli IVD il giorno 4. Raccogliere il tessuto del nucleo pulposo (NP) (parte gelosa nel mezzo dell'IVD) con un punzone di biopsia. Raccogliere il fibroso annulus esterno (AF) con una lama bisturi (n. 20).
NOTA: Per il riferimento di base al giorno 0, raccogliere i tessuti immediatamente dopo la dissezione per l'estrazione dell'RNA. - Utilizzare la quantità di tessuto NP o AF necessaria per l'analisi dell'espressione genica, a seconda del progetto sperimentale.
NOTA: Per gli esperimenti attuali è stato utilizzato circa 150 mg di tessuto. Il rapporto tra la soluzione di isolamento dell'RNA e la massa tissutale dovrebbe essere di almeno 2 mL per 100-150 mg di tessuto per un'estrazione efficiente. - Digerire il tessuto NP o AF con la soluzione di digestione (0,2% di pronasi in DMEM, filtro sterilizzato) e incubare per 1 h a 37 °C con agitazione magnetica18.
- Congelare flash i campioni di tessuto utilizzando azoto liquido e polverizzare in una polvere fine. Dividere equamente la polvere di tessuto polverizzato in due tubi da 2 ml ciascuno contenenti 1 ml di tiocianato di guanidina e fenolo in una soluzione monofase (soluzione di isolamento dell'RNA).
- Eseguire l'omogeneizzazione in tubi da 2 ml contenenti la soluzione di isolamento dell'RNA e la polvere di tessuto polverizzato. Omogeneizzare la polvere di tessuto 5x con una sfera di acciaio inossidabile da 8 mm e un tissutale a 30 Hz per 3 min. Centrifugare a 12.000 x g,4 °C per 10 min e trasferire il supernatante in un tubo fresco. I supernatanti possono essere conservati a -80 °C per almeno un mese.
- Aggiungere 0,1 mL di 1-bromo-3-cloropropano (BCP) per 1 mL di soluzione di isolamento dell'RNA e agitare vigorosamente per 15 s. Conservare la miscela risultante a temperatura ambiente su uno shaker orbitale per 15 minuti e centrifugare a 12.000 x g per 15 min a 4 °C.
NOTA: L'RNA rimane esclusivamente nella fase acquosa superiore. - Trasferire la fase acquosa in un tubo fresco e precipitare l'RNA con 0,25 mL di isopropanolo e 0,25 ml di soluzione ad alta precipitazione del sale per 1 ml di soluzione di isolamento dell'RNA utilizzata per l'omogeneizzazione iniziale. Conservare i campioni a temperatura ambiente per 15 minuti su uno shaker orbitale e centrifugare a 12.000 x g per 8 min a 4 °C.
NOTA: In alternativa, utilizzare il metodo di estrazione dell'RNA basato su colonne che generalmente porta a una maggiore purezza dell'RNA ma a una minore resa dell'RNA. - Rimuovere il supernatante e lavare il pellet di RNA con 1 mL di 75% di etanolo per 1 mL di soluzione di isolamento dell'RNA utilizzato per l'omogeneizzazione iniziale. Centrifuga a 7.500 x g per 5 min a 4 °C.
- Rimuovere il lavaggio dell'etanolo e asciugare brevemente il pellet di RNA ad aria compressa per 3-5 minuti. Sciogliere l'RNA in 20 μL di acqua trattata con dietilpirocarbonato (DEPC) passando la soluzione alcune volte attraverso una punta di pipetta e incubando per 10-15 min a 55-60 °C.
- Misurare l'assorbanza a 230 nm, 260 nm e 280 nm (rispettivamente A230, A260 e A280). A260 di 1,0 corrisponde a 40 μg/mL RNA. Si prevede un rapporto A260/A280 di 1,6-1,9, mentre la contaminazione si traduce in un rapporto A260/A280 di <1,6.
- Preparare una miscela di reazione inversa transcriptasi (RT) per un volume di reazione di 20 μL. La miscela contiene miscela enzimatica RT, inibitore della ribonucleasi, proteina aiutante, primer, dNTP, MgCl2, acqua libera di RNasi e 0,4 μg di campione di RNA.
- Centrifugare brevemente i tubi RT per mescolare tutti i componenti nella parte inferiore del tubo.
- Posizionare i campioni nello strumento termociclo. Selezionare il programma appropriato per l'RT. Eseguire l'RT per 10 min a 25 °C, seguito dalla fase di trascrizione inversa per 120 min a 42 °C e dall'inattivazione della transcriptasi inversa per 5 minuti a 85 °C, raffreddandola fino a 4 °C alla fine.
- Diluire il cDNA risultante con acido tris(idrossimetil)amminometano (Tris)-etilendiamminatetraacetico (EDTA) (TE) tampone (10 mM Tris con 1 mM EDTA) ad una concentrazione finale di 0,4 μg di RNA utilizzato per RT per soluzione di cDNA da 100 μL. Conservare i campioni di cDNA a -20 °C.
- Eseguire la reazione a catena della polimerasi in tempo reale (PCR) utilizzando un volume di reazione di 10 μL. Il volume di reazione contiene il mix principale (contenente DNA polimerasi, glicosilasi uracil-DNA, dNTP con dUTP, riferimento passivo e componenti tampone ottimizzati), primer in avanti 45 μM, primer inverso 45 μM, sonda 12,5 μM (contenente un colorante reporter collegato all'estremità 5 ' della sonda, un legante di scanalatura minore all'estremità 3 ' della sonda e un quencher non fluorescente all'estremità 3 ' della sonda) , 2 μL cDNA e acqua trattata con DEPC.
- Eseguire un controllo endogeno (RPLP0) per la quantificazione relativa con il metodo 2-ΔΔCT 19.
- Aggiungere esempi in duplicati ed eseguire un controllo senza modello aggiungendo TE-buffer anziché cDNA. Eseguire PCR in condizioni standard (2 min a 50 °C, 10 min a 95 °C, 40 cicli di 15 s a 95 °C e 1 min a 60 °C).
- Eseguire la quantificazione relativa degli obiettivi di mRNA seguendo il metodo ct comparativo. La quantità di mRNA normalizzata al campione di base è calcolata come 2-ΔΔCT, mentre ΔΔCT è la differenza tra il ΔCT (CT target- CT endogeno control) del campione e ΔCT (CT target e CT endogeno control) del campione di base.
5. Quantificazione del contenuto proteico nel mezzo IVD
- Raccogliere il mezzo condizionato dai campioni IVD per misurare il contenuto proteico nel mezzo. Eseguire test immunoassorbenti legati all'enzima (ELISA) secondo il protocollo della proteina bersaglio.
- L'interleuchina bovina-8 (IL-8) è quantificata con kit ELISA IL8 anti bovino secondo le istruzioni del produttore.
Risultati
Il carico degenerativo in mezzo a basso glucosio combinato con l'iniezione di TNF-α ha causato un aumento significativo dell'espressione genica dei marcatori proinfiammatori interleuchina 6 (IL-6) e interleuchina 8 (IL-8) rispetto al gruppo di controllo fisiologico nelle cellule NP dopo 4 giorni dicoltura (Figura 2). Al contrario, non abbiamo osservato cambiamenti significativi per i geni proinflammatori interleuchina 1β (IL-1β) e TNF-α nelle cellule NP (dati non mostrati). Inoltre, le c...
Discussione
Qui abbiamo fornito un protocollo dettagliato per simulare l'IVDD degenerativo e infiammatorio. Questo protocollo può essere applicato per esami dettagliati delle vie infiammatorie che portano agli effetti distruttivi sul disco. Inoltre, il protocollo può aiutare a determinare obiettivi terapeutici promettenti coinvolti nella progressione della malattia.
Recentemente abbiamo dimostrato che il TNF-α ricombinante umano potrebbe indurre infiammazione nelle cellule NP sia bovine che umane
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato da AO Foundation e AOSpine International. Babak Saravi ha ricevuto il sostegno della Fondazione tedesca Spine e della Fondazione tedesca per l'osteoartrite. Gernot Lang è stato sostenuto dal Programma Berta-Ottenstein per scienziati clinici avanzati, Facoltà di Medicina, Università di Friburgo, Germania.
Materiali
| Name | Company | Catalog Number | Comments |
| 1-Bromo-3-chloropropane(BCP) | Sigma-Aldrich, St. Louis, USA | B9673 | |
| Ascorbate-2-phosphate | Sigma-Aldrich, St. Louis, USA | A8960 | |
| Band saw | Exakt Apparatebau, Norderstedt, Germany | model 30/833 | |
| Betadine | Munndipharma, Frankfurt, Germany | ||
| Bovine IL-8 Do.it-Yourself ELISA | Kingfisher Biotech, St. Paul, USA | DIY1028B-003 | |
| Corning ITS Premix | Corning Inc., New York, USA | 354350 | |
| DMEM high glucose | Gibco by life technologies, Carlsbad, USA | 10741574 | |
| DMEM low glucose | Gibco by life technologies, Carlsbad, USA | 11564446 | |
| Ethanol for molecular biology | Sigma-Aldrich, St. Louis, USA | 09-0851 | |
| Fetal Bovine Serum (FBS) | Gibco by life technologies, Carlsbad, USA | A4766801 | |
| Non-essential amino acid solution | Gibco by life technologies, Carlsbad, USA | 11140050 | |
| Penicillin/Streptomycin(P/S) | gibco by life technologies, Carlsbad, USA | 11548876 | |
| Phosphate Buffer Solution, tablet | Sigma-Aldrich, St. Louis, USA | P4417 | |
| Pronase | Sigma-Aldrich, St. Louis, USA | 10165921001 | |
| Primocin | InvivoGen, Sandiego, USA | ant-pm-05 | |
| Pulsavac Jet Lavage System | Zimmer, IN,USA | ||
| TissueLyser II | Quiagen, Venlo, Netherlands | 85300 | |
| Streptavidinn-HRP | Kingfisher Biotech, St. Paul, USA | AR0068-001 | |
| Superscript VILO | Invitrogen by life Technologies, Carlsbad, USA | 10704274 | |
| cDNA Synthesis Kit | Applied Biosystems by life technologies | 10400745 | |
| TaqMan Universal Master Mix | Applied Biosystems by life technologies | ||
| TNF-alpha, recombinant human protein | R&D systems, Minnesota, USA | 210-TA-005 | |
| TRI Reagent | Molecular Research Center, Cincinnati, USA | TR 118 | |
| Tris-EDTA buffer solution | sigma-Aldrich, St. Louis, USA | 93283 | |
| Gene bIL-6 | Applied Biosystems by life technologies | Custom made probes | Primer fw (5′–3′) TTC CAA AAA TGG AGG AAA AGG A Primer rev (5′–3′) TCC AGA AGA CCA GCA GTG GTT Probe (5′FAM/3′TAMRA) CTT CCA ATC TGG GTT CAA TCA GGC GATT |
| Gene bIL8 | Applied Biosystems by life technologies | Bt03211906_m1 | |
| Gene bTNF-alpha | Applied Biosystems by life technologies | Custom made probes | Primer fw (5′–3′) CCT CTT CTC AAG CCT CAA GTA ACA A Primer rev (5′–3′) GAG CTG CCC CGG AGA GTT Probe (5′FAM/3′TAMRA) ATG TCG GCT ACA ACG TGG GCT ACC G |
| GENE bIL1beta | Applied Biosystems by life technologies | Custom made probes | Primer fw (5′–3′) TTA CTA CAG TGA CGA GAA TGA GCT GTT Primer rev (5′–3′) GGT CCA GGT GTT GGA TGC A Probe (5′FAM/3′TAMRA) CTC TTC ATC TGT TTA GGG TCA TCA GCC TCA A |
| RPLP0 | Applied Biosystems by life technologies | Bt03218086_m1 |
Riferimenti
- Vos, T., et al. Global, regional, and national incidence, prevalence, and years lived with disability for 328 diseases and injuries for 195 countries, 1990-2016: a systematic analysis for the Global Burden of Disease Study 2016. The Lancet. 390 (10100), 1211-1259 (2017).
- Hoy, D., et al. Measuring the global burden of low back pain. Best Practice & Research Clinical Rheumatology. 24 (2), 155-165 (2010).
- Thiese, M. S., et al. Prevalence of low back pain by anatomic location and intensity in an occupational population. BMC Musculoskeletal Disorders. 15 (1), 283 (2014).
- Katz, J. N. Lumbar Disc Disorders and Low-Back Pain: Socioeconomic Factors and Consequences. The Journal of Bone and Joint Surgery (American). 88, 21 (2006).
- Vlaeyen, J. W. S., et al. Low back pain. Nature Reviews Disease Primers. 4 (1), 52 (2018).
- Khan, A. N., et al. Inflammatory biomarkers of low back pain and disc degeneration: a review: Biomarkers of disc degeneration and back pain. Annals of the New York Academy of Sciences. 1410 (1), 68-84 (2017).
- Kim, H. S., Wu, P. H., Jang, I. T. Lumbar Degenerative Disease Part 1: Anatomy and Pathophysiology of Intervertebral Discogenic Pain and Radiofrequency Ablation of Basivertebral and Sinuvertebral Nerve Treatment for Chronic Discogenic Back Pain: A Prospective Case Series and Review of Literature. International Journal of Molecular Sciences. 21 (4), 1483 (2020).
- Adams, M. A., Roughley, P. J. What is Intervertebral Disc Degeneration, and What Causes It. Spine. 31 (18), 2151-2161 (2006).
- Wu, P. H., Kim, H. S., Jang, I. T. Intervertebral Disc Diseases Part 2: A Review of the Current Diagnostic and Treatment Strategies for Intervertebral Disc Disease. International Journal of Molecular Sciences. 21 (6), 2135 (2020).
- Lurie, J. D., et al. Surgical Versus Nonoperative Treatment for Lumbar Disc Herniation: Eight-Year Results for the Spine Patient Outcomes Research Trial. Spine. 39 (1), 3-16 (2014).
- Risbud, M. V., Shapiro, I. M. Role of cytokines in intervertebral disc degeneration: pain and disc content. Nature Reviews Rheumatology. 10 (1), 44-56 (2014).
- Ponnappan, R. K., et al. An organ culture system to model early degenerative changes of the intervertebral disc. Arthritis Research & Therapy. 13 (5), 171 (2011).
- O'Connell, G. D., Vresilovic, E. J., Elliott, D. M. Comparison of Animals Used in Disc Research to Human Lumbar Disc Geometry. Spine. 32 (3), 328-333 (2007).
- Stannard, J. T., et al. Development of a whole organ culture model for intervertebral disc disease. Journal of Orthopaedic Translation. 5, 1-8 (2016).
- Li, Z., et al. Preclinical ex-vivo Testing of Anti-inflammatory Drugs in a Bovine Intervertebral Degenerative Disc Model. Frontiers in Bioengineering and Biotechnology. 8, 583 (2020).
- Li, Z., et al. Development of an ex vivo cavity model to study repair strategies in loaded intervertebral discs. European Spine Journal. 25 (9), 2898-2908 (2016).
- Kazezian, Z., Li, Z., Alini, M., Grad, S., Pandit, A. Injectable hyaluronic acid down-regulates interferon signaling molecules, IGFBP3 and IFIT3 in the bovine intervertebral disc. Acta Biomaterialia. 52, 118-129 (2017).
- Caprez, S., Menzel, U., Li, Z., Grad, S., Alini, M., Peroglio, M. Isolation of high-quality RNA from intervertebral disc tissue via pronase predigestion and tissue pulverization. JOR Spine. 1 (2), 1017 (2018).
- Lopa, S., Ceriani, C., Cecchinato, R., Zagra, L., Moretti, M., Colombini, A. Stability of housekeeping genes in human intervertebral disc, endplate and articular cartilage cells in multiple conditions for reliable transcriptional analysis. European Cells & Materials. 31, 395-406 (2016).
- Lang, G., et al. An intervertebral disc whole organ culture system to investigate proinflammatory and degenerative disc disease condition. Journal of Tissue Engineering and Regenerative Medicine. 12 (4), 2051-2061 (2018).
- Du, J., et al. Proinflammatory intervertebral disc cell and organ culture models induced by tumor necrosis factor alpha. JOR Spine. 3, 1104 (2020).
- Purmessur, D., Walter, B. A., Roughley, P. J., Laudier, D. M., Hecht, A. C., Iatridis, J. A role for TNFα in intervertebral disc degeneration: A non-recoverable catabolic shift. Biochemical and Biophysical Research Communications. 433 (1), 151-156 (2013).
- Walter, B. A., Likhitpanichkul, M., Illien-Junger, S., Roughley, P. J., Hecht, A. C., Iatridis, J. C. TNFα Transport Induced by Dynamic Loading Alters Biomechanics of Intact Intervertebral Discs. PLOS One. 10 (3), 0118358 (2015).
- Gullbrand, S. E., et al. A large animal model that recapitulates the spectrum of human intervertebral disc degeneration. Osteoarthritis and Cartilage. 25 (1), 146-156 (2017).
- Willems, N., et al. Safety of intradiscal injection and biocompatibility of polyester amide microspheres in a canine model predisposed to intervertebral disc degeneration: intradiscal application of pea microspheres. Journal of Biomedical Materials Research Part B: Applied Biomaterials. 105 (4), 707-714 (2017).
- Michalek, A. J., Buckley, M. R., Bonassar, L. J., Cohen, I., Iatridis, J. C. The effects of needle puncture injury on microscale shear strain in the intervertebral disc annulus fibrosus. The Spine Journal. 10 (12), 1098-1105 (2010).
- Illien-Jünger, S., et al. The combined effects of limited nutrition and high-frequency loading on intervertebral discs with endplates. Spine. 35 (19), 1744-1752 (2010).
- Gantenbein, B., et al. Organ culture bioreactors--platforms to study human intervertebral disc degeneration and regenerative therapy. Current Stem Cell Research & Therapy. 10 (4), 339-352 (2015).
- Boubriak, O. A., Watson, N., Sivan, S. S., Stubbens, N., Urban, J. P. G. Factors regulating viable cell density in the intervertebral disc: blood supply in relation to disc height. Journal of Anatomy. 222 (3), 341-348 (2013).
- Maroudas, A., Stockwell, R. A., Nachemson, A., Urban, J. Factors involved in the nutrition of the human lumbar intervertebral disc: cellularity and diffusion of glucose in vitro. Journal of Anatomy. 120, 113-130 (1975).
- Beckstein, J. C., Sen, S., Schaer, T. P., Vresilovic, E. J., Elliott, D. M. Comparison of Animal Discs Used in Disc Research to Human Lumbar Disc: Axial Compression Mechanics and Glycosaminoglycan Content. Spine. 33 (6), 166-173 (2008).
- Walter, B. A., Illien-Jünger, S., Nasser, P. R., Hecht, A. C., Iatridis, J. C. Development and validation of a bioreactor system for dynamic loading and mechanical characterization of whole human intervertebral discs in organ culture. Journal of Biomechanics. 47 (9), 2095-2101 (2014).
- Rajan, N. E., et al. Toll-Like Receptor 4 (TLR4) Expression and Stimulation in a Model of Intervertebral Disc Inflammation and Degeneration. Spine. 38 (16), 1343-1351 (2013).
- vanden Akker, G. G., Rorije, A. J., Davidson, E. N. B., vander Kraan, P. M. Phenotypic marker genes distinguish inner and outer annulus fibrosus from nucleus pulposus tissue in the bovine intervertebral disc. Osteoarthritis and Cartilage. 25, 402 (2017).
- Du, J., et al. Functional cell phenotype induction with TGF-β1 and collagen-polyurethane scaffold for annulus fibrosus rupture repair. European Cells & Materials. 39, 1-17 (2020).
- Risbud, M. V., et al. Defining the phenotype of young healthy nucleus pulposus cells: recommendations of the Spine Research Interest Group at the 2014 annual ORS meeting. Journal of Orthopaedic Research: Official Publication of the Orthopaedic Research Society. 33 (3), 283-293 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon