È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento delle cellule muscolari lisce aortiche primarie specifiche del paziente e misurazioni semiquantitative di contrazione in tempo reale in vitro
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo documento descrive un metodo basato su colture di espianto per l'isolamento e la coltura di cellule muscolari lisce aortiche umane primarie e specifiche per il paziente e fibroblasti dermici. Inoltre, viene presentato un nuovo metodo per misurare la contrazione cellulare e la successiva analisi, che può essere utilizzato per studiare le differenze specifiche del paziente in queste cellule.
Abstract
Le cellule muscolari lisce (SMC) sono il tipo di cellula predominante nel mezzo aortico. Il loro meccanismo contrattile è importante per la trasmissione della forza nell'aorta e regola la vasocostrizione e la vasodilatazione. Le mutazioni nei geni che codificano per le proteine dell'apparato contrattile SMC sono associate a malattie aortiche, come gli aneurismi dell'aorta toracica. Misurare la contrazione SMC in vitro è impegnativo, specialmente in modo ad alta produttività, che è essenziale per lo screening del materiale del paziente. I metodi attualmente disponibili non sono adatti a questo scopo. Questo documento presenta un nuovo metodo basato sul rilevamento dell'impedenza del substrato elettrico della cellula (ECIS). In primo luogo, viene descritto un protocollo di espianto per isolare le SMC primarie umane specifiche del paziente dalle biopsie aortiche e dai fibroblasti dermici primari umani specifici del paziente per lo studio degli aneurismi aortici. Successivamente, viene fornita una descrizione dettagliata di un nuovo metodo di contrazione per misurare la risposta contrattile di queste cellule, compresa la successiva analisi e il suggerimento per confrontare diversi gruppi. Questo metodo può essere utilizzato per studiare la contrazione delle cellule aderenti nel contesto di studi traslazionali (cardiovascolari) e studi di screening di pazienti e farmaci.
Introduzione
Le cellule muscolari lisce (SMC) sono il tipo di cellula predominante nello strato mediale aortico, lo strato più spesso dell'aorta. All'interno della parete, sono orientati radialmente e sono coinvolti, tra le altre funzioni, nella vasocostrizione e nella vasodilatazione1. Il macchinario contrattile SMC è coinvolto nella trasmissione della forza nell'aorta attraverso il collegamento funzionale con la matrice extracellulare2. Mutazioni in geni che codificano per le proteine dell'apparato contrattile SMC, come la catena pesante della miosina muscolare liscia (MYH11) e l'actina della muscolatura liscia (ACTA2), sono state correlate a casi di aneurismi dell'aorta toracica familiare, sottolineando la rilevanza della contrazione della SMC nel mantenimento dell'integrità strutturale e funzionale dell'aorta 1,2 . Inoltre, le mutazioni nella via di segnalazione del TGFβ sono anche associate agli aneurismi aortici e i loro effetti nella fisiopatologia dell'aneurisma aortico possono essere studiati anche nei fibroblasti cutanei3.
La misurazione ad alto rendimento della contrazione SMC in vitro è impegnativa. Poiché la contrattilità SMC non può essere misurata in vivo nell'uomo, i saggi in vitro su cellule umane presentano un'alternativa fattibile. Inoltre, lo sviluppo dell'aneurisma dell'aorta addominale (AAA) in modelli animali è indotto chimicamente, ad esempio, dalla perfusione di elastasi o causato da una mutazione specifica. Pertanto, i dati sugli animali non sono paragonabili allo sviluppo di AAA nell'uomo, che per lo più ha una causa multifattoriale, come il fumo, l'età e / o l'aterosclerosi. In vitro La contrattilità SMC è stata finora misurata principalmente mediante microscopia a forzadi trazione 4,5, quantificazione dei flussi di calcio intracellulare a fluorescenza Fura-26 e saggi di rughe di collagene7. Mentre la microscopia con forza di trazione fornisce preziose informazioni numeriche sulle forze generate da una singola cellula, non è adatta per lo screening ad alto rendimento a causa della complessa elaborazione dei dati matematici e dell'analisi di una cellula alla volta, il che significa che è molto dispendioso in termini di tempo misurare un numero rappresentativo di cellule per donatore. I saggi di tintura fura-2 e di rughe di collagene consentono la determinazione superficiale della contrazione e non danno un risultato numerico preciso, rendendoli meno adatti a discriminare le differenze specifiche del paziente. La compromissione della contrazione della SMC nelle cellule derivate dall'aorta dei pazienti con aneurisma dell'aorta addominale è stata dimostrata per la prima volta ottimizzando un nuovo metodo per misurare la contrazione della SMC in vitro8. Ciò è stato fatto riutilizzando il metodo di rilevamento dell'impedenza del substrato elettrico della cella (ECIS). ECIS è un test in tempo reale a medio rendimento per la quantificazione del comportamento e della contrazione delle cellule aderenti 9,10,11 come la crescita e il comportamento SMC nei saggi di guarigione e migrazione delle ferite 12,13,14. Il metodo esatto è descritto nella sezione del protocollo. In questo modo ottimizzato, l'ECIS può anche essere utilizzato per studiare la contrazione dei fibroblasti a causa delle loro dimensioni e morfologia simili.
Lo scopo di questo documento è quello di fornire una descrizione graduale del metodo per misurare la contrazione SMC in vitro utilizzando ECIS8 e confrontando la contrazione tra controllo e SMC paziente. In primo luogo, viene spiegato l'isolamento e la coltura delle SMC primarie dalle biopsie aortiche di controllo e del paziente, che possono essere utilizzate per la misurazione della contrazione. In secondo luogo, vengono descritte le misurazioni e l'analisi della contrazione, insieme alla verifica dell'espressione del marcatore SMC. Inoltre, questo documento descrive il metodo per l'isolamento dei fibroblasti dermici specifici del paziente la cui contrazione può essere misurata utilizzando la stessa metodologia. Queste cellule possono essere utilizzate per studi specifici del paziente incentrati sull'aneurisma aortico o altre patologie cardiovascolari15 o studi prognostici utilizzando un protocollo di transdifferenziazione che consente la misurazione della contrazione prima dell'intervento chirurgico all'aneurisma16.
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Le biopsie aortiche sono state ottenute durante la riparazione dell'aneurisma aperto nei centri medici dell'Università di Amsterdam, nel centro medico dell'Università VU, Amsterdam, Zaans Medisch Centrum, Zaandam e l'ospedale Dijklander, Hoorn, Paesi Bassi. Il tessuto aortico di controllo è stato ottenuto dal pezzo dell'aorta attaccato all'arteria renale raccolta per i trapianti di rene. Sono stati inclusi solo i pazienti di età superiore ai 18 anni e tutti i pazienti hanno dato il loro consenso informato a partecipare allo studio. Tutto il materiale è stato raccolto in conformità con i regolamenti della Dichiarazione WMA di Helsinki e le linee guida istituzionali del Comitato etico medico del VU Medical Center. Tutti gli esperimenti e i protocolli sperimentali sono stati eseguiti in conformità con le linee guida istituzionali e approvati dal Comitato Etico Medico del VU Medical Center. Per informazioni complete sul controllo e sulle linee cellulari del paziente utilizzate, fare riferimento a 8.
1. Isolare le SMC umane primarie dalle biopsie aortiche
NOTA: Eseguire i seguenti passaggi sotto una cappa a flusso laminare sterile per colture tissutali. Indossare guanti e utilizzare tecniche asettiche standard quando si maneggiano campioni di sangue umano e tessuto umano. Le SMC sono coltivate in 231 Human Vascular Smooth Muscle Cell Basal Medium integrato con 100 U / mL di penicillina, 100 μg / mL di streptomicina e Smooth Muscle Growth Supplement indicato come SMC medium.
- Sterilizzare due paia di pinze chirurgiche e un bisturi immergendole in etanolo al 70% e successivamente asciugandole.
- Pipetta 2 mL di mezzo SMC in una capsula di Petri in cui verrà eseguita la dissezione del tessuto.
- Pipettare 2,5 mL di SMC medium in due palloni T25. Ruotare i palloni in modo che il piccolo volume di medio copra l'intera superficie.
- Trasportare la biopsia della parete aortica umana raccolta dalla sala operatoria al laboratorio su ghiaccio in un tubo di plastica sterile con soluzione di trasferimento tissutale fredda e sterile (vedere la tabella dei materiali) o 0,9% NaCl.
- Aprire il tubo con il tessuto all'interno del cappuccio di coltura del tessuto. Estrarre la biopsia dal tubo usando la pinza sterilizzata e metterla nella capsula di Petri (Figura 1A).
- Ispezionare visivamente la biopsia per identificare i tre strati aortici, la tunica intima (interna), media (al centro) e avventizia (strato esterno). Cerca la presenza di placche aterosclerotiche sul lato intima e tessuto connettivo viscido sul lato avventiziale (Figura 1B) per distinguere gli strati.
- Per isolare gli SMC dal supporto, rimuovere gli altri due livelli. Posizionare il tessuto con la placca intima rivolta verso l'alto per primo (Figura 1B). Rimuovere la placca solida allontanandola dal tessuto con una pinza mentre si spinge il tessuto verso il basso con l'altra coppia di pinze. Rimuovere gli strati successivi di placca fino a quando lo strato mediale rosa e uniforme è visibile.
- Capovolgere il tessuto (Figura 1C). Ripetere la stessa procedura, come nel passaggio 1.7, estraendo il livello avventizia (Figura 1D). Assicurati di rimuovere tutte le parti visibili in tutti i tentativi necessari, poiché questo livello non si stacca facilmente dal supporto.
NOTA: È essenziale che gli strati intimale e avventiziale siano rimossi correttamente per ottenere una popolazione SMC il più pulita possibile. - Una volta isolato lo strato mediale, tagliare il tessuto a cubetti di circa 1 mm x 1 mm x 1 mm. Premere il supporto verso il basso con una pinza e tagliare il tessuto in una direzione usando il bisturi. Non tagliare avanti e indietro; effettuare tagli puliti e unidirezionali per ridurre al minimo i danni. Cerca di creare il maggior numero possibile di cubi, date le dimensioni della biopsia (Figura 1E).
- Posizionare i pezzi di tessuto nel quarto superiore del pallone T25 usando la pinza. Posizionare 10-20 cubetti per pallone se la quantità di materiale lo consente (Figura 1F).
NOTA: utilizzare una pinza liscia per ridurre al minimo l'adesione del tessuto alle costole della pinza e staccare facilmente il tessuto nel pallone. - Incubare i cubetti di tessuto nei palloni T25 per circa 10 giorni al 5% di CO2 a 37 °C in un incubatore.
NOTA: la prima crescita cellulare è prevista in quel periodo. Le celle che inizialmente migrano potrebbero sembrare più piccole delle normali SMC. - Una volta osservata la crescita cellulare, aggiungere 2,5 ml di terreno al pallone, assicurandosi di non pipettarlo sui pezzi di tessuto per evitare che si stacchino.
- Dopo circa altri 5 giorni, quando si osservano gruppi di cellule intorno ai pezzi di tessuto, cambiare il terreno di coltura. Se i pezzi di tessuto si staccano, rimuoverli in quanto non si ricollegano.
- Una volta che le cellule sono confluenti all'80-90%, trasferirle in un pallone T75 e continuare a coltivare in questo formato.
NOTA: si consiglia un rapporto di divisione 1:2 o 1:3. Congelare un backup delle celle a un passaggio precoce. Le cellule generalmente mantengono le loro proprietà fino al passaggio 10; i passaggi successivi non dovrebbero essere usati per esperimenti.
2. Isolare i fibroblasti dermici primari dalle biopsie cutanee
NOTA: Eseguire i seguenti passaggi sotto una cappa a flusso laminare sterile per colture tissutali. Indossare guanti e utilizzare tecniche asettiche standard quando si maneggiano campioni di sangue umano e tessuto umano. I fibroblasti sono coltivati in basale media integrato con il 10% di siero bovino fetale, 100 U / mL di penicillina e 100 μg / mL di streptomicina, indicato come mezzo fibroblastico.
- Sterilizzare due paia di pinze chirurgiche e un bisturi immergendole in etanolo al 70% e successivamente asciugandole.
- Pipetta 2 mL di mezzo fibroblastico in una capsula di Petri in cui verrà eseguita la dissezione del tessuto.
- Pipettare 2,5 mL di fibroblasto medio in due palloni T25. Ruotare i palloni in modo che il piccolo volume di medio copra l'intera superficie.
- Trasportare la biopsia cutanea raccolta dalla sala operatoria al laboratorio su ghiaccio in soluzione di trasferimento tissutale fredda e sterile (vedere la tabella dei materiali) o 0,9% NaCl in un tubo di plastica sterile.
- Aprire il tubo con il tessuto all'interno del cappuccio di coltura del tessuto. Estrarre la biopsia dal tubo usando la pinza sterilizzata e metterla nella capsula di Petri (Figura 2A).
- Ispezionare visivamente la biopsia per identificare i tre strati della pelle, l'epidermide, il derma e il grasso sottocutaneo. Cerca una superficie della pelle riconoscibile, a volte con capelli visibili, per identificare l'epidermide. Sul lato opposto, cerca il grasso sottocutaneo, che è spesso giallo e viscido. Identificare lo strato tra l'epidermide e il grasso sottocutaneo come derma, la fonte di fibroblasti vitali (Figura 2B).
- Per isolare i fibroblasti dal derma, rimuovere gli altri due strati e posizionare il tessuto su un lato in modo che tutti e tre gli strati siano visibili.
NOTA: A differenza del tessuto aortico, gli strati della pelle non possono essere separati l'uno dall'altro; quindi, devono essere tagliati. Il tessuto è anche più gommoso, rendendo più difficile il taglio. Usa un bisturi affilato. - Tenere il tessuto verso il basso con una pinza. Tagliare in parallelo al confine tra epidermide e derma. Tagliare via l'intera epidermide. Cerca di tagliare in una linea pulita e non muoverti avanti e indietro per evitare danni ai tessuti.
- Capovolgere il tessuto. Ripetere la stessa procedura del passaggio 2.8; questa volta, tagliare all'interno del derma parallelamente al bordo con il grasso sottocutaneo.
- Una volta isolato il derma, tagliare il tessuto a cubetti di circa 1 x 1 x 1 mm3. Premere il tessuto verso il basso con una pinza e tagliare il tessuto in una direzione usando il bisturi. Non tagliare avanti e indietro; effettuare tagli puliti e unidirezionali per ridurre al minimo i danni. Cerca di creare il maggior numero possibile di cubi, date le dimensioni della biopsia (Figura 2C).
- Posizionare i pezzi di tessuto nel quarto superiore del pallone T25 usando la pinza. Posizionare 10-20 cubi per pallone se la quantità di materiale lo consente (Figura 2D).
NOTA: utilizzare una pinza liscia per ridurre al minimo l'adesione del tessuto alle costole della pinza e staccare facilmente il tessuto nel pallone. - Incubare i cubetti di tessuto nei palloni T25 per circa 10 giorni al 5% di CO2 a 37 °C in un incubatore.
NOTA: la prima crescita cellulare è prevista intorno a allora. Le cellule che inizialmente migrano potrebbero sembrare più piccole dei normali fibroblasti. - Una volta osservata la crescita cellulare, aggiungere 2,5 ml di terreno al pallone, assicurandosi di non pipettarlo sui pezzi di tessuto per evitare che si stacchino.
- Dopo circa altri 5 giorni, quando si possono osservare gruppi di cellule intorno ai pezzi di tessuto, cambiare il terreno di coltura. Se i pezzi di tessuto si staccano, rimuoverli in quanto non si ricollegano.
- Una volta che le cellule sono confluenti all'80-90%, trasferirle in un pallone T75 e continuare a coltivare in questo formato.
NOTA: si consiglia un rapporto di divisione 1:2 o 1:3. Congelare un backup delle celle a un passaggio precoce. Le cellule generalmente mantengono le loro proprietà fino al passaggio 10; i passaggi successivi non dovrebbero essere usati per esperimenti.
3. Misurazione della contrazione (esempio SMC)
- Preparare un array ECIS 96w10e (vedere la Figura 3A per un'immagine rappresentativa dell'array con elettrodi e celle ingranditi seminati sugli elettrodi).
NOTA: Eseguire i seguenti passaggi sotto una cappa a flusso laminare sterile per colture tissutali.- Rivestire un array sterile 96w10e con 200 μL di 10 mM di L-cisteina per pozzetto per 30 minuti a temperatura ambiente.
- Lavare il piatto due volte con acqua sterile. Lasciare asciugare la piastra per una notte nella cappa di coltura del tessuto con il coperchio leggermente aperto.
NOTA: Il rivestimento della piastra con L-cisteina è necessario solo quando si utilizza la piastra per la prima volta. - Il giorno successivo, sterilizzare UV piastra e coperchio per 30 minuti. Ruotare il coperchio verso l'alto in modo che l'interno sia sterilizzato. Una volta sterilizzata la piastra, non aprire la piastra all'esterno della cappa di flusso.
- Sezionamento di celle nella matrice ECIS
- Soluzione di gelatina sterile all'1% preriscaldata a bagnomaria per 30 min.
NOTA: la soluzione viene conservata in frigorifero e potrebbe solidificarsi, rendendo difficile la pipettatura. - Successivamente, rivestire i pozzetti con 100 μL della soluzione di gelatina all'1 % per pozzetto e incubare la piastra a 37 °C per almeno 1 ora.
- Aspirare la gelatina dai pozzetti.
- Contare le cellule utilizzando un contatore di cellule automatizzato e seminare gli SMC in triplice copia a una densità di semina di 30.000 cellule / pozzetto in 200 μL di mezzo SMC.
- Posizionare la piastra con gli SMC nel supporto del pozzetto ECIS 96 nell'incubatore di colture cellulari. Fare doppio clic sul software ECIS Applied Biophysics per aprire il programma e premere il pulsante Setup .
- Controllare se tutti gli elettrodi hanno contatto con il supporto (verde; rosso se nessuna connessione) nel pannello inferiore sinistro etichettato Well Configuration. Se gli elettrodi non sono collegati correttamente, regolare la piastra nel supporto prima di iniziare la misurazione.
- Selezionate il tipo di piastra (matrice a 96 pozzetti) nello stesso pannello facendo clic su Tipo matrice(Array type).
- Nel pannello superiore destro Data Collection Setup, fare clic su singola frequenza e modificare il valore di impedenza a 4000 Hz e l'intervallo a 8 s.
- Fare clic sul pulsante Start per avviare le misurazioni. Attendere l'apertura di un nuovo pannello, in cui è possibile salvare il file ECIS.
- Consentire alle celle di attaccarsi e stabilire un monostrato per 48 ore.
NOTA: l'attacco e la diffusione delle celle sugli elettrodi generano un valore di resistenza basale (Figura 3B).
- Soluzione di gelatina sterile all'1% preriscaldata a bagnomaria per 30 min.
- Stimolazione delle cellule per indurre la contrazione
- Indurre la contrazione SMC stimolando le cellule con 10 μg/mL di ionoforo di calcio, ionomicina.
NOTA: La ionomicina indurrà l'afflusso di Ca2+ extracellulare, attivando la cascata contrattile; vedere la Figura 4A per immagini rappresentative di cellule contratte non stimolate e stimolate. - Diluire 1 mg di ionomicina in polvere in 100 μL di dimetilsolfossido per ottenere una soluzione madre di 10 mg/mL e conservare aliquote da 10 μL a -20 °C. Prima dell'uso, diluire la soluzione di ionomicina 1:10 in mezzo SMC per preparare la soluzione di lavoro da aggiungere alle cellule.
- Eseguire la stimolazione cellulare mentre l'array è ancora nel supporto dell'array all'interno dell'incubatore di colture cellulari e gli elettrodi sono collegati al sistema.
- Per stimolare le cellule, rimuovere il coperchio e posizionarlo su una superficie sterile all'interno dell'incubatore. Preparare due pipette, impostate a 2 μL e 150 μL.
- Prima di iniziare la stimolazione, premere Mark nel software e inserire un commento.
NOTA: Questo renderà più facile trovare i tempi esatti della stimolazione durante l'analisi dei dati. - Pipettare 2 μL della soluzione di lavoro di ionomicina in ciascun pozzo il più rapidamente possibile. Una volta che tutte le cellule sono state stimolate, mescolare il mezzo nei pozzetti usando la seconda pipetta.
NOTA: a causa degli effetti rapidi, non è necessario modificare le punte tra diverse linee cellulari e condizioni. Lavorare rapidamente stimolando e mescolando perché la ionomicina ha un effetto acuto. Quando si stimola una piastra piena, stimolare un massimo di 3 colonne contemporaneamente. Dopo ogni stimolazione, attendere almeno 30 minuti fino alla stimolazione successiva per consentire alle cellule di riprendersi dalla temperatura e dal cambiamento di CO2 causato dall'apertura della porta dell'incubatore. - Subito dopo aver terminato la stimolazione, premere nuovamente Mark per aggiungere un commento che la stimolazione è stata eseguita.
- Terminare l'esperimento di stimolazione
- Registrare i valori di impedenza per circa 5 minuti dopo la stimolazione della ionomicina. Terminare la registrazione premendo Fine.
- Riutilizzare gli array ECIS fino a tre volte: lavare i pozzetti due volte con acqua sterile e incubare la piastra a 37 °C per 2-3 ore con tripsina o un reagente simile. Ripetere i passaggi 3.1.2 e 3.1.3.
- Indurre la contrazione SMC stimolando le cellule con 10 μg/mL di ionoforo di calcio, ionomicina.
- Esportazione dei dati
- Per esportare i dati, fare clic su File | Esportare i dati | In Excel (selezionato). Selezionare una cartella in cui salvare il file.
- Fare clic su Separa quando il programma chiede di combinare o separare i fogli.
- Analisi dell'output contrattile
- Per calcolare la risposta contrattile, utilizzare la seguente equazione (1), dove C indica la contrazione (espressa in % della variazione rispetto al basale), la prestimulazione (PrS) indica il valore di resistenza (espresso in Ω) poco prima della stimolazione della ionomicina e la post-stimolazione (PoS) indica la resistenza (espressa in Ω) 2 minuti dopo aver terminato la stimolazione.
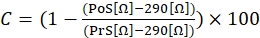 (1)
(1)
NOTA: Come mostrato nell'equazione (1), il valore di impedenza di un pozzo riempito con terreno di coltura senza cellule attaccate (valore di 290 Ω) viene sottratto da tutti i risultati nel calcolo finale. Si raccomanda di misurare le risposte contrattili in triplice copia in ogni esperimento ed eseguire tre esperimenti indipendenti con la stessa linea cellulare per tenere conto di qualsiasi variazione inter- e intra-sperimentale. - Una volta eseguiti i tre esperimenti indipendenti, raggruppare i dati calcolando la media delle tre misurazioni, inclusa la deviazione standard (SD).
- Per calcolare la risposta contrattile, utilizzare la seguente equazione (1), dove C indica la contrazione (espressa in % della variazione rispetto al basale), la prestimulazione (PrS) indica il valore di resistenza (espresso in Ω) poco prima della stimolazione della ionomicina e la post-stimolazione (PoS) indica la resistenza (espressa in Ω) 2 minuti dopo aver terminato la stimolazione.
- Confronto di diversi gruppi
- Per definire l'intervallo di contrazione normale e indagare le successive domande di ricerca specifiche, utilizzare il gruppo di controllo per definire la "contrazione normale" e confrontarla con la risposta contrattile dei pazienti o dei gruppi di trattamento.
- Calcolare la media e la SD dell'intero gruppo di controllo, cioè dei valori finali per tutte le linee cellulari, come descritto al punto 3.5.2.
- Utilizzare l'intervallo della media ± 2SD per identificare le cellule dell'altro gruppo che non rientrano in questo intervallo, indicando che hanno una risposta contrattile alterata.
NOTA: Il meccanismo alla base della risposta contrattile alterata è soggetto a domande di ricerca specifiche ed è dipendente dalle cellule e dalle condizioni e non sarà discusso in questo protocollo.
4. Rilevare la presenza di marcatori specifici SMC
- Isolamento dell'RNA
- Seminare le stesse linee cellulari specifiche del paziente utilizzate per le misurazioni di contrazione in piastre a 6 pozzetti, un pozzetto per linea cellulare. Contare le cellule utilizzando un contatore di celle automatizzato e seminare le cellule a una densità di semina di 200.000 cellule / pozzetto in mezzo SMC e consentire loro di attaccarsi durante la notte.
- Lavare le cellule una volta con PBS sterile.
- Lisare le cellule e isolare le celle secondo le istruzioni del produttore.
- Sintesi del cDNA
- Eseguire la sintesi del cDNA in 20 μL di una miscela di reazione a trascrizione inversa. Diluire le concentrazioni di RNA isolato totale secondo le istruzioni fornite dal produttore.
- qPCR
- Misurare l'espressione genica dei geni marcatori SMC, ad esempio ACTA2, CNN1, SMTN e TAGLN per confermare che le cellule isolate sono effettivamente SMC e rilevare i marcatori SMC. Verificare la correlazione dei risultati con l'output contrattile.
- Utilizzare almeno due geni di pulizia per normalizzare i dati, ad esempio YWHAZ e TBP.
NOTA: L'esecuzione e l'analisi qPCR possono essere eseguite utilizzando diverse macchine PCR e software compatibile, a seconda di ciò che è disponibile e ottimizzato in laboratorio. Vedere la tabella supplementare S1 per le sequenze di primer.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per testare la riproducibilità di questo metodo, il metodo è stato prima convalidato utilizzando solo SMC di controllo. Per determinare la riproducibilità della misurazione interesperimentale, due misurazioni indipendenti di tutte le linee cellulari di controllo e paziente incluse sono state tracciate come un grafico Bland-Altman (Figura 3B). Il grafico ha dimostrato che questo metodo non mostra variabilità al di fuori dell'intervallo di confidenza, ad eccezione di una linea cellulare an...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo documento presenta un metodo per misurare la contrazione SMC in vitro, basato sui cambiamenti nell'impedenza e nell'occupazione della superficie. In primo luogo, viene descritto l'isolamento, la coltura e l'espansione delle SMC umane primarie specifiche del paziente e dei fibroblasti cutanei, seguite da come usarle per le misurazioni della contrazione.
Una limitazione dello studio è legata all'ottenimento delle cellule attraverso un protocollo di espianto. Le cellule che prol...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Vorremmo ringraziare Tara van Merrienboer, Albert van Wijk, Jolanda van der Velden, Jan D. Blankensteijn, Lan Tran, Peter L. Hordijk, il team PAREL-AAA e tutti i chirurghi vascolari dell'ospedale UMC di Amsterdam, Zaans Medisch Centrum e Dijklander per aver fornito materiali e supporto per questo studio.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well Array | Applied Biophysics | 96W10idf PET | Array used to measure contraction in the ECIS setup |
| Custodiol | Dr. Franz Höhler Chemie GmbH | RVG 12801 | Solution used to transfer tissue in from surgery room to laboratorium |
| Dimethyl sulfoxide | Sigma-Aldrich | 472301 | Solution used to dilute ionomycin |
| Fetal Bovine Serum | Gibco | 26140079 | Addition to cell culture medium |
| Ham's F-10 Nutrient Mix | Gibco | 11550043 | Medium used to culture skin fibroblasts |
| Human Vascular Smooth Muscle Cell Basal Medium (formerly ''Medium 231'') | Gibco | M231500 | Medium used to culture smooth muscle cells |
| Invitrogen countess II | Thermo Fisher Scientific | AMQAX1000 | Automated cell counter |
| Ionomycin calcium salt from Streptomyces conglobatus | Sigma-Aldrich | I0634-1MG | Compound used for contraction stimulation |
| NaCl 0.9% | Fresenius Kabi | B230561 | Solution used to transfer tissue in from surgery room to laboratorium |
| Penicillin-Streptomycin | Gibco | 15140122 | Antibiotics used for cell culture medium |
| Phospathe buffered saline | Gibco | 10010023 | Used to wash cells |
| Quick-RNA Miniprep Kit | Zymo Research | R1055 | Kit used for RNA isolation |
| Smooth Muscle Growth Supplement (SMGS) | Gibco | S00725 | Supplement which is added to smooth muscle cell culture medium |
| SuperScript VILO cDNA Synthesis Kit | Thermo Fisher Scientific | 11754250 | Kit used for cDNA synthesis |
| SYBR Green PCR Master Mix | Thermo Fisher Scientific | 4309155 | Reagent for qPCR |
| Trypsin-EDTA | Gibco | 15400-054 | Used to trypsinize cells |
| ZTheta | Applied Biophysics | ZTheta | ECIS instrument used for contraction measurements |
Riferimenti
- Milewicz, D. M., et al. Genetic basis of thoracic aortic aneurysms and dissections: focus on smooth muscle cell contractile dysfunction. Annual Review of Genomics and Human Genetics. 9, 283-302 (2008).
- Milewicz, D. M., et al. Altered smooth muscle cell force generation as a driver of thoracic aortic aneurysms and dissections. Arteriosclerosis, Thrombosis, and Vascular Biology. 37 (1), 26-34 (2017).
- Groeneveld, M. E., et al. Betaglycan (TGFBR3) up-regulation correlates with increased TGF-β signaling in Marfan patient fibroblasts in vitro. Cardiovascular Pathology. 32, 44-49 (2018).
- Chen, J., Li, H., SundarRaj, N., Wang, J. H. C. Alpha-smooth muscle actin expression enhances cell traction force. Cell Motility and the Cytoskeleton. 64 (4), 248-257 (2007).
- Peyton, S. R., Putnam, A. J. Extracellular matrix rigidity governs smooth muscle cell motility in a biphasic fashion. Journal of Cellular Physiology. 204 (1), 198-209 (2005).
- Williams, D. A., Fogarty, K. E., Tsien, R. Y., Fay, F. S. Calcium gradients in single smooth muscle cells revealed by the digital imaging microscope using Fura-2. Nature. 318 (6046), 558-561 (1985).
- Wu, D., et al. NLRP3 (nucleotide oligomerization domain-like receptor family, pyrin domain containing 3)-caspase-1 inflammasome degrades contractile proteins: implications for aortic biomechanical dysfunction and aneurysm and dissection formation. Arteriosclerosis, Thrombosis, and Vascular Biology. 37 (4), 694-706 (2017).
- Bogunovic, N., et al. Impaired smooth muscle cell contractility as a novel concept of abdominal aortic aneurysm pathophysiology. Scientific Reports. 9 (1), 1-14 (2019).
- Hurst, V., Goldberg, P. L., Minnear, F. L., Heimark, R. L., Vincent, P. A. Rearrangement of adherens junctions by transforming growth factor-β1: role of contraction. American Journal of Physiology-Lung Cellular and Molecular Physiology. 276 (4), 582-595 (1999).
- Hu, N., et al. Comparison between ECIS and LAPS for establishing a cardiomyocyte-based biosensor. Sensors and Actuators B: Chemical. 185, 238-244 (2013).
- Peters, M. F., Lamore, S. D., Guo, L., Scott, C. W., Kolaja, K. L. Human stem cell-derived cardiomyocytes in cellular impedance assays: bringing cardiotoxicity screening to the front line. Cardiovascular Toxicology. 15 (2), 127-139 (2015).
- Zhang, S., Yang, Y., Kone, B. C., Allen, J. C., Kahn, A. M. Insulin-stimulated cyclic guanosine monophosphate inhibits vascular smooth muscle cell migration by inhibiting Ca/calmodulin-dependent protein kinase II. Circulation. 107 (11), 1539-1544 (2003).
- Halterman, J. A., Kwon, H. M., Zargham, R., Bortz, P. D. S., Wamhoff, B. R. Nuclear factor of activated T cells 5 regulates vascular smooth muscle cell phenotypic modulation. Arteriosclerosis, Thrombosis, and Vascular Biology. 31 (10), 2287-2296 (2011).
- Bass, H. M., Beard, R. S., Cha, B. J., Yuan, S. Y., Nelson, P. R. Thrombomodulin induces a quiescent phenotype and inhibits migration in vascular smooth muscle cells in vitro. Annals of Vascular Surgery. 30, 149-156 (2016).
- Burger, J., et al. Molecular phenotyping and functional assessment of smooth muscle like-cells with pathogenic variants in aneurysm genes ACTA2, MYH11, SMAD3 and FBN1. Human Molecular Genetics. , (2021).
- Yeung, K. K., et al. Transdifferentiation of human dermal fibroblasts to smooth muscle-like cells to study the effect of MYH11 and ACTA2 mutations in aortic aneurysms. Human Mutation. 38 (4), 439-450 (2017).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon