È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento scalabile e purificazione di vescicole extracellulari da Escherichia coli e altri batteri
In questo articolo
Riepilogo
I batteri secernono vescicole extracellulari (EV) di dimensioni nanometriche che trasportano molecole biologiche bioattive. La ricerca EV si concentra sulla comprensione della loro biogenesi, del ruolo nelle interazioni e malattie microbo-microbo e ospite-microbo, nonché sulle loro potenziali applicazioni terapeutiche. Viene presentato un flusso di lavoro per l'isolamento scalabile dei veicoli elettrici da vari batteri per facilitare la standardizzazione della ricerca sui veicoli elettrici.
Abstract
Diverse specie batteriche secernono ~ 20-300 nm vescicole extracellulari (EV), composte da lipidi, proteine, acidi nucleici, glicani e altre molecole derivate dalle cellule parentali. Le EV funzionano come vettori di comunicazione intra e interspecie, contribuendo anche all'interazione tra batteri e organismi ospiti nel contesto dell'infezione e della colonizzazione. Data la moltitudine di funzioni attribuite alle EV in salute e malattia, vi è un crescente interesse nell'isolamento delle EV per studi in vitro e in vivo . È stato ipotizzato che la separazione dei veicoli elettrici in base alle proprietà fisiche, vale a dire le dimensioni, faciliterebbe l'isolamento delle vescicole da diverse colture batteriche.
Il flusso di lavoro di isolamento consiste in centrifugazione, filtrazione, ultrafiltrazione e cromatografia ad esclusione dimensionale (SEC) per l'isolamento di EV da colture batteriche. È stata incorporata una fase di filtrazione a flusso tangenziale (TFF) azionata da una pompa per migliorare la scalabilità, consentendo l'isolamento del materiale da litri di coltura cellulare iniziale. Escherichia coli è stato utilizzato come sistema modello che esprime nanoluciferasi associata a EV e mCherry non associata a EV come proteine reporter. La nanoluciferasi è stata mirata alle EV fondendo il suo N-terminale con la citolisina A. Le prime frazioni cromatografiche contenenti EV 20-100 nm con citolisina A - nanoLuc associata erano distinte dalle frazioni successive contenenti le proteine libere. La presenza di nanoluciferasi associate a EV è stata confermata dalla marcatura immunogold e dalla microscopia elettronica a trasmissione. Questo flusso di lavoro di isolamento EV è applicabile ad altre specie batteriche gram-negative e gram-positive associate all'intestino umano. In conclusione, la combinazione di centrifugazione, filtrazione, ultrafiltrazione / TFF e SEC consente l'isolamento scalabile di EV da diverse specie batteriche. L'impiego di un flusso di lavoro di isolamento standardizzato faciliterà gli studi comparativi delle EV microbiche tra le specie.
Introduzione
Le vescicole extracellulari (EV) sono strutture simili a liposomi di dimensioni nanometriche composte da lipidi, proteine, glicani e acidi nucleici, secrete da cellule procariotiche ed eucariotiche1. Fin dai primi studi che visualizzavano il rilascio di EV da batteri gram-negativi2, il numero di funzioni biologiche attribuite alle EV batteriche (20-300 nm di diametro) è cresciuto costantemente negli ultimi decenni. Le loro funzioni includono il trasferimento della resistenza agli antibiotici3, la formazione di biofilm4, il quorum sensing5 e il rilascio di tossine6. C'è anche un crescente interesse per l'uso di EV batteriche come terapie, specialmente in vaccinologia7 e terapia del cancro8.
Nonostante il crescente interesse per la ricerca sui veicoli elettrici, ci sono ancora sfide tecniche per quanto riguarda i metodi di isolamento. In particolare, vi è la necessità di metodi di isolamento che siano riproducibili, scalabili e compatibili con diversi organismi produttori di veicoli elettrici. Per creare un insieme unificato di principi per la pianificazione e la segnalazione dei metodi di isolamento e ricerca dei veicoli elettrici, l'International Society for Extracellular Vesicles pubblica e aggiorna il documento di posizione MISEV9. Inoltre, il consorzio EV-TRACK fornisce una piattaforma aperta per la segnalazione di metodologie dettagliate per l'isolamento dei veicoli elettrici utilizzate nei manoscritti pubblicati per migliorare la trasparenza10.
In questo protocollo, le precedenti metodologie utilizzate per l'isolamento di EV da colture cellulari di mammifero sono state adattate11,12 per consentire l'isolamento di EV da colture cellulari batteriche. Abbiamo cercato di impiegare metodi che consentano l'isolamento dei veicoli elettrici da una varietà di microbi, che possono essere scalabili, e bilanciare la purezza e la resa dei veicoli elettrici (come discusso nel documento di posizione MISEV9). Dopo aver rimosso cellule batteriche e detriti mediante centrifugazione e filtrazione, il terreno di coltura viene concentrato mediante ultrafiltrazione del dispositivo centrifugo (per un volume fino a ~ 100 ml) o TFF azionato da pompa (per volumi maggiori). I veicoli elettrici vengono quindi isolati dalla SEC utilizzando colonne ottimizzate per la purificazione di piccoli veicoli elettrici.
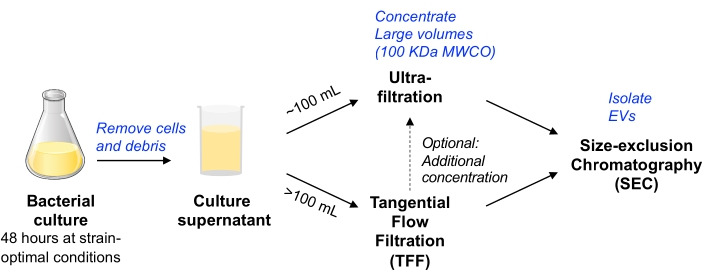
Figura 1: Panoramica schematica del flusso di lavoro di isolamento EV batterico. Abbreviazioni: EV = vescicola extracellulare; TFF = filtrazione tangenziale a flusso; SEC = cromatografia ad esclusione dimensionale; MWCO = cut-off del peso molecolare. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Un ceppo commensale di topo di Escherichia coli (cioè E. coli MP113) è stato utilizzato come organismo modello e modificato per esprimere nanoluciferasi associata a EV mediante fusione alla citolisina A, come precedentemente riportato14. I metodi utilizzati qui possono elaborare almeno fino a diversi litri di colture batteriche e separare efficacemente le proteine associate a EV da quelle non associate a EV. Infine, questo metodo può essere utilizzato anche per altre specie batteriche gram-positive e gram-negative. Tutti i dati rilevanti degli esperimenti riportati sono stati inviati alla knowledge base EV-TRACK (EV-TRACK ID: EV210211)10.
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Assicurarsi che tutto il lavoro che coinvolge batteri e DNA ricombinante segua le migliori pratiche per il contenimento della biosicurezza appropriate per il livello di pericolo per la biosicurezza di ciascun ceppo. Il lavoro dovrebbe essere svolto in conformità con le normative locali, nazionali e internazionali sulla biosicurezza.
1. Ceppi batterici e condizioni di coltura
NOTA: I ceppi batterici utilizzati in questo studio erano Escherichia coli MP113, Akkermansia mucinophila, Bacteroides thetaiotaomicron, Bifidobacterium breve e Bifidobacterium dentium.
- Per E. coli, utilizzare un ciclo sterile per inoculare singole colonie in 250-1.000 ml di brodo di Luria-Bertani (LB) e incubare aerobicamente in un incubatore di agitazione a 300 giri / min e 37 ° C per 48 ore prima di elaborare la coltura. Per il ceppo ricombinante di E. coli MP1 che ospita p114-mCherry-Clyluc (metodo supplementare e figura supplementare S1), aggiungere cloramfenicolo all'agar LB e al brodo ad una concentrazione finale di 17 μg/ml.
- Per A. mucinophila, B. thetaiotaomicron, B. breve e B. dentium, strisciare su piastre di agar per infusione cardiaca cerebrale (BHI) e incubare anaerobicamente all'interno di una camera anaerobica in vinile. Inoculare singole colonie in 100 ml di brodo BHI pre-ridotto e incubare per 48 ore anaerobicamente.
2. Isolamento EV
- Chiarificazione del terreno di coltura batterica mediante centrifugazione e filtrazione
- Trasferire le colture cellulari batteriche inoculate nella fase 1 per pulire i flaconi di centrifuga in polipropilene da 250 ml o 500 ml versando. Centrifugare le bottiglie in un rotore ad angolo fisso di grande capacità a 4 °C e 5.000 × g per 15 minuti. Trasferire il surnatante in bottiglie di centrifuga pulite versando accuratamente e centrifugare nuovamente a 10.000 × g per 15 minuti.
NOTA: Riutilizzare le bottiglie dopo la pulizia e la decontaminazione appropriate per la biosicurezza.- Se dopo la seconda centrifugazione sono presenti grandi pellet di cellule batteriche, ripetere la centrifugazione in una bottiglia pulita per rimuovere ulteriormente le cellule.
- Trasferire il surnatante in un dispositivo filtrante sottovuoto in polietersulfone da 0,22 μm di dimensioni appropriate mediante versamento. Filtrare collegando il dispositivo di filtrazione a un'alimentazione a parete sottovuoto. Se la velocità di filtrazione diminuisce in modo significativo, è sufficiente spostare qualsiasi materiale non filtrato su un nuovo dispositivo. Conservare il mezzo filtrato a 4 °C durante la notte e, se lo si desidera, continuare il protocollo il giorno successivo.
NOTA: Le centrifugazioni di cui sopra consentono in genere l'elaborazione di ~ 2 volte il volume indicato di coltura cellulare attraverso ciascun dispositivo. Ad esempio, un singolo dispositivo filtrante da 500 ml potrebbe filtrare ~ 1.000 ml di coltura precentrifugata. Questi dispositivi non vengono in genere riutilizzati. L'uso di filtri a siringa in questa fase non è raccomandato senza ottimizzazione, poiché sono state notate perdite significative con i modelli testati. Questo è un potenziale punto di arresto. - Verificare a questo punto la completa rimozione delle cellule vitali spargendo un'aliquota del surnatante filtrato su apposite piastre di agar e assicurarsi dell'assenza di colonie dopo l'incubazione in condizioni ottimali per il ceppo batterico. Se vengono rilevati batteri, ottimizzare ulteriormente la procedura di cui sopra eseguendo centrifugazioni e/o filtrazioni aggiuntive.
- Trasferire le colture cellulari batteriche inoculate nella fase 1 per pulire i flaconi di centrifuga in polipropilene da 250 ml o 500 ml versando. Centrifugare le bottiglie in un rotore ad angolo fisso di grande capacità a 4 °C e 5.000 × g per 15 minuti. Trasferire il surnatante in bottiglie di centrifuga pulite versando accuratamente e centrifugare nuovamente a 10.000 × g per 15 minuti.
- Concentrazione del mezzo filtrato
- Se si lavora con volumi significativamente >100 ml, procedere al punto 2.2.2. Se si lavora con volumi di ~100 ml, caricare 90 ml di terreno di coltura filtrato sul serbatoio di un dispositivo di ultrafiltrazione centrifuga MWCO (Molecular weight Cutoff) da 100 kDa di capacità rispettiva utilizzando pipette sierologiche. Bilanciare sempre con un dispositivo di ultrafiltrazione corrispondente e centrifugare nel rotore della benna oscillante a 4 °C e 2.000 × g per intervalli di 15-30 minuti, fino a quando il volume del fluido nel serbatoio superiore è stato concentrato a <0,5 ml.
- Riempire il serbatoio con qualsiasi terreno di coltura filtrato rimanente. In caso di "rabbocco", rimuovere il flusso nella parte inferiore del dispositivo e riequilibrare tutti i dispositivi.
NOTA: È stato osservato che il volume massimo di terreno di coltura filtrato che può essere concentrato utilizzando questi dispositivi è <2 volte il volume raccomandato. - Se la viscosità del mezzo concentrato nel serbatoio è visibilmente aumentata (materiale scuro e viscoso), diluire con soluzione salina tamponata fosfato (PBS) e riconcentrare mediante centrifugazione per diluire eventuali proteine non EV inferiori all'MWCO di 100 kDa.
NOTA: questo è un potenziale punto di arresto. - Trasferire il terreno concentrato in una provetta a basso legame proteico, conservare a 4 °C durante la notte e continuare il protocollo il giorno seguente, se lo si desidera.
- Riempire il serbatoio con qualsiasi terreno di coltura filtrato rimanente. In caso di "rabbocco", rimuovere il flusso nella parte inferiore del dispositivo e riequilibrare tutti i dispositivi.
- Se si lavora con volumi significativamente >100 ml, selezionare un dispositivo TFF di dimensioni appropriate (100 kDa MWCO) per ospitare il volume da elaborare.
NOTA: I dispositivi di filtrazione per la lavorazione da 100 ml a >1.000 ml sono disponibili in commercio. La disponibilità locale, i costi e la compatibilità con la pompa e i tubi/connessioni determineranno quali particolari modelli saranno più utili. Fino a 2 litri di terreno di coltura sono stati lavorati con il dispositivo indicato nella tabella dei materiali prima di dover pulire il filtro (vedere il passaggio 2.3 di seguito per il protocollo di pulizia).- Assemblare un circuito di filtrazione con tubi #16 a bassa legatura/bassa lisciviazione, adattatori da 1/8 di pollice a Luer, il dispositivo TFF e una pompa peristaltica, come indicato nella figura supplementare S2.
NOTA: Eseguire TFF all'interno di un armadio di biosicurezza per ridurre al minimo il rischio di contaminare la preparazione EV con batteri ambientali. - A temperatura ambiente, iniziare a far circolare il mezzo filtrato e condizionato a circa 200 ml/min (minimo 100 ml/min). Determinare il numero di giri appropriato corrispondente alla portata desiderata pompando 200 mL di PBS in un recipiente graduato. Quando si circola un mezzo filtrato e condizionato, raccogliere le molecole <100 kDa che attraversano la membrana di ultrafiltrazione come rifiuti in un recipiente separato.
NOTA: L'esempio seguente presuppone un volume iniziale di 2 L di cultura. - Continuare a far circolare il mezzo condizionato fino a quando il suo volume è stato ridotto a ~ 100-200 ml. Spostarsi su navi più piccole, se necessario. Diluire 2 volte con PBS e continuare a circolare con la pompa, concentrandosi fino a 75-100 ml. Diluire 2 volte con PBS e continuare a circolare fino a un volume finale di 25 ml. Diluire 2 volte con PBS e continuare a circolare fino a <10 ml.
- Sollevare il tubo di alimentazione dal serbatoio del campione e continuare a pompare per spurgare il filtro e recuperare la quantità massima di campione.
NOTA: questo è un potenziale punto di arresto. - Trasferire il campione concentrato in un tubo conico e conservare per una notte a 4 °C, se lo si desidera. In alternativa, continuare con il protocollo.
- Spostare il campione concentrato in un dispositivo di ultrafiltrazione centrifuga MWCO da 15 mL con capacità di 100 kDa. Centrifugare in un rotore a benna oscillante a 4 °C e 2.000 × g per intervalli di 15-30 minuti fino a quando il volume del fluido nel serbatoio superiore è stato concentrato a <2 ml.
NOTA: questo è un potenziale punto di arresto. - Trasferire il terreno concentrato in una provetta a basso legame proteico e conservare a 4 °C durante la notte, continuando il protocollo il giorno seguente, se lo si desidera.
- Assemblare un circuito di filtrazione con tubi #16 a bassa legatura/bassa lisciviazione, adattatori da 1/8 di pollice a Luer, il dispositivo TFF e una pompa peristaltica, come indicato nella figura supplementare S2.
- Se si lavora con volumi significativamente >100 ml, procedere al punto 2.2.2. Se si lavora con volumi di ~100 ml, caricare 90 ml di terreno di coltura filtrato sul serbatoio di un dispositivo di ultrafiltrazione centrifuga MWCO (Molecular weight Cutoff) da 100 kDa di capacità rispettiva utilizzando pipette sierologiche. Bilanciare sempre con un dispositivo di ultrafiltrazione corrispondente e centrifugare nel rotore della benna oscillante a 4 °C e 2.000 × g per intervalli di 15-30 minuti, fino a quando il volume del fluido nel serbatoio superiore è stato concentrato a <0,5 ml.
- Pulizia del dispositivo TFF (opzionale)
NOTA: la velocità di filtrazione diminuisce quando il dispositivo TFF inizia a "intasarsi" durante il processo (incrostazioni). Se necessario, il dispositivo filtrante può essere pulito per facilitare la filtrazione di campioni aggiuntivi nella stessa corsa di purificazione. Sebbene teoricamente possibile, un filtro TFF pulito non è stato utilizzato per un diverso ciclo di purificazione per evitare la contaminazione incrociata.- Per pulire, rimuovere tutti i tubi e i tappi dal dispositivo TFF e scaricare il liquido residuo.
- Utilizzare la pompa peristaltica e i tubi per inondare sia i compartimenti interni che quelli esterni del dispositivo TFF (cioè tramite le porte parallele e perpendicolari nel modello elencato nella tabella dei materiali) con ~ 100 ml di acqua distillata. Rimuovere tutti i tubi/tappi e scaricare il dispositivo TFF.
- Tappare le porte esterne (perpendicolari, filtrate) e far circolare 250 ml di etanolo al 20% in acqua distillata a >200 ml/min per 10 minuti attraverso il compartimento interno. Scolare, inondare con acqua distillata e scolare di nuovo come sopra.
- Far circolare 250 mL di soluzione fresca di NaOH 0,5 N per 30 minuti attraverso lo scomparto interno e scolare nuovamente.
- Ricollegare tutti i tubi e i tappi alle porte di ingresso, uscita e filtrato, come nella figura supplementare S2, e far circolare nuovamente la soluzione di NaOH 0,5 N fino a quando un volume di superficie del filtro > 1 mL/cm2 permea attraverso la membrana filtrante e viene raccolto come filtrato/rifiuto.
- Risciacquare il dispositivo TFF con acqua distillata come sopra. Utilizzare immediatamente il dispositivo TFF o inondare il dispositivo con ~ 100 ml di etanolo al 20% e conservare per una notte a 4 ° C.
NOTA: Se conservato in etanolo, assicurarsi di drenare, risciacquare con acqua, scolare e far circolare 250 ml di PBS attraverso il dispositivo fino a quando un volume di superficie del filtro di >1 ml / cm2 permea attraverso la membrana del filtro e viene raccolto come filtrato / rifiuto per rimuovere l'etanolo residuo prima della lavorazione del campione.
- Cromatografia ad esclusione dimensionale (SEC)
NOTA: SEC viene utilizzato per aumentare la purezza delle EV e rimuovere le proteine non vescicolari.- Utilizzare una piccola colonna SEC (10 mL di volume del letto) per l'isolamento dei veicoli elettrici da <100 mL di materiale di partenza e una colonna più grande (47 mL di volume del letto) per l'isolamento dei veicoli elettrici da >100 mL di materiale di partenza.
Nota : l'esempio seguente elencherà i volumi per la colonna più grande, con i volumi per la colonna più piccola tra parentesi. - Portare la colonna SEC e PBS a temperatura ambiente per diverse ore. Stabilizzare la colonna SEC in posizione verticale utilizzando un supporto da laboratorio standard e un supporto. In alternativa, utilizzare supporti per colonne per cromatografia commerciale.
- Prima di collegarlo alla colonna SEC, idratare il serbatoio del campione consentendo a 5 ml di PBS di fluire attraverso la fritta e in un contenitore per rifiuti. Svitare il tappo di ingresso della colonna SEC, aggiungere 2 mL di PBS al serbatoio del campione e collegare con attenzione il serbatoio alla colonna mentre il PBS gocciola attraverso la fritta (non applicabile per piccole colonne SEC).
NOTA: questo passaggio precedente impedisce che eventuali bolle d'aria rimangano intrappolate nella parte superiore della colonna SEC. Se l'aria è intrappolata, rimuovere il serbatoio, toccare la colonna per estrarre la bolla d'aria e ripetere la procedura di connessione. Per la colonna più piccola, è sufficiente aprire la parte superiore della colonna SEC e collegare la tramoggia del campione. - Aggiungere 47 mL (10 mL) di PBS al serbatoio del campione e aprire la parte inferiore della colonna SEC. Consentire a tutto il buffer di campionamento caricato di fluire attraverso la colonna per l'equilibrio. Eliminare il flusso in corso.
- Caricare un massimo di 2 mL (0,5 mL) di campione sul serbatoio del campione, eliminare il flusso e consentire al campione di entrare completamente nella colonna.
- Aggiungere immediatamente PBS al serbatoio del campione o alla tramoggia con un volume di 14,25 mL meno il volume del campione (3 mL meno il volume del campione, per la colonna piccola). Lasciare che la soluzione fluisca attraverso la colonna e scartare questa quantità uguale al volume vuoto della colonna.
NOTA: per un tipico campione da 2 ml, la quantità di PBS da aggiungere al serbatoio del campione o alla tramoggia sarà di 12,25 ml. - Posizionare un microtubo a basso legame da 2 mL direttamente sotto la colonna SEC. Aggiungere immediatamente 2 mL (0,5 mL) di PBS al serbatoio del campione e lasciarlo entrare nella colonna. Etichettare questi primi 2 mL (0,5 mL) di flusso in transito come Frazione 1. Continuare ad aggiungere 2 mL (0,5 mL) alla volta al serbatoio del campione per raccogliere ogni frazione successiva.
NOTA: La maggior parte dei veicoli elettrici batterici si eluisce nelle prime 5 frazioni. Durante l'ottimizzazione, sono state raccolte le prime 12 frazioni. - Conservare le frazioni a 4 °C per la conservazione a breve termine (giorni) o a -80 °C per la conservazione a lungo termine.
- Pulizia e stoccaggio delle colonne SEC riutilizzabili
NOTA: Le colonne SEC descritte in questo protocollo possono essere riutilizzate fino a 5 volte a seconda del produttore. Se la portata delle colonne SEC diminuisce dopo <5 utilizzi, il produttore consiglia di centrifugare i campioni concentrati a 10.000 x g per 10 minuti per eliminare eventuali aggregati prima di SEC. Quindi caricare il surnatante di questa centrifugazione sulla colonna SEC per l'isolamento EV.- Per pulire e conservare la colonna SEC dopo ogni utilizzo, aggiungere 2 mL (0,5 mL) di 0,5 M NaOH e lasciarla entrare completamente nella colonna. Far passare 100 mL (20 mL) di etanolo al 20% attraverso la colonna e conservarlo a 4 °C fino al successivo utilizzo. Prima dell'uso successivo, equilibrare l'etanolo a temperatura ambiente come sopra e scambiarlo con tampone PBS facendo scorrere altri 150 ml (30 ml) di PBS attraverso la colonna.
- Per pulire e riutilizzare immediatamente la colonna SEC dopo ogni utilizzo, aggiungere 2 mL (0,5 mL) di 0,5 M NaOH e lasciarla entrare completamente nella colonna. Eseguire circa 150 mL (30 mL) di tampone PBS per lavare via NaOH. Quando il pH dell'eluato è uguale a PBS (~7), può essere caricato un nuovo campione.
- Utilizzare una piccola colonna SEC (10 mL di volume del letto) per l'isolamento dei veicoli elettrici da <100 mL di materiale di partenza e una colonna più grande (47 mL di volume del letto) per l'isolamento dei veicoli elettrici da >100 mL di materiale di partenza.
3. Controllo di qualità della preparazione EV
- Test di sterilità
NOTA: Poiché questi veicoli elettrici provengono da colture batteriche, è fondamentale garantire la sterilità prima dell'uso a valle.- Ottenere 100 μL (20 μL) delle frazioni da utilizzare nei saggi e inoculare 3 mL del terreno utilizzato per far crescere i batteri di origine. Cultura nelle rispettive condizioni ottimali per almeno 3 giorni e osservare per torbidità. In alternativa, applicare i campioni di frazione a piastre di agar contenenti il mezzo utilizzato per far crescere i batteri produttori e cercare la formazione di colonie.
NOTA: se viene rilevata una contaminazione batterica, non è consigliabile utilizzare la preparazione EV per la sperimentazione. Invece, ripetere l'isolamento, avendo cura di ridurre al minimo il rischio di contaminazione batterica (a) eseguendo una sufficiente centrifugazione / filtrazione del mezzo di coltura cellulare batterica condizionato, (b) utilizzando bottiglie pulite, tubi, filtri e colonne cromatografiche e (c) impiegando tecniche asettiche appropriate.
- Ottenere 100 μL (20 μL) delle frazioni da utilizzare nei saggi e inoculare 3 mL del terreno utilizzato per far crescere i batteri di origine. Cultura nelle rispettive condizioni ottimali per almeno 3 giorni e osservare per torbidità. In alternativa, applicare i campioni di frazione a piastre di agar contenenti il mezzo utilizzato per far crescere i batteri produttori e cercare la formazione di colonie.
- Quantificazione delle proteine
NOTA: È stato utilizzato un kit di quantificazione delle proteine ad alta sensibilità basato sulla fluorescenza (vedere la tabella dei materiali). Il kit funziona con un fluorimetro proprietario corrispondente a lunghezze d'onda di eccitazione/emissione di 485/590 nm.- Portare tutti i reagenti, gli standard e i campioni a temperatura ambiente.
- Preparare una miscela principale di reagente proteico e tampone aggiungendo 1 μL di reagente a 199 μL di tampone per ciascun campione e standard da analizzare. Utilizzando provette PCR da 0,5 mL a parete sottile, aggiungere 10 μL standard + 190 μL di master mix a ciascuna provetta standard.
NOTA: Per rientrare nell'intervallo del test, la quantità di ciascuna frazione da aggiungere a ciascuna provetta dipende dalla resa proteica attesa della purificazione. Tipicamente, sono stati utilizzati 5 μL di ciascuna frazione + 195 μL di master mix. Il volume finale di campione + master mix deve essere di 200 μL. - Vortice le provette di analisi e incubare per almeno 15 minuti a temperatura ambiente al buio.
- Misurare gli standard sul fluorimetro proprietario appropriato (vedere la tabella dei materiali) selezionando l'opzione Saggio delle proteine utilizzando i pulsanti freccia e premendo il pulsante GO per confermare. Seguire le istruzioni sullo schermo, inserendo ogni tubo standard e premendo GO.
- Inserire la provetta sperimentale; premere GO per leggere; e notare il risultato visualizzato, che è la concentrazione proteica effettiva nella miscela tampone/campione del test. Per ottenere la concentrazione proteica nel campione, utilizzare i tasti freccia per selezionare l'opzione Calcola concentrazione campione, premere GO e utilizzare i tasti freccia per selezionare il volume del campione aggiunto al buffer di analisi per il campione specificato. Premere GO e registrare la concentrazione proteica del campione. Ripetere questo passaggio per ogni campione da analizzare.
- Conteggio delle particelle e distribuzione granulometrica
NOTA: il rilevamento di impulsi resistivi microfluidici (MRPS) è stato utilizzato per quantificare la concentrazione di EV e la distribuzione delle dimensioni.- Diluire i campioni in PBS integrato con 1% Tween-20 che è stato filtrato attraverso un filtro a siringa da 0,02 μm a una concentrazione proteica di circa 0,1 μg / ml.
NOTA: L'obiettivo della diluizione è raggiungere una concentrazione di particelle attesa nell'intervallo di 1010 particelle/ml nelle frazioni contenenti EV. Potrebbe essere necessario determinare empiricamente la diluizione ottimale. Pochi EV sono attesi per le frazioni successive (oltre la frazione 6). Pertanto, la concentrazione di particelle sarà probabilmente <1010 particelle / ml nonostante l'analisi a basse diluizioni. - Caricare 3 μL di ciascun campione nella cartuccia microfluidica monouso con una micropipetta, inserire la cartuccia nello strumento MRPS e premere il pulsante metallico con un bordo illuminato blu.
- Fare clic su Vai! sul software di acquisizione e attendere che il campione venga analizzato dallo strumento. Acquisire da 1.000 a 10.000 eventi di particelle per ridurre al minimo l'errore statistico tecnico dell'analisi. A questo punto, fare clic su Interrompi e termina esecuzione per completare l'acquisizione di esempio.
NOTA: Insieme ai file di dati grezzi, lo strumento produce un foglio di calcolo riassuntivo che elenca la concentrazione di particelle nel campione. Correggere questo valore in base alla diluizione del campione effettuata. - Utilizzando un software di analisi, caricare i dati grezzi e generare grafici personalizzati della distribuzione delle dimensioni.
- Diluire i campioni in PBS integrato con 1% Tween-20 che è stato filtrato attraverso un filtro a siringa da 0,02 μm a una concentrazione proteica di circa 0,1 μg / ml.
4. Stoccaggio EV
- Aliquote singole o aggregate al 25-50% della dimensione della singola frazione (a seconda delle dimensioni della colonna utilizzata) in provette a basso legame proteico e conservarle a -80 °C per evitare cicli di congelamento-disgelo.
NOTA: applicazioni diverse possono richiedere aliquote più piccole o più grandi a seconda della quantità prevista utilizzata in ciascun esperimento. Questo dovrà essere determinato empiricamente. Le frazioni non contenenti EV possono essere scartate se non applicabili agli obiettivi di ricerca.
5. Microscopia elettronica a trasmissione
- Colorazione negativa
- Aggiungere 5 μL del campione EV alla griglia a 400 maglie di rame rivestita di carbonio e incubare a temperatura ambiente per 10 minuti. Lavare il lato del campione con 5 gocce di tampone Tris da 5 mM (pH 7,1) e poi con 5 gocce di acqua distillata.
- Colorare il campione lato con 5 gocce di acetato di uranile al 2%. Asciugare qualsiasi quantità extra di macchia con carta da filtro e lasciare asciugare completamente la griglia per diverse ore o durante la notte. Visualizza i campioni con un microscopio elettronico funzionante a 80 kV.
- Etichettatura Immunogold
- Applicare 10 μL della sospensione EV su una griglia a maglia formvar/carbonio 400 e incubare a temperatura ambiente per 1 ora. Lavare la griglia in PBS tre volte, quindi applicare il 4% di paraformaldeide per 10 minuti per fissare il campione. Lavare le griglie cinque volte con PBS.
- Bloccare la griglia con tre lavaggi di PBS contenenti lo 0,1% di albumina sierica bovina (BSA). Quindi, applicare 10 μL di un anticorpo primario per 40 minuti a temperatura ambiente (qui, 1 μg / ml di anticorpo nluc). Lavare tre volte di nuovo con PBS contenente 0,1% BSA.
- Aggiungere 10 μL di anticorpo secondario marcato con oro alla griglia e incubare per 40 minuti a temperatura ambiente. Lavare le griglie tre volte con PBS.
NOTA: Qui, un anticorpo anti-topo di capra coniugato con nanoparticelle d'oro a 10 nm è stato utilizzato dopo aver diluito 1:10 nel tampone di blocco. Se l'etichettatura dell'oro oscura la visualizzazione EV, è possibile utilizzare anticorpi secondari con nanoparticelle d'oro più piccole (ad esempio 5 nm). - Post-fissare la griglia con 10 μL di glutaraldeide al 2,5% per 10 minuti a temperatura ambiente. Lavare tre volte in PBS. Eseguire la colorazione negativa con acetato di uranile al 2% (10 μL) per 15 minuti. Incorporare i campioni in 10 μL di acetato di uranile allo 0,5% e soluzione di metilcellulosa allo 0,13% per 10 minuti.
- Lasciare asciugare le griglie del campione durante la notte a temperatura ambiente prima di eseguire l'imaging al microscopio elettronico.
- Sul software di acquisizione del microscopio, determinare empiricamente l'esposizione per ottenere la qualità ottimale dell'immagine (ad esempio, 0,80851 s in questa particolare configurazione) e regolarla digitando questo valore nella casella di opzione del tempo di esposizione . Selezionare l'opzione 80 kV e fare clic su Avvia acquisizione per acquisire l'immagine.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per valutare quali frazioni cromatografiche SEC sono state arricchite per EV, la colonna SEC è stata caricata con 2 ml di terreno di coltura condizionato da E. coli MP1 che era stato concentrato 1.000 volte dal TFF e sono state raccolte frazioni sequenziali. Utilizzando MRPS, è stato riscontrato che le frazioni 1-6 contenevano il maggior numero di veicoli elettrici (Figura 2A). Le frazioni successive contenevano pochissime EV, comprendenti invece proteine prive di EV (
Access restricted. Please log in or start a trial to view this content.
Discussione
Nel protocollo di cui sopra, viene descritto un metodo scalabile e che isola in modo affidabile le EV da vari batteri gram-negativi / positivi e aerobici / anaerobici. Ha diversi potenziali punti di arresto durante la procedura, anche se è meglio evitare di impiegare più di 48 ore per isolare gli EV dai terreni di coltura batterica condizionati.
In primo luogo, consiste nel coltivare batteri per generare terreno di coltura batterica condizionato. È stato riscontrato che aumentare il tempo d...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
La ricerca sopra descritta è stata supportata dalla borsa di formazione NIH TL1 TR002549-03. Ringraziamo i dottori John C. Tilton e Zachary Troyer (Case Western Reserve University) per aver facilitato l'accesso allo strumento analizzatore di dimensioni delle particelle; Lew Brown (Spectradyne) per l'assistenza tecnica nell'analisi dei dati di distribuzione granulometrica; Dr. David Putnam presso la Cornell University per aver fornito pClyA-GFP plasmide14; e il Dr. Mark Goulian dell'Università della Pennsylvania per averci fornito l'E. coli MP113.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.5 mL flat cap, thin-walled PCR tubes | Thermo Scientific | 3430 | it is important to use thin-walled PCR tubes to obtain accurate readings with Qubit |
| 16% Paraformaldehyde (formaldehyde) aqueous solution | Electron microscopy sciences | 15700 | |
| 250 mL Fiberlite polypropylene centrifuge bottles | ThermoFisher | 010-1495 | |
| 500 mL Fiberlite polypropylene centrifuge bottles | ThermoFisher | 010-1493 | |
| 65 mm Polypropylene Round-Bottom/Conical Bottle Adapter | Beckman Coulter | 392077 | Allows Vivacell to fit in rotor |
| Akkermansia mucinophila | ATCC | BAA-835 | |
| Amicon-15 (100 kDa MWCO) | MilliporeSigma | UFC910024 | |
| Avanti J-20 XPI centrifuge | Beckman Coulter | No longer sold by Beckman. Avanti J-26XP is closest contemporary model. | |
| Bacteroides thetaiotaomicron VPI 5482 | ATCC | 29148 | |
| Bifidobacterium breve | NCIMB | B8807 | |
| Bifidobacterium dentium | ATCC | 27678 | |
| Brain Heart infusion (BHI) broth | Himedia | M2101 | After autoclaving, Both BHI broth and agar were introduced into the anaerobic chamber, supplemented with Menadione (1 µg/L), hematin (1.2 µg/L), and L-Cysteine Hydrochloride (0.05%). They were then incubated for at least 24 h under anaerobic conditions before inoculation with the anaerobic bacterial strains. |
| C-300 microfluidics cartridge | Spectradyne | ||
| Chloramphenicol | MP Biomedicals | ICN19032105 | |
| Electron microscope | FEI company | Tecnai G2 SpiritBT | |
| Escherichia coli HST08 (Steller competent cells) | Takara | 636763 | |
| Escherichia coli MP1 | Dr. Mark Goulian (gift) | commensal bacteria derived from mouse gut | |
| Fiberlite 500 mL to 250 mL adapter | ThermoFisher | 010-0151-05 | used with Fiberlite rotor to enable 250 mL bottles to be used for smaller size of starting bacterial culture |
| Fiberlite fixed-angle centrifuge rotor | ThermoFisher | F12-6x500-LEX | fits 6 x 500 mL bottles |
| Formvar Carbon Film 400 Mesh, Copper | Electron microscopy sciences | FCF-400-CU | |
| Glutaraldehyde (EM-grade, 10% aqeous solution) | Electron microscopy sciences | 16100 | |
| Hematin | ChemCruz | 207729B | Stock solution was made in 0.2 M L-histidine solution as 1.2 mg/mL |
| Infinite M Nano+ Microplate reader | Tecan | This equibment was used to measure the mCherry fluorescence | |
| In-Fusion HD Cloning Plus | Takara | 638909 | For cloning of the PCR fragements into the PCR-lineraized vectors |
| JS-5.3 AllSpin Swinging-Bucket Rotor | Beckman Coulter | 368690 | |
| Lauria Bertani (LB) broth, Miller | Difco | 244620 | |
| L-Cysteine Hydrochloride | J.T. Baker | 2071-05 | It should be weighed and added directly to the autoclaved BHI media inside the anaerobic chamber |
| Masterflex Fitting, Polypropylene, Straight, Female Luer to Hose Barb Adapter, 1/8" ID; 25/PK | cole-parmer - special | HV-30800-08 | connection adapters for filtration tubing circuit |
| Masterflex Fitting, Polypropylene, Straight, Male Luer to Hose Barb Adapter, 1/8" ID; 25/PK | cole-parmer - special | HV-30800-24 | connection adapters for filtration tubing circuit |
| Masterflex L/S Analog Variable-Speed Console Drive, 20 to 600 rpm | Masterflex | HV-07555-00 | |
| Masterflex L/S Easy-Load Head for Precision Tubing, 4-Roller, PARA Housing, SS Rotor | Masterflex | EW-07514-10 | |
| Masterflex L/S Precision Pump Tubing, PharmaPure, L/S 16; 25 ft | Cole Palmer | EW-06435-16 | low-binding/low-leaching tubing |
| Menadione (Vitamin K3) | MP | 102259 | Stock solution was made in ethanol as 1 mg/mL |
| MIDIKROS 41.5CM 100K MPES 0.5MM FLL X FLL 1/PK | Repligen | D04-E100-05-N | TFF device we have used to filter up to 2 L of E. coli culture supernatant |
| Nano-Glo Luciferase Assay System | Promega | N1110 | This assay kit was used to measure the luminescence of the nluc reporter protein |
| NanoLuc (Nluc) Luciferase Antibody, clone 965808 | R&D Systems | MAB10026 | |
| nCS1 microfluidics resistive pulse sensing instrument | Spectradyne | ||
| nCS1 Viewer | Spectradyne | Analysis software for particle size distribution | |
| OneTaq 2x Master Mix with Standard Buffer | NEB | M0482 | DNA polymerase master mix used to perform the routine PCR reactions for colony checking |
| Protein LoBind, 2.0 mL, PCR clean tubes | Eppendorf | 30108450 | |
| Q5 High-Fidelity 2x Master Mix | NEB | M0492 | DNA polymerase master mix used to perform the PCR reactions needed for cloning |
| qEV original, 35 nm | Izon | maximal loading volume of 0.5 mL | |
| qEV rack | Izon | for use with the qEV-original SEC columns | |
| qEV-2, 35 nm | Izon | maximal loading volume of 2 mL | |
| Qubit fluorometer | ThermoFisher | Item no longer available. Closest available product is Qubit 4.0 Fluorometer (cat. No. Q33238) | |
| Qubit protein assay kit | ThermoFisher | Q33211 | Store kit at room temperature. Standards are stored at 4 °C. |
| Sorvall Lynx 4000 centrifuge | ThermoFisher | 75006580 | |
| SpectraMax i3x Microplate reader | Molecular Devices | This equipment was used to measure the nanoluciferase bioluminescence | |
| Stericup Quick-release-GP Sterile Vacuum Filtration system (150, 250, or 500 mL) | MilliporeSigma | S2GPU01RE S2GPU02RE S2GPU05RE | One or multiple filters can be used to accommodate working volumes. In our experience, you can filter twice the volume listed on the product size. |
| Uranyl acetate | Electron microscopy sciences | 22400 | |
| Vinyl anaerobic chamber | Coy Lab | ||
| Vivacell 100, 100,000 MWCO PES | Sartorius | VC1042 | |
| Whatman Anotop 10 Plus syringe filters (0.02 micron) | MilliporeSigma | WHA68093002 | to filter MRPS diluent |
Riferimenti
- Yanez-Mo, M., et al. Biological properties of extracellular vesicles and their physiological functions. Journal of Extracellular Vesicles. 4, 27066(2015).
- Chatterjee, S. N., Das, J. Electron microscopic observations on the excretion of cell-wall material by Vibrio cholerae. Journal of General Microbiology. 49 (1), 1-11 (1967).
- Ciofu, O., Beveridge, T. J., Kadurugamuwa, J., Walther-Rasmussen, J., Hoiby, N. Chromosomal beta-lactamase is packaged into membrane vesicles and secreted from Pseudomonas aeruginosa. Journal of Antimicrobial Chemotherapy. 45 (1), 9-13 (2000).
- Yonezawa, H., et al. Outer membrane vesicles of Helicobacter pylori TK1402 are involved in biofilm formation. BMC Microbiology. 9, 197(2009).
- Mashburn, L. M., Whiteley, M. Membrane vesicles traffic signals and facilitate group activities in a prokaryote. Nature. 437 (7057), 422-425 (2005).
- Kato, S., Kowashi, Y., Demuth, D. R. Outer membrane-like vesicles secreted by Actinobacillus actinomycetemcomitans are enriched in leukotoxin. Microbial Pathogenesis. 32 (1), 1-13 (2002).
- Petousis-Harris, H., et al. Effectiveness of a group B outer membrane vesicle meningococcal vaccine against gonorrhoea in New Zealand: a retrospective case-control study. Lancet. 390 (10102), 1603-1610 (2017).
- Kim, O. Y., et al. Bacterial outer membrane vesicles suppress tumor by interferon-gamma-mediated antitumor response. Nature Communications. 8 (1), 626(2017).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750(2018).
- Consortium, E. -T., et al. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14 (3), 228-232 (2017).
- Watson, D. C., et al. Efficient production and enhanced tumor delivery of engineered extracellular vesicles. Biomaterials. 105, 195-205 (2016).
- Watson, D. C., et al. Scalable, cGMP-compatible purification of extracellular vesicles carrying bioactive human heterodimeric IL-15/lactadherin complexes. Journal of Extracellular Vesicles. 7 (1), 1442088(2018).
- Lasaro, M., et al. Escherichia coli isolate for studying colonization of the mouse intestine and its application to two-component signaling knockouts. Journal of Bacteriology. 196 (9), 1723-1732 (2014).
- Kim, J. Y., et al. Engineered bacterial outer membrane vesicles with enhanced functionality. Journal of Molecular Biology. 380 (1), 51-66 (2008).
- Beveridge, T. J. Structures of gram-negative cell walls and their derived membrane vesicles. Journal of Bacteriology. 181 (16), 4725-4733 (1999).
- Reimer, S. L., et al. Comparative analysis of outer membrane vesicle isolation methods with an Escherichia coli tolA mutant reveals a hypervesiculating phenotype with outer-inner membrane vesicle content. Frontiers in Microbiology. 12, 628801(2021).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon