È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutazione dell'ubiquitilazione del substrato mediante ubiquitina-ligasi E3 nei lisati a cellule di mammifero
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Forniamo un protocollo dettagliato per un test di ubiquitilazione di un substrato specifico e un'ubiquitina-ligasi E3 in cellule di mammifero. Le linee cellulari HEK293T sono state utilizzate per la sovraespressione proteica, il substrato poliubiquitilato è stato purificato dai lisati cellulari mediante immunoprecipitazione e risolto in SDS-PAGE. L'immunoblotting è stato utilizzato per visualizzare questa modifica post-traduzionale.
Abstract
L'ubiquitilazione è una modifica post-traduzionale che si verifica nelle cellule eucariotiche che è fondamentale per la regolazione di diversi percorsi biologici, tra cui la sopravvivenza cellulare, la proliferazione e la differenziazione. È un processo reversibile che consiste in un attaccamento covalente di ubiquitina al substrato attraverso una reazione a cascata di almeno tre diversi enzimi, composti da E1 (enzima di attivazione dell'ubiquitina), E2 (enzima coniugante dell'ubiquitina) ed E3 (enzima Ubiquitina-ligasi). Il complesso E3 svolge un ruolo importante nel riconoscimento del substrato e nell'ubiquitilazione. Qui, viene descritto un protocollo per valutare l'ubiquitilazione del substrato nelle cellule di mammifero utilizzando la co-trasfezione transitoria di un plasmide che codifica il substrato selezionato, una ubiquitina ligasi E3 e un'ubiquitina marcata. Prima della lisi, le cellule trasfettate vengono trattate con l'inibitore del proteasoma MG132 (carbobenzossi-leu-leu-leucinale) per evitare la degradazione proteasomiale del substrato. Inoltre, l'estratto cellulare viene sottoposto a immunoprecipitazione su piccola scala (IP) per purificare il substrato poliubiquitilato per la successiva rilevazione mediante western blotting (WB) utilizzando anticorpi specifici per il tag ubiquitina. Quindi, viene descritto un protocollo coerente e semplice per il test di ubiquitilazione nelle cellule di mammifero per aiutare gli scienziati ad affrontare l'ubiquitilazione di substrati specifici e le ligasi di ubiquitina E3.
Introduzione
Le modificazioni post-traduzionali (PTM) sono un meccanismo importante per quanto riguarda la regolazione delle proteine, che è essenziale per l'omeostasi cellulare. L'ubiquitilazione proteica è una modifica dinamica e intricata che crea un assortimento di segnali diversi con conseguenti diversi risultati cellulari negli organismi eucariotici. L'ubiquitilazione è un processo reversibile che consiste nell'attaccamento di una proteina di ubiquitina contenente 76 amminoacidi al substrato, che si verifica in una cascata enzimatica composta da tre reazioni distinte1. Il primo passo è caratterizzato dall'attivazione dell'ubiquitina, che dipende da un'idrolisi dell'ATP per formare un'ubiquitina legata al tioestere ad alta energia tra l'ubiquitina C-terminus e il residuo di cisteina presente nel sito attivo dell'enzima E1. Successivamente, l'ubiquitina viene trasferita all'enzima E2 formando un complesso simile al tioestere con l'ubiquitina. Successivamente, l'ubiquitina viene attaccata covalentemente al substrato dall'E2, o più spesso, dall'enzima E3, che riconosce e interagisce con il substrato 2,3. Occasionalmente, gli enzimi E4 (fattori di allungamento della catena dell'ubiquitina) sono necessari per promuovere l'assemblaggio della catena multiubiquitina3.
L'ubiquitina ha sette residui di lisina (K6, K11, K27, K29, K33, K48 e K63), consentendo la formazione di catene di poliubiquitina che generano legami distinti per produrre diverse strutture tridimensionali che saranno riconosciute da diverse proteine effettrici 4,5. Quindi, il tipo di catena di poliubiquitina introdotta nel substrato è essenziale per decidere il suo destino cellulare 6,7,8. Inoltre, il substrato potrebbe anche essere ubiquitinato attraverso i suoi residui N-terminali chiamati N-degrons. Specifiche ubiquitina-ligasi E3 sono responsabili del riconoscimento dell'N-degron, consentendo la poliubiquitilazione del vicino residuo di lisina9.
Al giorno d'oggi, ci sono più di 40 diversi substrati specifici SCF caratterizzati. Tra questi, i regolatori chiave di diversi percorsi biologici, tra cui la differenziazione e lo sviluppo cellulare, nonché la sopravvivenza e la morte cellulare, possono essere trovati 10,11,12,13. Pertanto, l'identificazione di substrati specifici di ciascuna ubiquitina-ligasi E3 è essenziale per progettare una mappa completa di vari eventi biologici. Anche se l'identificazione di veri substrati è biochimicamente impegnativa, l'uso di metodi basati sulla biochimica è molto adatto per valutare la specificità della catena e la distinzione tra mono- e poliubiquitilazione14. Questo studio descrive un protocollo completo per il test di ubiquitilazione utilizzando la linea cellulare di mammiferi HEK293T sovraesprimendo il substrato UXT-V2 (Ubiquitously expressed prefoldin-like chaperone isoform 2) con il complesso E3 ubiquitina-ligasi SCF (Fbxo7). UXT-V2 è un co-fattore essenziale per la segnalazione di NF-κB e, una volta che questa proteina viene abbattuta nelle cellule, inibisce l'attivazione NF-κB indotta da TNF-α11. Pertanto, per rilevare UXT-V2 poliubiquitilato, viene utilizzato l'inibitore del proteasoma MG132 poiché ha la capacità di bloccare l'attività proteolitica della subunità 26S del complesso proteasoma15. Inoltre, l'estratto cellulare viene sottoposto a un IP su piccola scala per purificare il substrato, utilizzando un anticorpo specifico immobilizzato alla resina di agarosio per la successiva rilevazione da parte di WB utilizzando anticorpi selezionati. Questo protocollo è molto utile per convalidare l'ubiquitilazione del substrato nell'ambiente cellulare e può anche essere adattato per diversi tipi di cellule di mammifero e altri complessi E3 ubiquitina-ligasi. Tuttavia, è necessario convalidare il substrato testato anche attraverso un test di ubiquitilazione in vitro, poiché entrambi i protocolli si completano a vicenda per quanto riguarda l'identificazione di substrati reali.
Protocollo
NOTA: Una panoramica del protocollo di analisi dell'ubiquitilazione nelle cellule di mammifero è rappresentata nella Figura 1.
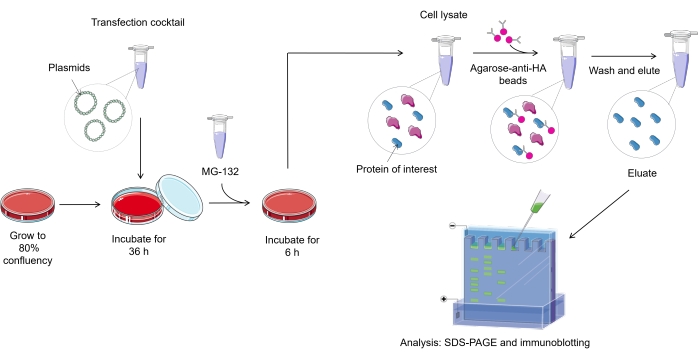
Figura 1. Panoramica della procedura di analisi dell'ubiquitilazione. Fare clic qui per visualizzare una versione più grande di questa figura.
1. Coltura cellulare
- Coltiva la linea cellulare HEK293T in un piatto di coltura trattato tc da 100 mm fino all'80% -90% di confluenza nei mezzi di crescita (Dulbecco's Modified Eagle's Medium (DMEM) ad alto glucosio integrato con il 10% di siero bovino fetale (FBS) e penicillina (100 unità), streptomicina (100 μg) e L-glutammina (0,292 mg / mL)). Incubare la coltura a 37 °C in un incubatore di colture cellulari umidificate al 5% di CO2.
- Per passare le cellule, aspirare il mezzo dal piatto di coltura usando una pipetta sierologica. Lavare le cellule una volta con 1 mL di soluzione salina sterilizzata 1x tamponata con fosfato (PBS 1x).
- Staccare le cellule aggiungendo 1 mL di soluzione di tripsina/EDTA (acido etilendiamminotetraacetico). Incubare il piatto a 37 °C per 5 min. Risospesso le cellule utilizzando una pipetta sierologica in 2 ml di terreno di crescita.
- Trasferire la sospensione cellulare in un tubo fresco e pulito da 15 ml e centrifugare a 500 x g per 5 minuti a temperatura ambiente (RT). Rimuovere il surnatante versandolo con cura. Risospesciare delicatamente il pellet cellulare in 3 ml di terreno di crescita pipettando su e giù per ottenere una sospensione cellulare omogenea.
- Trasferire 1 mL della sospensione cellulare in una capsula di coltura da 100 mm trattata con TC contenente 9 mL di terreno di crescita.
NOTA: Se le cellule sono in buone condizioni, ogni piatto di coltura di HEK293T, in cui la confluenza è dell'80% -90%, può generare tre piatti di coltura con confluenza dell'80% 2 giorni dopo il passaggio.
2. Trasfezione cellulare
NOTA: Non è consigliabile trasfettare la coltura cellulare se la confluenza raggiunta è inferiore all'80%.
- Prima della trasfezione, certificare se le cellule sono esenti da contaminazione e ad una confluenza adeguata per le trasfezioni transitorie.
- Per ogni campione di trasfezione, preparare i complessi DNA-Polietilenimina (PEI 1 μg/μL a pH 7,2) come segue:
- Diluire 3 μg di ciascun plasmide in 100 μL di opti-MEM I mezzo sierico ridotto senza integrazione e mescolarlo delicatamente pipettando la soluzione su e giù.
NOTA: Qui, le cellule sono state trasfettate con 4 μg di ciascun plasmide: vettori vuoti (pcDNA3) o costrutti FLAG-Fbxo7 e UXT-V2-HA, con o senza 6xHis-myc-ubiquitina. Il contenuto totale di DNA era di 12 μg. - Scongelare il PEI a RT e aggiungerlo nella soluzione seguendo una proporzione di 3 μL di PEI per 1 μg di DNA. Omogeneizzare la soluzione tubando su e giù. Quindi incubarlo per 15 minuti a RT per consentire la formazione di complessi DNA-PEI.
NOTA: La proporzione ottimale di volume di PEI per quantità di DNA varia a seconda della linea cellulare selezionata.
- Diluire 3 μg di ciascun plasmide in 100 μL di opti-MEM I mezzo sierico ridotto senza integrazione e mescolarlo delicatamente pipettando la soluzione su e giù.
- Aggiungere il volume totale dei complessi DNA-PEI a ciascun piatto contenente la coltura cellulare e mescolarlo delicatamente facendo oscillare la piastra avanti e indietro. Incubare le cellule a 37 °C in un incubatore di colture cellulari umidificate al 5% di CO2.
- Sostituire il mezzo di crescita dopo 5 ore, poiché l'esposizione prolungata al PEI può essere tossica per le cellule HEK293T. Incubare le cellule a 37 °C in un incubatore di colture cellulari umidificate al 5% di CO2 per 36 ore.
3. Lisi cellulare e immunoprecipitazione
- Dopo il periodo di incubazione e 6 ore prima della lisi cellulare, trattare le cellule trasfettate con 10 μM dell'inibitore del proteasoma MG-132. Ancora una volta, incubare le cellule a 37 °C in un incubatore di colture cellulari umidificate al 5% di CO2.
- Aspirare il materiale da ogni piatto di coltura usando una pipetta sierologica e lavarlo una volta con 1 ml di 1x PBS. Staccare le cellule aggiungendo 1 mL di tripsina e incubando il piatto a 37 °C per 5 min. Risospendare le cellule in 1 mL di mezzi di crescita.
- Trasferire la sospensione cellulare in un tubo da 15 ml fresco e pulito e centrifugarla a 500 x g per 5 minuti a RT.
- Rimuovere il surnatante versandolo con cura. Risospenare delicatamente il pellet cellulare in 200 μL di tampone di lisi NP-40 ghiacciato (50 mM Tris-HCl pH 7,2, 225 mM KCl e 1% NP-40), integrato con il cocktail inibitori della proteasi e della fosfatasi (10 mM NaF e 1 mM Na3VO4) e trasferire la soluzione in un microtubo pulito da 1,5 mL.
- Incubare il lisato cellulare per 30 minuti sul ghiaccio. Dopo l'incubazione, centrifugare i lisati cellulari a 16.900 x g per 20 minuti a 4 °C.
- Nel frattempo, equilibrare le perle di agarosio-anti-HA con un tampone di lisi NP-40 ghiacciato. Utilizzare 15 μL di perline di agarosio-anti-HA per ogni campione. Lavare le perline con 200 μL di tampone di lisi NP-40 pulsandolo in un tubo microcentrifuga a 3.000 x g per 1 minuto a 4 °C. Con una pipetta, aspirare e scartare il surnatante con molta attenzione; ripetere questo processo tre volte. Successivamente, mantenere le perline equilibrate sul ghiaccio fino all'uso.
- Dopo aver centrifugato i lisati cellulari, recuperare il surnatante. Quantificare il contenuto proteico nel lisato totale utilizzando il metodo Bradford16.
- Assicurarsi che ogni campione sottoposto a immunoprecipitazione presenti una quantità uguale di proteine. Incubare il volume necessario di lisato cellulare con le perle equilibrate di agarosio-anti-HA per 4 ore, ruotando delicatamente in un incubatore rotante a 4 °C, che consente all'UXT-V2-HA di legarsi alle perle di agarosio-anti-HA.
- Raccogliere le perle di agarosio-anti-HA pulsandole in un tubo microcentrifuga a 3.000 x g per 1 minuto a 4 °C. Aspirare con cura ed scartare il surnatante. Lavare le perline tre volte con tampone di lisi cellulare NP-40 ghiacciato e due volte con tampone FLAG/HA ghiacciato (10 mM Hepes pH 7,9, 15 mM MgCl2, 225 mM KCl e 0,1% NP-40).
- Dopo il lavaggio finale, rimuovere accuratamente tutto il surnatante utilizzando una pipetta ed eluire la proteina poliubiquitilata con peptide HA (300 μg/mL) diluito su tampone FLAG/HA. Incubare le perle di agarosio-anti-HA con peptide HA per 1 ora a 4 °C in una piattaforma shaker a dondolo.
- Abbassare le perline a 3.000 x g per 2 minuti a 4 °C e pipettare con cura il surnatante contenente le proteine poliubiquitinate. Se necessario, conservare l'eluato in un microtubo fresco e pulito a -20 °C.
- Risolvere gli eluati e i lisati cellulari in 10% SDS-PAGE (elettroforesi su gel di sodio dodecilsolfato-poliacrilammide)17 e immunoblotting.
- In questo studio, è stato eseguito un WB a trasferimento umido. Per questo tipo di trasferimento, posizionare il gel in un sandwich di trasferimento composto da carta da filtro-gel-membrana-carta da filtro, ammortizzarlo con pastiglie e premerlo insieme da una griglia di supporto. Posizionare questo sistema verticalmente in un serbatoio pieno di buffer di trasferimento e tra elettrodi a filo di acciaio inossidabile / platino. Il trasferimento avviene per 90 min a 150 V in tampone di trasferimento umido (glicina 192 mM, tris-base 25 mM, 0,025% SDS, 20% metanolo).
NOTA: Poiché gli estratti cellulari sono stati quantificati (fase 3.7), eseguire una SDS-PAGE con una quantità uguale di proteine per ciascun campione. Inoltre, per risolvere l'eluato, eseguire volumi uguali di ciascun campione. - Sondare la membrana immunoblot utilizzando anticorpi selezionati 11. Assicurarsi che venga rilevato un segnale di striscio nell'eluato dal substrato poliubiquitilato estratto nel processo IP utilizzando anticorpi anti-myc nei campioni contenenti la legasi E3 wild-type, il substrato e myc-ub. Nel lisato cellulare (input), assicurarsi che venga rilevato il segnale proveniente dal substrato scelto, la proteina Fbxo7, le proteine ubiquitilate e una proteina di pulizia (ad esempio, GAPDH e β-actina) per garantire la stessa quantità di proteine in ogni corsia.
NOTA: La diluizione per ciascun anticorpo utilizzato è stata preparata secondo le istruzioni del produttore.
Risultati
UXT (trascrizione ubiquitariamente espressa) è una proteina simile alla prefoldina che forma complessi di ripiegamento proteico ubiquitariamente espressi nei tessuti di topo e umani come cuore, cervello, muscolo scheletrico, placenta, pancreas, rene e fegato18. Due isoforme di giunzione di UXT, che sono denominate UXT-V1 e UXT-V2, sono state descritte mentre svolgono funzioni distinte e posizioni subcellulari. UXT-V1 è prevalentemente localizzato nel citoplasma e all'interno dei mitocondri, ed ?...
Discussione
L'ubiquitilazione è una modificazione post-traduzionale essenziale che regola i livelli di diverse proteine e svolge un ruolo cruciale in molte vie di segnalazione e processi biologici, garantendo un ambiente intracellulare sano. Il sistema ubiquitina-proteasoma (UPS) è uno dei principali focus della recente ricerca farmaceutica, fornendo la possibilità di stabilizzare i soppressori tumorali o indurre la degradazione dei prodotti oncogeni22. Ad esempio, la proliferazione aberrante delle neoplas...
Divulgazioni
Gli autori dichiarano che non vi è alcun conflitto di interessi.
Riconoscimenti
F.R.T è supportato dal numero di sovvenzione FAPESP 2020/15771-6 e CNPq Universal 405836/2018-0. P.M.S.P e V.S sono supportati da CAPES. C.R.S.T.B.C è stato supportato dalla borsa di studio FAPESP numero 2019/23466-1. Ringraziamo Sandra R. C. Maruyama (FAPESP 2016/20258-0) per il supporto materiale.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
Riferimenti
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon