È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Riattivazione di modelli cellulari demembranati in Chlamydomonas reinhardtii
In questo articolo
Riepilogo
La riattivazione in vitro delle cellule mobili è un esperimento cruciale per comprendere i meccanismi della motilità cellulare. Il protocollo descrive la riattivazione dei modelli cellulari demembranati di Chlamydomonas reinhardtii, un organismo modello per lo studio delle ciglia/flagelli.
Abstract
Dall'esperimento storico sulla contrazione del muscolo glicerinato con l'aggiunta di ATP, che Szent-Györgyi ha dimostrato a metà del 20 ° secolo, la riattivazione in vitro delle cellule demembranate è stata un modo tradizionale e potente per esaminare la motilità cellulare. Il vantaggio fondamentale di questo metodo sperimentale è che la composizione della soluzione di riattivazione può essere facilmente modificata. Ad esempio, un ambiente ad alta concentrazione di Ca2+ che si verifica solo temporaneamente a causa dell'eccitazione della membrana in vivo può essere replicato in laboratorio. Le ciglia eucariotiche (note anche come flagelli) sono elaborati meccanismi di motilità i cui meccanismi di regolazione devono ancora essere chiariti. L'alga verde unicellulare Chlamydomonas reinhardtii è un eccellente organismo modello nel campo della ricerca delle ciglia. Gli esperimenti di riattivazione utilizzando modelli cellulari demembranati di C. reinhardtii e loro derivati, come gli assonemi demembranati di ciglia isolate, hanno contribuito in modo significativo alla comprensione dei meccanismi molecolari della motilità ciliare. Questi esperimenti hanno chiarito che l'ATP energizza la motilità ciliare e che vari segnali cellulari, tra cui Ca2 +, cAMP e specie reattive dell'ossigeno, modulano i movimenti ciliari. Il metodo preciso per la demembranazione delle cellule di C. reinhardtii e la riattivazione dei modelli cellulari è descritto qui.
Introduzione
La riattivazione in vitro di cellule mobili demembranate è un valido strumento per lo studio delle basi molecolari del meccanismo regolatore della motilità cellulare. Szent-Györgyi ha dimostrato per la prima volta la contrazione in vitro delle fibre muscolari scheletriche di coniglio estratte con il 50% di glicerolo aggiungendo adenosina trifosfato (ATP)1. Questo esperimento è stato il primo a dimostrare che l'ATP energizza la contrazione muscolare. La metodologia è stata presto applicata allo studio della motilità ciliare/flagellare energizzata dall'ATP, come i flagelli2 degli spermatozoi, le ciglia paramecium 3 e le ciglia di Chlamydomonas reinhardtii (chiamate anche flagelli)4 utilizzando detergenti non ionici per la demembranazione.
L'alga verde unicellulare C. reinhardtii è un organismo modello per lo studio delle ciglia: nuota con due ciglia battendole come la rana di un essere umano5. Il movimento ciliare è guidato dalla dineina, una proteina motoriaa base di microtubuli diretti all'estremità inferiore 6,7. Le dineine ciliari possono essere classificate in dineine del braccio esterno e dineine del braccio interno. I mutanti privi di ogni tipo di dineina sono stati isolati come mutanti a nuoto lento con diverse anomalie della motilità. L'analisi dettagliata della motilità in vitro di questi mutanti ha fatto progredire significativamente la ricerca sulla dineina8.
Molti risultati importanti sono stati raggiunti utilizzando questo metodo e i suoi derivati da quando è stato stabilito l'esperimento di riattivazione in vitro di cellule demembranate di C. reinhardtii (modelli cellulari). La riattivazione di modelli cellulari in una serie di tamponi Ca2+, ad esempio, ha mostrato9 che due ciglia sono regolate in modo diverso da Ca2+ submicromolare, e questo controllo asimmetrico delle ciglia consente l'orientamento fototattico di C. reinhardtii10. Inoltre, entrambe le ciglia mostrano la conversione della forma d'onda dalla modalità di nuoto in avanti (chiamata forma d'onda asimmetrica) alla modalità di nuoto all'indietro (chiamata forma d'onda simmetrica che appare per un breve periodo quando le cellule sono foto- o meccano-shockate)11,12. Questa conversione della forma d'onda è regolata dal Ca2+ submillimolare, che è stato dimostrato dalla riattivazione del cosiddetto apparato nucleoflagellare (un complesso contenente due ciglia, i corpi basali, le strutture che collegano i corpi basali con il nucleo e il residuo del nucleo)11 o assonomi demembranati di ciglia isolate13. Oltre a Ca2+, l'equilibrio redox (riduzione-ossidazione) è un segnale che regola la frequenza di battito ciliare, che è stata dimostrata dalla riattivazione di modelli cellulari in tamponi redox contenenti diversi rapporti di glutatione ridotto vs glutatione ossidato14. Inoltre, l'adenosina monofosfato ciclico (cAMP) regola asimmetricamente due ciglia, come dimostrato dalla riattivazione degli assonemi con cAMP15 in gabbia fotoccolarivibile. Questi risultati in vitro, combinati con i risultati genetici, hanno portato a una comprensione più profonda dei meccanismi molecolari della regolazione delle ciglia in C. reinhardtii.
Un protocollo per riattivare i modelli cellulari è descritto qui. Il metodo è semplice, consente varie modifiche e può essere applicato a più organismi che si muovono con le ciglia. Tuttavia, poiché le cellule demembranate sono fragili, sono necessari alcuni suggerimenti per riattivare la motilità dei modelli cellulari con una buona efficienza prevenendo la deciliazione.
Protocollo
Un ceppo wild-type di Chlamydomonas reinhardtii, CC-125, è stato utilizzato per il presente studio. CC-125 è stato ottenuto dal Chlamydomonas Resource Center (vedi Tabella dei materiali) e mantenuto su un mezzo tris-acetato-fosfato (TAP)16, 1,5% di agarosio a 20-25 °C.
1. Coltura cellulare
- Coltura Chlamydomonas reinhardtii (CC-125) in tap medio16 in un periodo luce-buio di 12 h/12 h (condizioni di luce per il periodo di luce: ~50 μmol fotoni m−2 s−1 luce bianca) a 20-25 °C per 2 giorni (Figura 1).
NOTA: le cellule devono trovarsi in una fase di crescita logaritmica media (Filmato 1). La coltura lunga (>4 giorni, nella crescita logaritmica tardiva o nella fase stazionaria) diminuisce l'efficienza di riattivazione dei modelli cellulari demembranati.

Figura 1: Coltura liquida dopo 2 giorni di coltivazione. Da una piastra di agar TAP-1,5%, un pezzo di cellule wild-type per riempire il loop di platino è stato inoculato a ~ 150 ml di mezzo liquido TAP in un pallone. La densità cellulare dopo coltura di 2 giorni è stata di 2,3 × 106 cellule/ml. Fare clic qui per visualizzare una versione più grande di questa figura.
Film 1: Nuoto di cellule vive. Le cellule sono state osservate al microscopio con una lente obiettivo 10x e un condensatore a campo scuro ad immersione in olio. Barra della scala = 100 μm. Fare clic qui per scaricare questo filmato.
2. Preparazione di modelli cellulari demembranati
NOTA: Prima di iniziare l'esperimento, tenere il tampone di lavaggio a temperatura ambiente e la demembranazione, la diluizione, i tamponi di riattivazione e la soluzione di ATP sul ghiaccio. La composizione di tali buffer è fornita nella tabella supplementare 1.
- Centrifugare ~10 mL di coltura liquida a 1000 × g a 20 °C per 3 min.
NOTA: in tutto il protocollo da questo passaggio, non utilizzare plastica autoclavata (tubi, punte di pipette, ecc.), Diminuendo l'efficienza di riattivazione. Per i tubi conici, quelli riutilizzati sono preferibili. La densità cellulare dopo coltura di 2 giorni può essere in genere 1,0-5,0 × 106 cellule / ml. Quando la densità cellulare è inferiore a questa, prendere abbastanza volume di coltura per contenere ~ 5 × 107 cellule. - Scartare il surnatante, prima per decantazione e poi con una pipetta Pasteur, e risospescere i precipitati in ~ 5 ml di tampone di lavaggio.
- Centrifugare le celle nel tampone di lavaggio a 1000 × g per 3 min a 20 °C.
- Scartare accuratamente il surnatante con una pipetta (Figura 2).
NOTA: le pipette Pasteur sono preferibili. Quando si utilizzano micropipette, evitare di utilizzare punte di pipette che sono state autoclavate. - Sovrapporre ~ 0,5 ml di tampone di demembranazione su un pellet cellulare, scuotere delicatamente il tubo a mano per sospendere approssimativamente le cellule nel tampone e mettere il tubo sul ghiaccio (Figura 3A).
NOTA: Non è necessario sospendere completamente il pellet in questo momento. Aumentare la concentrazione di MgSOda 4 a 15 mM nei tamponi di demembranazione e riattivazione quando i modelli cellulari vengono riattivati, con una concentrazione finale di ATP >1 mM per una riattivazione stabile17. - Sospendere delicatamente il pellet cellulare rimanente con una pipetta e posizionare nuovamente il tubo sul ghiaccio (Figura 3B).
- Prendi 5-10 μL dei modelli cellulari, diluisci 10 volte con il tampone di diluizione e osserva al microscopio (Film 2) per confermare che tutti i modelli cellulari sono demembranati e non nuotano (Figura 4).
NOTA: se alcune cellule stanno ancora nuotando (vive), aggiungere il detergente non ionico utilizzato nel tampone di demembranazione direttamente alla soluzione del modello cellulare alla concentrazione finale di ~ 0,15%. In alternativa, ripetere i passaggi 2.1-2.5 con la soluzione di demembranazione contenente lo 0,15% di detergente.
Figura 2: Scartare il surnatante. Il resto del surnatante è stato accuratamente rimosso con una pipetta Pasteur dopo che il surnatante è stato rimosso per decantazione del tubo. Fare clic qui per visualizzare una versione più grande di questa figura.
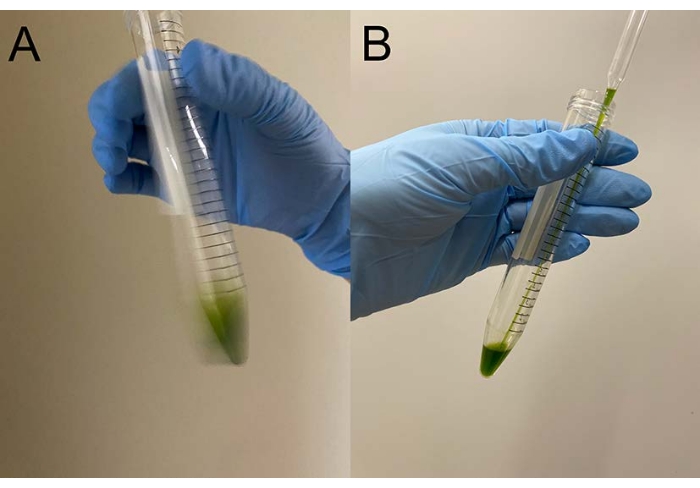
Figura 3: Demembranazione. (A) Dopo aver sovrapposto 0,5 ml di soluzione di demembranazione sul pellet cellulare, la soluzione è stata miscelata con una mano per sospendere approssimativamente le cellule. (B) Dopo la miscelazione, il resto del pellet cellulare è stato interamente sospeso nella soluzione da una pipetta Pasteur. Fare clic qui per visualizzare una versione più grande di questa figura.
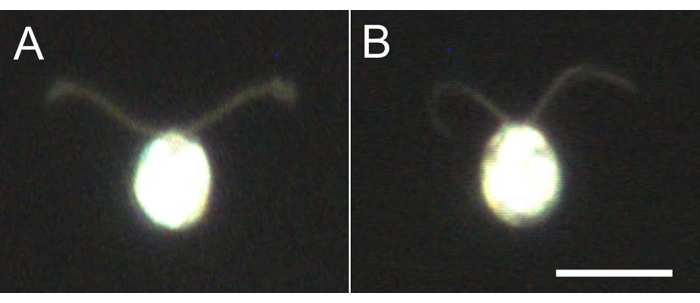
Figura 4: Effetti della demembranazione. (A) Una cellula viva bloccata su un vetrino. (B) Un modello di cella incollato su un vetrino. Si noti che nel modello cellulare, le ciglia sono diventate leggermente più sottili. Le immagini sono state osservate al microscopio con una lente obiettivo 20x e un condensatore a campo scuro ad immersione in olio. Barra della scala = 10 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Film 2: Conferma della demembranazione. La sospensione del modello cellulare è stata osservata al microscopio con una lente obiettivo 10x e un condensatore a campo scuro ad immersione in olio. Nessuna cellula stava nuotando. Barra della scala = 100 μm. Fare clic qui per scaricare questo filmato.
3. Riattivazione di modelli cellulari demembranati
- Mescolare 80 μL di soluzione di riattivazione, 10 μL di soluzione di ATP e 10 μL di modelli cellulari in un tubo da 0,5 mL toccando il tubo (Figura 5).
NOTA: La concentrazione finale di ATP deve essere <3 mM perché la frequenza di battitura ciliare, un parametro per il moto ciliare, aumenta con la concentrazione di ATP e si satura a 2-3 mM di ATP17.
ATTENZIONE: La miscelazione mediante pipettaggio o vortice provoca la deciliazione e diminuisce l'efficienza di riattivazione.- Per riattivare con <0,2 mM di ATP, aggiungere un sistema di rigenerazione dell'ATP, come 70 U/mL di creatina chinasi e 5 mM di creatina fosfato (vedi Tabella dei materiali).
NOTA: La soluzione di riattivazione viene preparata ad una concentrazione più elevata (1,125x, Tabella Supplementare 1) rispetto alla soluzione di diluizione in modo che dopo aver miscelato l'ATP disciolto in acqua, il contenuto raggiunga le seguenti concentrazioni finali: 30 mM di Hepes (pH 7,4), 5 mM di MgSO4, 1 mM di ditiotreitolo (DTT), 1 mM di EGTA e 50 mM di acetato di potassio (vedere Tabella dei materiali).
- Per riattivare con <0,2 mM di ATP, aggiungere un sistema di rigenerazione dell'ATP, come 70 U/mL di creatina chinasi e 5 mM di creatina fosfato (vedi Tabella dei materiali).
- Mettere ~ 30 μL della soluzione mista su un vetrino e posizionare delicatamente un coperchio su di esso con distanziali per evitare shock meccanici ai modelli di cella (Figura 6).
NOTA: Il petrolio bianco o i nastri biadesivi possono essere utilizzati per realizzare i distanziali. - Osservare i modelli cellulari riattivati al microscopio (Film 3).

Figura 5: Soluzione di miscelazione toccando il tubo. (A) A 80 μL della soluzione di riattivazione, sono stati aggiunti 10 μL di soluzione di ATP e 10 μL di modelli cellulari in ordine sequenziale. (B) La soluzione è stata miscelata picchiettando il tubo con un dito. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 6: Realizzazione di distanziali sui bordi delle coperture. (A) Un sottile strato di petrolio bianco è stato applicato sul dorso di una mano. (B) Una piccola quantità di petrolio bianco è stata raschiata via con un bordo di una copertura. (C) Un distanziatore è stato realizzato sul bordo di un coperchio. (D) Un altro distanziatore è stato realizzato sul bordo opposto. Fare clic qui per visualizzare una versione più grande di questa figura.
Film 3: Nuoto di modelli cellulari riattivati. La motilità dei modelli cellulari è stata riattivata aggiungendo ATP ad una concentrazione finale di 1 mM e osservata al microscopio con una lente obiettivo 10x e un condensatore a campo scuro ad immersione in olio. Barra della scala = 100 μm. Fare clic qui per scaricare questo filmato.
Risultati
Il processo di demembranazione e riattivazione nel ceppo wild type C. reinhardtii (CC-125) è mostrato qui. La coltura 2 giorni dopo l'inoculazione è diventata di un colore verde chiaro (passo 1.1) (Figura 1). Le cellule sono state raccolte (fase 2.1), lavate (fase 2.2) e demembranate (fase 2.5). Dopo la demembranazione, tutti i modelli cellulari sono diventati immotili (passo 2.7). Le ciglia demembranate (chiamate assonemi) rimangono attaccate al corpo cellulare, il che dimostra ...
Discussione
Ci sono due passaggi critici in questo protocollo. Il primo è un processo noto come demembranazione, che deve essere eseguito delicatamente ma accuratamente. La deciliazione (cioè il distacco delle ciglia dal corpo cellulare) è indotta da un vigoroso pipettaggio o vortice, rendendo i modelli cellulari immobili anche dopo l'aggiunta di ATP. Tipicamente, 5 × 107 cellule sono sospese in ~ 0,5 mL di tampone di demembranazione (densità cellulare finale: 1 × 108 celle / mL). Il tasso di riattivazion...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo studio è stato supportato da sovvenzioni della Japan Society for the Promotion of Science KAKENHI (https://www.jsps.go.jp/english/index.html) a N.U. (19K23758, 21K06295) e K.W. (19H03242, 20K21420, 21H00420), dalla Ohsumi Frontier Science Foundation (https://www.ofsf.or.jp/en/) a K.W., e dalla Dynamic Alliance for Open Innovation Bridging Human, Environment and Materials (http://alliance.tagen.tohoku.ac.jp/english/) a N.U. e K.W. Ringraziamo la signora Miyuki Shinohara (Hosei Univ.) per il suo aiuto nella preparazione delle figure.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.5 mL plastic tube | QSP | 502-PLN-Q | |
| 15 mL conical tube | SARSTEDT | 62.554.502 | |
| Adenosine 5'-triphosphate disodium salt hydrate (ATP) | Sima-Aldrich | A2383 | |
| Centrifuge | KUBOTA | 2800 | |
| Chlamydomonas strain CC-125 | Chlamydomonas Resource Center | https://www.chlamycollection.org/ | |
| Creatine kinase | Merck | CK-RO | |
| Creatine phosphate | Merck | CRPHO-RO | |
| Dithiothreitol (DTT) | Nakalai tesque | 14128-46 | |
| GEDTA(EGTA) | Dojindo | G002 | |
| Hepes | Dojindo | GB70 | |
| Igepal CA-630 | Sigma-Aldrich | I8896 | IUPAC name is octylphenoxypolyethoxyethanol: IGEPAL CA-630 is a substitute for Nonidet P-40 (NP-40); NP-40 is no longer available in Sigma-Aldrich. |
| MgSO4-7H2O | Nakalai tesque | 21002-85 | |
| Microscope | Olympus | BX-53 | |
| Pasteur pipette | fisher scientific | 13-678-20C | |
| Polyethylene glycol, Mr 20,000 | Merck | 8.18897.1000 | |
| Pottasium acetate | Nakalai tesque | 28434-25 | |
| Sodium Hydroxide | Nacalai | 31511-05 | |
| Sucrose | FUJIFILM Wako Pure Chemical Corporation | 196-00015 |
Riferimenti
- Szent-Gyorgyi, A. Free-energy relations and contraction of actomyosin. Biological Bulletin. 96 (2), 140-161 (1949).
- Hoffman-Berling, H. Adenosintriphosphat als betriebsstoff von zellbewegungen. Biochimica et Biophysica Acta. 14, 182-194 (1954).
- Naitoh, Y., Kaneko, H. Reactivated Triton-extracted models of Paramecium: modification of ciliary movement by calcium ions. Science. 176 (4034), 523-524 (1972).
- Witman, G. B., Plummer, J., Sander, G. Chlamydomonas flagellar mutants lacking radial spokes and central tubules. Structure, composition, and function of specific axonemal components. Journal of Cell Biology. 76 (3), 729-747 (1978).
- Rüffer, U., Nultsch, W. Flagellar coordination in Chlamydomonas cells held on micropipettes. Cell Motility and the Cytoskeleton. 41 (4), 297-307 (1998).
- Sale, W. S., Satir, P. Direction of active sliding of microtubules in Tetrahymena cilia. Proceedings of the National Academy of Sciences of the United States of America. 74 (5), 2045-2049 (1977).
- Fox, L. A., Sale, W. S. Direction of force generated by the inner row of dynein arms on flagellar microtubules. Journal of Cell Biology. 105 (4), 1781-1787 (1987).
- Kamiya, R., Yagi, T. Functional diversity of axonemal dyneins as assessed by in vitro and in vivo motility assays of Chlamydomonas mutants. Zoolog Science. 31 (10), 633-644 (2014).
- Kamiya, R., Witman, G. B. Submicromolar levels of calcium control the balance of beating between the two flagella in demembranated models of Chlamydomonas. Journal of Cell Biology. 98 (1), 97-107 (1984).
- Okita, N., Isogai, N., Hirono, M., Kamiya, R., Yoshimura, K. Phototactic activity in Chlamydomonas 'non-phototactic' mutants deficient in Ca2+-dependent control of flagellar dominance or in inner-arm dynein. Journal of Cell Science. 118, 529-537 (2005).
- Hyams, J. S., Borisy, G. G. Isolated flagellar apparatus of Chlamydomonas: characterization of forward swimming and alteration of waveform and reversal of motion by calcium ions in vitro. Journal of Cell Science. 33, 235-253 (1978).
- Fujiu, K., Nakayama, Y., Yanagisawa, A., Sokabe, M., Yoshimura, K. Chlamydomonas CAV2 encodes a voltage- dependent calcium channel required for the flagellar waveform conversion. Current Biology. 19 (2), 133-139 (2009).
- Bessen, M., Fay, R. B., Witman, G. B. Calcium control of waveform in isolated flagellar axonemes of Chlamydomonas. Journal of Cell Biology. 86 (2), 446-455 (1980).
- Wakabayashi, K., King, S. M. Modulation of Chlamydomonas reinhardtii flagellar motility by redox poise. Journal of Cell Biology. 173 (5), 743-754 (2006).
- Saegusa, Y., Yoshimura, K. cAMP controls the balance of the propulsive forces generated by the two flagella of Chlamydomonas. Cytoskeleton. 72 (8), 412-421 (2015).
- Gorman, D. S., Levine, R. P. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. Proceedings of the National Academy of Sciences of the United States of America. 54 (6), 1665-1669 (1965).
- Takano, W., Hisabori, T., Wakabayashi, K. Rapid estimation of cytosolic ATP concentration from the ciliary beating frequency in the green alga Chlamydomonas reinhardtii. Journal of Biological Chemistry. 296, 100156 (2021).
- Wakabayashi, K., Yagi, T., Kamiya, R. Ca2+-dependent waveform conversion in the flagellar axoneme of Chlamydomonas mutants lacking the central-pair/radial spoke system. Cell Motility and the Cytoskeleton. 38 (1), 22-28 (1997).
- Yueh, Y. G., Crain, R. C. Deflagellation of Chlamydomonas reinhardtii follows a rapid transitory accumulation of inositol 1,4,5-trisphosphate and requires Ca2+ entry. Journal of Cell Biology. 123 (4), 869-875 (1993).
- Wakabayashi, K., Ide, T., Kamiya, R. Calcium-dependent flagellar motility activation in Chlamydomonas reinhardtii in response to mechanical agitation. Cell Motility and the Cytoskeleton. 66 (9), 736-742 (2009).
- Ueki, N., Wakabayashi, K. Detergent-extracted Volvox model exhibits an anterior-posterior gradient in flagellar Ca2. Proceedings of the National Academy of Sciences of the United States of America. 115 (5), 1061-1068 (2018).
- Tanno, A., et al. The four-celled Volvocales green alga Tetrabaena socialis exhibits weak photobehavior and high-photoprotection ability. PLoS One. 16 (10), 0259138 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon