È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Una pipeline per studiare le strutture e le vie di segnalazione dei recettori della sfingosina 1-fosfato
In questo articolo
Riepilogo
S1P esercita i suoi diversi effetti fisiologici attraverso la sottofamiglia dei recettori S1P (S1PR). Qui viene descritta una pipeline per illustrare le strutture e la funzione degli S1PR.
Abstract
I lisofosfolipidi (LPL) sono lipidi bioattivi che includono sfingosina 1-fosfato (S1P), acido lisofosfatidico, ecc. S1P, un prodotto metabolico degli sfingolipidi nella membrana cellulare, è uno degli LPL meglio caratterizzati che regola una varietà di risposte fisiologiche cellulari tramite vie di segnalazione mediate dai recettori della sfingosina 1-fosfato (S1PR). Ciò implicava che il sistema di segnalazione S1P-S1PRs è un notevole potenziale bersaglio terapeutico per disturbi, tra cui la sclerosi multipla (SM), i disturbi autoimmuni, il cancro, l'infiammazione e persino COVID-19. Gli S1PR, un piccolo sottoinsieme della famiglia dei recettori accoppiati alla proteina G di classe A (GPCR), sono composti da cinque sottotipi: S1PR1, S1PR2, S1PR3, S1PR4 e S1PR5. La mancanza di informazioni strutturali dettagliate, tuttavia, impedisce la scoperta di farmaci mirati agli S1PR. Qui, abbiamo applicato il metodo della microscopia crioelettronica per risolvere la struttura del complesso S1P-S1PRs e abbiamo chiarito il meccanismo di attivazione, riconoscimento selettivo dei farmaci e accoppiamento della proteina G utilizzando saggi funzionali basati su cellule. Altri recettori lisofosfolipidi (LPLR) e GPCR possono anche essere studiati utilizzando questa strategia.
Introduzione
La sfingosina-1-fosfato (S1P), un prodotto metabolico degli sfingolipidi nella membrana cellulare, è una molecola di segnalazione lisofosfatidica ubiquitaria che coinvolge varie attività biologiche, tra cui il traffico linfocitario, lo sviluppo vascolare, l'integrità endoteliale e la frequenza cardiaca 1,2,3. S1P esercita i suoi diversi effetti fisiologici attraverso cinque sottotipi di recettori S1P (S1PRs 1-5); Le S1PR si trovano in una varietà di tessuti e mostrano preferenze uniche per le proteine G a valle 4,5. S1PR1 è principalmente accoppiato con la proteina Gi, che successivamente inibisce la produzione di cAMP; S1PR2 e S1PR3 sono accoppiati con Gi, Gq e G12/13, e S1PR4 e S1PR5 trasducono il segnale attraverso Gi e G12/136.
La segnalazione S1P-S1PR è un bersaglio terapeutico critico per molteplici malattie, tra cui i disturbi autoimmuni7, l'infiammazione8, il cancro9 e persino COVID-1910. Nel 2010, fingolimod (FTY720) è stato autorizzato come farmaco di prima classe mirato agli S1PR per il trattamento della sclerosi multipla recidivante (SM)11. Tuttavia, è in grado di legarsi a tutti gli S1PR tranne S1PR2, mentre il legame non specifico a S1PR3 provoca edema della corteccia cerebrale, costrizione vascolare e bronchiale e perdita epiteliale polmonare12. Come strategia alternativa per aumentare la selettività terapeutica, sono stati prodotti ligandi sottotipo-specifici per il recettore. Siponimod (BAF312) è stato approvato nel 2019 per il trattamento della SM recidivante13; si rivolge efficacemente a S1PR1 e S1PR5, mentre non ha affinità per S1PR3, mostrando meno effetti collaterali nella pratica clinica14. Nel 2020, la Food and Drug Administration degli Stati Uniti ha autorizzato ozanimod per la terapia della SM15. È stato riportato che ozanimod detiene una selettività 25 volte maggiore per S1PR1 rispetto a S1PR516. In particolare, nel contesto dell'attuale pandemia di COVID-19, è stato scoperto che i farmaci agonisti che hanno come bersaglio le S1PR possono essere utilizzati per trattare COVID-19 utilizzando tecniche di terapia immunomodulatoria17. Rispetto a fingolimod, ozanimod ha mostrato superiorità nel ridurre i sintomi nei pazienti COVID-19 ed è ora in fase di sperimentazione clinica10. La comprensione delle basi strutturali e della funzione delle S1PR pone una base significativa per lo sviluppo di un farmaco che colpisca selettivamente le S1PR18.
Molte tecniche sono utilizzate per studiare le informazioni strutturali delle biomacromolecole, tra cui la cristallografia a raggi X, la risonanza magnetica nucleare (NMR) e la microscopia elettronica (EM). A partire da marzo 2022, ci sono più di 180.000 strutture depositate sulla Protein Databank (PDB) e la maggior parte di esse è stata risolta dalla cristallografia a raggi X. Tuttavia, con la prima struttura di risoluzione quasi atomica di TPRV1 (risoluzione 3,4 Å) riportata da Yifan Cheng e David Julius nel 201319, la microscopia crioelettronica (crio-EM) è diventata una tecnica mainstream per le strutture proteiche e il numero totale di strutture PDB EM era superiore a 10.000. Le aree critiche di svolta sono lo sviluppo di nuove telecamere per l'imaging note come telecamere a rilevamento diretto di elettroni e nuovi algoritmi di elaborazione delle immagini. Cryo-EM ha rivoluzionato la biologia della struttura e la scoperta di farmaci basati sulla struttura nell'ultimo decennio20. Poiché comprendere come i complessi macromolecolari svolgono i loro complicati ruoli nella cellula vivente è un tema centrale nelle scienze biologiche, la crio-EM ha il potenziale per rivelare conformazioni di complessi molecolari dinamici, in particolare per le proteine transmembrana21. I recettori accoppiati alle proteine G (GPCR) sono la più grande superfamiglia di proteine transmembrana e il bersaglio di oltre il 30% dei farmaci attualmente commercializzati22. Lo sviluppo della crio-EM ha contribuito a un'esplosione di strutture ad alta risoluzione di complessi proteici GPCR-G, consentendo la determinazione strutturale di bersagli "intrattabili" che non sono ancora accessibili all'analisi cristallografica a raggi X nella progettazione di farmaci23. Quindi, l'applicazione crio-EM offre la possibilità di determinare la struttura tridimensionale dei GPCR in condizioni quasi native a una risoluzione atomica vicina a24. I progressi nella crio-EM rendono possibile visualizzare le basi meccanicistiche della stimolazione o dell'inibizione della GPCR e ulteriori benefici nello scoprire i nuovi siti di legame per la creazione di farmaci mirati al GPCR25.
Basandosi sugli enormi progressi della tecnologia crio-EM, abbiamo identificato strutture di complessi di segnalazione agonizzati S1PR1-, S1PR3- e S1PR5-Gi recentemente26,27. Nell'uomo, gli S1PR si trovano in vari tessuti e mostrano preferenze uniche per le proteine G a valle 4,5. S1PR1 è principalmente accoppiato con la proteina Gi, che successivamente inibisce la produzione di 3′,5′-adenosina monofosfato ciclico (cAMP). S1PR3 e S1PR5 sono anche in grado di accoppiarsi con Gi 6,28. Poiché l'attivazione del recettore accoppiato a Gi diminuisce la produzione di cAMP29, è stato introdotto un test di inibizione del cAMP per misurare gli effetti di inibizione del cAMP per catturare le alterazioni funzionali26,27. Utilizzando una versione mutante di Photinus pyralis luciferasi in cui è stata inserita una porzione proteica legante il cAMP, questo test cAMP offre un metodo semplice e affidabile per monitorare l'attività GPCR attraverso cambiamenti nella concentrazione intracellulare di cAMP30. Si tratta di un saggio funzionale sensibile e non radioattivo e può essere applicato per monitorare la segnalazione a valle in tempo reale di un'ampia gamma di GPCR ai fini della scoperta di farmaci31.
Qui, viene fornito un riepilogo dei metodi critici per risolvere le modalità di attivazione e riconoscimento dei farmaci di S1PR, includendo principalmente manipolazioni crio-EM e un test di inibizione Gi-cAMP. Questo articolo ha lo scopo di fornire una guida sperimentale completa per ulteriori esplorazioni nelle strutture e nelle funzioni dei GPCR.
Protocollo
1. Purificazione del complesso proteico S1PRs-G
- Per purificare il complesso proteico umano S1PRs-G, clonare i cDNA di S1PR1 privi di residui C-terminali 338-382, il wild-type S1PR3, S1PR5 troncato con 345-398 al C-terminale, e il wild-type Gi1 nel vettore pFastBac1 e i cDNA del wild-type Gβ1 e Gγ2 nel vettore pFastBacdual (Table of Materials).
NOTA: Tutti i costrutti per S1PR contengono anche la sequenza di segnali emoagglutinina (HA) seguita da un tag epitopo Flag al N-terminale e un tag 10x his al C-terminale. Inoltre, una sequenza di DNA sintetico (Table of Materials) per la traduzione del lisozima T4 (T4L) è stata inserita nel N-terminale degli S1PR al fine di facilitare l'espressione e la purificazione del recettore. - Preparazione del baculovirus che codifica S1PRs, Gi1 e Gβ1γ2
- Aggiungere i vettori ricombinanti a 50 μL di Escherichia coli competente DH5α (E. coli) conservato in una provetta da 1,5 ml a -80 °C e incubare su ghiaccio per 30 minuti.
- Shock termico delle celle a 42 °C per 90 secondi, trasferirle immediatamente nel ghiaccio e raffreddarle per 2 minuti.
- Agitare il tubo a 37 °C per 3-5 ore dopo aver completato con 300 μL di terreno di brodo lisogenico (LB). Placcare 100 μL di cellule nella piastra agar LB e incubare a 37 °C mantenendo al buio per 48 ore.
- Inoculare la colonia bianca in 5 mL di terreno LB contenente 50 μg/mL di kanamicina, 10 μg/mL di tetraciclina e 7 μg/mL di gentamicina, e coltivare a 37 °C per 16 ore.
- Isolare il batteride ricombinante con il kit miniprep plasmidico (Table of Materials) seguendo le istruzioni del produttore, per la produzione del baculovirus P0.
NOTA: Prima dell'uso, il bacmide purificato è stato analizzato mediante PCR con primer pUC / M13 avanti e indietro. Per la PCR, numero di cicli = 30 cicli, temperatura di fusione = 58 °C e tempo di estensione = 1 min per 1 Kb. - Preparare il baculovirus P0 come descritto in un precedente protocollo32.
- Coltura delle celle sf9 (ESF921 medium) in piastre a sei pozzetti e verificare che le cellule siano nella fase logaritmica (1,0-1,5 x 106 celle/ml).
- Diluire 8 μL di reagente di trasfezione baculovirus in 100 μL di terreno di Grace e incubare per 30 minuti a temperatura ambiente. Diluire 10 μg del bacmide ricombinante in 100 μL di terreno di Grace e mescolare delicatamente. Combinare il batteride diluito con il reagente di trasfezione del baculovirus diluito, mescolare delicatamente e incubare per 30 minuti a temperatura ambiente.
- Aggiungere la miscela (bacmide e reagente) sulle cellule (punto 1.2.6.1) e placcarle a 27 °C per 3 ore.
- Rimuovere il terreno di coltura di Grace e sostituirlo con 2 ml di terreno di coltura cellulare ESF921. Placcare le piastre a sei pozzetti a 27 °C e raccogliere il terreno di coltura cellulare ESF921 dopo 5 giorni dalla trasfezione.
- Centrifugare a 500 x g a 4 °C per 10 minuti per rimuovere le cellule e i detriti. Trasferire il surnatante in provette da 2 ml. Questo è lo stock di baculovirus P0.
- Isolare lo stock di virus P1
- Trasferire 30 mL di cellule sf9 in un flacone conico, coltivare a 27 °C agitando a 270 rpm e verificare che le cellule raggiungano una fase logaritmica (1,0-1,5 x 106 celle/ml).
- Aggiungere 2 mL di materiale di riserva di virus P0 al flacone e agitare a 270 giri/min per 4 giorni a 27 °C.
- Trasferire le cellule in una provetta da 50 ml, centrifugare a 1.800 x g per 10 minuti a 4 °C per rimuovere le cellule e i detriti e trasferire il surnatante in provette da 50 ml. Questo è lo stock di baculovirus P1.
- Amplifica lo stock di baculovirali
- Ripetere il punto 1.2.7 usando 50 mL di celle sf9 nella fase logaritmica (1,0-1,5 x 106 celle/ml) e 1 mL di stock P1.
- Conservare lo stock di baculovirus P2 risultante a 4 °C, proteggendolo dalla luce.
NOTA: Non amplificare il baculovirus indefinitamente, poiché i mutanti deleteri sono stati prodotti ad ogni passaggio.
- Espressione del complesso proteico S1PRs-G
- Coltura di cellule di insetti sf9 per raggiungere 2,5 x 106 cellule/mL di densità, co-infettare con il baculovirus P2 codificante S1PRs, Gi1 e Gβ1γ2 con un rapporto di volume di 1:2:1, e coltura di nuovo a 27 °C per 48 ore.
- Raccogliere le cellule centrifugando a 700 x g a 4 °C per 15 minuti, congelarle in azoto liquido e conservarle a -80 °C per l'uso.
- Purificazione delle proteine
- Scongelare il pellet cellulare ottenuto nella fase 1.3 a temperatura ambiente, quindi risospenderlo in tampone di lisi (20 mM HEPES pH 7,5, 50 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2) integrato con 100 μg/mL di benzamidina, 100 μg/mL di leupeptina, 100 μg/mL di aprotinina, 25 mU/mL di apirasi e 10 μM agonista. Agitare la sospensione cellulare a temperatura ambiente per 2 ore per indurre la formazione del complesso proteico S1PRs-G.
NOTA: L'apirasi è una difosfoidrolasi ATP. Catalizza la rimozione del fosfato gamma dall'ATP e del fosfato beta dall'ADP. - Trasferire la soluzione nei tubetti, centrifugare a 70.000 x g per 10 minuti e rimuovere accuratamente il surnatante . Risospendere il pellet in tampone solubilizzante (20 mM HEPES pH 7,5, 100 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2, 0,5% (p/v) LMNG, 0,1% (p/v) CHS, 1% (p/v) colato di sodio, 10% (v/v) glicerolo).
- Trasferire la sospensione in un bicchiere Dounce e omogeneizzare completamente il pellet. Aggiungere alla sospensione 10 μM di agonista, 4 mg di scFv16, 100 μg/mL di benzamidina, 100 μg/mL di leupeptina, 100 μg/mL di aprotinina e 25 mU/mL di apirasi e agitare a 4 °C per 2 ore.
NOTA: Le fasi di omogeneizzazione dei pellet sono cruciali per la produzione del complesso proteico GPCR-G. - Trasferire la soluzione nei tubi e centrifugare a 100.000 x g per 30 minuti a 4 °C.
- Pre-equilibrare la resina flag con il tampone di lavaggio (20 mM HEPES pH 7,5, 100 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2, 10 μM agonista, 0,0375% (p/v) LMNG, 0,0125% (p/v) GDN, 0,01% (p/v) CHS).
- Trasferire il surnatante nei tubi e incubare con la resina bandiera a 4 °C per 2 ore.
- Caricare la miscela di cui sopra sulla colonna di vetro e lavare la colonna con 50 ml di tampone di lavaggio integrato con 100 μg/mL di benzamidina, 100 μg/mL di leupeptina e 100 μg/mL di aprotinina.
- Eluire la colonna con 10 mL di tampone di eluizione contenente 20 mM HEPES pH 7,5, 100 mM NaCl, 5 mM EDTA, 200 μg/mL Flag peptide, 10 μM agonista, 0,0375% (p/v) LMNG, 0,0125% (p/v) GDN, 0,01% (p/v) CHS, 100 μg/mL di benzamidina, 100 μg/mL di leupeptina e 100 μg/mL di aprotinina.
- Raccogliere il complesso proteico S1PRs-G e concentrarsi a 1 mL utilizzando un concentratore cut-off da 100 kDa a 1.300 x g a 4 °C. Filtrare attraverso un filtro da 0,22 μM e centrifugare a 13.000 x g per 10 minuti a 4 °C per rimuovere gli aggregati.
- Caricare il complesso proteico S1PRs-G su una colonna di filtrazione su gel per cromatografia ad esclusione dimensionale (SEC) pre-equilibrata con il tampone SEC contenente 20 mM HEPES pH 7,5, 100 mM NaCl, 10 μM agonista, 100 Μm TCEP, 0,00375% (p/v) LMNG, 0,00125% (p/v) GDN e 0,001% (p/v) CHS ad una portata di 0,5 mL/min a 4 °C.
- Raccogliere le frazioni di picco e concentrare utilizzando un concentratore di taglio da 100 kDa a 1.300 x g a 4 °C per la crio-EM.
- Scongelare il pellet cellulare ottenuto nella fase 1.3 a temperatura ambiente, quindi risospenderlo in tampone di lisi (20 mM HEPES pH 7,5, 50 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2) integrato con 100 μg/mL di benzamidina, 100 μg/mL di leupeptina, 100 μg/mL di aprotinina, 25 mU/mL di apirasi e 10 μM agonista. Agitare la sospensione cellulare a temperatura ambiente per 2 ore per indurre la formazione del complesso proteico S1PRs-G.
2. Microscopia elettronica per risolvere la struttura delle S1PR
- Raccolta dei dati
- Per preparare le griglie crio-EM, tenere griglie Au R1.2/1.3 a 300 maglie per 10 s e scaricare a bagliore per 60 s a 15 mA utilizzando un sistema di pulizia a scarica luminosa.
- Eseguire la vetrificazione del campione come descritto in precedenza33,34. Nella console di congelamento a tuffo, impostare la temperatura su 4 °C e l'umidità relativa su 100% per l'ambiente di lavoro della camera. Utilizzare la forza di macchia per 0, il tempo di attesa di 0 s, il tempo di macchia di 2 o 3 s e il tempo di drenaggio di 0 s. Di solito richiede solo 3 μL del campione a concentrazioni di 5-10 mg / ml per la vitrificazione singola.
- Ritagliare e caricare le griglie nell'assemblaggio automatico della griglia, caricare l'assemblaggio automatico della griglia in Nanocab e caricare Nanocab nel microscopio tramite caricatore automatico come descritto in precedenza35. Qualità del campione dello schermo con il software EPU234. Di solito, i dati raccolti nelle aree di spessore del ghiaccio adatto erano migliori.
- Raccogliere dati crio-EM come descritto in dettaglio in precedenza35. Per il recettore S1P, impostare l'offset di sfocatura tra -1,0 μm e -1,8 μm con la dose di elettroni di esposizione di 50-65 e-/Å2. Per i complessi S1PR1-Gi, raccogliere automaticamente lo stack di filmati utilizzando il software EPU2 in modalità di conteggio con il rilevatore K2 a un tempo di esposizione totale di 2 s, una velocità di registrazione di cinque fotogrammi grezzi al secondo e una dose totale di 56 e-/Å2 per produrre 35 fotogrammi per stack.
NOTA: Di solito, sono necessari più di 5.000 film per ricostruire la struttura del recettore.
- Elaborare i dati utilizzando una combinazione di RELION36 e cryoSPARC37 per ottenere una mappa di densità crio-EM ideale. Utilizzare RELION-3.1_gpu_ompi4 per elaborare inizialmente i dati che comportano operazioni simili a quelle descritte in precedenza34.
- Nel terminale di sistema Linux, immettere la directory padre della directory di archiviazione dei dati.
- Immettere il comando relion nel terminale per aprire l'interfaccia utente grafica (GUI) RELION.
NOTA: se è la prima volta che una GUI RELION è stata aperta in questa directory, verrà visualizzata una finestra di richiesta; fare clic su Sì. - Fare clic su Importa nella barra delle funzioni sul lato destro della GUI RELION per importare i dati grezzi in RELION.
- Nell'opzione Filmati/microfoni, selezionare Sì per l'importazione di filmati o micrografie non elaborati?, immettere il percorso dei dati nel campo File di input non elaborati (consigliati caratteri jolly) e selezionare Sì per Filmati multifotogramma?. Immettere la dimensione in pixel dei filmati nel campo Dimensione pixel (Angstrom), la tensione di funzionamento del microscopio (in kV) nel campo Tensione (kV) e l'aberrazione sferica del microscopio nel campo Aberrazione sferica (mm). Questi sono i parametri che sono stati registrati al momento della raccolta dei dati.
- Nell'opzione In esecuzione, modificare il nome della coda in base al server in cui è in esecuzione il programma (anche altre funzioni devono modificare questo parametro). Lasciare gli altri parametri ai valori predefiniti impostati da RELION. Infine, quando tutti i parametri vengono rilevati correttamente, fare clic su ESEGUI! per eseguire il programma.
- Utilizzare la funzione di correzione del movimento per l'allineamento di tutti i fotogrammi38.
- Nell'opzione I / O , fai clic su Sfoglia e scegli l'output della funzione di importazione denominata movies.star come input del file STAR dei filmati di input. Immettere la dose per fotogramma nel campo Dose per fotogramma (e/A2) che equivale alla dose totale divisa per il numero di fotogrammi. Selezionare No per Salva somma degli spettri di potenza?.
- Nell'opzione Movimento, immettete 250 per il fattore B, 5,5 per Numero di patch X,Y e 2 per i fotogrammi di gruppo (assicuratevi della dose del gruppo >3). Se i dati non sono stati referenziati durante il periodo di raccolta, è necessaria un'immagine di riferimento del guadagno che può essere ottenuta acquisendo un'area vuota della griglia. Selezionare No for Use RELION's own implementation? e inserire la directory contenente il file eseguibile di MOTIONCOR2 39nel campo eseguibile MOTIONCOR2.
- Nell'opzione In esecuzione , scegliere l'MPI e il numero di thread appropriati in base alla potenza di calcolo del server; qui sono stati utilizzati MPI = 8 e thread = 3.
- Utilizzare la funzione di stima CTF per modulare l'immagine crio-EM del campione vetrificato40. Nell'opzione I/O, fare clic su Sfoglia e scegliere l'output di Motion cor denominato corrected micrographs.star come input del file STAR dei filmati di input. Nell'opzione Gctf, selezionare Sì per UseGctf?.
- Utilizzare la funzione di selezione dei sottoinsiemi per rimuovere le micrografie con un valore di rlnCtfMaxResolutin >4.
- Nell'opzione I/O , fare clic su Sfoglia situato a destra di OR selezionare da micrographs.star e scegliere l'output di CtfFind denominato micrographs_ctf.star come input. Nell'opzione Sottoinsieme , selezionare Sì per Seleziona in base ai valori dei metadati? e immettere 4 per il valore massimo dei metadati per rimuovere i dati non validi.
- Prelievo manuale: seleziona manualmente alcune immagini per la selezione preliminare e la classificazione.
- Nell'opzione I/O , fare clic su Sfoglia e scegliere micrographs_selected.star dalla directory Select precedente (Passo 2.2.6) come input.
- Fare clic su ESEGUI! (viene visualizzata una finestra). Fare clic su File nell'angolo in alto a sinistra della nuova finestra e fare clic su Inverti selezione per annullare la selezione di tutte le immagini. Seleziona la casella di selezione più a sinistra nella riga corrispondente di ogni voce e fai clic su scegli per controllare le immagini e selezionare ~ 500 buone immagini. Fare clic su File > Salva selezione per salvare le immagini selezionate e chiudere la finestra.
- Auto-picking: i pacchetti software automatizzati di prelievo delle particelle sono utili e potenti41.
- Nell'opzione I/O , fare clic su Sfoglia a destra di Micrografie di input per autopick e scegliere micrographs_selected.star dalla directory ManualPick precedente (Passo 2.2.7) come input. L'algoritmo laplaciano-di-gaussiano viene utilizzato all'inizio, quindi selezionare Sì per OR: utilizzare Laplaciano-di-Gaussiano.
- Nell'opzione Laplacian, impostare Min.diameter per il filtro LoG (A) su 80 e Max.diameter per il filtro LoG (A) su 130. Nell'opzione Selezione automatica, impostare Distanza minima tra le particelle (A) su 65 e selezionare Sì per Usa accelerazione GPU se è possibile accedere alla GPU.
- Estrai particelle per i passaggi successivi.
- Nell'opzione I/O , fare clic su Sfoglia a destra del file STAR della micrografia e scegliere micrographs_selected.star dal passaggio 2.2.6. Fare clic su Sfoglia a destra delle coordinate di input e scegliere cords_suffix_autopick.star dal passaggio 2.2.8.
- Nell'opzione Estrai , selezionate Sì per Riscala particelle e impostate le dimensioni ridimensionate (pixel) su 128 per velocizzare i passaggi successivi.
- Classificazione 2D per la classificazione preliminare delle particelle
- Nell'opzione I/O , fare clic su Sfoglia a destra del file STAR delle immagini di input e scegliere particles.star dal passaggio 2.2.9. Nell'opzione Ottimizzazione , impostare Numero di classi su 100 e Diametro maschera (A) su 140.
- Nell'opzione Calcola , impostare Numero di particelle raggruppate su 10, immettere la directory che si trova su un'unità locale veloce (ad esempio, un'unità SSD) nel campo Copia particella nella directory scratch e selezionare Sì per Usa accelerazione GPU? per una maggiore velocità di elaborazione.
- Selezione di sottoinsiemi per selezionare buoni risultati 2D come modelli per scegliere le particelle
- Nell'opzione I/O , fare clic su Sfoglia a destra di Seleziona classi da model.star e scegliere l'output del passaggio 2.2.10 denominato run_it025_model.star come input. Clicca su RUN!. Nella finestra pop-up, seleziona Ordina immagini su e Inverti ordina? e fai clic su Visualizza!.
- Scegliete buoni risultati 2D rappresentativi come riferimento per Riferimento della funzione di prelievo automatico .
NOTA: i risultati selezionati sono contrassegnati in rosso. I risultati della classificazione 2D buoni e cattivi vengono mostrati più avanti. - Fare clic con il pulsante destro del mouse e selezionare Salva classi selezionate.
- Utilizzare il modello per il secondo round di selezione automatica. Nell'opzione I/O , fare clic su Sfoglia a destra di Micrografie di input per il prelievo automatico e scegliere micrographs_selected.star dal passaggio 2.2.6. Fare clic su Sfoglia a destra dei riferimenti 2D, scegliere class_averages.star dal passaggio 2.2.11 e selezionare No per OR: utilizzare Laplacian-of-Gaussian.
- Eseguire nuovamente l'estrazione delle particelle utilizzando coord_suffix_autopick.star dal passaggio 2.2.12 e micrographs_selected.star dal passaggio 2.2.6.
- Eseguire nuovamente la classificazione 2D utilizzando particles.star dal passaggio 2.2.13.
- Eseguire nuovamente la selezione dei sottoinsiemi utilizzando run_it025_optimiser.star dal passaggio 2.2.14.
NOTA: tutte le immagini 2D con contorni chiari e forme corrette devono essere scelte. - Eseguire l'estrazione delle particelle come segue. Nell'opzione I/O , fare clic su Sfoglia a destra del file STAR della micrografia, scegliere micrographs_selected.star dal passaggio 2.2.6 e selezionare Sì per OR riestrarre particelle raffinate?. Fare clic su Sfoglia a destra del file STAR delle particelle raffinate e scegliere particles.star dal passaggio 2.2.15.
- Modello iniziale 3D e generazione di una mappa di riferimento: nell'opzione I/O , fare clic su Sfoglia a destra del file STAR delle immagini di input e scegliere particles.star dal passaggio 2.2.16. Impostare Numero di classi su 1 e Diametro maschera (A) su 140 nell'opzione Ottimizzazione .
- Classificazione 3D e generazione di una mappa 3D preliminare: nell'opzione I/O , fare clic su Sfoglia a destra del file STAR delle immagini di input e scegliere particles.star dal passaggio 2.2.16. Fare clic su Sfoglia a destra della mappa di riferimento e scegliere il file initial_model.mrc dal passaggio 2.2.17. Impostare Numero di classi su 4-6 e Diametro maschera (A) su 140 nell'opzione Ottimizzazione .
- Generazione maschera: selezionare una o più mappe 3D valide dal passaggio 2.2.17 come input nell'opzione I/O . Impostate la soglia di binarizzazione iniziale su 0,05 (regolate in base all'output), Estendi mappa binario di questo numero di pixel a 3 e Aggiungi soft-edge di questo numero di pixel a 8 nell'opzione Maschera .
- Utilizzare cryoSPARC per la prossima elaborazione.
- Creare una nuova area di lavoro e fare clic su Job Builder per il primo processo.
- Per importare la pila di particelle, immettete il percorso della particella (Passo 2.2.16) nel campo Meta percorso particella e il percorso del filmato (Passo 2.2.16) nel campo Percorso dati particelle .
NOTA: i parametri Tensione di accelerazione (kV), Aberrazione sferica (mm) e Dimensione pixel (Angstrom) sono gli stessi di prima. Il valore di contrasto (frazione) di ampiezza è 0,1. - Importare volumi 3D immettendo il percorso del miglior volume 3D nel passaggio 2.2.18 nel percorso dati volume e selezionando Mappa per Tipo di volume importato.
- Importare maschera immettendo il percorso maschera (Passo 2.2.19) nel percorso Dati volume e selezionando Maschera per Tipo di volume da importare.
- Perfezionamento non uniforme: prendere come input gli output dei passaggi 2.2.22, 2.2.23 e 2.2.24.
NOTA: Questa funzione è molto utile per le proteine di membrana.- Trascinate l'output del Passo 2.2.22 (imported_particles) come input delle particelle di Raffinamento non uniforme (particella), l'output del Passo 2.2.23 (imported_volume_1) come input del volume (volume) del Raffinamento non uniforme e l'output del Passo 2.2.24 (imported_mask_1) come input della maschera di Raffinamento non uniforme (maschera).
NOTA: A volte è possibile ottenere risultati migliori senza maschera. - Fare clic su Coda per avviare l'elaborazione.
- Trascinate l'output del Passo 2.2.22 (imported_particles) come input delle particelle di Raffinamento non uniforme (particella), l'output del Passo 2.2.23 (imported_volume_1) come input del volume (volume) del Raffinamento non uniforme e l'output del Passo 2.2.24 (imported_mask_1) come input della maschera di Raffinamento non uniforme (maschera).
- Eseguire i passaggi 2.2.18-2.2.25 per ottenere risultati migliori. Attraverso la serie di elaborazione di cui sopra, è possibile ottenere una buona risoluzione S1PR-Gi mappa 3D.
3. S1PRs-Gi mediato test di inibizione cAMP
NOTA: L'esperimento di inibizione cAMP mediata da S1PRs-Gi è stato diviso in diverse parti, e le seguenti sono procedure sperimentali dettagliate. Il principio sperimentale e il processo sperimentale generale sono mostrati sotto forma di diagramma di flusso nella figura 1.
- Costruzione di plasmidi
- Sub-clonare i cDNA di wild-type S1PR1, S1PR3 e S1PR5 nel vettore pcDNA3.1+ con una sequenza di segnali HA seguita da un tag Flag al N-terminus (Table of Materials).
NOTA: Le mutazioni per tutti i recettori sono state generate utilizzando il kit di mutagenesi (Tabella dei materiali).
- Sub-clonare i cDNA di wild-type S1PR1, S1PR3 e S1PR5 nel vettore pcDNA3.1+ con una sequenza di segnali HA seguita da un tag Flag al N-terminus (Table of Materials).
- Preparazione del plasmide
- Aggiungere i vettori ricombinanti pcDNA3.1+ o il plasmide del sensore (Table of Materials) a 50 μL di Escherichia coli competente DH5α (E. coli) conservato separatamente in una provetta da 1,5 mL a -80 °C e incubare su ghiaccio per 30 minuti. Shock termico delle celle a 42 °C per 90 secondi, trasferirle immediatamente nel ghiaccio e raffreddarle per 2 minuti.
NOTA: Il plasmide del sensore fornito dal kit di analisi cAMP di inibizione Gi (Table of Materials) esprime un gene della luciferasi modificato fuso con un dominio di legame cAMP e aumenta l'attività di luminescenza quando cAMP è legato. - Agitare il tubo a 37 °C per 1 ora dopo aver completato con 300 μL di terreno di brodo lisogenico (LB). Placcare 100 μL di cellule nella piastra di agar LB e incubare a 37 °C tenendole al buio per 48 ore. Inoculare la colonia bianca in 5 mL di terreno LB contenente 100 μg/mL di ampicillina e coltivare a 37 °C per 16 ore.
- Isolare il DNA utilizzando il kit miniprep plasmidico (Table of Materials) seguendo le istruzioni del produttore; il plasmide era ad una concentrazione di oltre 350 ng/μL con il valore di A260/A280 tra 1,7 e 1,9.
- Aggiungere i vettori ricombinanti pcDNA3.1+ o il plasmide del sensore (Table of Materials) a 50 μL di Escherichia coli competente DH5α (E. coli) conservato separatamente in una provetta da 1,5 mL a -80 °C e incubare su ghiaccio per 30 minuti. Shock termico delle celle a 42 °C per 90 secondi, trasferirle immediatamente nel ghiaccio e raffreddarle per 2 minuti.
- Coltura cellulare
- Placcare le cellule CHO-K1 in una capsula di Petri da 10 cm, coltivarle in un incubatore a 37 °C con CO2 al 5% e raccoglierle quando il monostrato è all'80% -90% di confluenza.
- Aspirare il terreno di coltura delle cellule CHO-K1, aggiungere 4 mL di tripsina-EDTA allo 0,05% preriscaldato a 37 °C sulla capsula di Petri delicatamente e incubare per 15 s. Quindi, aggiungere 4 ml di terreno di crescita costituito da mezzo F12 + 10% FBS.
- Rimuovere le cellule dalla superficie della piastra di Petri ondeggiando delicatamente e toccando il lato della piastra di Petri. Riempire un tubo conico con sospensione cellulare. Mescolare e pipettare lentamente per rimuovere delicatamente i grumi cellulari.
- Centrifugare le celle a 250 x g per 5 minuti a temperatura ambiente, aspirare il surnatante e rianimare con 3 ml di PBS. Ripetere questo passaggio.
- Determinare il numero di cellule con l'ematocitometro e centrifugare le cellule a 250 x g per 5 minuti a temperatura ambiente.
- Aspirare il tampone PBS e risospendere le cellule CHO-K1 con 3 ml di terreno di crescita costituito da mezzo F12 e 10% FBS.
- Aggiungere 2 ml del terreno di crescita costituito da mezzo F12 e 10% FBS in ciascun pozzetto della piastra a sei pozzetti e seminare 150 μL di sospensione cellulare in ciascun pozzetto per mantenere le cellule alla densità di 1,5 x 105 cellule / ml. Incubare la piastra a sei pozzetti in un incubatore per colture tissutali a 37 °C con CO2 al 5% per circa 24 ore.
- Trasfezione transitoria
- Diluire 2 μg di DNA (Fase 3.2.3) in 200 μL di tampone reagente di trasfezione (Tabella dei materiali). Mescolare a vortice per 10 s e girare brevemente prima dell'uso.
NOTA: Qui, i 2 μg di DNA contengono 0,5 μg di vettore recettore (S1PR1, S1PR3 o S1PR5) e 1,5 μg del plasmide sensore. - Aggiungere 4 μL del reagente di trasfezione (Tabella dei materiali), vortice per 10 s e centrifugare brevemente prima dell'uso. Incubare per 15 minuti a temperatura ambiente.
- Rilasciare lentamente 200 μL di miscela di trasfezione in ciascun pozzetto (contenente cellule CHO-K1) per distribuire uniformemente. Agitare delicatamente la piastra a sei pozzetti per garantire una miscelazione accurata.
- Sostituire il mezzo di trasfezione dopo 4-6 ore con il mezzo di crescita cellulare costituito da mezzo F12 + 10% FBS e riportare la piastra a sei pozzetti all'incubatore.
- Raccogliere le cellule 24-48 h dopo la trasfezione.
- Digerire le cellule CHO-K1 sul pozzetto con 500 μL di tripsina-EDTA allo 0,05% (preriscaldato a 37 °C) per 15 s e aggiungere 1 mL di terreno di crescita costituito da terreno F12 + 10% FBS. Rimuovere le cellule dalla superficie del pozzo dondolando e picchiettando delicatamente il lato del pozzo.
- Trasferire la sospensione cellulare in un tubo conico e centrifugare a 250 x g per 5 minuti a temperatura ambiente. Versare il surnatante e raccogliere le cellule trasfettate.
NOTA: Prima della determinazione del segnale di fluorescenza, controllare i livelli di espressione superficiale cellulare dei recettori mediante ELISA come descritto in precedenza26.
- Diluire 2 μg di DNA (Fase 3.2.3) in 200 μL di tampone reagente di trasfezione (Tabella dei materiali). Mescolare a vortice per 10 s e girare brevemente prima dell'uso.
- Equilibrazione con sale di D-luciferina-potassio (Tabella dei materiali)
- Sospendere immediatamente le cellule raccolte (24-48 ore dopo la trasfezione) con 3 ml di tampone di dosaggio (cioè la soluzione salina bilanciata di Hank (HBSS) contenente 10 mM HEPES pH 7,4), con un'ulteriore diluizione del 3% v / v del sale D-luciferina-potassio.
- Aggiungere 90 μL di sospensione cellulare per pozzetto di una piastra a 96 pozzetti utilizzando una pipetta multicanale e mescolare delicatamente.
- Incubare per 40 minuti a temperatura ambiente.
- Determinazione del segnale di fluorescenza
- Preparare in anticipo 10 mM di soluzioni madre di Siponimod disciolto in DMSO ed effettuare una diluizione seriale utilizzando tampone HBSS contenente 25 μM di forskolina prima della stimolazione del ligando.
NOTA: Ad eccezione del gruppo di controllo senza ligando, i restanti hanno un intervallo di gradiente di concentrazione di 10-11-10-5 mol/L. - Stimolare con 10 μL (per pozzetto) di soluzione agonista a diverse concentrazioni per 30 minuti.
- Contare i segnali di luminescenza su un lettore di micropiastre utilizzando i parametri software associati (Table of Materials) come segue. Selezionare Luminescenza per Metodo di rilevamento, Endpoint per Tipo di lettura e Fibra di luminescenza per Tipo ottica. Impostare il guadagno ottico su 255.
NOTA: Ogni misurazione è stata ripetuta in almeno tre esperimenti indipendenti, ciascuno in triplice copia. - Ottenere i valori del segnale di fluorescenza, importare i dati in un programma di foglio di calcolo ed elaborare i dati utilizzando la funzione dose-risposta di regressione non lineare (adattamento alla curva).
- Preparare in anticipo 10 mM di soluzioni madre di Siponimod disciolto in DMSO ed effettuare una diluizione seriale utilizzando tampone HBSS contenente 25 μM di forskolina prima della stimolazione del ligando.
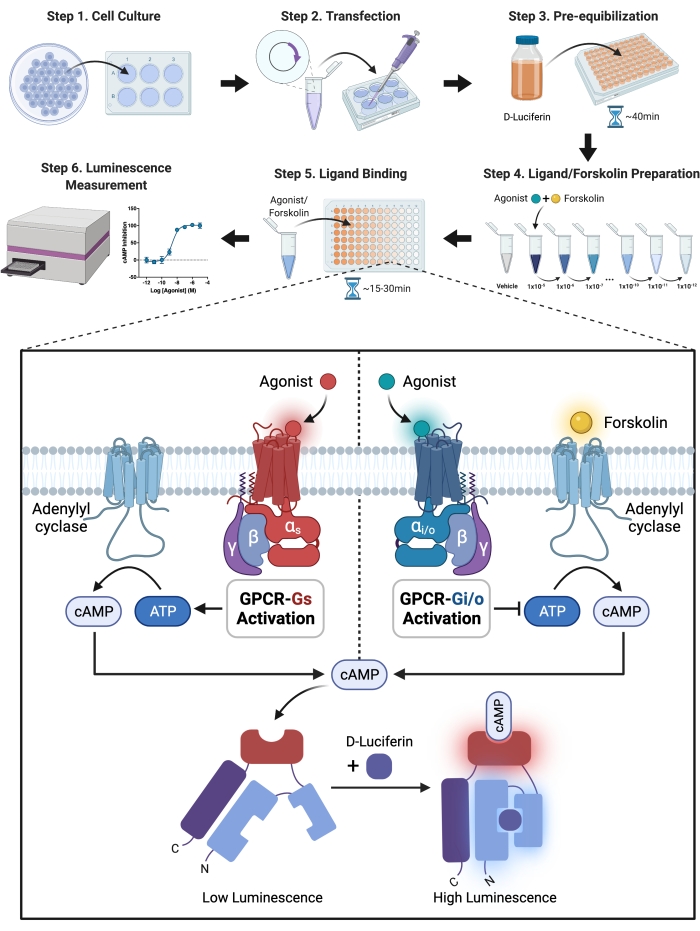
Figura 1: Illustrazione schematica dell'esperimento. Una guida dettagliata per la configurazione e l'esecuzione sperimentale. In breve, il recettore e la luciferasi modificata sono stati transitoriamente co-espressi nelle cellule CHO-K1 trasfettando il recettore e il plasmide sensore nelle cellule con reagente di trasfezione. Le cellule sono state sospese in soluzione HBSS con sale di D-luciferina-potassio, il substrato della luciferasi, e seminate in una piastra a 96 pozzetti dopo 24 ore. Per consentire la permeazione nelle cellule, la D-luciferina deve essere pre-equilibrata con le cellule. L'enzima ossidativo luciferasi trasforma la luciferina in ossiluciferina ed emette luce. La luciferasi modificata, d'altra parte, genera luce attraverso una reazione chimica solo quando legata al cAMP e l'intensità della luce ha un'associazione positiva con i livelli di cAMP nelle cellule. I livelli di cAMP sono stati regolati con GPCR attivato dall'agonista. I recettori accoppiati a Gi hanno ridotto i livelli di cAMP, rendendo necessaria l'aggiunta di forskolina per attivare l'adenilil ciclasi nell'esperimento di inibizione del Gi cAMP. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Risultati
Prima di congelare il campione del complesso S1PRs-Gi, il campione purificato deve essere separato mediante cromatografia ad esclusione dimensionale (SEC) e analizzato con cromatografia a filtrazione su gel. La Figura 2 mostra il complesso S1PR3-Gi come esempio. La frazione di picco del complesso proteico omogeneo GPCR-G era solitamente localizzata a ~ 10,5 ml della cromatografia di esclusione dimensionale (Figura 2A). L'analisi della pagina SDS del complesso S1...
Discussione
Questo protocollo descrive una pipeline primaria per determinare le strutture degli S1PR mediante crio-EM e misurare la potenza di attivazione degli S1PR mediante il saggio di inibizione del cAMP mediato da Gi. Alcuni passaggi sono cruciali per il successo dell'esperimento.
Per purificare il complesso S1PRs-Gi, la qualità del virus e la salute delle cellule sf9 dovrebbero essere prestate maggiore attenzione. L'espressione del recettore è drasticamente ridotta nelle cellule sf9
Divulgazioni
Gli autori non hanno conflitti di interesse.
Riconoscimenti
I dati del complesso S1PRs-Gi sono stati raccolti presso il West China Cryo-EM Center dell'Università del Sichuan e il Cryo-EM Center presso la Southern University of Science and Technology (SUSTech) ed elaborati presso il Duyu High-Performance Computing Center dell'Università del Sichuan. Questo lavoro è stato sostenuto dalla Natural Science Foundation of China (32100965 a L.C., 32100988 a W.Y., 31972916 a Z.S.) e dal Fondo di ricerca post-dottorato a tempo pieno dell'Università del Sichuan (2021SCU12003 a L.C.)
Materiali
| Name | Company | Catalog Number | Comments |
| 0.05% trypsin-EDTA | GIBCO | Cat# 25300054 | |
| 0.22 µM filter | Thermo Fisher Scientific | Cat# 42213-PS | |
| 100 kDa cut-off concentrator | Thermo Fisher Scientific | Cat# 88533 | |
| 6-well plate | Corning | Cat# 43016 | |
| 96-well plate | Corning | Cat# 3917 | |
| Aprotinin | Sigma-Aldrich | Cat# 9087-70-1 | |
| Apyrase | NEB | Cat# M0398S | |
| Baculovirus transfection reagent | Thermo Fisher Scientific | Cat# 10362100 | For the preparation of P0 baculovirus |
| Benzamidine | Sigma-Aldrich | Cat# B6506 | |
| CHO-K1 | ATCC | N/A | |
| CHS | Sigma-Aldrich | Cat# C6512 | |
| CryoSPARC | Punjani, A., et al.,2017 | https://cryosparc.com/ | |
| DH5α competent E.coli | Thermo Fisher Scientific | Cat# EC0112 | |
| D-Luciferin-Potassium Salt | Sigma- Aldrich | Cat# 50227 | |
| DMSO | Sigma- Aldrich | Cat# D2438 | |
| EDTA | Thermo Fisher Scientific | Cat# S311-500 | |
| ESF921 cell culture medium | Expression Systems | Cat# 96-001 | |
| Excel | microsoft | N/A | |
| F12 medium | Invitrogen | Cat# 11765 | |
| FBS | Cell Box | Cat# SAG-01U-02 | |
| Flag resin | Sigma- Aldrich | Cat# A4596 | |
| Forskolin | APExBIO | Cat# B1421 | |
| Gctf | Zhang, 2016 | https://www.mrc-lmb.cam.ac.uk/kzhang/Gctf/ | |
| GDN | Anatrace | Cat# GDN101 | |
| Gel filtration column | GE healthcare | Cat# 28990944 | |
| Gen5 3.11 | BIO-TEK | N/A | |
| Gentamicin | Solarbio | Cat# L1312 | |
| GloSensor cAMP assay kit | Promega | Cat# E1291 | Gi-inhibition cAMP assay kit |
| GloSensor plasmid | Promega | Cat# E2301 | Sensor plasmid |
| Grace’s medium | GIBCO | Cat# 11595030 | |
| GraphPad Prism 8 | Graphpad | N/A | |
| HBSS | Thermo Fisher Scientific | Cat# 88284 | |
| HEPES | Sigma- Aldrich | Cat# H4034 | |
| jetPRIME Reagent | Polyplus Transfection | Cat# 114-15 | transfection reagent |
| Janamycin | Solarbio | Cat# K1030 | |
| LB medium | Invitrogen | Cat# 12780052 | |
| Leupeptin | Sigma-Aldrich | Cat# L2884 | |
| LMNG | Anatrace | Cat# NG310 | |
| MotionCor2 | (Zheng et al., 2017) | https://emcore.ucsf.edu/ucsf-software | |
| NanoCab | Thermo Fisher Scientific | Cat# 1121822 | |
| PBS | Invitrogen | Cat# 14190-144 | |
| pcDNA3.1-HA-FLAG-S1PRs | GenScript | N/A | |
| pFastBac1-Gαi | GenScript | N/A | |
| pFastBac1-HA-FLAG-T4L-S1PRs-His10 | GenScript | N/A | |
| pFastBacdual-Gβ1γ2 | GenScript | N/A | |
| PureLink HiPure Plasmid Miniprep Kit | Invitrogen | Cat# K210003 | For the preparation of plasmids and P0 baculovirus |
| Q5 site-Directed Mutagenesis kit | NEB | Cat# E0554S | For the preparation of plasmids |
| Quantifoil | Quantifoil | Cat# 251448 | |
| RELION-3.1 | (Zivanov et al., 2018) | https://www2.mrc-lmb.cam.ac.uk/relion | |
| S1PRs cDNA | addgene | N/A | |
| scFv16 | Invitrogen | Cat# 703976 | |
| Sf9 | Expression Systems | N/A | |
| Siponimod | Selleck | Cat# S7179 | |
| sodium cholate | Sigma-Aldrich | Cat# C1254 | |
| Synergy H1 microplate reader | BIO-TEK | N/A | |
| Synthetic T4L DNA (sequence) | N/A | N/A | Aacatcttcgagatgctgcgcatcgacgaagg cctgcgtctcaagatttacaagaataccgaagg ttattacacgattggcatcggccacctcctgaca aagagcccatcactcaacgctgccaagtctga actggacaaagccattggtcgcaacaccaac ggtgtcattacaaaggacgaggcggagaaac tcttcaaccaagatgtagatgcggctgtccgtgg catcctgcgtaatgccaagttgaagcccgtgt atgactcccttgatgctgttcgccgtgcagcctt gatcaacatggttttccaaatgggtgagaccgg agtggctggttttacgaactccctgcgcatgctcc agcagaagcgctgggacgaggccgcagtga atttggctaaatctcgctggtacaatcagacacc taaccgtgccaagcgtgtcatcactaccttccg tactggaacttgggacgcttac |
| TCEP | Thermo Fisher Scientific | Cat# 77720 | |
| Tetracycline | Solarbio | Cat# T8180 | |
| Vitrobot Mark IV | Thermo Fisher Scientific | N/A |
Riferimenti
- Verstockt, B., et al. Sphingosine 1-phosphate modulation and immune cell trafficking in inflammatory bowel disease. Nature Reviews: Gastroenterology & Hepatology. , 1-16 (2022).
- Rosen, H., Stevens, R. C., Hanson, M., Roberts, E., Oldstone, M. B. Sphingosine-1-phosphate and its receptors: structure, signaling, and influence. Annual Review of Biochemistry. 82, 637-662 (2013).
- Cartier, A., Hla, T. Sphingosine 1-phosphate: Lipid signaling in pathology and therapy. Science. 366 (6463), 5551 (2019).
- Jozefczuk, E., Guzik, T. J., Siedlinski, M. Significance of sphingosine-1-phosphate in cardiovascular physiology and pathology. Pharmacological Research. 156, 104793 (2020).
- Kihara, Y., Maceyka, M., Spiegel, S., Chun, J. Lysophospholipid receptor nomenclature review: IUPHAR Review 8. British Journal of Pharmacology. 171 (15), 3575-3594 (2014).
- Bryan, A. M., Del Poeta, M. Sphingosine-1-phosphate receptors and innate immunity. Cellular Microbiology. 20 (5), 12836 (2018).
- Pelletier, D., Hafler, D. A. Fingolimod for multiple sclerosis. New England Journal of Medicine. 366 (4), 339-347 (2012).
- Obinata, H., Hla, T. Sphingosine 1-phosphate and inflammation. International Immunology. 31 (9), 617-625 (2019).
- Pyne, N. J., Pyne, S. Sphingosine 1-phosphate and cancer. Nature Reviews: Cancer. 10 (7), 489-503 (2010).
- Abu-Farha, M., et al. The role of lipid metabolism in COVID-19 virus infection and as a drug target. International Journal of Molecular Sciences. 21 (10), 3544 (2020).
- Chun, J., Kihara, Y., Jonnalagadda, D., Blaho, V. A. Fingolimod: lessons learned and new opportunities for treating Multiple Sclerosis and other disorders. Annual Review of Pharmacology and Toxicology. 59, 149-170 (2019).
- Murakami, A., et al. Sphingosine 1-phosphate (S1P) regulates vascular contraction via S1P3 receptor: investigation based on a new S1P3 receptor antagonist. Molecular Pharmacology. 77 (4), 704-713 (2010).
- Cao, L., et al. Siponimod for multiple sclerosis. Cochrane Database of Systematic Reviews. 11, (2021).
- Scott, L. J. Siponimod: a review in secondary progressive Multiple Sclerosis. CNS Drugs. 34 (11), 1191-1200 (2020).
- Lamb, Y. N. Ozanimod: first approval. Drugs. 80 (8), 841-848 (2020).
- Scott, F. L., et al. Ozanimod (RPC1063) is a potent sphingosine-1-phosphate receptor-1 (S1P1) and receptor-5 (S1P5) agonist with autoimmune disease-modifying activity. British Journal of Pharmacology. 173 (11), 1778-1792 (2016).
- McGowan, E. M., Haddadi, N., Nassif, N. T., Lin, Y. Targeting the SphK-S1P-SIPR pathway as a potential therapeutic approach for COVID-19. International Journal of Molecular Sciences. 21 (19), 7189 (2020).
- O'Sullivan, C., Dev, K. K. The structure and function of the S1P1 receptor. Trends in Pharmacological Sciences. 34 (7), 401-412 (2013).
- Liao, M., Cao, E., Julius, D., Cheng, Y. Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature. 504 (7478), 107-112 (2013).
- Bai, X. C., McMullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- Murata, K., Wolf, M. Cryo-electron microscopy for structural analysis of dynamic biological macromolecules. Biochimica et Biophysica Acta General Subjects. 1862 (2), 324-334 (2018).
- Zhang, M., et al. Cryo-EM structure of an activated GPCR-G protein complex in lipid nanodiscs. Nature Structural & Molecular Biology. 28 (3), 258-267 (2021).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews: Drug Discovery. 17 (7), 471-492 (2018).
- Ishchenko, A., Gati, C., Cherezov, V. Structural biology of G protein-coupled receptors: new opportunities from XFELs and cryoEM. Current Opinion in Structural Biology. 51, 44-52 (2018).
- Yang, D., et al. G protein-coupled receptors: structure- and function-based drug discovery. Signal Transduction and Targeted Therapy. 6 (1), 7 (2021).
- Yuan, Y., et al. Structures of signaling complexes of lipid receptors S1PR1 and S1PR5 reveal mechanisms of activation and drug recognition. Cell Research. 31 (12), 1263-1274 (2021).
- Zhao, C., et al. Structural insights into sphingosine-1-phosphate recognition and ligand selectivity of S1PR3-Gi signaling complexes. Cell Research. 32 (2), 218-221 (2022).
- Xu, Z., et al. Structural basis of sphingosine-1-phosphate receptor 1 activation and biased agonism. Nature Chemical Biology. 18, 281-288 (2022).
- Liu, Y. F., Ghahremani, M. H., Rasenick, M. M., Jakobs, K. H., Albert, P. R. Stimulation of cAMP synthesis by Gi-coupled receptors upon ablation of distinct Galphai protein expression. Gi subtype specificity of the 5-HT1A receptor. Journal of Biological Chemistry. 274 (23), 16444-16450 (1999).
- Buccioni, M., et al. Innovative functional cAMP assay for studying G protein-coupled receptors: application to the pharmacological characterization of GPR17. Purinergic Signalling. 7 (4), 463-468 (2011).
- Wang, F. I., Ding, G., Ng, G. S., Dixon, S. J., Chidiac, P. Luciferase-based GloSensor cAMP assay: Temperature optimization and application to cell-based kinetic studies. Methods. , (2021).
- Audet, M., et al. Small-scale approach for precrystallization screening in GPCR X-ray crystallography. Nature Protocols. 15 (1), 144-160 (2020).
- Sgro, G. G., Costa, T. R. D. Cryo-EM grid preparation of membrane protein samples for single particle analysis. Frontiers in Molecular Biosciences. 5, 74 (2018).
- White, J. B. R., et al. Single particle cryo-electron microscopy: from sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Thompson, R. F., Iadanza, M. G., Hesketh, E. L., Rawson, S., Ranson, N. A. Collection, pre-processing and on-the-fly analysis of data for high-resolution, single-particle cryo-electron microscopy. Nature Protocols. 14 (1), 100-118 (2019).
- Fernandez-Leiro, R., Scheres, S. H. W. A pipeline approach to single-particle processing in RELION. Acta Crystallographica Section D. 73 (6), 496-502 (2017).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Brilot, A. F., et al. Beam-induced motion of vitrified specimen on holey carbon film. Journal of Structural Biology. 177 (3), 630-637 (2012).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14 (4), 331-332 (2017).
- Zhang, K. Gctf: Real-time CTF determination and correction. Journal of Structural Biology. 193 (1), 1-12 (2016).
- Scheres, S. H. Semi-automated selection of cryo-EM particles in RELION-1.3. Journal of Structural Biology. 189 (2), 114-122 (2015).
- Liu, S., et al. Differential activation mechanisms of lipid GPCRs by lysophosphatidic acid and sphingosine 1-phosphate. Nature Communications. 13 (1), 731 (2022).
- Duan, J., et al. Cryo-EM structure of an activated VIP1 receptor-G protein complex revealed by a NanoBiT tethering strategy. Nature Communications. 11 (1), 4121 (2020).
- DiIorio, M. C., Kulczyk, A. W. A robust single-particle cryo-electron microscopy (cryo-EM) processing workflow with cryoSPARC, RELION, and Scipion. Journal of Visualized Experiments. (179), e63387 (2022).
- Pradelles, P., Grassi, J., Chabardes, D., Guiso, N. Enzyme immunoassays of adenosine cyclic 3',5'-monophosphate and guanosine cyclic 3',5'-monophosphate using acetylcholinesterase. Analytical Chemistry. 61 (5), 447-453 (1989).
- Jiang, L. I., et al. Use of a cAMP BRET sensor to characterize a novel regulation of cAMP by the sphingosine 1-phosphate/G13 pathway. Journal of Biological Chemistry. 282 (14), 10576-10584 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon