È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi della forma della linea di spettri NMR dinamici per caratterizzare i riarrangiamenti delle sfere di coordinazione in un complesso di poliidruro di renio chirale
In questo articolo
Riepilogo
L'analisi della forma della linea degli spettri NMR raccolti su un intervallo di temperature serve come guida per il riarrangiamento degli atomi della sfera di coordinazione interna in un complesso chirale, a otto coordinate, poliidruro di renio (V), ReH5 (PPh3) 2 (ammina sec-butile). L'analisi della forma della linea viene anche utilizzata per determinare i parametri di attivazione ΔH‡, ΔS‡ e ΔG‡ per questi riarrangiamenti atomici.
Abstract
La spettroscopia di risonanza magnetica nucleare (NMR) a soluzione dinamica è il metodo tipico per caratterizzare i riarrangiamenti dinamici degli atomi all'interno della sfera di coordinazione per i complessi di poliidruro dei metalli di transizione. L'adattamento della forma della linea degli spettri NMR dinamici può portare a stime per i parametri di attivazione dei processi di riarrangiamento dinamico. Una combinazione di spettroscopia NMR dinamica 31 P-{1 H} di atomi di fosforo legati al metallo con spettroscopia NMR dinamica 1H-{31P} di ligandi idruriche può identificare riarrangiamenti di leganti idruro che si verificano in concomitanza con un riarrangiamento atomico di fosforo. Per le molecole che presentano una tale coppia accoppiata di riarrangiamenti, la spettroscopia NMR dinamica può essere utilizzata per testare modelli teorici per i riarrangiamenti del ligando. La spettroscopia NMR dinamica 1H-{31P} e il line shape fitting possono anche identificare la presenza di un processo di scambio che sposta uno specifico ligando dell'idruro oltre la sfera di coordinazione interna del metallo attraverso uno scambio protonico con una molecola di solvente come l'acqua avventizia. La preparazione di un nuovo composto, ReH5 (PPh3) 2 (sec-butililammina), che esemplifica molteplici processi di riarrangiamento dinamico è presentata insieme all'adattamento della forma della linea degli spettri NMR dinamici del complesso. I risultati del fitting della forma della linea possono essere analizzati dall'equazione di Eyring per stimare i parametri di attivazione per i processi dinamici identificati.
Introduzione
La spettroscopia NMR è comunemente usata per caratterizzare i processi dinamici che si verificano all'interno o tra le molecole. Per molti semplici riarrangiamenti intramolecolari, la stima di ΔG‡ è semplice come misurare la differenza di frequenza, Δν, tra due risonanze al limite di scambio lento e determinare la temperatura di coalescenza per quelle stesse risonanze (Figura 1)1. La relazione,
ΔG‡ = 4,575 x 10-3 kcal/mol x T c [9,972 + log (Tc/Δν)]
dove Tc è la temperatura di coalescenza per una coppia di risonanze che rappresentano la forma di scambio lento di un campione dinamico, può essere usata per risolvere l'energia libera di attivazione per tale riarrangiamento dinamico. Sistemi dinamici più complessi richiedono l'adattamento a forma di linea di spettri NMR dinamici o un'altra tecnica NMR come la spettroscopia di scambio bidimensionale (2D-EXSY) o la spettroscopia ad effetto Overhauser a telaio rotante bidimensionale (2D-ROESY) per stimare i parametri di attivazione.
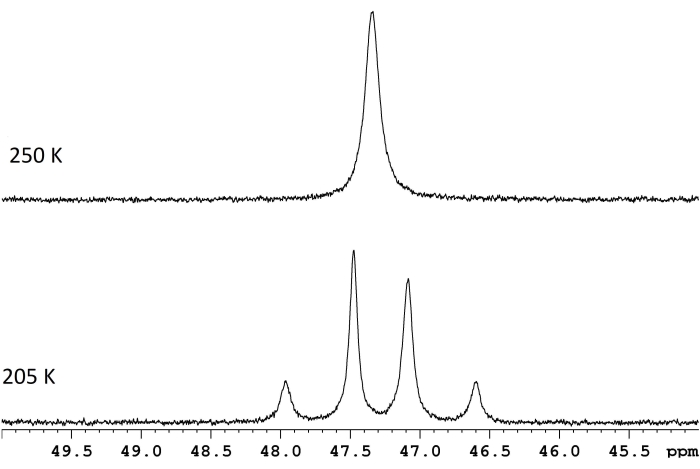
Figura 1: Spettri NMR per una soluzione d 8-toluene di ReH5(PPh3)2(sec-butililammina) a due temperature. La differenza di frequenza tra i due doppietti a scambio lento (traccia inferiore, 117,8 Hz) e una temperatura di coalescenza di 250 K (traccia superiore) corrispondono a una barriera di energia (ΔG‡) di 11,8 kcal/mol. Fare clic qui per visualizzare una versione più grande di questa figura.
Il Line Shape Fitting degli spettri NMR dinamici è una tecnica comune che è stata a lungo utilizzata per la stima dei parametri di attivazione che descrivono i riarrangiamenti dinamici per sostanze con un'energia di attivazione di circa 5-25 kcal/mol 2,3,4,5. La determinazione delle barriere energetiche allo scambio protonico tra acqua e molecole amminiche6, la barriera energetica alla rotazione attorno al legame C-N nella dimetilformammide7, o la dimensione generale delle frazioni organiche8 sono solo alcuni esempi delle molte proprietà che sono state valutate attraverso il line shape fitting di spettri NMR dinamici. Questo manoscritto dimostra l'uso del line shape fitting per caratterizzare i processi dinamici intermolecolari e intramolecolari che si verificano per il complesso ReH5 (PPh3) 2 (sec-butililammina). Gli obiettivi di questo e di altri simili esperimenti NMR sono: 1) caratterizzare tutti i processi di scambio atomico dinamico intramolecolare osservabili NMR se presenti, 2) identificare e caratterizzare i processi di scambio atomico dinamico intramolecolare osservabili NMR se presenti, 3) identificare gli scambi atomici intramolecolari correlati che si verificano, in questo esempio, sia per gli atomi di idrogeno che di fosforo, e 4) per l'esempio qui presentato, confrontare due modelli pubblicati per i processi dinamici che si verificano nel complesso ReH5 (PPh3) 2 (sec-butililammina).
I sistemi di poliidruro di renio (V) a otto coordinate sono sistemi dinamici complessi in cui i ligandi partecipano a più processi dinamici e gli atomi di fosforo possono partecipare a un singolo processo dinamico che è un secondo aspetto di un processo di scambio di liganti idruri. 9,10,11,12,13,14,15,16,17,18 ,19,20,21,22,23,24,25,26,
27,28,29. I complessi di poliidruro di renio(V) pseudododecaedrali a otto coordinate, pseudododecaedrali adottano una geometria molecolare (Figura 2), che può essere descritta come una coppia di trapezi ortogonali di ligandi17,26. I vertici sui bordi lunghi dei trapezi sono comunemente etichettati come siti B e, nei complessi di poliidruro di renio, sono solitamente i siti occupati da ligandi donatori neutri a due elettroni come fosfine terziarie o ligandi amminici. I vertici sui bordi corti dei trapezi sono comunemente etichettati come siti A e sono tipicamente occupati da ligandi anionici, donatori di due elettroni, idruro. Gli spettri NMR a temperatura ambiente dei complessi di poliidruro di renio (V) sono, tipicamente, ingannevolmente semplici a causa dei numerosi processi dinamici che si verificano nelle soluzioni a temperatura ambiente.
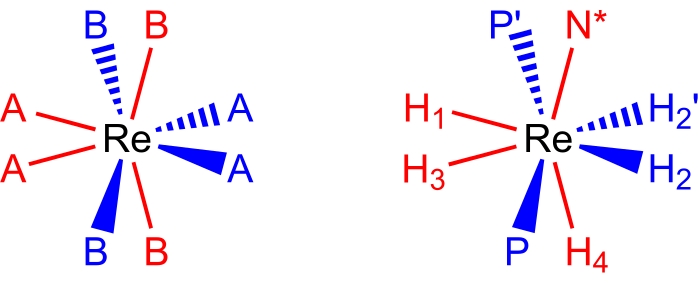
Figura 2: Un insieme di coordinazione dodecaedrica (a sinistra) e il complesso ReH5 (PPh3) 2 (sec-butililammina) dalla stessa prospettiva (a destra). I siti di colore rosso rappresentano i siti di coordinamento che formano un trapezio verticale e i siti di colore blu rappresentano i siti di coordinamento che formano un trapezio orizzontale. Fare clic qui per visualizzare una versione ingrandita di questa figura.
I complessi della forma ReH5(PPh3)2(ammina) sono la classe più studiata di complessi di poliidruro di renio rispetto ai processi dinamici 9,10,12,13,16,30,31. Sono stati identificati tre processi dinamici (Figura 3) per complessi ReH5(PPh3)2(ammina): 1) uno scambio protonico tra l'unico ligando dell'idruro del sito B e un protone da una molecola d'acqua (accidentale o intenzionale)9,13, 2) uno scambio del tornello di una coppia di ligandi di idruro del sito A con un ligando adiacente all'idruro del sito B 9, 11,13,30,31 e 3) un'inversione sterica (o pseudorotazione) che si manifesta come uno scambio a coppie dei ligandi idruro del sito A e un movimento a coppie degli atomi del sito B sul lato opposto del centro del renio (come illustrato nella Figura 4)4,5,6,8,26,27 . Il movimento degli atomi del sito B sul lato opposto del renio è osservabile mediante spettroscopia NMR dinamica come: 1) un processo che rende i protoni inequivalenti 3 e 5 di N = equivalente piridina a temperatura ambiente10,30,31, 2) un processo che fa sì che gli isomeri E e Z di N = leganti amminici aromatici sostituiti in modo asimmetrico subiscano uno scambio rapido a temperatura ambiente9, 10,13,30,31, o 3) un processo che provoca un rapido scambio delle prospettive steriche di una coppia diastereotopica di atomi di fosforo rispetto ad un centro chirale situato sul ligando amminico 9,30,31. Il complesso chirale ReH5 (PPh3) 2 (sec-butililammina) precedentemente non riportato offre l'opportunità di descrivere in generale i metodi che possono essere utilizzati per identificare e caratterizzare i riarrangiamenti dinamici dei complessi di poliidruro di renio.
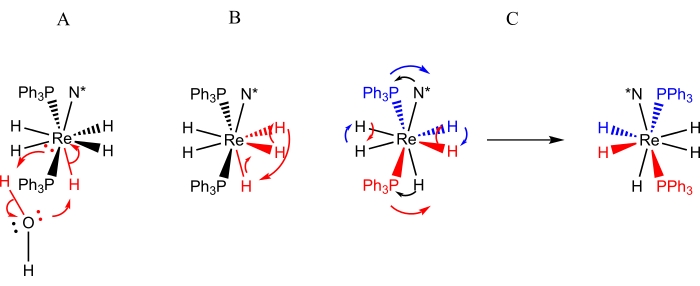
Figura 3: Rappresentazioni dei processi dinamici osservati dalla spettroscopia NMR per soluzioni di ReH5(PPh3)2(sec-butililammina). La rappresentazione A raffigura lo scambio di un singolo protone di acqua avventizia per l'unico ligando dell'idruro del sito B. La rappresentazione B raffigura lo scambio del tornello di tre ligandi idruro adiacenti, due dei quali risiedono nel sito A mentre il terzo è l'unico ligando idruro del sito B. La rappresentazione C rappresenta sia lo scambio a coppie di ligandi di idruro del sito A sia l'inversione sterica degli atomi di fosforo rispetto al ligando amminico chirale (N*). Va notato che lo scambio a coppie del ligando dell'idruro del sito A non richiede uno spostamento dei ligandi dell'idruro del sito A sul lato opposto del centro del renio. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Per sistemi chimici come i complessi di poliidruro di renio, che presentano un insieme complesso di processi dinamici, il line shape fitting degli spettri NMR dinamici è la tecnica NMR più utilizzata per caratterizzare i processi 9,11,13,16,21,29. EXSY 9,32 o 2D-ROESY11 bidimensionali sono tecniche NMR dinamiche alternative che possono essere utilizzate anche per caratterizzare quantitativamente i processi dinamici. Gli spettri EXSY bidimensionali sono tipicamente misurati nel dominio della temperatura a scambio lento; gli spettri ROESY bidimensionali sono tipicamente misurati nel dominio della temperatura di scambio rapido. Entrambe le tecniche bidimensionali possono richiedere molto tempo nello spettrometro per l'acquisizione dei dati, in quanto ciascuna delle tecniche sta acquisendo un set di dati molto più grande, a una data temperatura, rispetto ai set di dati unidimensionali necessari per l'analisi del line shape fitting. Semplici processi dinamici che sono ben compresi, come lo scambio dinamico dei due gruppi metilici della dimetilformammide, possono essere facilmente caratterizzati da una qualsiasi delle tre tecniche NMR. Sistemi più complessi, come ReH5 (PPh3) 2 (sec-butilammina), in cui i singoli ligandi dell'idruro partecipano a molteplici processi dinamici, o sistemi che non sono necessariamente ben compresi, come un nuovo complesso di poliidruro di metalli di transizione che può o non può scambiare protoni tra un ligando di idruro e acqua avventizia, sono più facilmente caratterizzati quantitativamente dal metodo NMR di adattamento della linea che dai metodi NMR bidimensionali. A differenza dei metodi NMR bidimensionali, il metodo line shape fitting fornisce una visualizzazione facilmente interpretabile della corrispondenza tra un modello testato e i dati sperimentali, nonché la prova visiva di uno scambio che sposta un ligando idruro oltre la sfera di coordinazione interna del renio. Sulla base delle altezze dei picchi e delle forme dei picchi negli spettri di scambio lento, anche un sistema dinamico complesso come ReH5 (PPh3) 2 (sec-butililammina) può portare a una serie iniziale di modelli di scambio facilmente testati. Inoltre, quando sono stati riportati più modelli teorici per una trasformazione molecolare, l'adattamento della forma della linea degli spettri NMR dinamici può consentire un confronto visivo di ciascun modello rispetto agli spettri osservati.
Oltre alle tre tecniche NMR sopra menzionate, esperimenti NMR di sostituzione isotopica che coinvolgono D2O o HD sono stati utilizzati per dimostrare qualitativamente lo scambio intermolecolare di atomi per complessi sistemi di poliidruro di renio, ma non sono stati utilizzati per caratterizzazioni quantitative 9,33,34,35. I calcoli teorici presentano un metodo aggiuntivo per caratterizzare i processi dinamici di sistemi dinamici complessi30,31,36. I calcoli teorici hanno il vantaggio rispetto al line shape fitting in quanto possono essere utilizzati per differenziare tra possibilità che non possono essere distinte dall'analisi line shape fitting. Ad esempio, calcoli teorici sono stati utilizzati per descrivere uno scambio che coinvolge tre ligandi idruro adiacenti su determinati complessi di renio (V) come uno scambio del tornello di tutti e tre i ligandi dell'idruro, piuttosto che una coppia alternata di scambi a coppie con ogni scambio a coppie comprendente un unico legante dell'idruro e uno dei due ligandi dell'idruro chimicamente equivalenti30, 31. I risultati dei calcoli teorici sono tipicamente confrontati con caratterizzazioni quantitative osservate sperimentalmente da una delle tre tecniche NMR sopra menzionate come controllo sulla validità dei risultati calcolati.
L'adattamento della forma della linea degli spettri NMR dinamici sfrutta il cambiamento nell'aspetto degli spettri NMR che si verifica quando i nuclei NMR-attivi si spostano tra diversi ambienti chimici durante una misurazione NMR. Gli spettri NMR a scambio lento (spettri con risonanze lorentziane indipendenti per ogni insieme di nuclei in scambio) si verificano a temperature in cui la differenza di frequenza tra le risonanze per i nuclei che scambiano è grande rispetto alla velocità di scambio dei nuclei37. Gli spettri NMR a scambio rapido (spettri con una singola risonanza lorentziana per lo scambio di nuclei) si verificano a temperature in cui la velocità di scambio dei nuclei è molto maggiore della differenza di frequenza tra le risonanze a scambio lento37. I tassi di cambio intermedi si verificano per le temperature tra il dominio della temperatura di scambio lento e il dominio della temperatura di scambio rapido37. Se sono noti i parametri fondamentali della frequenza di Larmor, dello spostamento chimico dei nuclei in scambio, delle costanti di accoppiamento (se presenti) per i nuclei in scambio e delle popolazioni relative di ciascun tipo di nucleo, le costanti di velocità per gli scambi putativi tra nuclei possono essere determinate confrontando spettri simulati con spettri osservati a diverse temperature intermedie. Buoni adattamenti per simulazioni a diverse temperature si traducono in dati costanti di temperatura e velocità che possono essere utilizzati con l'equazione di Eyring per stimare i parametri di attivazione per gli scambi putativi. I risultati del metodo sono risultati accurati e riproducibili.
Protocollo
1. Preparazione del campione
- Preparazione di ReH7(PPh3)235
- Combinare 0,15 g di boroidruro di sodio e 0,41 g di ReOCl 3(PPh3)2 in un matraccio a fondo tondo da 100 mL a due o tre colli munito di setto di gomma e porta per gas, oppure in un matraccio Kjeldahl da 100 mL (con una porta per gas a braccio laterale) munito di un setto di gomma (figura supplementare 1).
- Aggiungete una barra di rotazione al recipiente di reazione.
- In una cappa aspirante, utilizzare un pezzo di tubo di pressione di gomma per collegare la porta del gas del recipiente di reazione con uno dei rubinetti di arresto di un doppio collettore in vetro per vuoto e azoto gassoso. Collegare il collettore del vuoto in vetro a una pompa per vuoto con tubo di pressione in gomma e collegare il collettore di azoto in vetro a una bombola di azoto gassoso regolata.
- Collegare il gas di uscita dal collettore del gas azoto a un rubinetto che può essere utilizzato per dirigere il gas sfiatato attraverso una colonna di olio minerale di 2 cm o una colonna di mercurio di 2 cm.
- Aprire il rubinetto sulla bombola di azoto e regolare la pressione sul gas che scorre a 34 libbre per pollice quadrato. Sfiatare il flusso di azoto gassoso attraverso il gorgogliatore di mercurio.
- Evacuare il gas all'interno del recipiente di reazione regolando il rubinetto sul collettore di vetro per collegare il recipiente al collettore a vuoto. Riempire il recipiente di reazione con azoto gassoso cambiando il rubinetto del collettore di vetro in modo che colleghi il collettore del gas con il recipiente di reazione.
- Ripetere i passaggi 1.1.5 e 1.1.6 altre due volte per sostituire completamente l'aria nel recipiente di reazione con azoto gassoso. Raffreddare il pallone e il suo contenuto in un bagno di ghiaccio.
- Aggiungere 8 mL di acqua deossigenata e 8 mL di tetraidrofurano deossigenato ai solidi nel recipiente di reazione tramite una siringa. Commutare il rubinetto di sfiato del gas in modo che il gas si sfiati attraverso il gorgogliatore di olio minerale. Mescolare leggermente la sospensione nel bagno di ghiaccio per 15 minuti. Rimuovere il recipiente di reazione dal bagno di ghiaccio dopo i primi 15 minuti di agitazione.
- Lasciare che la miscela continui a mescolare per altri 45 minuti. Notare il colore della miscela di reazione come indicatore di quando la reazione è stata completata. Un colore della miscela di reazione da marrone ad arancione (Figura supplementare 1) indica che la reazione ha raggiunto il suo punto finale.
- Dopo aver ottenuto un colore da arancione a marrone chiaro per la miscela di reazione, filtrare la miscela attraverso un imbuto di vetro sinterizzato medio da 30 ml. Lavare il solido recuperato tre volte ciascuna con 15 ml di porzioni di acqua, metanolo ed etere etilico. Asciugare il solido sotto vuoto per rimuovere qualsiasi solvente adsorbito.
NOTA: La reazione produce generalmente tra 0,20 g e 0,25 g di prodotto.
- Preparazione di ReH5(PPh3)2(sec-butililammina)
- Pesare 0,070 g di ReH7(PPh3)2 e trasferirlo in un matraccio a collo singolo a fondo tondo da 50 mL contenente una barra di rotazione. Montare il pallone su un condensatore dotato di una porta per gas. Deossigenare il recipiente di reazione utilizzando il metodo pompa e riempimento dai punti 1.1.3-1.1.7.
- Aggiungere un volume di 8 mL di tetraidrofurano deossigenato al recipiente di reazione attraverso una siringa rompendo il giunto tra il matraccio a fondo rotondo e il condensatore. Aggiungere un volume di 0,2 mL di sec-butile ammina in modo simile. Passare il rubinetto di sfiato del gas in modo che il gas si sfiati sul gorgogliatore di olio minerale.
- Riscaldare la miscela di reazione a riflusso a 65 °C con un mantello riscaldante collegato a un trasformatore AC variabile impostato a 40 su una scala da 0 a 140 per 40 min. Raffreddare la miscela di reazione a una temperatura che consenta una comoda manipolazione del pallone.
- Versare la miscela di reazione in 25 mL di metanolo in un matraccio Erlenmeyer da 125 ml. Mescolare energicamente il composto per 5 minuti. Aggiungere 5 ml di acqua per indurre la formazione di un precipitato giallo flocculante.
- Raccogliere il precipitato giallo mediante filtrazione sottovuoto in un imbuto di vetro sinterizzato. Lavare il solido con 15 ml di metanolo. Asciugare il solido sotto vuoto. Dopo questo processo, la resa tipica del prodotto è di 0,035 g.
2. Acquisizione e analisi di spettri NMR
- Misura di spettri NMR dinamici
- Preparare un campione NMR con circa 8 mg del complesso ReH5 (PPh3) 2 (sec-butililammina) in circa 0,8 ml di d8-toluene. Inserire il campione nello strumento.
- Fare clic sulla scheda File e selezionare Nuovo dalle opzioni visualizzate per aprire una finestra di dialogo utilizzata per creare un esperimento NMR.
- Crea un esperimento di 1ora completando i passaggi seguenti.
- Assegna un nome di cartella per il nuovo esperimento completando la casella di input Nome con un nome file univoco. Assegna un numero di esperimento, ad esempio 1, per l'esperimento 1H nella casella EXPNO .
- Assegnare un numero di processo pari a 1 per l'esperimento nella casella PROCNO . Assegnare la cartella in una directory utilizzando l'elenco a discesa per DIR. Identificare il solvente su cui lo strumento si bloccherà dalle scelte del solvente a discesa.
- Scegliere la directory che contiene i parametri per l'esperimento 1H dall'elenco a discesa delle directory in Dirs esperimento. Seleziona l'esperimento Proton dalle opzioni nell'elenco a discesa Esperimento e (facoltativo) aggiungi un titolo per i dati nella casella Riempimento titolo .
- Immettere un comando Eda nella riga di comando e regolare i parametri in base alle esigenze per soddisfare le descrizioni dell'esperimento fornite nel secondo paragrafo della sezione Discussione riportata di seguito.
- Fare clic sulla scheda Finestra, selezionare Nuova finestra dall'elenco e ripetere i passaggi 2.1.3.1-2.1.3.8 per preparare un esperimento 1 H-{31P} utilizzando un valore EXPNO pari a 2 per differenziare l'esperimento dall'esperimento 1H creato in precedenza.
- Fare clic sulla scheda Finestra, selezionare Nuova finestra dall'elenco e ripetere i passaggi 2.1.3.1-2.1.3.8 per preparare un esperimento 31 P-{1 H} utilizzando un valore EXPNO pari a 3 per differenziare l'esperimento dagli esperimenti 1 H e 1 H-{31P} costruiti in precedenza (vedere la Tabella supplementare 1per informazioni dettagliate sui parametri).
- Immettere un comando Blocca nella riga di comando e selezionare la scelta d 8-toluene dall'elenco. Fare clic su OK per accettare la scelta del solvente. Immettere un comando Atma nella riga di comando, se necessario, a causa di una sonda in banda X del nucleo variabile, per ridurre al minimo l'energia riflessa alle frequenze di Larmor per 1H e 31P sullo strumento.
- Immettere un comando Ro nella riga di comando, digitare il valore 20 nella casella e fare clic sul pulsante Avvia rotazione . Immettere un comando Shim nella riga di comando. Scegliere una routine di shim automatica appropriata, ad esempio Topshim , dall'elenco delle routine di shim e fare clic sul pulsante Start .
- Immettere un comando Rga nella riga di comando. Scegliere la selezione Regolazione automatica ricevitore e fare clic su OK. A sua volta, misurare i tre spettri del campione a temperatura ambiente utilizzando 64 scansioni per ogni spettro con un comando Go sulla riga di comando.
- Trasforma i dati di un esperimento in uno spettro con un comando Efp immesso nella riga di comando.
- Regolare la fase dello spettro utilizzando i seguenti comandi.
- Fare clic sulla scheda Fase seguita da un clic sulla scheda Regola fase. Passare il cursore sul pulsante 0 sulla barra degli strumenti di selezione e tenere premuto il pulsante sinistro del mouse in modo che il pulsante 0 diventi verde.
- Con il tasto sinistro del mouse tenuto premuto, ruotare il mouse in avanti o indietro fino a quando la linea di base è piatta su tutto lo spettro e tutte le risonanze vengono visualizzate come assorbanze (i picchi salgono al di sopra della linea di base).
- Se la linea di base non può essere resa piatta con il solo pulsante 0, regolare il pulsante 1 come descritto nei passaggi 2.1.10.1 e 2.1.10.2 e il pulsante 0, finché la linea di base non è piatta per l'intera finestra spettrale.
- Salvate la regolazione di fase con i dati facendo clic sul pulsante Salva e ritorna sulla barra degli strumenti di phasing.
- Regolare il numero di scansioni per ogni misurazione, se necessario, in base al rapporto segnale-rumore nello spettro, tenendo presente che il rapporto segnale-rumore diminuisce tipicamente a temperature più basse a causa della de-coalescenza dei segnali in singole risonanze (Figura 4).
- Preparare lo spettrometro per il controllo della temperatura secondo le istruzioni del fornitore. Immettere una portata di 200 L/h per il gas di raffreddamento e una temperatura target di 290 K per la sonda. Lasciare stabilizzare lo spettrometro alla temperatura target per 2 minuti. Aumentare la portata del gas di raffreddamento, se necessario, a 210 o 220 L/h per stabilizzare la temperatura.
- Eseguire lo spessore del campione a 290 K come indicato al punto 2.1.7. Modificare il nome del file per ciascuno degli spettri misurati in precedenza aggiungendo la temperatura alla fine del nome del file (punti 2.1.2 e 2.1.3.1) e acquisire un insieme di tre spettri a 290 K.
- Aumentare la portata del gas di raffreddamento di ≥ 30 L/h, se necessario per stabilizzare alla temperatura successiva, e diminuire la temperatura target di 10 K. Lasciare stabilizzare lo spettrometro alla temperatura successiva per 2 minuti e quindi eseguire lo spessore del campione come al punto 2.1.7. Misurare l'insieme di tre spettri.
- Ripetere i punti 2.1.13 e 2.1.14 secondo necessità per acquisire spettri fino alla temperatura più bassa desiderata.
NOTA: Una temperatura di 200 K è solitamente sufficiente per un set completo di dati adatto a determinare i parametri di attivazione per i processi dinamici del campione. - Riscaldare il campione a temperatura ambiente con incrementi di 10 K. Stabilizzare la temperatura per 2 minuti a ciascuna temperatura prima di riscaldare nuovamente il campione per evitare danni al rivestimento in vetro della sonda.
- Analisi della forma della linea degli spettri misurati
- All'interno del programma NMR fare clic sulla barra dei comandi in alto a sinistra della finestra e selezionare Apri dal menu a discesa. Selezionare Apri dati NMR memorizzati in formato standard. Fare clic su OK per aprire la finestra Esplora file per il programma.
- Passare alla cartella per i dati da analizzare in base al fitting della forma della linea. Selezionare il numero di file corrispondente allo spettro da analizzare e fare clic sul pulsante Visualizza . Lo spettro (se precedentemente elaborato) o la curva di decadimento ad induzione libera (FID) viene visualizzata nel software NMR.
- Elaborare il FID, se necessario, immettendo un comando Efp (moltiplicazione esponenziale, trasformazione di Fourier e correzione di fase) nella riga di comando. Regolare la fase dello spettro (Passo 2.1.10).
- Regolare la linea di base dello spettro; Se non è piatto su tutto lo spettro, livellare con la linea di intensità 0, come segue.
- Fare clic sulla scheda Processo e quindi sulla scheda Baseline . Passare il cursore sul pulsante A . Premere il pulsante sinistro del mouse e ruotare il mouse avanti o indietro per livellare la linea di regolazione rossa con l'estremità sinistra (downfield) dello spettro.
- Se la linea di base non è ancora all'altezza della linea di regolazione rossa, ripetere il processo con i pulsanti delle lettere rimanenti finché la linea di regolazione rossa non si adatta alla linea di base dello spettro. Utilizzare il pulsante Salva e ritorna per salvare la regolazione quando la baseline rossa corretta corrisponde alla baseline effettiva.
- Selezionare la scheda Analizza all'interno del software NMR. All'interno delle opzioni di analisi, selezionare la scelta Forme linea (Line Shapes ), seguita dall'opzione Adatta modelli NMR dinamici .
- Lo spettro viene ora visualizzato nella finestra del modulo di adattamento della forma della linea. Utilizzare le barre degli strumenti sopra lo spettro per regolare la modalità di visualizzazione dello spettro. La finestra a sinistra dello spettro gestisce il raccordo della forma della linea dello spettro.
- Regolare la visualizzazione dello spettro con lo strumento Zoom uniforme in modo che la parte dello spettro da adattare venga visualizzata nella finestra dello spettro. Utilizzate il pulsante della barra degli strumenti Sposta spettro a sinistra e a destra per centrare una parte dello spettro nella finestra di visualizzazione.
- Accedere alla finestra di spostamento chimico per l'adattamento della forma della linea selezionando la scheda Spettro nella finestra di adattamento della forma della linea.
- Fare clic sul pulsante Modifica intervallo . Immettete gli spostamenti chimici superiore e inferiore per il raccordo della forma della linea e fate clic sul pulsante OK per accettare tali limiti.
- Avviate un modello per il raccordo di forma di linea facendo clic sulla scheda Sistema di rotazione (Spin System ) nella finestra Adattamento forma linea. Fare clic sul pulsante Aggiungi per consentire la creazione di un sistema di rotazione del modello.
- Deselezionate LB (per l'allargamento delle linee) e immettete manualmente il valore per l'allargamento della linea con il mouse e il pulsante LB sulla barra degli strumenti di adattamento della forma della linea.
- Aggiungere il primo nucleo nel modello facendo clic sulla scheda Nucleo seguita da un clic sul pulsante Aggiungi . Apparirà un set di valori predefiniti per Nucleus 1. Regolate lo spostamento chimico per il nucleo 1 immettendo un valore per lo spostamento chimico nella casella Nu(iso) o con lo strumento spostamento chimico sulla barra degli strumenti di adattamento della forma della linea.
NOTA: se la casella di selezione viene lasciata nella forma selezionata, lo spostamento chimico di questo nucleo verrà variato per ottenere la migliore adattabilità. Le variabili non controllate non verranno variate nel processo di adattamento della linea. - Usa la casella Pseudospin per il nucleo 1 per inserire il numero di nuclei equivalenti per il nucleo 1 con ogni nucleo di spin 1/2 equivalente a 0,5 nel conteggio. Immettete la somma degli spin nella casella Pseudospin per tenere conto di tutti i nuclei equivalenti.
- Utilizzare la casella In molecola per includere modelli che richiedono più di una singola molecola per partecipare a un processo dinamico. Assegna risonanze che derivano da molecole diverse a molecole separate usando designazioni come 1, 2, ecc. per molecole diverse. Per le risonanze che derivano da una singola molecola, assegnare 1 per tutti i valori di In Molecola .
- Aggiungere il secondo nucleo e tutti i nuclei successivi al modello facendo clic sulla scheda Nucleo seguita facendo clic sul pulsante Aggiungi . Includere l'accoppiamento spin-spin tra nuclei inserendo l'accoppiamento nella casella JN appropriata (dove N è il nucleo con cui è accoppiato il nucleo che viene aggiunto, N = 1, 2, ...) o regolando il pulsante di accoppiamento scalare sulla barra degli strumenti di adattamento della forma della linea.
- Inizia il processo di descrizione degli scambi atomici facendo clic sulla scheda Reazione . Fare clic sulla casella di controllo se la costante di velocità per lo scambio deve essere variata in base all'adattamento della forma della linea. Immettere il numero di nuclei da scambiare (numero rispetto alle relative schede identificative come Nucleo 1 e Nucleo 2) nella casella Scambi per il primo scambio nel modello.
- Descrivere gli scambi da testare nelle caselle sotto la casella Scambi . Definisci gli scambi tra le schede Nucleo nelle caselle sottostanti. Uno scambio di due nuclei verrebbe inserito come Nucleo 1 a Nucleo 2 e Nucleo 2 a Nucleo 1. Assicurarsi che gli scambi siano ciclici in quanto se un nucleo viene spostato dal nucleo 1, un altro nucleo deve essere spostato nel nucleo 1.
- Utilizzare il pulsante Velocità di Exchange sulla barra degli strumenti Adattamento forma linea per modificare il valore iniziale di k e regolare iterativamente il valore di k, anche se la casella di controllo è selezionata per la costante di velocità.
- Aggiungi altri scambi al modello facendo clic sulla scheda Reazione seguita dal pulsante Aggiungi . Aggiungere scambi al modello in base alle esigenze. Utilizzare gli strumenti sulla barra degli strumenti di adattamento della forma della linea per regolare le variabili iniziali, inclusa l'intensità dello spettro, in modo che corrispondano bene allo spettro da adattare.
- Iniziare l'adattamento iterativo della forma della linea facendo clic sul pulsante Avvia adattamento spettro sulla barra degli strumenti di adattamento della forma della linea. Continuate il fitting iterativo fino a quando non viene rilevata alcuna modifica nella migliore sovrapposizione tra spettro e modello o fino al raggiungimento di 1000 iterazioni. Se il fitting si ferma a 1000 iterazioni, continuate con il pulsante Avvia adattamento Spectrum . Lo spettro del modello viene visualizzato con lo spettro effettivo per il confronto.
- Registrare i valori di adattamento migliori dalle schede appropriate. Salva lo spettro di adattamento migliore facendo clic sulla scheda Spettro nella finestra di adattamento della forma della linea, quindi facendo clic sul pulsante Salva .
NOTA: lo spettro di adattamento migliore verrà salvato nella stessa cartella utilizzata per raccogliere i dati. Lo spettro di adattamento migliore si distinguerà dai dati originali per essere salvato con un numero di elaborazione diverso che viene inserito quando si verifica il salvataggio. - Salvate il modello utilizzato per il raccordo della forma della linea facendo clic sulla scheda Principale e facendo clic sul pulsante Salva. Immettere un nome per il modello.
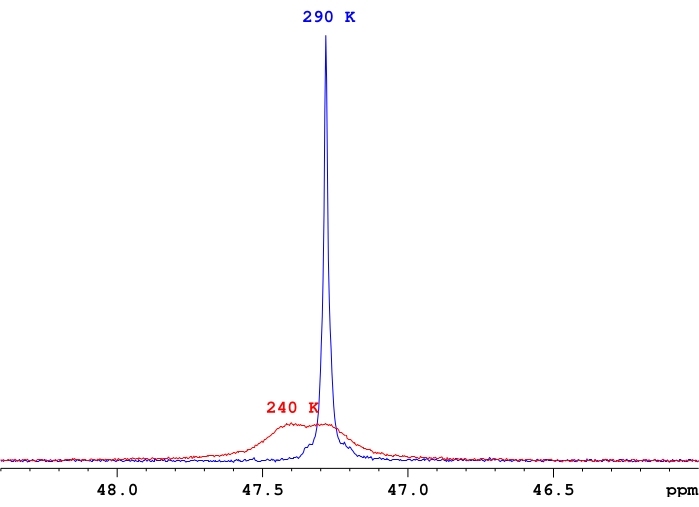
Figura 4: Un confronto delle intensità del segnale 31P-{1H} per un singolo campione di ReH5(PPh3)2(sec-butililammina) in d8-toluene. Una dimostrazione rappresentativa della differenza di intensità del segnale tra una singola risonanza di fosforo a scambio rapido e una coppia di risonanze di fosforo vicino alla temperatura di coalescenza per tali risonanze. Fare clic qui per visualizzare una versione ingrandita di questa figura.
3. Determinazione dei parametri di attivazione da un diagramma di Eyring 1
- Immettere i dati dal fitting della forma della linea per un processo dinamico modellato in un foglio di calcolo con la variabile indipendente immessa come 1/T e la variabile dipendente immessa come ln(k/T).
- Inserire un grafico a dispersione dei dati nel foglio di calcolo. Aggiungere una linea di tendenza attraverso i dati. Usa la pendenza e l'intercetta della linea di tendenza per risolvere ΔH‡ e ΔS‡. La pendenza della linea di tendenza è -ΔH‡/R mentre l'intercetta della linea di tendenza è ΔS‡/R + 23,76.
- Risolvere per ΔG‡ ad una data temperatura usando la relazione
ΔG‡(T) = ΔH‡ - TΔS‡.
NOTA: Per un semplice scambio di due nuclei con risonanze che si fondono, è possibile effettuare un controllo dei valori di ΔH‡ e ΔS‡ confrontando ΔG‡ calcolato alla temperatura di coalescenza con il valore di ΔG‡ che deriva dalla differenza di frequenza di scambio lento tra le risonanze e la temperatura di coalescenza.
Risultati
Le caratterizzazioni di entrambi i prodotti di poliidruro di renio descritte in questo manoscritto sono meglio realizzate mediante spettroscopia NMR1 H-{31 P} e 31P-{1H}. In una soluzione di d 6-benzene a temperatura ambiente, la risonanza del ligando dell'idruro di ReH7(PPh3)2 appare come una tripletta binomiale a δ = -4,2 ppm con 2 JPH = 18 Hz per spettroscopia NMR 1H (Figura supplementare 2)....
Discussione
Ci sono quattro elementi nella preparazione di ReH7 (PPh3) 2 che possono influire sulla quantità e sulla purezza del materiale prodotto. In primo luogo, l'uso di un bagno di ghiaccio durante i primi 15 minuti della reazione è importante per rimuovere il calore dalla reazione che si verifica tra boroidruro di sodio e acqua. Temperature iniziali più elevate portano ad una diminuzione della resa del prodotto ReH7(PPh 3)2 a causa della formazione del prodotto di decomposizione t...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Gli autori ringraziano il Dipartimento di Chimica e Fisica e il Creativity and Research Grant Program (Naik, Moehring) della Monmouth University per il sostegno finanziario di questo lavoro.
Materiali
| Name | Company | Catalog Number | Comments |
| Bruker Avance II 400 MHz NMR spectrometer | Bruker Biospin | The instrument includes a two channel probe (1H and X) with the X channel tunable from 162 MHz to 10 Mhz. The instrument is also VT capable with a dewar and heat exchanger for VT work. | |
| d8-toluene | MilliporeSigma | 434388 | |
| Powerstat variable transformer | Powerstat | ||
| sec-butyl amine | MilliporeSigma | B89000 | |
| Sodium borohydride | MilliporeSigma | 452882 | |
| Tetrahydrofuran | MilliporeSigma | 186562 | |
| Thermowell C3AM 100 mL | Thermowell | ||
| Topspin 3.0 or 4.1.4 with dNMR | Bruker Biospin | Data was acquired with Topspin version 3.0 and data handling was performed on a second computer that was running Topspin version 4.1.4.. | |
| Trichlorooxobis(triphenylphosphine) rhenium(V) | MilliporeSigma | 370193 | |
| Vacuubrand PC3000 vacuum pump with a CVC 3000 controller | Vacuubrand |
Riferimenti
- Zimmer, K. D., Shoemaker, R., Ruminski, R. R. Synthesis and characterization of a fluxional Re(I) carbonyl complex fac-[Re(CO)3(dpop')Cl] with the nominally tri-dentate ligand dipyrido(2,3-α:3',2'-j)phenazine (dpop). Inorganica Chimica Acta. 359 (5), 1478-1484 (2006).
- McGlinchey, M. J. Symmetry breaking in NMR spectroscopy: the elucidation of hidden molecular rearrangement processes. Symmetry. 6 (3), 622-654 (2014).
- Casarini, D., Luazzi, L., Mazzanti, A. Recent advances in stereodynamics and conformational analysis by dynamic NMR and theoretical calculations. European Journal of Organic Chemistry. 2010 (11), 2035 (2010).
- Palmer, A. G., Williams, J., McDermott, A. Nuclear magnetic resonance studies of biopolymer dynamics. Journal of Physical Chemistry. 100 (31), 13293-13310 (1996).
- Kern, D., Kern, G., Scherer, G., Fischer, G., Drakenberg, T. Kinetic analysis of cyclophilin-catalyzed prolyl cis/trans isomerization by dynamic NMR spectroscopy. Biochemistry. 34 (41), 13594-13602 (1995).
- Menger, F. M., Lynn, J. L. Fast proton transfer at a micelle surface. Journal of the American Chemical Society. 97 (4), 948-949 (1975).
- Pines, A., Rabinovitz, M. A nuclear magnetic resonance total line-shape treatment of internal rotation in dimethylformamide. Tetrahedron Letters. 9 (31), 3529-3532 (1968).
- Mancinelli, M., Bencivenni, G., Pecorari, D., Mazzanti, A. Stereochemistry and recent applications of axially chiral organic molecules. European Journal of Organic Chemistry. 2020 (27), 4070-4086 (2020).
- Streisel, D. J., et al. Fluxionality, substitution, and hydrogen exchange at eight-coordinate rhenium(V) polyhydride centers. Inorganica Chimica Acta. 496 (1), 119028 (2019).
- Jimenez, Y., Strepka, A. M., Borgohain, M. D., Hinojosa, P. A., Moehring, G. A. Ortho-metalation, rotational isomerization, and hydride-hydride coupling at rhenium(V) polyhydride complexes stabilized by aromatic amine ligands. Inorganica Chimica Acta. 362 (9), 3259-3266 (2009).
- Lee, J. C., Yao, W., Crabtree, R. H., Ruegger, H. Fluxionality in [ReH5(PPh3)2(pyridine)]. Inorganic Chemistry. 35 (3), 695-699 (1996).
- Patel, B. P., Kavallieratos, K., Crabtree, R. H. Effects of dihydrogen bonding on fluxionality in ReH5(PPh3)2L. Journal of Organometallic Chemistry. 528 (1), 205-207 (1997).
- Geetha, B., et al. Chiral amine ligands at rhenium(V) pentahydride complexes allow for characterization of an energetically accessible and reversible steric inversion of diastereotopic phosphorus atoms. Inorganica Chimica Acta. 531 (1), 120741 (2022).
- Paulo, A., Ascenso, J., Domingos, A., Galvao, A., Santos, I. Rhenium-(III) and -(V) hydride complexes with modified poly(pyrazolyl)borates. Journal of the Chemical Society, Dalton Transactions. 1999 (8), 1293-1300 (1999).
- Bianchini, C., et al. Synthesis and characterization of rhenium polyhydrides stabilized by the tripodal ligand MeC(CH2PPh2)3. Journal of Organometallic Chemistry. 451 (1), 97-106 (1993).
- Scorzelli, A. G., Macalush, B. E., Naik, D. V., Moehring, G. A. Comparative study of fluxional processes at two different classes of eight-coordinate rhenium(V) polyhydride complexes. Inorganica Chimica Acta. 516 (1), 120120 (2021).
- Luo, X. -. L., Crabtree, R. H. Synthesis and spectroscopic characterization of rhenium complexes ReH5(triphos)] and [ReH6(triphos)]+ [triphos = PPh(CH2CH2PPh2)2]. Journal of the Chemical Society. 1991 (5), 587-590 (1991).
- Kim, Y., Deng, H., Gallucci, J. C., Wojcicki, A. Rhenium polyhydride complexes containing PhP(CH2CH2CH2PCy2)2 (Cyttp): protonation, insertion, and ligand substitution reactions of ReH5(Cyttp) and structural characterization of ReH5(Cyttp) and [ReH4(η2-H2)(Cyttp)]SbF6. Inorganic Chemistry. 35 (24), 7166-7173 (1996).
- Bolano, S., et al. Synthesis, characterization, protonation studies and X-ray crystal structure of ReH5(PPh3)2(PTA) (PTA = 1,3,5-triaza-7-phosphaadamantane). Journal of Organometallic Chemistry. 691 (4), 629-637 (2006).
- Ginsberg, A. P., Abrahams, S. C., Jamieson, P. B. Nonrigid stereochemistry in eight-coordinate pentahydridorhenium complexes. Journal of the American Chemical Society. 95 (14), 4751-4752 (1973).
- Bolano, S., Bravo, J., Garcia-Fontan, S. Mono- and dinuclear rhenium polyhydride complexes bearing the chelating ligand 1,2-bis(dicyclohexylphosphinanyloxy)ethane. European Journal of Inorganic Chemistry. 2004 (24), 4812-4819 (2004).
- Leeaphon, M., Rohl, K., Thomas, R. J., Fanwick, P. E., Walton, R. A. Reactions of the polyhydride complex ReH7(PPh3)2 with quinoline, 2-hydroxyquinoline, and 2-mercaptoquinoline. The preparation and characterization of hydrido complexes of rhenium(V) and chloro complexes of rhenium(III). Inorganic Chemistry. 32 (24), 5562-5568 (1993).
- Mejia, E., Togni, A. Rhenium complexes containing the chiral tridentate ferrocenyl ligand pigiphos. Organometallics. 30 (17), 4765-4770 (2011).
- Moehring, G. A., Walton, R. A. Reactions of heptahydrobis(triphenylphosphine)rhenium with bidentate aromatic heterocycles. Inorganic Chemistry. 26 (17), 2910-2912 (1987).
- Kosanovich, A. J., Reibenspies, J. H., Ozerov, A. V. Complexes of high-valent rhenium supported by the PCP pincer. Organometallics. 35 (4), 513-519 (2016).
- Emge, T. J., Koetzle, T. F., Bruno, J. W., Caulton, K. G. Pentahydridorhenium: crystal and molecular structure of ReH5(PMePh2)3. Inorganic Chemistry. 23 (24), 4012-4017 (1984).
- Costello, M. T., Fanwick, P. E., Green, M. A., Walton, R. A. Reactions of Heptahydridobis(triphenylphosphine)rhenium with 1-(diphenylphosphino)-2-(diphenylarsino)ethane (arphos) and 1,2-bis(diphenylarsino)ethane (dpae). Structural characterization of ReH5(PPh3)2(arphos-As) and ReH5(PPh3)2(dpae-As). Inorganic Chemistry. 30 (4), 861-864 (1991).
- Alvarez, D., Lundquist, E. G., Ziller, J. W., Evans, W. J., Caulton, K. G. Synthesis, structure and applications of transition-metal polyhydride anions. Journal of the American Chemical Society. 111 (22), 8392-8398 (1989).
- Albinati, A., et al. Synthesis, characterization, and interconversion of the rhenium polyhydrides ReH3(η4-NP3)] and [ReH4(η4-NP3)]+ {NP3 = tris[2-(diphenylphosphanyl)ethyl]amine}. European Journal of Inorganic Chemistry. 2002 (6), 1530-1539 (2002).
- Bosque, R., et al. Site preference energetics, fluxionality, and intramolecular M−H···H−N hydrogen bonding in a dodecahedral transition metal polyhydride. Inorganic Chemistry. 36 (24), 5505-5511 (1997).
- Tao, Y., Sou, W., Luo, G. -. G., Kraka, E. Describing polytopal rearrangement processes of octacoordinate structures. I. renewed insights into fluxionality of the rhenium polyhydride complex ReH5(PPh3)2(Pyridine). Inorganic Chemistry. 60 (4), 2492-2502 (2021).
- Beringhelli, T., D'Alfonso, G., Minoja, A. P. Rhenium-platinum mixed metal clusters. Characterization in solution and dynamic behavior of the isomers of [Re3Pt(µ-H3)(CO)14]. An example of a labile metal fragment that undergoes intermolecular exchange. Organometallics. 13 (2), 663-668 (1994).
- Grieco, G., Blacque, O. Solution and solid-state structure of the first NHC-substituted rhenium heptahydrides. European Journal of Inorganic Chemistry. 2019 (34), 3810-3819 (2019).
- Wazio, J. A., Jimenez, V., Soparawalla, S., John, S., Moehring, G. A. Hydrogen exchange of rhenium(VII) heptahydridobis(triphenylphosphine) with water, aniline, methanol, and itself. Inorganica Chimica Acta. 362 (1), 159-165 (2009).
- Chatt, J., Coffey, R. S. Hydrido-complexes of rhenium-containing tertiary phosphines. Journal of the Chemical Society, A. 1969, 1963-1972 (1969).
- Tao, Y., Wang, X., Zou, W., Luo, G. -. G., Kraka, E. Unusual intramolecular motion of ReH92- in K2ReH9 crystal: circle dance and three-arm turnstile mechanisms revealed by computational study. Inorganic Chemistry. 61 (2), 1041-1050 (2022).
- Berger, X., Braun, S. . 200 and More NMR Experiments a Practical Course. , (2004).
- He, G., Chen, J., Sung, H. H. -. Y., Williams, I. D., Jia, G. Substituent effect on reactions of ReH5(PMe2Ph)3 with propargyl alcohols. Inorganica Chimica Acta. 518 (1), 120239 (2021).
- Donnelly, L. J., Parsons, S., Morrison, C. A., Thomas, S. P., Love, J. B. Synthesis and structures of anionic rhenium polyhydride complexes of boron-hydride ligands and their application in catalysis. Chemical Science. 11 (9), 9994-9999 (2020).
- Donnelly, L. J., et al. C-H borylation catalysis of heteroaromatics by a rhenium boryl polyhydride. ACS Catalysis. 11 (12), 7394-7400 (2021).
- Jin, H., et al. CO-enabled rhenium hydride catalyst for directed C(sp2)-H bond alkylation with olefins. Organic Chemistry Frontiers. 2 (4), 378-382 (2015).
- Takaya, H., Ito, M., Murahashi, S. -. I. Rhenium-catalyzed addition of carbonyl compounds to the carbon−nitrogen triple bonds of nitriles: α-C−H activation of carbonyl compounds. Journal of the American Chemical Society. 131 (31), 10824-10825 (2009).
- Carr, S. W., Fowles, E. H., Fontaine, X. L. R., Shaw, B. L. Multihydride complexes of rhenium, osmium or iridium containing monodentate ditertiary phosphine ligands: selective hydrogen-deuterium exchanges of the rhenium multihydrides. Journal of the Chemical Society, Dalton Transactions. 1990 (2), 573-579 (1990).
- Jin, H., et al. Rhenium-catalyzed acceptorless dehydrogenative coupling via dual activation of alcohols and carbonyl compounds. ACS Catalysis. 3 (10), 2195-2198 (2013).
- Loza, M. L., de Gala, S., Crabtree, R. H. Steric crowding in a rhenium polyhydride induced by a chelating disilyl ligand: synthesis, characterization, and reactivity of ReH5(disil)(PPh3)2 (disil = 1,2-Bis(dimethylsilyl)benzene and 1,2-Bis(dimethylsilyl)ethane). Inorganic Chemistry. 33 (22), 5073-5078 (1994).
- Lin, Y., Zhu, X., Xiang, M. Transition metal polyhydrides-catalyzed addition of activated nitriles to aldehydes and ketones via Knoevenagel condensation. Journal of Organometallic Chemistry. 448 (1-2), 215-218 (1993).
- Abdukader, A., Jin, H., Cheng, Y., Zhu, C. Rhenium-catalyzed amination of alcohols by hydrogen transfer process. Tetrahedron Letters. 55 (30), 4172-4174 (2014).
- Lin, Y., Zhou, Y. Selective transfer hydrogenation catalyzed by transition metal polyhydrides. Fenzi Cuihua. 5 (2), 119-124 (1991).
- Green, M. A., Huffman, J. C., Caulton, K. G., Rybak, W. K., Ziolkowski, J. J. Ligand scavenging and catalytic utilization of the phototransient ReH5(PMe2Ph)2. Journal of Organometallic Chemistry. 218 (2), 39-43 (1981).
- Komiya, S., Chigira, T., Suzuki, T., Hirano, M. Polymerization of alkyl methacrylate catalyzed by hydridorhenium complexes. Chemistry Letters. 4 (4), 347-348 (1999).
- Michos, D., Luo, X. L., Faller, J. W., Crabtree, R. H. Tungsten(VI) hexahydride complexes supported by chelating triphosphine ligands: protonation to give η2-dihydrogen complexes and catalytic dehydrogenation of cyclooctane to cyclooctene. Inorganic Chemistry. 32 (8), 1370-1375 (1993).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon