È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sonda β-galattosidasi associata alla senescenza fluorescente Far-Red per l'identificazione e l'arricchimento delle cellule tumorali senescenti mediante citometria a flusso
In questo articolo
Riepilogo
Viene presentato un protocollo per la quantificazione fluorescente citometrica a flusso di cellule tumorali senescenti indotte da farmaci chemioterapici in colture cellulari o in modelli tumorali murini. Le procedure opzionali includono la co-immunocolorazione, la fissazione del campione per facilitare l'analisi di grandi lotti o punti temporali e l'arricchimento di cellule senescenti vitali mediante selezione citometrica a flusso.
Abstract
La senescenza cellulare è uno stato di arresto proliferativo indotto da un danno biologico che normalmente si accumula nel corso degli anni nelle cellule che invecchiano, ma può anche emergere rapidamente nelle cellule tumorali come risposta al danno indotto da vari trattamenti antitumorali. La senescenza delle cellule tumorali è generalmente considerata indesiderabile, poiché le cellule senescenti diventano resistenti alla morte e bloccano la remissione del tumore, esacerbando la malignità del tumore e la resistenza al trattamento. Pertanto, l'identificazione di cellule tumorali senescenti è di continuo interesse per la comunità di ricerca sul cancro. Esistono vari saggi di senescenza, molti basati sull'attività del noto marcatore di senescenza, la beta-galattosidasi associata alla senescenza (SA-β-Gal).
Tipicamente, il saggio SA-β-Gal viene eseguito utilizzando un substrato cromogenico (X-Gal) su cellule fisse, con l'enumerazione lenta e soggettiva delle cellule senescenti "blu" mediante microscopia ottica. Saggi migliorati che utilizzano substrati fluorescenti SA-β-Gal permeanti alle cellule, tra cui C12-FDG (verde) e DDAO-Galactoside (DDAOG; far-red), hanno permesso l'analisi di cellule viventi e consentito l'uso di piattaforme di analisi fluorescenti ad alto rendimento, compresi i citometri a flusso. C12-FDG è una sonda ben documentata per SA-β-Gal, ma la sua emissione fluorescente verde si sovrappone all'autofluorescenza cellulare intrinseca (AF) che si verifica durante la senescenza a causa dell'accumulo di aggregati lipofuscinici. Utilizzando la sonda SA-β-Gal DDAOG, l'autofluorescenza cellulare verde può essere utilizzata come parametro secondario per confermare la senescenza, aggiungendo affidabilità al test. I restanti canali di fluorescenza possono essere utilizzati per la colorazione della vitalità cellulare o per l'immunomarcatura fluorescente opzionale.
Utilizzando la citometria a flusso, dimostriamo l'uso di DDAOG e autofluorescenza lipofuscina come saggio a doppio parametro per l'identificazione di cellule tumorali senescenti. Viene eseguita la quantificazione della percentuale di cellule senescenti vitali. Se lo si desidera, può essere inclusa una fase di immunomarcatura opzionale per valutare gli antigeni di superficie cellulare di interesse. Le cellule senescenti identificate possono anche essere selezionate citometricamente a flusso e raccolte per l'analisi a valle. Le cellule senescenti raccolte possono essere immediatamente lisate (ad esempio, per saggi immunologici o analisi omiche) o ulteriormente coltivate.
Introduzione
Le cellule senescenti normalmente si accumulano negli organismi nel corso degli anni durante il normale invecchiamento biologico, ma possono anche svilupparsi rapidamente nelle cellule tumorali come risposta ai danni indotti da vari trattamenti antitumorali, tra cui radiazioni e chemioterapia. Sebbene non siano più proliferanti, le cellule tumorali senescenti indotte dalla terapia (TIS) possono contribuire alla resistenza al trattamento e guidare la recidiva 1,2,3. I fattori secreti dalle cellule TIS possono esacerbare la malignità tumorale promuovendo l'evasione immunitaria o le metastasi 4,5. Le cellule TIS sviluppano fenotipi complessi e specifici del contesto, profili metabolici alterati e risposte immunitarie uniche 6,7,8. Pertanto, l'identificazione e la caratterizzazione delle cellule tumorali TIS indotte da vari approcci terapeutici del cancro è un argomento di continuo interesse per la comunità di ricerca sul cancro.
Per rilevare le cellule tumorali TIS, i saggi di senescenza convenzionali sono ampiamente utilizzati, principalmente basati sulla rilevazione di una maggiore attività dell'enzima marcatore di senescenza, la beta-galattosidasi lisosomiale GLB19. Il rilevamento a un pH lisosomiale quasi neutro (piuttosto che acido) consente il rilevamento specifico della beta-galattosidasi associata alla senescenza (SA-β-Gal)10. Un saggio standard SA-β-Gal che è stato utilizzato per diversi decenni utilizza X-Gal (5-bromo-4-cloro-3-indolil-β-D-galattopiranoside), un substrato cromogenico blu della beta-galattosidasi, per rilevare SA-β-Gal in cellule fisse mediante microscopia ottica11. Il test X-Gal consente la conferma visiva qualitativa di TIS utilizzando reagenti comunemente disponibili e apparecchiature di laboratorio. Un microscopio a luce trasmessa di base è l'unica strumentazione necessaria per valutare la presenza del cromogeno blu. Tuttavia, la procedura di colorazione X-Gal può mancare di sensibilità, a volte richiedendo più di 24 ore per lo sviluppo del colore. La colorazione è seguita da un punteggio soggettivo a basso rendimento delle singole cellule senescenti basato sul conteggio delle cellule che mostrano un certo livello di intensità del cromogeno blu al microscopio ottico. Poiché X-Gal è impermeabile alle cellule, questo test richiede celle fissate con solvente, che non possono essere recuperate per l'analisi a valle. Quando si lavora con campioni limitati di animali o pazienti, questo può essere un grosso svantaggio.
Saggi migliorati di SA-β-Gal utilizzando substrati enzimatici fluorescenti permeanti alle cellule, tra cui C 12-FDG (5-dodecanoylaminofluorescein Di-β-D-Galactopyranoside, verde) e DDAOG (9H-(1,3-dicloro-9,9-dimetilacridin-2-one-7-yl) β-D-Galactopyranoside, far-red) sono stati precedentemente pubblicati in letteratura12,13,14,15. La struttura chimica della sonda e le caratteristiche ottiche del DDAOG sono mostrate nella figura supplementare S1. Queste sonde cellulari consentono l'analisi di cellule viventi (piuttosto che fisse) e le sonde fluorescenti piuttosto che cromogeniche facilitano l'uso di piattaforme di analisi fluorescenti rapide ad alto rendimento, compresi strumenti di screening ad alto contenuto e citometri a flusso. I citometri a flusso di selezione consentono il recupero di popolazioni arricchite di cellule senescenti viventi da colture cellulari o tumori per l'analisi a valle (ad esempio, western blotting, ELISA o 'omics). L'analisi della fluorescenza fornisce anche un segnale quantitativo, consentendo una determinazione più accurata della percentuale di cellule senescenti all'interno di un determinato campione. Ulteriori sonde fluorescenti, tra cui sonde di vitalità e anticorpi marcati con fluorofori, possono essere facilmente aggiunte per l'analisi multiplex di bersagli oltre SA-β-Gal.
Simile a DDAOG, C12-FDG è una sonda fluorescente per SA-β-Gal, ma la sua emissione fluorescente verde si sovrappone alla FA cellulare intrinseca, che si verifica durante la senescenza a causa dell'accumulo di aggregati di lipofuscina nelle cellule16. Utilizzando la sonda DDAOG rosso, la fibrillazione atriale cellulare verde può essere utilizzata come parametro secondario per confermare la senescenza17. Ciò migliora l'affidabilità del test utilizzando un secondo marcatore in aggiunta a SA-β-Gal, che spesso può essere inaffidabile come singolo marcatore per la senescenza18. Poiché la rilevazione della fibrillazione atriale endogena nelle cellule senescenti è un approccio label-free, è un modo rapido e semplice per espandere la specificità del nostro test basato su DDAOG.
In questo protocollo, dimostriamo l'uso di DDAOG e AF come test di citometria a flusso rapido e a doppio parametro per l'identificazione di cellule tumorali TIS vitali da colture in vitro o isolate da tumori trattati con farmaci stabiliti nei topi (Figura 1). Il protocollo utilizza fluorofori compatibili con un'ampia gamma di analizzatori e selezionatori di citometria a flusso commerciali standard (Tabella 1). È abilitata la quantificazione della percentuale di cellule senescenti vitali utilizzando l'analisi citometrica a flusso standard. Se lo si desidera, può essere eseguita una fase di immunomarcatura opzionale per valutare gli antigeni della superficie cellulare di interesse in concomitanza con la senescenza. Le cellule senescenti identificate possono anche essere arricchite utilizzando la metodologia standard di selezione cellulare attivata dalla fluorescenza (FACS).
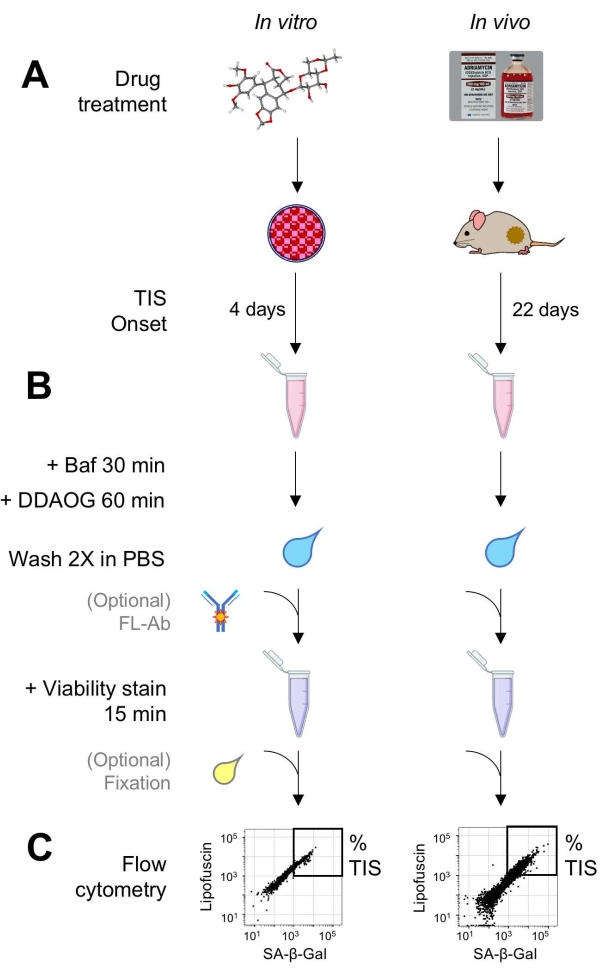
Figura 1: Flusso di lavoro sperimentale. Uno schema che riassume i punti chiave del test DDAOG. (A) Un farmaco che induce TIS viene aggiunto a cellule coltivate di mammiferi o somministrato a topi portatori di tumore. Il tempo è quindi concesso per l'insorgenza della TIS: per le cellule, 4 giorni dopo il trattamento; per i topi, 22 giorni totali, con tre trattamenti ogni 5 giorni più 7 giorni di recupero. Le cellule vengono raccolte o i tumori vengono dissociati in sospensione. (B) I campioni sono trattati con Baf per regolare il pH lisosomiale per la rilevazione di SA-β-Gal per 30 minuti; quindi, la sonda DDAOG viene aggiunta per 60 minuti per rilevare SA-β-Gal. I campioni vengono lavati 2 volte in PBS e viene aggiunta brevemente una macchia di vitalità (15 min). Opzionalmente, i campioni possono essere colorati con anticorpi fluorescenti in canali di fluorescenza aperti e/o fissati per analisi successive. (C) I campioni vengono analizzati utilizzando un citometro a flusso standard. Le cellule vitali sono visualizzate in grafici a punti che mostrano DDAOG rosso (che indica SA-β-Gal) rispetto all'autofluorescenza verde (lipofuscina). Un gate per determinare la percentuale di cellule TIS viene stabilito sulla base di campioni di controllo non trattati (non mostrati). Se viene utilizzato un citometro di selezione (FACS), le cellule TIS possono essere raccolte e rimesse in coltura per ulteriori test in vitro o lisate ed elaborate per saggi di biologia molecolare. Abbreviazioni: DDAO = 9H-(1,3-dicloro-9,9-dimetilacridina-2-one); DDAOG = DDAO-galattoside; TIS = senescenza indotta dalla terapia; FL-Ab = anticorpo coniugato con fluorofori; Baf = Bafilomicina A1; SA-β-Gal = beta-galattosidasi associata alla senescenza; PBS = soluzione salina tamponata fosfato; FACS = selezione cellulare attivata dalla fluorescenza. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Fluoroforo | Rileva | Ex/Em (nm) | Laser citometrico (nm) | Rilevatore citometrico / filtro passa banda (nm) |
| DDAOG | SA-β-Gal | 645/6601 | 640 | 670 / 30 |
| AF | Lipofuscina | < 600 | 488 | 525 / 50 |
| CV450 | Vitalità | 408/450 | 405 | 450 / 50 |
| PE | Marcatore anticorpo/superficie | 565/578 | 561 | 582 / 15 |
Tabella 1: Specifiche ottiche dei fluorofori e dei citometri. Le specifiche del citometro utilizzate in questo protocollo sono elencate per uno strumento con un totale di 4 laser e 15 rilevatori di emissioni. DDAOG rilevato a 645/660 nm è la forma della sonda scissa da SA-β-Gal1. DDAOG non scisso può presentare fluorescenza di basso livello a 460/610 nm, ma viene rimosso mediante fasi di lavaggio nel protocollo. Abbreviazioni: DDAO = 9H-(1,3-dicloro-9,9-dimetilacridina-2-one); DDAOG = DDAO-galattoside; AF = autofluorescenza; PE = ficoeritrina; SA-β-Gal = beta-galattosidasi associata alla senescenza.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutto il lavoro sugli animali descritto è stato approvato dall'Institutional Animal Care and Use Committee dell'Università di Chicago.
1. Preparazione e stoccaggio delle soluzioni stock
NOTA: Se le celle saranno selezionate in flusso, tutte le soluzioni devono essere preparate utilizzando tecniche sterili e filtrate attraverso un dispositivo filtrante da 0,22 μm.
- Preparare una soluzione madre di DDAO-Galactoside a 5 mg/ml in DMSO. Aliquote a 50 μL per provetta (o volume desiderato). Conservare a -20 °C al buio per un massimo di 1 anno.
- Preparare una soluzione madre di Bafilomicina A1 a 1 mM in DMSO. Aliquote a 50 μL per provetta (o volume desiderato). Conservare a -20 °C per un massimo di 6 mesi.
- Preparare una soluzione madre di Calcein Violet 450 AM a 1 mM in DMSO. Aliquote a 50 μL per provetta (o volume desiderato). Conservare a -20 °C al buio per un massimo di 1 anno.
- Per il trattamento di cellule in coltura in vitro, preparare 10 mM di soluzioni madre concentrate di agenti che inducono la senescenza nel solvente appropriato e sterilizzare utilizzando filtri a siringa da 0,2 μm. Conservare a -20 °C o secondo le indicazioni del produttore.
NOTA: Per la somministrazione di agenti chemioterapici che inducono senescenza in vivo (a topi con tumori accertati), gli agenti devono essere di grado USP e diluiti in soluzione salina da stock concentrato appena prima dell'iniezione. - Preparare un terreno di coltura completo per le linee cellulari utilizzate.
NOTA: Ad esempio, preparare il terreno per le cellule B16-F10 o A549 con DMEM 1x + 10% FBS + 1x sostituto della glutammina + 1x penicillina / streptomicina. I supporti devono essere mantenuti sterili. Possono essere utilizzate altre formulazioni di terreni specifici per linea cellulare. Alcuni componenti come il glutatione possono, in alcuni casi, interferire con l'inizio della senescenza. Il test empirico di varie formulazioni di terreni deve essere condotto se l'insorgenza della senescenza è inferiore al previsto o non osservata con agenti chemioterapici di controllo. - Preparare tamponi di colorazione e lavaggio.
- Preparare l'albumina sierica bovina all'1% (BSA) in 1x PBS per l'uso nelle procedure di colorazione. Sciogliere 2 g di BSA in 200 ml di PBS e mescolare per 10 minuti, o fino a completa dissoluzione, a temperatura ambiente.
- Preparare lo 0,5% di BSA in 1x PBS come tampone di lavaggio. Diluire 100 mL dell'1% di BSA preparato nella fase 1.6.1 in 100 mL di 1x PBS per lo 0,5% di BSA.
- Conservare i tamponi a 4 °C per un massimo di 1 mese.
- Preparare il 4% di paraformaldeide in 1x PBS. Utilizzare fiale di paraformaldeide sigillate disponibili in commercio (ad esempio, 16% v / v) per comodità e stabilità: 2,5 ml di 16% PFA + 7,5 ml di 1x PBS (= 10 ml di 4% PFA). Regolare il volume preparato in base al volume totale necessario per esperimento.
NOTA: Preparare se necessario solo per la fissazione cellulare; Preparare fresco ogni volta. - Preparare il tampone di selezione FACS: 1x PBS, 1 mM EDTA, 25 mM HEPES, 1% BSA (pH 7,2). Filtrare sterile attraverso un dispositivo di filtrazione da 0,22 μm e conservare a 4 °C per un massimo di 1 mese.
NOTA: preparare se necessario solo per lo smistamento FACS. Le formulazioni tampone di smistamento del flusso possono variare tra gli strumenti FACS. La formulazione di cui sopra è compatibile con lo strumento utilizzato in questo studio (vedi Tabella dei materiali). Consultare le linee guida del produttore. - Preparare la soluzione di dissociazione tumorale: 20 μg/mL Liberase TL + 100 μg/mL DNAse I in supporti RPMI-1640 (senza FBS o altri integratori). Le soluzioni madre sono utili per tenere a portata di mano Liberase TL (preparato e conservato secondo le indicazioni del produttore) e DNAse I (100 mg/ml in acqua bidistillata [ddH2O], conservato a -20 °C).
NOTA: Preparare se necessario se si utilizzano solo tumori; Preparare fresco ogni volta.
2. Induzione della senescenza da parte di farmaci chemioterapici in cellule tumorali in coltura
NOTA: tutte le fasi di manipolazione cellulare in questa sezione devono essere eseguite in un armadio di biosicurezza utilizzando pratiche sterili. Questa sezione è scritta per i tipi di cellule aderenti. Le celle di sospensione possono essere utilizzate con le opportune modifiche, come indicato.
- Coltivare linee cellulari tumorali secondo il protocollo standard del fornitore o del laboratorio che ha fornito le linee cellulari specifiche utilizzate.
NOTA: Le cellule a basso passaggio (p < 10) sono generalmente preferite in quanto vi saranno livelli più bassi di senescenza replicativa, cioè uno sfondo inferiore, nei campioni di cellule non trattate. - Un giorno prima dell'induzione della senescenza da parte di farmaci, raccogliere cellule con tripsina-EDTA 0,25% (o come raccomandato). Neutralizzare la tripsina aggiungendo un volume uguale di terreno di coltura completo e trasferire la sospensione cellulare in un tubo conico sterile.
NOTA: questo passaggio non è necessario per le celle di sospensione. - Contare le cellule utilizzando il metodo ematocitometrico standard e registrare le cellule / ml. Celle a piastre a 1 × 10 3-10 × 103 celle/cm2 in piatti di coltura standard in plastica a 6 pozzetti.
NOTA: La densità di placcatura ottimale dipende dal tasso di proliferazione delle cellule e deve essere determinata dall'utente. Le cellule devono essere in fase logaritmica di crescita a circa il 10%-20% di confluenza al momento del trattamento (cioè, dopo 18-24 ore di incubazione dopo la placcatura). La densità iniziale (celle/ml) delle celle di sospensione deve essere determinata dall'utente. Le piastre a sei pozzetti producono in genere abbastanza cellule senescenti per pozzetto per un campione standard di analisi della citometria a flusso. In caso di selezione a flusso, dovrebbe essere utilizzata un'area superficiale molto più ampia (ad esempio, più piastre P150) per consentire il recupero di un numero sufficiente di cellule senescenti per i saggi a valle (≥1 × 106). - Incubare le cellule placcate durante la notte (18-24 ore) in un incubatore a 37 °C con il 5% di CO2 e un vaso di umidità.
- Trattare le cellule placcate con farmaci chemioterapici che inducono senescenza. Includere almeno un controllo positivo, ad esempio etoposide (ETO) o bleomicina (BLM). Preparare pozzetti duplicati per farmaco. Includi un set trattato con solo veicolo come controllo.
NOTA: Una curva di dose per ciascun agente sperimentale deve essere testata dall'utilizzatore per determinare la concentrazione ottimale per l'induzione della senescenza nelle linee cellulari utilizzate. - Incubare le cellule per 4 giorni in un incubatore a 37 °C con il 5% di CO2 e un vaso di umidità per consentire l'inizio della senescenza. Esaminare quotidianamente i cambiamenti morfologici previsti utilizzando un microscopio ottico.
NOTA: I tempi di incubazione da 3-5 giorni possono essere accettabili a seconda del tasso di insorgenza della senescenza. I mezzi possono essere cambiati e l'agente riapplicato (o meno), come desiderato, per promuovere condizioni di crescita sane raggiungendo una percentuale accettabile di cellule senescenti. - Dopo l'inizio della senescenza, raccogliere le cellule aggiungendo tripsina-EDTA 0,25% per 5 minuti a 37 °C. Quando le cellule sono dissociate in sospensione, neutralizzare la tripsina con un volume uguale di mezzo completo.
NOTA: Questo passaggio non è necessario per le cellule che crescono in sospensione. Se verrà condotta la colorazione dei marcatori superficiali, evitare l'uso di tripsina-EDTA in quanto può distruggere temporaneamente gli antigeni di superficie sulle cellule. Invece, dissociare delicatamente il monostrato usando un raschietto per cellule di plastica sterile (o un reagente di dissociazione alternativo progettato per preservare gli antigeni di superficie). - Contare le cellule in ogni campione utilizzando un ematocitometro. Calcolare le celle/ml per ogni campione.
NOTA: Il blu di Trypan può essere aggiunto per valutare la percentuale di cellule morte a questo punto (cioè a causa del trattamento farmacologico), ma la morte cellulare sarà determinata anche con un colorante fluorescente di vitalità durante il flusso di colorazione DDAOG. - Aliquot ≥0,5 × 106 cellule per campione in provette da microcentrifuga da 1,7 ml.
NOTA: il numero di celle per campione deve essere standardizzato in tutti i campioni. - Centrifugare i tubi per 5 minuti a 1.000 × g in una microcentrifuga a 4 °C. Rimuovere il surnatante.
NOTA: Se una microcentrifuga refrigerata non è disponibile, può essere accettabile eseguire centrifugazioni a temperatura ambiente per alcuni tipi di celle resilienti. - Procedere alla colorazione DDAOG nella sezione 4.
3. Induzione della senescenza da parte di farmaci chemioterapici in tumori stabiliti nei topi
NOTA: Se le cellule tumorali saranno classificate FACS, garantire la sterilità in ogni fase lavorando in un armadio di biosicurezza e lavorando con strumenti, procedure e reagenti sterili.
- Creare modelli tumorali murini iniettando cellule tumorali per via sottocutanea, secondo metodi standard (ad esempio, Appelbe et al.19).
NOTA: Il numero di cellule tumorali da iniettare, il sito di iniezione e il ceppo di topo appropriato devono essere ottimizzati per ciascun protocollo. Qui, le cellule B16-F10 sono state iniettate per via sottocutanea a 1 × 106 cellule in 0,1 ml di soluzione salina (1 × 107 cellule / ml).- Verificare che la vitalità delle cellule utilizzando il blu di tripano sia del >90% prima di eseguire le iniezioni. Anestetizzare i topi con isoflurano.
- Anestetizzare topi femmina C57/BL6 di 6-7 settimane con una miscela di isoflurano al 3% e aria e mantenerli in queste condizioni in una camera di induzione posta all'interno di un armadio di biosicurezza sterile. Confermare l'anestesia pizzicando delicatamente il piede del topo. Applicare un unguento veterinario sterile su entrambi gli occhi per prevenire l'essiccazione corneale durante la procedura. Durante la procedura, mantenere la temperatura corporea del mouse utilizzando una lampada riscaldante.
- Lavorando all'interno di un armadio sterile di biosicurezza, rimuovere il mouse dalla camera di induzione e metterlo a contatto con un cono nasale che fornisce un apporto di isoflurano al 3%. Rasare l'area del fianco nel sito di iniezione utilizzando un rasoio elettrico pulito. Mescolare brevemente la sospensione cellulare preparata capovolgendo manualmente il tubo appena prima dell'iniezione e iniettare la sospensione cellulare per via sottocutanea nel fianco rasato utilizzando una siringa sterile da 0,5 mL dotata di un ago sterile da 27 G. Rimuovere il mouse dal cappuccio e trasferirlo nella gabbia di recupero.
- Nella gabbia di recupero, monitorare continuamente i segni vitali dei topi fino a quando non hanno riacquistato sufficiente coscienza per mantenere la recumenza sternale, dimostrare il riflesso raddrizzante e sono in grado di muoversi in sicurezza nella gabbia. Non lasciare topi incustoditi o restituire animali che hanno subito l'inoculazione di cellule tumorali alla compagnia di altri animali fino a quando non si sono completamente ripresi. Monitorare tutti i topi inoculati quotidianamente per perdita di peso, ridotta attività / mobilità e sintomi neurologici; eutanasia topi che presentano sintomi gravi in qualsiasi categoria. Per i topi che presentano sintomi dolorosi dopo l'inoculazione, somministrare buprenorfina (0,1-0,2 mg/kg) una volta per via sottocutanea.
NOTA: I topi che mostrano dolore persistente dopo buprenorfina dovrebbero essere eutanasiati.
- A partire da 5-7 giorni dopo l'inoculazione delle cellule tumorali, misurare la crescita del tumore con pinze ogni 2-3 giorni. Iniziare il trattamento prosenescente quando il tumore ha raggiunto 50 mm 3 ± 10 mm3 di volume.
NOTA: In questo lavoro, dosi di doxorubicina cloridrato di grado USP (DOX) o doxorubicina liposomiale PEGilata (PLD) a 10 mg/kg sono state somministrate in iniezione di cloruro di sodio allo 0,9% (USP). I farmaci sono stati iniettati per via intraperitoneale 3 volte, una volta ogni 5 giorni, a partire da quando i tumori hanno raggiunto 50 mm 3 ± 10 mm3. I topi si sono ripresi per 7 giorni dopo il trattamento finale per consentire l'insorgenza della TIS nei tumori. I dosaggi e le condizioni di induzione della senescenza per altri trattamenti e/o modelli tumorali devono essere ottimizzati. - A 7 giorni dopo il trattamento farmacologico finale, sacrificare i topi per sovradosaggio di CO2 e lussazione cervicale o altri metodi approvati in conformità con le linee guida sul lavoro degli animali da laboratorio. Asportare i tumori e raccoglierli in tubi sterili o piastre a 6 pozzetti riempiti con terreno di crescita RPMI sterile (per preservare la vitalità durante la lavorazione).
NOTA: Se si esegue un esame istologico (ad esempio, X-Gal o immunoistochimica), i tumori possono essere tagliati in due qui, con una metà congelata a scatto in O.C.T. embedding medium e criosezionata utilizzando procedure standard per l'istologia dei tessuti congelati. La restante metà del tumore dovrebbe produrre materiale abbondante per la dissociazione e la colorazione DDAOG. - Trasferire un tumore in un piatto di plastica P100 contenente 5 ml di terreno RPMI. Tritare il tumore in pezzi usando un bisturi.
- Trasferire 5 ml della sospensione di pezzi tumorali contenenti cellule sospese e detriti in un tubo conico da 15 ml. Utilizzare la punta più larga di un pipettuccio sierologico da 25 ml per trasferire questa sospensione se sono presenti detriti di grandi dimensioni. Risciacquare il piatto con un volume aggiuntivo di RPMI sterile per raccogliere materiali. Tappare e posizionare il tubo conico sul ghiaccio.
- Ripetere i passaggi 3,45-3,5 per i tumori rimanenti. Utilizzare una piastra separata e bisturi per ogni tumore per evitare la contaminazione incrociata, o risciacquare bene con PBS tra i tumori. Utilizzare 5 ml di terreno fresco per tritare ogni tumore.
- Preparare la soluzione di dissociazione tumorale: 20 μg/mL Liberase TL + 100 μg/mL DNAse I in supporti RPMI-1640 (senza FBS).
NOTA: Esistono molte formulazioni efficaci per soluzioni di dissociazione tumorale e possono includere una varietà di enzimi e altri componenti di diversi produttori. Le concentrazioni ottimali di componenti possono variare notevolmente tra i tipi di tumore. Se i globuli rossi sono altamente presenti nel tumore, la lisi dei globuli rossi può essere condotta in aggiunta; Se le cellule morte sono un problema, è possibile utilizzare un kit di rimozione delle cellule morte. Si raccomanda vivamente all'utente di determinare in modo indipendente le condizioni ottimali di dissociazione del tumore che forniscono un'elevata vitalità e una bassa presenza di cellule contaminanti, materiale connettivo e detriti. - Centrifugare tutti i campioni tumorali in provette coniche per 5 minuti a 1.000 × g (4 °C). Rimuovere il surnatante.
- Aggiungere 1-5 ml di soluzione di dissociazione tumorale a ciascun campione tumorale, a seconda del volume del materiale tumorale. Assicurarsi che ci siano 1-2 ml in eccesso del pellet di materiale tumorale nel tubo. Vortice a velocità moderata per mescolare.
- Collocare i campioni in un incubatore a 37 °C con rotazione rapida per 45 minuti. Vortice brevemente ogni 15 minuti.
- Filtrare ogni campione attraverso un filtro cellulare da 100 μm in un tubo conico da 50 ml. Se i campioni sono troppo viscosi per passare attraverso il filtro, aggiungere 10 ml di terreno RPMI-1640 per diluire. Risciacquare i filtri con mezzo RPMI per raccogliere le celle residue.
- Utilizzare un ematocitometro per contare le cellule/ml per ogni campione.
- Aliquote due o più repliche di 5 × 106 cellule per campione tumorale.
- Centrifugare per 5 minuti a 1.000 × g (4 °C). Rimuovere il surnatante.
- (Facoltativo) Crioconservare i campioni tumorali per la successiva colorazione DDAOG, se lo si desidera.
- Risospendere il pellet di cellule tumorali dissociate in terreno di crioconservazione: 50% FBS, 40% RPMI-1640, 10% DMSO, preparato in condizioni sterili a 5 × 106 cellule/ml.
- Aliquot 1 mL di sospensione cellulare in ciascun crioviale.
- Congelare i crioviali per 24 ore in un contenitore di congelamento a celle isopropanoli a -80 °C; Quindi, trasferire al criostoccaggio di azoto liquido per la conservazione a lungo termine (>1 settimana).
- Quando si desidera colorare, scongelare i crioviali sul ghiaccio e procedere alla colorazione DDAOG nella sezione 4.
NOTA: Alcuni tumori potrebbero non rimanere vitali attraverso la crioconservazione e la resilienza a questo processo dovrebbe essere valutata dall'utente per il modello tumorale di interesse.
- Procedere alla sezione 4 per la colorazione DDAOG.
4. Colorazione DDAOG di SA-β-Gal in campioni cellulari o tumorali
- Diluire 1 mM di Bafilomicina A1 a 1:1.000x in mezzo DMEM (senza FBS) per una concentrazione finale di 1 μM.
- Aggiungere la soluzione Baf-DMEM preparata ai campioni di pellet cellulare (dal punto 2.11 o dal punto 3.16) per una concentrazione di 1 × 106 cellule/ml.
NOTA: Ad esempio, se si utilizzano 0,5 × 106 cellule per campione, aggiungere 0,5 ml di Baf-DMEM. Per i tumori, 5 × 106 cellule possono essere colorate in 5 ml di Baf-DMEM. - Incubare per 30 minuti a 37 °C (senza CO2) su un rotatore/agitatore impostato a bassa velocità.
NOTA: Evitare incubatori di CO2 per il processo di colorazione, che possono acidificare le soluzioni e quindi interferire con la colorazione di Baf e DDAOG. - Senza lavare, aggiungere la soluzione madre DDAOG (5 mg/ml) a 1:500x (10 μg/mL finale) a ciascun campione. Pipetta da miscelare. Sostituire su un rotatore/agitatore a 37 °C (senza CO2) per 60 minuti. Proteggere dalla luce diretta.
- Centrifugare i tubi per 5 minuti a 1.000 x g a 4 °C. Rimuovere il surnatante.
- Lavare con 1 mL di BSA ghiacciato allo 0,5% per tubo e pipetta da miscelare. Centrifugare i tubi per 5 minuti a 1.000 x g a 4 °C e rimuovere il surnatante. Ripetere questo passaggio 2x per lavare accuratamente le celle. Rimuovere il surnatante e procedere.
NOTA: è importante eseguire le fasi di lavaggio al punto 4.6 per rimuovere il DDAOG non scisso, che può presentare un'emissione di fluorescenza indesiderata (460/610 nm).
NOTA: In caso di immunocolorazione per marcatori di superficie cellulare, procedere al paragrafo 5 di seguito. - (Facoltativo) Fissazione e conservazione delle cellule colorate DDAOG per analisi successive
- Aggiungere 0,5 ml di paraformaldeide ghiacciata al 4% a goccia per ogni campione lavato. Pipetta da miscelare.
- Incubare per 10 minuti a temperatura ambiente.
- Lavare le celle 2x con 1 mL di PBS.
- Conservare i campioni per un massimo di 1 settimana a 4 °C prima dell'analisi citometrica a flusso.
NOTA: per i campioni fissi, saltare il passaggio 4.8.
- Diluire il brodo Calcein Violet 450 AM (1 mM) a 1:1.000x in BSA-PBS all'1% (1 μM finale). Aggiungere 300 μL (per campioni di cellule coltivate) o 1.000 μL (per campioni tumorali) ai pellet di cellule lavate dal punto 4.6. Incubare per 15 minuti sul ghiaccio al buio.
- Procedere alla configurazione della citometria a flusso (paragrafo 6).
5. (Opzionale) Immunocolorazione per marcatori di superficie cellulare in combinazione con DDAOG
NOTA: Come con qualsiasi esperimento di citometria a flusso, i campioni di controllo a colorazione singola con solo DDAOG e solo anticorpo fluorescente devono essere preparati per determinare la diafonia (se presente) attraverso i canali di fluorescenza. Se si osserva una diafonia, deve essere eseguita la compensazione standard della citometria a flusso20.
- Risospendere i pellet cellulari ottenuti al punto 4.6 in 100 μL di tampone colorante (1% BSA in 1x PBS).
- Aggiungere il reagente bloccante del recettore Fc appropriato per la specie cellulare (topo o uomo) alla titolazione raccomandata dal produttore. Incubare per 10 minuti a 24 °C.
- Aggiungere anticorpi coniugati con fluorofori alla titolazione raccomandata dal produttore (o determinata dall'utente). Incubare per 20 minuti su ghiaccio, al riparo dalla luce.
- Centrifugare i tubi per 5 minuti a 1.000 × g a 4 °C. Rimuovere il surnatante.
- Lavare con 1 mL di tampone di lavaggio ghiacciato (0,5% BSA-PBS) per provetta e pipetta da miscelare. Centrifugare i tubi per 5 minuti a 1.000 × g a 4 °C e rimuovere il surnatante. Ripetere questo passaggio 2x per lavare accuratamente le celle.
- Diluire 1 mM Calcein Violet 450 AM a 1:1.000x in 1% BSA-PBS. Aggiungere 300 μL ai pellet di celle lavate dal punto 5.5. Incubare per 15 minuti sul ghiaccio al buio.
- Procedere all'analisi citometrica a flusso (paragrafi 6-7).
6. Configurazione del citometro a flusso e acquisizione dati
- Trasferire i campioni di cellule in provette compatibili con lo strumento di citometria a flusso. Posizionare i tubi sul ghiaccio e tenerli protetti dalla luce.
NOTA: Se si osservano aggregati nelle sospensioni cellulari, passare la sospensione attraverso i filtri cellulari da 70-100 μm prima dell'analisi. Non utilizzare filtri da 40 μm perché possono escludere alcune delle cellule senescenti più grandi. - Nel software di riferimento (vedere la tabella dei materiali), aprire i seguenti grafici: 1) FSC-A vs SSC-A dot plot, 2) istogramma del canale viola, 3) canale rosso lontano (ad esempio, APC-A) rispetto al dot plot del canale verde (ad esempio, FITC-A).
NOTA: è possibile utilizzare anche grafici di esclusione del doppietto e istogrammi a canale singolo, ma non sono strettamente necessari. - Avviare l'acquisizione dei dati del citometro.
- Posizionare il campione di controllo solo veicolo colorato con DDAOG sulla porta di aspirazione. A bassa velocità di aspirazione, iniziare ad acquisire dati campione.
- Regolare le tensioni FSC e SSC in modo che il >90% degli eventi sia contenuto all'interno del grafico. Se le celle non si adattano bene al grafico, abbassare l'impostazione di ridimensionamento dell'area a 0,33-0,5 unità.
- Rimuovere il campione solo veicolo senza registrare i dati.
- (Facoltativo) Aggiungere una goccia di microsfere di calibrazione arcobaleno a un tubo citometrico con 1 mL di PBS. Posizionare il tubo sulla porta di aspirazione del citometro. Iniziare ad acquisire dati di esempio.
- Regolare le tensioni dei canali viola, verdi e rossi in modo che il picco superiore della microsfera arcobaleno sia nell'intervallo di 104-10 5 unità di fluorescenza relativa in ciascun canale e tutti i picchi siano ben separati in ciascun canale. Record di 10.000 eventi. Rimuovere il tubo.
- Posizionare il campione di controllo positivo (ad esempio, BLM, ETO) colorato con DDAOG sulla porta di aspirazione. A bassa velocità, acquisire dati di esempio. Osserva gli eventi nei canali FSC, SSC, viola, verde e rosso per assicurarti che oltre il 90% degli eventi sia contenuto all'interno di tutti i grafici. Cerca un aumento del segnale AF e DDAOG rispetto al controllo del solo veicolo.
- Se si utilizza un citometro di ordinamento, avviare l'ordinamento in questo passaggio.
- Ai fini della conservazione dei registri, registrare 10.000 celle per il campione di controllo e ogni campione ordinato.
- Ordinare la quantità desiderata di cellule (≥1 × 106 è in genere adatto) in una provetta di raccolta appropriata allo strumento con 3-5 ml di terreno di coltura.
- Dopo l'ordinamento, procedere alla coltura o all'analisi a valle.
- Passare alla sezione 7 per l'analisi di routine dei campioni ordinati.
- Se si utilizzano anticorpi fluorescenti, ottimizzare le tensioni del canale utilizzando i campioni non colorati, monocolorati e a doppia colorazione preparati nella sezione 5.
NOTA: Per il citometro a flusso calibrato utilizzato nel presente documento, le tensioni ottimali del canale in genere sono scese tra 250 e 600 (fascia media), ma le tensioni ottimali e gli intervalli di tensione del canale variano tra gli strumenti. Evitare l'uso di tensioni a intervalli molto bassi o alti, che potrebbero sopprimere il segnale o amplificare il rumore. - Dopo aver completato i passaggi 6.1-6.5 e aver apportato le modifiche necessarie alle impostazioni del citometro, registrare i dati per tutti i campioni. Assicurarsi che le impostazioni rimangano uniformi per tutte le registrazioni di esempio. Registrare ≥10.000 eventi per campione di cellule in coltura o ≥100.000 eventi per campione di cellule tumorali.
NOTA: Sebbene il gating e l'analisi possano essere eseguiti utilizzando software di acquisizione dati (ad esempio, FACSDiva), un flusso di lavoro completo di gating e analisi da condurre dopo l'acquisizione utilizzando un software di analisi separato (FlowJo) è descritto nella sezione 7 di seguito. L'analisi post-acquisizione è preferibile per ridurre i tempi presso la postazione di lavoro del citometro e sfruttare gli strumenti aggiuntivi inclusi nel software di analisi dedicato. - Salvare i dati di esempio nel formato di file con estensione fcs. Esportare i file su un computer workstation dotato di software di analisi della citometria a flusso (ad esempio, FlowJo). Procedere alla sezione 7.
7. Analisi dei dati di citometria a flusso
NOTA: Il flusso di lavoro presentato utilizza il software FlowJo. È possibile utilizzare software alternativi di analisi dei dati di citometria a flusso se i passaggi chiave descritti in questa sezione sono seguiti in modo simile.
- Utilizzando il software FlowJo, aprire i file di dati .fcs dal passaggio 6.7.
- Aprire la finestra del layout.
- Trascinare e rilasciare tutti gli esempi nella finestra del layout.
- Cancelli le cellule vitali.
- Per prima cosa fare doppio clic sui dati di esempio per il controllo solo veicolo per aprire la finestra dei dati.
- Visualizza i dati come istogramma del canale viola. Identificare le cellule vitali colorate da CV450 in base alla loro fluorescenza più luminosa rispetto alle cellule morte.
- Disegna un cancello usando lo strumento istogramma a porta singola per includere solo le celle vitali. Assegna il nome al cancello praticabile.
- Quindi, dalla finestra del layout di esempio, trascinare la porta praticabile sugli altri campioni di cella per applicare la porta in modo uniforme.
- Nella finestra del layout, visualizzare tutti i campioni come istogrammi del canale viola (vitalità). Verificare che il gating cellulare vitale sia appropriato tra i campioni prima di procedere; In caso contrario, apportare le modifiche necessarie.
NOTA: La colorazione di vitalità può presentare variazioni tra trattamenti o tumori.
- Cancelliamo le cellule senescenti.
- Fare doppio clic sui dati della cella praticabile gated per il controllo solo veicolo per aprire la finestra dei dati.
- Visualizza i dati come un dot plot per il canale rosso lontano (DDAOG) rispetto al canale verde (AF).
- Disegna un cancello usando lo strumento di gating rettangolare per includere il <5% delle celle che sono DDAOG+ e AF+ (quadrante in alto a destra). Nomina il cancello senescente.
- Quindi, dalla finestra del layout di esempio, trascinare la porta senescente sui sottoinsiemi vitali degli altri campioni di cella per applicare la porta in modo uniforme.
- Nella finestra del layout, trascinare e rilasciare tutti i sottoinsiemi di celle vitali inclusi nella sezione 7.4. Visualizza tutti i campioni vitali come grafici a punti rosso (ad esempio, APC-A) rispetto al canale verde (ad esempio, FITC-A).
- Assicurarsi che il cancello senescente disegnato al punto 7.5.3 sia visibile su tutti i grafici e che il cancello per il controllo del solo veicolo presenti celle senescenti del ≤5%-10%.
- Una volta determinata la percentuale di cellule senescenti utilizzando i passaggi precedenti, presentare i dati risultanti utilizzando i grafici FlowJo, riassunti in una tabella dati e / o analizzati statisticamente utilizzando software standard.
Access restricted. Please log in or start a trial to view this content.
Risultati
Sono stati condotti diversi esperimenti per dimostrare la comparabilità di DDAOG con X-Gal e C12-FDG per la rilevazione della senescenza da parte di SA-β-Gal. In primo luogo, X-Gal è stato utilizzato per colorare le cellule di melanoma senescenti B16-F10 indotte da ETO (Figura 2A). Un colore blu intenso si è sviluppato in un sottogruppo di cellule trattate con ETO, mentre altre cellule hanno mostrato una colorazione blu meno intensa. La morfologia è stata ingrandita nella mag...
Access restricted. Please log in or start a trial to view this content.
Discussione
Nell'ultimo decennio circa, la citometria a flusso è diventata una piattaforma di analisi più comune nella ricerca sul cancro a causa della popolarità emergente dell'immunologia tumorale, dello sviluppo di citometri a flusso a basso costo e del miglioramento delle strutture di strumentazione condivise presso le istituzioni accademiche. I saggi multicolore sono ora standard, poiché la maggior parte degli strumenti più recenti sono dotati di array ottici viola, blu-verde e da rosso a rosso lontano. Pertanto, questo pr...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare per questo studio.
Riconoscimenti
Ringraziamo la Cytometry and Antibody Core Facility dell'Università di Chicago per il supporto sulla strumentazione di citometria a flusso. L'Animal Research Center dell'Università di Chicago ha fornito alloggi per animali.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Bafilomycin A1 | Research Products International | B40500 | |
| Bleomycin sulfate | Cayman | 13877 | |
| Bovine serum albumin (BSA) | US Biological | A1380 | |
| Calcein Violet 450 AM viability dye | ThermoFisher Scientific | 65-0854-39 | eBioscience |
| DPP4 antibody, PE conjugate | Biolegend | 137803 | Clone H194-112 |
| Cell line: A549 human lung adenocarcinoma | American Type Culture Collection | CCL-185 | |
| Cell line: B16-F10 mouse melanoma | American Type Culture Collection | CRL-6475 | |
| Cell scraper | Corning | 3008 | |
| Cell strainers, 100 µm | Falcon | 352360 | |
| DDAO-Galactoside | Life Technologies | D6488 | |
| DMEM medium 1x | Life Technologies | 11960-069 | |
| DMSO | Sigma | D2438 | |
| DNAse I | Sigma | DN25 | |
| Doxorubicin, hydrochloride injection (USP) | Pfizer | NDC 0069-3032-20 | |
| Doxorubicin, PEGylated liposomal (USP) | Sun Pharmaceutical | NDC 47335-049-40 | |
| EDTA 0.5 M | Life Technologies | 15575-038 | |
| Etoposide | Cayman | 12092 | |
| FBS | Omega | FB-11 | |
| Fc receptor blocking reagent | Biolegend | 101320 | Anti-mouse CD16/32 |
| Flow cytometer (cell analyzer) | Becton Dickinson (BD) | Various | LSRFortessa |
| Flow cytometer (cell sorter) | Becton Dickinson (BD) | Various | FACSAria |
| GlutaMax 100x | Life Technologies | 35050061 | |
| HEPES 1 M | Lonza | BW17737 | |
| Liberase TL | Sigma | 5401020001 | Roche |
| Paraformaldehyde 16% | Electron Microscopy Sciences | 15710 | |
| Penicillin/Streptomycin 100x | Life Technologies | 15140122 | |
| Phosphate buffered saline (PBS) 1x | Corning | MT21031CV | Dulbecco's PBS (without calcium and magnesium) |
| Rainbow calibration particles, ultra kit | SpheroTech | UCRP-38-2K | 3.5-3.9 µm, 2E6/mL |
| RPMI-1640 medium 1x | Life Technologies | 11875-119 | |
| Sodium chloride 0.9% (USP) | Baxter Healthcare Corporation | 2B1324 | |
| Software for cytometer data acquisition, "FACSDiva" | Becton Dickinson (BD) | n/a | Contact BD for license |
| Software for cytometer data analysis, "FlowJo" | TreeStar | n/a | Contact TreeStar for license |
| Trypsin-EDTA 0.25% | Life Technologies | 25200-114 |
Riferimenti
- Saleh, T., Tyutyunyk-Massey, L., Gewirtz, D. A. Tumor cell escape from therapy-induced senescence as a model of disease recurrence after dormancy. Cancer Research. 79 (6), 1044-1046 (2019).
- Wang, B., Kohli, J., Demaria, M. Senescent cells in cancer therapy: friends or foes. Trends in Cancer. 6 (10), 838-857 (2020).
- Prasanna, P. G., et al. Therapy-induced senescence: Opportunities to improve anticancer therapy. Journal of the National Cancer Institute. 113 (10), 1285-1298 (2021).
- Velarde, M. C., Demaria, M., Campisi, J. Senescent cells and their secretory phenotype as targets for cancer therapy. Interdisciplinary Topics in Gerontology and Geriatrics. 38, 17-27 (2013).
- Ou, H. L., et al. Cellular senescence in cancer: from mechanisms to detection. Molecular Oncology. 15 (10), 2634-2671 (2021).
- Hernandez-Segura, A., Nehme, J., Demaria, M. Hallmarks of cellular senescence. Trends in Cell Biology. 28 (6), 436-453 (2018).
- Bojko, A., Czarnecka-Herok, J., Charzynska, A., Dabrowski, M., Sikora, E. Diversity of the senescence phenotype of cancer cells treated with chemotherapeutic agents. Cells. 8 (12), 1501(2019).
- Mikuła-Pietrasik, J., Niklas, A., Uruski, P., Tykarski, A., Książek, K. Mechanisms and significance of therapy-induced and spontaneous senescence of cancer cells. Cellular and Molecular Life Sciences. 77 (2), 213-229 (2020).
- Lee, B. Y., et al. Senescence-associated beta-galactosidase is lysosomal beta-galactosidase. Aging cell. 5 (2), 187-195 (2006).
- Itahana, K., Itahana, Y., Dimri, G. P. Colorimetric detection of senescence-associated β galactosidase. Methods in Molecular Biology. 965, 143-156 (2013).
- Dimri, G. P., et al. A biomarker that identifies senescent human cells in culture and in aging skin in vivo. Proceedings of the National Academy of Sciences of the United States of America. 92 (20), 9363-9367 (1995).
- Debacq-Chainiaux, F., Erusalimsky, J. D., Campisi, J., Toussaint, O. Protocols to detect senescence-associated beta-galactosidase (SA-betagal) activity, a biomarker of senescent cells in culture and in vivo. Nature Protocols. 4 (12), 1798-1806 (2009).
- Noppe, G., et al. Rapid flow cytometric method for measuring senescence associated beta-galactosidase activity in human fibroblasts. Cytometry A. 75 (11), 910-916 (2009).
- Tung, C. -H., et al. In vivo imaging of β-galactosidase activity using far red fluorescent switch. Cancer Research. 64 (5), 1579-1583 (2004).
- Gong, H., et al. beta-Galactosidase activity assay using far-red-shifted fluorescent substrate DDAOG. Analytical Biochemistry. 386 (1), 59-64 (2009).
- Terman, A., Brunk, U. T. Lipofuscin: Mechanisms of formation and increase with age. APMIS. 106 (2), 265-276 (1998).
- Georgakopoulou, E. A., et al. Specific lipofuscin staining as a novel biomarker to detect replicative and stress-induced senescence. A method applicable in cryo-preserved and archival tissues. Aging. 5 (1), 37-50 (2013).
- Wang, B., Demaria, M. The quest to define and target cellular senescence in cancer. Cancer Research. 81 (24), 6087-6089 (2021).
- Appelbe, O. K., Zhang, Q., Pelizzari, C. A., Weichselbaum, R. R., Kron, S. J. Image-guided radiotherapy targets macromolecules through altering the tumor microenvironment. Molecular Pharmaceutics. 13 (10), 3457-3467 (2016).
- Maciorowski, Z., Chattopadhyay, P. K., Jain, P. Basic multicolor flow cytometry. Current Protocols in Immunology. 117, 1-38 (2017).
- Fan, Y., Cheng, J., Zeng, H., Shao, L. Senescent cell depletion through targeting BCL-family proteins and mitochondria. Frontiers in Physiology. 11, 593630(2020).
- Kim, K. M., et al. Identification of senescent cell surface targetable protein DPP4. Genes and Development. 31 (15), 1529-1534 (2017).
- Flor, A. C., Kron, S. J. Lipid-derived reactive aldehydes link oxidative stress to cell senescence. Cell Death Discovery. 7 (9), 2366(2016).
- Jochems, F., et al. The Cancer SENESCopedia: A delineation of cancer cell senescence. Cell reports. 36 (4), 109441(2021).
- Fallah, M., et al. Doxorubicin and liposomal doxorubicin induce senescence by enhancing nuclear factor kappa B and mitochondrial membrane potential. Life Sciences. 232, 116677(2019).
- Kasper, M., Barth, K. Bleomycin and its role in inducing apoptosis and senescence in lung cells - modulating effects of caveolin-1. Current Cancer Drug Targets. 9 (3), 341-353 (2009).
- Muthuramalingam, K., Cho, M., Kim, Y. Cellular senescence and EMT crosstalk in bleomycin-induced pathogenesis of pulmonary fibrosis-an in vitro analysis. Cell Biology International. 44 (2), 477-487 (2020).
- Flor, A. C., Wolfgeher, D., Wu, D., Kron, S. J. A signature of enhanced lipid metabolism, lipid peroxidation and aldehyde stress in therapy-induced senescence. Cell Death Discovery. 3, 17075(2017).
- Burd, C. E., et al. Monitoring tumorigenesis and senescence in vivo with a p16(INK4a)-luciferase model. Cell. 152 (1-2), 340-351 (2013).
- Liu, J. Y., et al. Cells exhibiting strong p16 (INK4a) promoter activation in vivo display features of senescence. Proceedings of the National Academy of Sciences of the United States of America. 116 (7), 2603-2611 (2019).
- Wang, L., Lankhorst, L., Bernards, R. Exploiting senescence for the treatment of cancer. Nature Reviews Cancer. 22 (6), 340-355 (2022).
- Baek, K. -H., Ryeom, S. Detection of oncogene-induced senescence in vivo. Methods in Molecular Biology. 1534, 185-198 (2017).
- González-Gualda, E., Baker, A. G., Fruk, L., Muñoz-Espín, D. A guide to assessing cellular senescence in vitro and in vivo. The FEBS Journal. 288 (1), 56-80 (2021).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon