È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misura della comprimibilità di cellula e nucleo basata su microdispositivo acustofluidico
In questo articolo
Riepilogo
Qui viene presentato un protocollo per costruire un sistema veloce e non distruttivo per misurare la comprimibilità di cellule o nuclei basato su microdispositivo acustofluidico. Sono stati studiati i cambiamenti nelle proprietà meccaniche delle cellule tumorali dopo transizione epiteliale-mesenchimale o radiazioni ionizzanti, dimostrando la prospettiva applicativa di questo metodo nella ricerca scientifica e nella pratica clinica.
Abstract
La meccanica cellulare svolge un ruolo importante nelle metastasi tumorali, nella trasformazione maligna delle cellule e nella radiosensibilità. Durante questi processi, studiare le proprietà meccaniche delle cellule è spesso impegnativo. I metodi di misurazione convenzionali basati sul contatto, come la compressione o lo stiramento, sono inclini a causare danni alle cellule, influenzando l'accuratezza della misurazione e la successiva coltura cellulare. Le misurazioni in stato aderente possono anche influenzare l'accuratezza, specialmente dopo l'irradiazione poiché le radiazioni ionizzanti appiattiscono le cellule e migliorano l'adesione. Qui è stato sviluppato un sistema di misura della meccanica cellulare basato sul metodo acustofluidico. La comprimibilità della cella può essere ottenuta registrando la traiettoria di movimento della cella sotto l'azione della forza acustica, che può realizzare misure rapide e non distruttive in stato sospeso. Questo documento riporta in dettaglio i protocolli per la progettazione di chip, la preparazione dei campioni, la registrazione della traiettoria, l'estrazione e l'analisi dei parametri. La comprimibilità di diversi tipi di cellule tumorali è stata misurata sulla base di questo metodo. La misurazione della comprimibilità del nucleo è stata ottenuta anche regolando la frequenza di risonanza della ceramica piezoelettrica e la larghezza del microcanale. In combinazione con la verifica a livello molecolare degli esperimenti di immunofluorescenza, è stata confrontata la comprimibilità cellulare prima e dopo la transizione epiteliale-mesenchimale indotta da farmaci (EMT). Inoltre, è stato rivelato il cambiamento di comprimibilità cellulare dopo l'irradiazione a raggi X con dosi diverse. Il metodo di misurazione della meccanica cellulare proposto in questo articolo è universale e flessibile e ha ampie prospettive di applicazione nella ricerca scientifica e nella pratica clinica.
Introduzione
Le proprietà meccaniche cellulari svolgono un ruolo importante nelle metastasi tumorali, nella trasformazione maligna delle cellule e nella radiosensibilità 1,2. Per ottenere una comprensione approfondita del ruolo delle proprietà meccaniche delle cellule nel processo di cui sopra, la misurazione accurata della meccanica cellulare è fondamentale e la misurazione non dovrebbe causare danni alle cellule per la successiva coltura e analisi. Il processo di misurazione dovrebbe essere il più veloce possibile, altrimenti la vitalità cellulare potrebbe essere compromessa se le cellule vengono rimosse dall'ambiente di coltivazione per lungo tempo.
I metodi di misurazione della meccanica cellulare esistenti devono affrontare alcune limitazioni. Alcuni metodi, come la citometria a torsione magnetica, le pinzette magnetiche e la microreologia a tracciamento delle particelle, causano danni cellulari a causa dell'introduzione di particelle nelle cellule 3,4,5. I metodi che misurano per contatto con le cellule, come il microscopio a forza atomica (AFM), l'aspirazione con micropipette, la microcostrizione e la tecnica a piastre parallele, sono anche soggetti a danni cellulari e il rendimento è difficile da aumentare 6,7,8. Inoltre, le radiazioni ionizzanti appiattiranno le cellule e aumenteranno la loro adesione9; È quindi necessario misurare la meccanica dell'intera cellula in sospensione.
In risposta alle sfide di cui sopra, è stato sviluppato un sistema di misura della meccanica cellulare basato sul metodo acustofluidico 10,11,12,13,14. La larghezza del canale è abbinata alla mezza lunghezza d'onda acustica, creando così un nodo d'onda stazionario sulla linea mediana del microcanale. Sotto l'azione della forza della radiazione acustica, le celle o le sfere standard possono spostarsi verso il nodo di pressione acustica. Poiché le proprietà fisiche delle sfere standard (dimensioni, densità e comprimibilità) sono note, è possibile determinare la densità di energia acustica. Quindi, la comprimibilità cellulare può essere ottenuta registrando le traiettorie di movimento delle cellule nel campo acustico. È possibile ottenere misurazioni non distruttive ad alta produttività di celle in stato di sospensione. Questo documento introdurrà la progettazione del chip microfluidico, la creazione del sistema e le fasi di misurazione. La misurazione di vari tipi di cellule tumorali è stata effettuata per verificare l'accuratezza del metodo. L'ambito di applicazione di questo metodo era stato esteso alle strutture subcellulari (come il nucleo) regolando la frequenza di risonanza della ceramica piezoelettrica e la larghezza del microcanale. Inoltre, sono stati studiati i cambiamenti nella comprimibilità cellulare dopo EMT indotta da farmaci o irradiazione a raggi X con dosi diverse. I risultati dimostrano l'ampia applicabilità di questo metodo come potente strumento per studiare la correlazione tra cambiamenti biochimici e proprietà meccaniche cellulari.
Protocollo
1. Fabbricazione e assemblaggio del microdispositivo acustofluidico
- Fabbricazione del chip microfluidico.
- Progettare un chip a canale singolo con un solo ingresso e uscita, come illustrato nella Figura 1. Per le celle di misura, mantenere la sezione trasversale rettangolare del microcanale a 740 μm di larghezza e 100 μm di profondità. Per misurare il nucleo della cella, modificare la larghezza e la profondità del microcanale rispettivamente a 250 μm e 100 μm.
- Preparare il microcanale su wafer di silicio tramite incisione ionica reattiva. Sigillare la parte superiore del microcanale con un pezzo di vetro trasparente resistente al calore mediante incollaggio anodico15. Lavare le patatine con un pulitore ad ultrasuoni per 10 minuti. Asciugarli in forno di essiccazione a 50 °C per un uso successivo.
- Fabbricare blocchi di polidimetilsilossano (PDMS).
- Aggiungere 30 ml di prepolimero in un piatto di vetro da 100 mm (di diametro). Aggiungere 3 ml di agente polimerizzante al prepolimero con una siringa.
NOTA: il rapporto volumetrico dell'agente di polimerizzazione e del prepolimero è 1:10. - Mescolare vigorosamente il prepolimero PDMS e l'agente di polimerizzazione con una bacchetta di vetro per circa 10 minuti. Cerca bolle d'aria piccole e uniformemente separate nella soluzione, che indicano che il prepolimero PDMS e l'agente polimerizzante sono ben miscelati.
- Mettere il piatto di vetro in un essiccatore sottovuoto ed evacuare per 15-25 s. Ripetere questo processo fino a quando non ci sono bolle d'aria nella miscela.
- Mettere la pirofila in un forno di essiccazione impostato a 50 °C per 1 ora per consentire alla miscela di polimerizzare. Dopo l'incubazione, utilizzare un bisturi per tagliare il PDMS in blocchi di dimensioni adeguate lunghi circa 1,2 cm e larghi 1 cm.
NOTA: la lunghezza del blocco PDMS è coerente con la larghezza del chip e la larghezza viene selezionata per garantire che vi sia spazio sufficiente nel mezzo per la ceramica piezoelettrica quando due blocchi PDMS sono aderenti sul chip.
- Aggiungere 30 ml di prepolimero in un piatto di vetro da 100 mm (di diametro). Aggiungere 3 ml di agente polimerizzante al prepolimero con una siringa.
- Associare il blocco PDMS al chip.
- Fori di punzonatura nel blocco PDMS per porte di ingresso e uscita con un ago cavo di 1 mm di diametro. Mettere i blocchi PDMS e il chip (lato posteriore verso l'alto) in un pulitore al plasma per 1 minuto.
- Allineare i fori sui blocchi PDMS con l'ingresso e l'uscita del truciolo. Premere delicatamente i blocchi PDMS sul chip per 15 s. Ciò dovrebbe causare il legame tra i blocchi PDMS e la superficie del chip.
- Collegare il catetere in politetrafluoroetilene (PTFE) al chip (Figura 2B).
- Tagliare due pezzi di catetere in PTFE con un diametro interno di 0,8 mm e una lunghezza di 10 cm. Piegare un ago in acciaio inossidabile con un diametro interno di 0,7 mm e una lunghezza di 1,5 cm per 90° in una forma a L. Collegarlo a un'estremità del catetere. Preparare due di questi cateteri con aghi.
- Inserire gli aghi in acciaio inossidabile nei fori dei blocchi PDMS. Per l'ingresso, collegare un ago erogatore da 19 G all'altra estremità del catetere come connettore per una siringa.
- Dopo aver completato i passaggi precedenti, iniettare acqua deionizzata per testare la tenuta del canale complessivo. Impermeabile all'acqua significa una buona tenuta.
- Gruppo ceramico piezoelettrico (Figura 2C)
- Utilizzare una taglierina diamantata per tagliare fogli di ceramica piezoelettrica con un diametro di 2 cm in quattro strisce con una larghezza di 5 mm.
- Assicurarsi che la frequenza di risonanza della ceramica piezoelettrica corrisponda alla larghezza del microcanale del chip. Per il microcanale largo 740 μm e 250 μm, utilizzare ceramiche piezoelettriche con frequenze di risonanza rispettivamente di 1 MHz e 3 MHz.
- Saldare i fili su entrambi i lati della ceramica piezoelettrica ad un'estremità.
- Incollare la ceramica piezoelettrica al centro del retro del chip con colla cianoacrilica.
- Per distribuire la colla in modo uniforme, posizionare una goccia di colla sulla ceramica piezoelettrica, lisciare la colla con uno stuzzicadenti e rimuovere la colla in eccesso. Quindi, premerlo rapidamente sul chip e continuare a premere per circa 1 minuto. Assicurarsi che la ceramica piezoelettrica e il chip siano saldamente legati e contattati uniformemente.
- Montare il microdispositivo (Figura 2D).
- Tagliare un pezzo di PDMS (circa 1,5 cm di lunghezza e 1 cm di larghezza) come base del microdispositivo. Utilizzando del nastro biadesivo, incollare un lato della base ai blocchi PDMS di ingresso e uscita e l'altro lato a un vetrino trasparente. Fissare l'intero microdispositivo allo stadio del microscopio per mantenere il chip su un piano focale.
2. Preparazione del campione
- Preparazione di soluzioni di particelle standard di polistirene.
- Aggiungere 0,05 mL di soluzione di particelle di polistirene (6 μm di diametro) (2,1 x 108 particelle/ml) a 10 mL di soluzione salina tamponata fosfato (PBS) e mescolare bene.
NOTA: Al fine di ridurre l'errore di misura causato dalla variazione della densità di energia acustica, la soluzione di particelle di polistirene è stata miscelata con la soluzione campione in ciascun esperimento come calibrazione.
- Aggiungere 0,05 mL di soluzione di particelle di polistirene (6 μm di diametro) (2,1 x 108 particelle/ml) a 10 mL di soluzione salina tamponata fosfato (PBS) e mescolare bene.
- Preparazione di sospensioni cellulari.
- Lavare le cellule aderenti (ad esempio, MCF7, MDA-MB-231, HCT116) al 90% di confluenza (~5 x 105 cellule) con PBS. Aggiungere 500 μL di tripsina allo 0,25% (1x) per 1-2 minuti a temperatura ambiente (25 °C). Rimuovere la tripsina, aggiungere 1 mL di terreno completo e formare una sospensione cellulare mediante pipettaggio.
- Centrifugare la sospensione cellulare a 100 x g per 5 minuti. Rimuovere il surnatante e risospendere in 0,5-1 mL di PBS per ottenere una sospensione cellulare. Le cellule sono state contate con un emocitometro e la concentrazione era di circa 3-5 x 105 cellule / ml.
- Preparazione della sospensione del nucleo cellulare
- Eseguire il passaggio 2.2. Quindi, rimuovere il surnatante e aggiungere 200 μL di reagente di estrazione della proteina citoplasmatica A (integrato con PMSF all'1%) per pellet cellulare da 20 μL (circa 5 milioni di cellule) e mescolare bene.
- Vortice la miscela di cui sopra a 220 x g per 5 s, quindi mettere sul bagno ghiacciato per 10 minuti. Dopo l'incubazione, aggiungere 10 μL di reagente B per l'estrazione della proteina citoplasmatica alla soluzione.
- Vortice a 220 x g per 5 s. Mettere sul bagno ghiacciato per 1 minuto e vortice di nuovo a 220 x g per 5 s. Quindi, infine centrifugare a 1.000 x g per 5 minuti a 4 °C.
NOTA: Il rapporto di volume dei reagenti di estrazione delle proteine citoplasmatiche A e B è 20:1. - Rimuovere il surnatante e risospendere il pellet in 1 mL di PBS. Quindi, centrifugare a 1.000 x g a 4 °C per 4 min. Rimuovere il surnatante e risospendere in 100 μL di PBS come sospensione del nucleo cellulare.
- Aggiungere il blu di tripano alla sospensione del nucleo cellulare sopra indicato e colorare a temperatura ambiente (25 °C) per 4 minuti. Il rapporto volumetrico tra la soluzione di tripano e la sospensione del nucleo è 1:1. Conta il numero di nuclei al microscopio invertito con un obiettivo 10x.
NOTA: Per identificare chiaramente i nuclei cellulari al microscopio, è necessaria la colorazione blu tripano. La soluzione di blu di Trypan deve essere a bagnomaria a 37 °C per 10 minuti prima dell'uso per una colorazione efficace. - Diluire la sospensione del nucleo cellulare di cui sopra con tampone PBS ad una concentrazione di 2-3 x 105 nucleo/ml. Filtrare la sospensione del nucleo cellulare attraverso un setaccio da 70 μm.
3. Misurare la comprimibilità della cellula e del nucleo
- Impostazione del sistema di misurazione (Figura 3)
- Accendere la sorgente luminosa del microscopio e aprire il software della fotocamera. Usa l'obiettivo 4x per trovare la posizione centrale del microcanale, cioè la posizione della ceramica piezoelettrica.
- Collegare i fili e saldarli ai terminali positivi e negativi dell'uscita del generatore di segnale sulla ceramica piezoelettrica, rispettivamente.
- Posizionare la siringa sulla pompa di microiniezione e collegarla al catetere di ingresso. Posizionare un piccolo contenitore all'estremità del catetere di uscita per trattenere il fluido che fuoriesce dal microcanale.
- Determinare i parametri di misurazione
- Aspirare la soluzione di particelle di polistirene con la siringa e iniettarla nel microcanale del chip. Evitare bolle d'aria nel microcanale del chip per garantire una misurazione accurata. Assicurarsi che le particelle siano distribuite uniformemente nel microcanale del chip.
NOTA: La misurazione può essere condotta senza flusso o pompa a siringa. Se necessario, la portata della pompa di microiniezione deve essere impostata su un valore corretto. Qui, l'intervallo di portata è 0-20 μL / h. - Impostare l'uscita del generatore di segnale su un segnale sinusoidale con una frequenza di 1 MHz (3 MHz per la misurazione del nucleo della cella) e una tensione picco-picco (Vpp) di 10 V.
- Regolare la frequenza del segnale fino a quando non si osserva che le particelle si muovono verso la linea mediana del microcanale e rimangono in movimento in avanti lungo la linea mediana dopo aver raggiunto la linea mediana (Figura 4).
NOTA: La velocità delle particelle che si muovono verso la linea mediana è determinata dall'ampiezza della tensione, che può essere regolata tra 5 Vpp e 20 Vpp.
- Aspirare la soluzione di particelle di polistirene con la siringa e iniettarla nel microcanale del chip. Evitare bolle d'aria nel microcanale del chip per garantire una misurazione accurata. Assicurarsi che le particelle siano distribuite uniformemente nel microcanale del chip.
- Misurare cellule e nuclei
- Mescolare 1 mL di sospensione di cellule o nucleo con la soluzione di particelle standard nel rapporto di 1:1 e iniettarla nel microcanale con una siringa.
- Avviare la registrazione con la telecamera CCD quando le cellule o i nuclei entrano nel campo visivo. Quindi, accendere il generatore di segnale. Interrompere la registrazione quando le cellule o i nuclei raggiungono la linea mediana.
- Risciacquare il microcanale con acqua deionizzata, alcool al 75% e acqua deionizzata in sequenza per un uso successivo.
4. Trattamento dei dati
- Mappare le traiettorie delle particelle o delle cellule.
- Importare il video acquisito nel software ImageJ: File > Apri> Seleziona cartella. Fare clic sulla forma ellittica nella barra degli strumenti del software ImageJ per scegliere una cella di interesse e la relativa particella adiacente (Figura 5).
- Come illustrato nella Figura 5, i parametri di misurazione preimpostati nel software ImageJ sono Analyze→ Set Measurement →Area, Centroid, Display Label.
- Prendendo il fotogramma in cui la cellula o la particella bersaglio subisce uno spostamento longitudinale come fotogramma iniziale; Registrare la posizione dei pixel e le dimensioni della cella o della particella in ciascun fotogramma fino a raggiungere la linea mediana del microcanale. Esportare i dati come file di foglio di calcolo e ripetere il passaggio fino a ottenere traiettorie per tutte le celle di interesse.
- Coordinare la trasformazione e la correzione.
- Registrare le coordinate dei pixel dei quattro angoli del microcanale in questo campo visivo come (0, y1), (0, y2), (x3, y3), (x4, y4). Qui x3 = x4.
- Per ogni punto di misura (xi, yi), calcolare la nuova coordinata (xi', yi') dopo la correzione della rotazione utilizzando le seguenti formule:
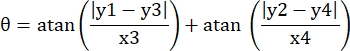
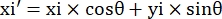

- Converti le coordinate pixel in coordinate di dimensioni reali. Le coordinate effettive possono essere ottenute moltiplicando le coordinate dei pixel per il rapporto. Il rapporto era la larghezza effettiva del microcanale divisa per la larghezza dei pixel (H) del microcanale.
- Trasformare e correggere le coordinate dei pixel delle celle e delle particelle ottenute nel passo 4.1 nei dati finali della traiettoria del movimento. Tutte le coordinate meno le coordinate dell'angolo in basso a sinistra, cioè (0, y2). Il frame rate del video è di 40 fotogrammi al secondo, quindi moltiplicare il numero di fotogrammi corrispondenti a ciascuna coordinata per 0,025 s per ottenere il tempo di movimento delle particelle, ottenendo così il cambiamento della posizione nella direzione y con il tempo.
- Calcolare la densità di energia acustica (Figura 6A,B).
- Il moto della cella o della particella nella direzione Y è guidato dalla forza acustica F ac e dalla forza idrodinamica FD. Calcolate la traiettoria del movimento utilizzando le seguenti formule:
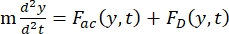 (1)
(1)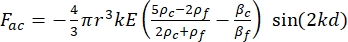 (2)
(2)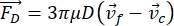 (3)
(3)
dove r e D sono il raggio e il diametro della cella o della particella, ρ e β sono la densità e la comprimibilità, ν è il vettore velocità. I pedice c e f indicano rispettivamente la cellula e il fluido. d è la distanza dal nodo di pressione acustica più vicino, μ è la viscosità dinamica del fluido, k è il numero d'onda ed E è la densità di energia acustica.
NOTA: La densità di MCF7, HCT116, A549 e dei nuclei cellulari era rispettivamente 1068 kg/m 3, 1077 kg/m 3, 1073 kg/m 3 e 1155 kg/m 3, rispettivamente 12,16,17. - Secondo le formule descritte al punto 4.3.1, utilizzare il software MATLAB per ottenere la soluzione numerica per la traiettoria standard delle particelle sotto il metodo del campo acustico con differenza finita.
- All'interno dell'intervallo di campo acustico preimpostato, modificare la densità di energia acustica e adattare la soluzione numerica (ottenuta al punto 4.3.2) e la traiettoria di movimento misurata (ottenuta al punto 4.2) per la particella standard. Selezionare il miglior risultato di adattamento in base all'errore quadratico medio di montaggio. La densità di energia acustica ottenuta qui viene utilizzata come parametro per il successivo calcolo della comprimibilità cellulare.
- Il moto della cella o della particella nella direzione Y è guidato dalla forza acustica F ac e dalla forza idrodinamica FD. Calcolate la traiettoria del movimento utilizzando le seguenti formule:
- Calcolare la comprimibilità delle celle (Figura 6C,D).
- Impostare la densità di energia acustica sul valore ottenuto al punto 4.3.3.
- Secondo le formule descritte al punto 4.3.1, utilizzare il software MATLAB per ottenere la soluzione numerica per la traiettoria della cella sotto il campo acustico con metodo alle differenze finite.
- Analogamente al punto 4.3.3, all'interno dell'intervallo di comprimibilità preimpostato, modificare la comprimibilità e adattare la soluzione numerica (ottenuta al punto 4.4.2) e la traiettoria di movimento misurata per la cella (ottenuta al punto 4.2). Utilizzare il coefficiente di comprimibilità corrispondente al risultato più adatto come comprimibilità della cella misurata.
Risultati
Qui, il lavoro ha presentato un protocollo per la costruzione di un sistema di misura della comprimibilità cellulare veloce e non distruttivo basato su microdispositivo acustofluidico e ha dimostrato i suoi vantaggi per misurare cella e nucleo in diverse situazioni. La figura 1 mostra lo schema del canale microfluidico. I componenti e l'assemblaggio del microdispositivo acustofluidico sono mostrati nella Figura 2. La figura 3 mostr...
Discussione
I metodi di misurazione della meccanica cellulare comunemente usati sono AFM, aspirazione di micropipette, metodi di microfluidica, tecnica a piastre parallele, pinzette ottiche, barelle ottiche e metodi acustici20. I metodi di microfluidica possono funzionare con tre approcci: micro-costrizione, flusso estensionale e flusso di taglio. Tra questi, barella ottica, pinzette ottiche, metodi acustici, flusso estensionale e approcci di flusso di taglio sono misurazioni senza contatto. A differenza dell...
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti o altri conflitti di interesse.
Riconoscimenti
Questo studio è stato sostenuto dalla National Natural Science Foundation of China (numeri di sovvenzione 12075330 e U1932165) e dalla Natural Science Foundation della provincia del Guangdong, Cina (numero di sovvenzione 2020A1515010270).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% trypsin(1x) | GIBCO | 15050-065 | |
| 502 glue | Evo-bond | cyanoacrylate glue | |
| A549 | ATCC | CCL-185 | lung adenocarcinoma |
| Cytonucleoprotein and cytoplasmic protein extraction kit | Beyotime | P0027 | Contains cytoplasmic protein extraction reagents A and B |
| Dulbecco’s modified Eagle medium (DMEM) | corning | 10-013-CVRC | |
| Fetal Bovine Srum(FBS) | AUSGENEX | FBS500-S | |
| HCT116 | ATCC | CCL247 | colorectal carcinoma |
| Heat-resistant glass | Pyrex | ||
| Leibovitz’s L-15 medium | GIBCO | 11415-064 | |
| MCF-7 | ATCC | HTB-22 | breast Adenocarcinoma |
| MDA-MB-231 | ATCC | HTB-26 | breast Adenocarcinoma |
| Minimum Essential Medium (MEM) | corning | 10-010-CV | |
| Penicillin-Streptomycin | GIBCO | 15140-122 | |
| Phosphate buffer | corning | 21-040-cvc | |
| PMSF | Beyotime | ST506 | 100mM |
| Polybead Polystyrene Red Dyed Microsphere | polysciences | 15714 | The diameter of microshpere is 6.00µm |
| propidium iodide(PI) | Sigma-Aldrich | P4170 | |
| SYLGARD 184Silicone ELASTOMER | Dow-Corning | 1673921 | Contains prepolymers and curing agents |
| Trypan Blue | Beyotime | C0011 |
Riferimenti
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nature Reviews. Cancer. 11 (7), 512-522 (2011).
- Frame, F. M., et al. HDAC inhibitor confers radiosensitivity to prostate stem-like cells. British Journal of Cancer. 109 (12), 3023-3033 (2013).
- Tseng, Y., Kole, T. P., Wirtz, D. Micromechanical mapping of live cells by multiple-particle-tracking microrheology. Biophysical Journal. 83 (6), 3162-3176 (2002).
- Möller, W., Brown, D. M., Kreyling, W. G., Stone, V. Ultrafine particles cause cytoskeletal dysfunctions in macrophages: role of intracellular calcium. Particle and Fibre Toxicology. 2, 7 (2005).
- Wang, X., et al. A three-dimensional magnetic tweezer system for intraembryonic navigation and measurement. IEEE Transactions on Robotics. 34 (1), 240-247 (2018).
- Machida, S., et al. Direct manipulation of intracellular stress fibres using a hook-shaped AFM probe. Nanotechnology. 21 (38), 385102 (2010).
- Bufi, N., et al. Human primary immune cells exhibit distinct mechanical properties that are modified by inflammation. Biophysical Journal. 108 (9), 2181-2190 (2015).
- Hogan, B., Babataheri, A., Hwang, Y., Barakat, A. I., Husson, J. Characterizing cell adhesion by using micropipette aspiration. Biophysical Journal. 109 (2), 209-219 (2015).
- Jung, J. -. W., et al. Ionising radiation induces changes associated with epithelial-mesenchymal transdifferentiation and increased cell motility of A549 lung epithelial cells. European Journal of Cancer. 43 (7), 1214-1224 (2007).
- Hartono, D., et al. On-chip measurements of cell compressibility via acoustic radiation. Lab-on-a-Chip. 11 (23), 4072-4080 (2011).
- Sitters, G., et al. Acoustic force spectroscopy. Nature Methods. 12 (1), 47-50 (2015).
- Augustsson, P., Karlsen, J. T., Su, H. -. W., Bruus, H., Voldman, J. Iso-acoustic focusing of cells for size-insensitive acousto-mechanical phenotyping. Nature Communications. 7 (1), 11556 (2016).
- Cushing, K. W., et al. Ultrasound characterization of microbead and cell suspensions by speed of sound measurements of neutrally buoyant samples. Analytical Chemistry. 89 (17), 8917-8923 (2017).
- Riaud, A., Wang, W., Thai, A. L. P., Taly, V. Mechanical characterization of cells and microspheres sorted by acoustophoresis with in-line resistive pulse sensing. Physical Review Applied. 13 (3), 034058 (2020).
- Petersson, F., Aberg, L., Swärd-Nilsson, A. -. M., Free Laurell, T. flow acoustophoresis: microfluidic-based mode of particle and cell separation. Analytical Chemistry. 79 (14), 5117-5123 (2007).
- Griwatz, C., Brandt, B., Assmann, G., Zänker, K. S. An immunological enrichment method for epithelial cells from peripheral blood. Journal of Immunological Methods. 183 (2), 251-265 (1995).
- Katholnig, K., Poglitsch, M., Hengstschläger, M., Weichhart, T. Lysis gradient centrifugation: a flexible method for the isolation of nuclei from primary cells. Methods in Molecular Biology. 1228, 15-23 (2015).
- Fu, Q., Zhang, Y., Huang, T., Liang, Y., Liu, Y. Measurement of cell compressibility changes during epithelial-mesenchymal transition based on acoustofluidic microdevice. Biomicrofluidics. 15 (6), 064101 (2021).
- Zhang, Y., et al. Ionizing radiation-induced DNA damage responses affect cell compressibility. Biochemical and Biophysical Research Communications. 603, 116-122 (2022).
- Hao, Y., et al. Mechanical properties of single cells: Measurement methods and applications. Biotechnology Advances. 45, 107648 (2020).
- Yousafzai, M., et al. Effect of neighboring cells on cell stiffness measured by optical tweezers indentation. Journal of Biomedical Optics. 21 (5), 057004 (2016).
- Wei, M. -. T., et al. A comparative study of living cell micromechanical properties by oscillatory optical tweezers. Optics Express. 16 (12), 8594-8603 (2008).
- Khan, Z. S., Vanapalli, S. A. Probing the mechanical properties of brain cancer cells using a microfluidic cell squeezer device. Biomicrofluidics. 7 (1), 011806 (2013).
- Hirawa, S., Masudo, T., Okada, T. Acoustic recognition of counterions in ion-exchange resins. Analytical Chemistry. 79 (7), 3003-3007 (2007).
- Joosse, S. A., Gorges, T. M., Biology Pantel, K. detection, and clinical implications of circulating tumor cells. EMBO Molecular Medicine. 7 (1), 1-11 (2015).
- Martin, O. A., Anderson, R. L., Narayan, K., MacManus, M. P. Does the mobilization of circulating tumour cells during cancer therapy cause metastasis. Nature Reviews Clinical Oncology. 14 (1), 32-44 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon