È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento e caratterizzazione del microbiota naturale del nematode modello Caenorhabditis elegans
In questo articolo
Riepilogo
Caenorhabditis elegans è una delle principali specie modello in biologia, ma quasi tutte le ricerche vengono eseguite in assenza dei suoi microbi naturalmente associati. I metodi qui descritti aiuteranno a migliorare la nostra comprensione della diversità dei microbi associati come base per la futura ricerca funzionale di C. elegans.
Abstract
Il nematode Caenorhabditis elegans interagisce con una grande varietà di microrganismi in natura. In generale, C. elegans si trova comunemente nella materia vegetale marcia, in particolare nei frutti marci come le mele o sui cumuli di compost. È anche associato a determinati ospiti invertebrati come lumache e woodlice. Questi habitat sono ricchi di microbi, che servono da cibo per C. elegans e che possono anche colonizzare persistentemente l'intestino del nematode. Ad oggi, l'esatta diversità e consistenza del microbiota nativo di C. elegans tra habitat e posizioni geografiche non è completamente compresa. Qui, descriviamo un approccio adatto per isolare C. elegans dalla natura e caratterizzare il microbiota dei vermi. I nematodi possono essere facilmente isolati dal materiale di compost, mele in decomposizione, lumache o attratti mettendo mele su cumuli di compost. Il momento migliore per trovare C. elegans nell'emisfero settentrionale va da settembre a novembre. I vermi possono essere lavati via dal materiale del substrato raccolto immergendo il substrato in una soluzione tampone, seguita dalla raccolta dei nematodi e dal loro trasferimento sul mezzo di crescita dei nematodi o sul tampone PCR per le successive analisi. Illustriamo inoltre come i campioni possono essere utilizzati per isolare e purificare i microrganismi associati ai vermi e per elaborare i vermi per l'analisi dell'RNA ribosomiale 16S della composizione della comunità del microbiota. Nel complesso, i metodi descritti possono stimolare nuove ricerche sulla caratterizzazione del microbiota di C. elegans in habitat e posizioni geografiche, contribuendo così a ottenere una comprensione completa della diversità e della stabilità del microbiota del nematode come base per la futura ricerca funzionale.
Introduzione
In natura, C. elegans si trova comunemente nella materia vegetale marcia, in particolare frutti marci come mele o cumuli di compost1. È anche associato ad alcuni ospiti invertebrati come lumache e woodlice 2,3. Questi habitat sono ricchi di microbi, che non solo servono come cibo per il verme, ma possono anche formare associazioni stabili con esso. Le informazioni sulla diversità dei microrganismi naturalmente associati sono state pubblicate solo nel 2016 4,5,6. Da allora, questi e solo pochi studi più recenti hanno rivelato che C. elegans è associato a una varietà di batteri e funghi, più comunemente tra cui batteri del genere Pseudomonas, Enterobacter, Ochrobactrum, Erwinia, Comamonas, Gluconobacter, e molti altri 6,7,8. Diversi batteri associati possono colonizzare stabilmente l'intestino del verme, anche se non tutti 6,9,10,11,12. È probabile che siano di fondamentale importanza per la nostra comprensione della biologia di C. elegans perché possono fornire nutrimento, proteggere dagli agenti patogeni e possibilmente da altri fattori di stress e influenzare i tratti centrali della storia della vita come il tasso riproduttivo, lo sviluppo o le risposte comportamentali.
Ad esempio, isolati naturalmente associati dei generi Pseudomonas, Ochrobactrum e anche Enterobacter o Gluconobacter possono proteggere il verme dall'infezione patogena e dall'uccisione in modi distinti 5,6,11,13,14. Un isolato specifico del genere Comamonas influenza la risposta dietetica dei nematodi, lo sviluppo, la durata della vita e la fertilità15,16,17. I batteri Providencia producono la tiramina neuromodulatrice e quindi modulano l'attività del sistema nervoso ospite e le conseguenti risposte comportamentali18. Un insieme di diversi batteri naturalmente associati hanno dimostrato di influenzare il tasso di crescita della popolazione, la fertilità e le risposte comportamentali 5,6,9,11,19.
Ad oggi, l'esatta diversità e consistenza del microbiota nativo di C. elegans tra gli habitat e le posizioni geografiche non sono completamente comprese, e ulteriori associazioni tra il verme e i microbi del suo ambiente rimangono da scoprire. Diversi studi precedenti hanno utilizzato ceppi batterici isolati da alcuni ambienti del suolo, habitat naturali di C. elegans o da esperimenti sul mesocosmo (cioè ambienti di laboratorio che ricreano habitat naturali) con ceppi di laboratorio di C. elegans 4,5,20. Anche se questi studi hanno ottenuto nuove intuizioni sull'influenza dei microbi su specifici tratti dei nematodi (ad esempio, il metabolismo dei nematodi21), la rilevanza di queste interazioni per la biologia di C. elegans in natura non è chiara. Pertanto, questo manoscritto descrive i metodi per isolare direttamente C. elegans dalla natura e per isolare e successivamente caratterizzare i microbi naturalmente associati sia da singoli vermi che da gruppi di vermi. I metodi descritti sono una versione aggiornata e migliorata delle procedure utilizzate in precedenza per l'isolamento e la caratterizzazione di C. elegans naturale e del suo microbiota nativo 2,6,7. Considerando che C. elegans è ampiamente presente nella decomposizione della materia vegetale in tutto il mondo (specialmente nei frutti in decomposizione, nelle regioni temperate e in autunno)1,2,22,23,24,25, questo protocollo può essere applicato da qualsiasi laboratorio ogni volta che c'è interesse a mettere in relazione C. elegans tratti di microbi naturalmente associati e quindi un contesto più naturalmente rilevante. Quest'ultimo è fondamentale per una piena comprensione della biologia del nematode perché è noto da una varietà di altri sistemi ospiti che il microbiota associato può influenzare diverse caratteristiche della storia di vita26, un aspetto che è attualmente ampiamente trascurato nella moltitudine di studi di C. elegans in quasi tutte le discipline delle scienze della vita.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparazione di buffer e supporti
- Preparare S-buffer aggiungendo 5,85 g di NaCl, 1,123 g di K 2 HPO 4, 5,926 g di KH 2 PO4 e 1 L di H2O deionizzato in un matraccio e in autoclaveper 20 minuti a 121 °C.
- Preparare un mezzo viscoso aggiungendo tampone S contenente 1,2% (p/v) idrossimetilcellulosa (la sostanza che causa la viscosità del mezzo), 5 mg/ml di colesterolo, 1 mM MgSO4, 1 mM CaCl2 e 0,1% (v/v) acetone. Autoclavare e agitare il mezzo viscoso fino a renderlo completamente omogeneo.
NOTA: questa operazione può richiedere più ore. Inoltre, il tampone S può essere preparato direttamente e aggiunto al mezzo viscoso senza previa sterilizzazione. - Preparare M9-tampone aggiungendo 3 g di KH 2 PO 4, 6 g di NA 2 HPO4·2 H 2 O, 5 g di NaCl e 1 L di H2O deionizzato in un matraccio da 1 L. Autoclavare la soluzione e, dopo raffreddamento, aggiungere 1 mL di 1 M MgSO 4 (123,24 g di MgSO4·7H 2 O in 500 mL di H2O deionizzato, filtro sterilizzato).
- Preparare la soluzione madre di Triton X-100 al 10% (v/v) mescolando 5 mL di Triton X-100 con 45 mL di tampone M9. Filtrare-sterilizzare la soluzione utilizzando un filtro da 0,2 μm.
- Preparare il tampone M9 con Triton X-100 (M9-T) aggiungendo 2,5 mL della soluzione madre Triton X-100 al 10% (v/v) a 1 L di tampone M9 dopo l'autoclave per ottenere lo 0,025% (v/v) di M9-T.
- Preparare il 30% (v/v) di glicerolo in S-buffer mescolando 15 mL di glicerolo sterile al 100% e 35 mL di tampone S sterile in una provetta da 50 ml.
2. Preparazione di campioni ambientali (Figura 1)
- Raccogli campioni ambientali come compost o frutta marcia e metti ogni campione in un singolo sacchetto di plastica, tubo o altro contenitore pulito.
NOTA: Per attirare i nematodi, le mele possono essere poste sul compost diverse settimane prima del campionamento. - Distribuire uniformemente i pezzi del campione ambientale in una capsula di Petri vuota e sterile di 9 cm.
NOTA: I campioni con livelli più elevati di decadimento hanno maggiori probabilità di contenere C. elegans. Opzionalmente, una capsula di Petri riempita con agar medium (PFM) senza peptone può essere utilizzata per aumentare il contrasto. - Coprire accuratamente il campione con circa 20 ml di mezzo viscoso sterile.
NOTA: I nematodi galleggiano in superficie entro 1-2 ore. Il mezzo viscoso rallenta il movimento dei vermi e rende più facile il campionamento. Il buffer M9 sterile può essere utilizzato come alternativa, tuttavia, il movimento del worm sarà più veloce.
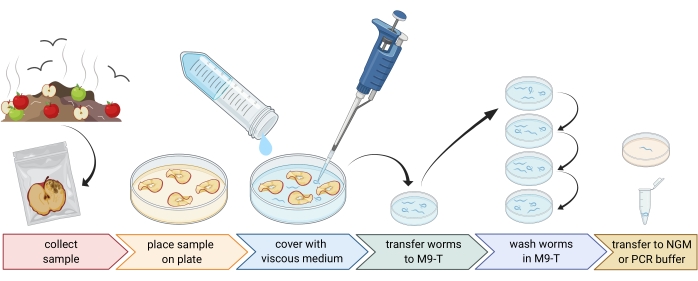
Figura 1: Isolamento dei nematodi dai substrati. I campioni di substrato vengono posti in piastre di Petri vuote e ricoperti con mezzo viscoso per scovare i nematodi. I nematodi vengono trasferiti a M9-T e ripetutamente lavati per rimuovere i batteri dall'esterno. I singoli nematodi possono essere utilizzati per l'isolamento del DNA, l'isolamento dei batteri associati o posizionati su piastre di agar per colture di popolazioni di vermi. Figura creata con BioRender.com. Fare clic qui per visualizzare una versione ingrandita di questa figura.
3. Isolamento dei nematodi Caenorhabditis (Figura 1)
- Ricerca di nematodi Caenorhabditis utilizzando un microscopio da dissezione, seguendo le linee guida presentate nel capitolo WormBook sull'isolamento di C. elegans e nematodi correlati da Barrière e Félix27.
NOTA: Gli ermafroditi / femmine di Caenorhabditis hanno un intestino di colore marrone chiaro (sotto illuminazione trasmessa). Inoltre, le cellule intestinali hanno grandi nuclei cellulari, che sono visibili come punti bianchi. Al contrario, altre specie di nematodi hanno spesso un intestino marrone scuro e possono mostrare asimmetria anteroposteriore nell'intensità del pigmento e di solito nessun nucleo visibile. La vulva delle femmine adulte di Caenorhabditis ermafrodite/femmine si trova nel mezzo dell'animale. Questa localizzazione della vulva non è sempre mostrata da altri taxa di nematodi. I nematodi Caenorhabditis possiedono due bulbi faringei, che sono entrambi visibili con sufficiente ingrandimento e illuminazione trasmessa e contrasto con molti altri taxa di nematodi. I nematodi Caenorhabditis hanno una coda appuntita, mentre alcuni altri nematodi hanno una coda rotonda. - Raccogliere i nematodi Caenorhabditis al microscopio da dissezione nel minor liquido possibile utilizzando una pipetta da 20-100 μL. Trasferire i nematodi raccolti direttamente su 1-3 ml di M9-T sterile in una capsula di Petri sterile da 3 cm per lavare i vermi per rimuovere i microbi non attaccati (fase 3.3) o, in alternativa, trasferire singoli vermi su una piastra con terreno di crescita nematode (NGM) per stabilire una popolazione di vermi (fase 3.4).
- Lavare i nematodi per rimuovere i microbi dall'esterno del nematode.
NOTA: Questo protocollo arricchisce i batteri intestinali ma non elimina completamente i batteri che si attaccano alla cuticola del verme.- Incubare i nematodi per 10-15 minuti in M9-T.
- Pipettare i nematodi nel minor liquido possibile in 1-3 ml di M9-T fresco in una nuova capsula di Petri sterile da 3 cm.
- Ripetere l'incubazione e trasferire i nematodi su M9-T fresco altre due volte.
NOTA: i seguenti passaggi possono essere eseguiti con singoli worm o con popolazioni di worm. I vermi possono ora essere utilizzati per la caratterizzazione di C. elegans e microbi associati, nonché per l'isolamento dei batteri (fasi 4 e 5). In alternativa, possono essere trasferiti singolarmente alle piastre NGM per stabilire una popolazione di vermi (fase 3.4).
- Per ottenere una popolazione di vermi, pipettare un singolo nematode su una piastra NGM.
NOTA: Solo i nematodi ermafroditi come C. elegans o C . briggsae sono in grado di produrre prole da singoli vermi. Tuttavia, singoli vermi di altre specie possono ancora produrre prole se sono già stati accoppiati.- I nematodi naturalmente isolati di solito contengono batteri nel loro intestino, che versano sulle piastre NGM, dove questi batteri crescono e sono disponibili come cibo per C. elegans. Non aggiungere organismi alimentari separati come l'organismo alimentare standard da laboratorio, E. coli ceppo OP50.
NOTA: L'allevamento di vermi su piastre influenza la composizione della comunità batterica associata, ma la composizione è ancora paragonabile a quella degli isolati naturali di C. elegans 6,7. - Lasciare proliferare i nematodi fino a 10 giorni alla temperatura appropriata (ad esempio, 15-20 °C per le località temperate). Congelare questi nematodi (fase 3.5) o usarli per la caratterizzazione di C. elegans e microbi associati, nonché per l'isolamento dei microbi (fasi 4 e 5).
- I nematodi naturalmente isolati di solito contengono batteri nel loro intestino, che versano sulle piastre NGM, dove questi batteri crescono e sono disponibili come cibo per C. elegans. Non aggiungere organismi alimentari separati come l'organismo alimentare standard da laboratorio, E. coli ceppo OP50.
- Congelamento dei nematodi per la conservazione a lungo termine
- Lasciare i nematodi sui piatti fino a quando i batteri alimentari sono scomparsi, e ci sono principalmente piccoli stadi larvali sui piatti. Lavare i vermi dalle piastre in 1,5 ml di tampone S.
- Mescolare accuratamente 500 μL di tampone S contenente verme con 500 μL di glicerolo al 30% (v/v) in tampone S in una provetta sterile da 2 ml. Congelare prontamente i tubi a -80 °C per la conservazione a lungo termine, altrimenti il glicerolo potrebbe danneggiare i nematodi.
4. Preparazione dei vermi per l'identificazione molecolare di C. elegans e microbi
- Per un'identificazione imparziale dei microbi associati ai nematodi, preparare una piastra a 96 pozzetti con tre perle sterili da 1 mm, 19,5 μL di tampone PCR e 0,5 μL di proteinasi K (20 mg/ml) per pozzetto. Pipetta un individuo, nematode lavato ad ogni pozzetto, trasferendo meno liquido possibile.
- Possono essere utilizzate anche popolazioni di vermi. Per questo, lavare i vermi dalle piastre con M9-buffer e trasferire ~ 300 μL di worm-contenente M9-buffer a 2 mL tubi con 10-15 perline.
- Rompere i nematodi usando un omogeneizzatore di perline (ad esempio, battito di perline per 3 minuti a 30 Hz). Centrifugare brevemente la piastra o i tubi per portare il liquido sul fondo (ad esempio, per 10 s a 8000 x g a temperatura ambiente [RT]).
- Identificazione di C. elegans
- Isolare il DNA dei singoli nematodi riscaldando i campioni in un ciclatore PCR per 1 ora a 50 °C e 15 minuti a 95 °C. Isolare il DNA delle popolazioni di vermi utilizzando qualsiasi metodo di isolamento scelto (esempi di protocolli di diversi metodi di isolamento utilizzando kit commerciali 7,9). Congelare il DNA a -20 °C per una conservazione a lungo termine.
- Per l'identificazione di C. elegans, utilizzare il DNA e la coppia di primer nlp30-F (Tabella 1, 5'-ACACATACAACTGATCACTCA-3') e nlp30-R (Tabella 1, 5'-TACTTTCCCCCCATCCGTATC-3') in una PCR, seguendo le istruzioni di un fornitore Taq di scelta.
- Utilizzare le seguenti condizioni di PCR: fase iniziale di denaturazione a 95 °C per 2 minuti, seguita da 35 cicli di 95 °C per 45 s, 55 °C per 30 s, 72 °C per 1 minuto e una fase di allungamento finale a 72 °C per 5 min. C. elegans produce un prodotto PCR da 154 bp.
- Caratterizzare i batteri associati ai nematodi attraverso il sequenziamento dell'amplicone 16S della regione V3-V4, utilizzando il DNA isolato.
- Preparare una libreria 16S con i primer 16S di scelta e seguire il protocollo del kit di preparazione della libreria. Un'opzione è quella di utilizzare i primer 341F (Tabella 1, 5'-CCTACGGGNGGCWGCAG-3') e 806R (Tabella 1, 5'-GACTACHVGGGTATCTAATCC-3') che coprono la regione V3-V4 del gene rRNA 16S, che si traduce in sequenze che possono essere classificate con database standard con buona risoluzione7.
NOTA: La quantità di DNA di input è fondamentale in questa fase. Il DNA acquisito da singoli vermi sarà molto inferiore a quello ottenuto dalle popolazioni di vermi. Per i singoli vermi, potrebbe essere necessario aumentare la quantità di DNA di input o aumentare la quantità di cicli di PCR7. - Le librerie possono essere sequenziate su una piattaforma di sequenziamento utilizzando un kit di sequenziamento adatto.
NOTA: Qui, la piattaforma MiSeq viene utilizzata con un kit di reagenti MiSeq adatto. I reagenti vengono costantemente migliorati e dovrebbero essere scelti secondo gli standard più recenti.
- Preparare una libreria 16S con i primer 16S di scelta e seguire il protocollo del kit di preparazione della libreria. Un'opzione è quella di utilizzare i primer 341F (Tabella 1, 5'-CCTACGGGNGGCWGCAG-3') e 806R (Tabella 1, 5'-GACTACHVGGGTATCTAATCC-3') che coprono la regione V3-V4 del gene rRNA 16S, che si traduce in sequenze che possono essere classificate con database standard con buona risoluzione7.
5. Isolamento e coltivazione di batteri associati ai nematodi (Figura 2)
- Per isolare i batteri, preparare una piastra da 96 pozzetti con tre sfere sterili da 1 mm, 20 μL di tampone M9 per pozzetto e pipettare un nematode individuale lavato in ciascun pozzetto, trasferendo meno liquido possibile.
NOTA: In alternativa, le popolazioni di vermi possono essere lavate dalle piastre con tampone M9 e ~ 300 μL di tampone M9 contenente vermi possono essere trasferiti in tubi da 2 ml con 10-15 perline. In tutti i casi, i vermi dovrebbero provenire da una coltura che è stata identificata come C. elegans utilizzando i passaggi 4.1-4.4. - Rompere i nematodi usando un omogeneizzatore di perline (ad esempio, battito di perline per 3 minuti a 30 Hz). Centrifugare brevemente la piastra o i tubi per portare il liquido sul fondo (ad esempio, per 10 s a 8000 x g a RT).
NOTA: L'uso di questo metodo ha portato all'isolamento di taxa batterici simili a quelli rivelati dal sequenziamento dell'amplicone del rDNA 16S7, suggerendo che il battito delle perline descritto lascia intatta la maggior parte delle cellule batteriche. - Raccogliere il surnatante, diluirlo in serie a 1:10 e placcare fino a 100 μL su piastre di agar da 9 cm.
- Per garantire che la maggior parte dei batteri possa essere coltivata, utilizzare una varietà di mezzi alternativi con diverse composizioni nutritive, tra cui agar di soia tripticasi diluito (TSA, diluizione 1:10), agar MacConkey, agar glucosio Sabouraud, agar destrosio patate o agar destrosio peptone lievito.
- Incubare le piastre alle condizioni di temperatura media del luogo di campionamento (ad esempio, temperature comprese tra 15-20 °C per le località temperate) per 24-48 ore.
- Utilizzare la tecnica standard a tre strisce e un anello sterile per ottenere colture batteriche pure (Figura 2).
- Prelevare una singola colonia da una piastra usando un cappio sterile o uno stuzzicadenti e strisciarla su una nuova piastra di agar contenente lo stesso mezzo di agar utilizzato per la purificazione. Assicurati di utilizzare solo circa 1/3 della piastra.
- Utilizzare un nuovo anello sterile o sterilizzare un anello riutilizzabile e trascinarlo attraverso la prima striscia per creare una seconda striscia su un altro 1/3 della stessa piastra.
- Ripetete questo passaggio trascinando un ciclo sterile attraverso la seconda striscia.
- Incubare la piastra nelle stesse condizioni di crescita utilizzate per l'isolamento. Questa tecnica deve portare alla crescita di singole colonie nell'area della terza striscia.
NOTA: Potrebbe essere necessario ripetere la fase di purificazione più volte poiché gli isolati naturali tendono a formare biofilm e/o aggregati.
- Coltivare le colonie pure in un mezzo liquido (dello stesso tipo del mezzo agar) utilizzando la stessa temperatura e lo stesso tempo di crescita di cui sopra (punto 5.4)
- Preparare le scorte di batteri aggiungendo 300 μL di coltura batterica a 200 μL di glicerolo all'86% (v/v) (nel rispettivo mezzo di crescita, ad esempio TSB) e pipetta su e giù per miscelare correttamente. In alternativa, preparare le scorte di DMSO mescolando 50 μL di coltura batterica con 50 μL di DMSO al 7% (v/v). Congelare a -80 °C per una conservazione a lungo termine.
- Caratterizzazione dei batteri mediante sequenziamento dell'intero gene RNA ribosomiale 16S
- Estrarre il DNA batterico da colture liquide pure utilizzando una tecnica adatta (ad esempio, un kit di estrazione del DNA; per esperienza, un protocollo di estrazione basato su CTAB funziona molto bene22).
- Amplificare il gene dell'rRNA 16S utilizzando i primer 27F (Tabella 1, 5'-GAGAGTTTGATCCTGGCTCAG-3') e 1495R (Tabella 1, 5'-CTACGGCTACCTTGTTACGA -3')28 e le seguenti condizioni PCR: 95 °C, 2 min, 22x (95 °C, 30 s; 55 °C, 30 s; 72 °C, 100 s), e un periodo di estensione finale a 72 °C, 5 min.
- Per acquisire le sequenze complete, utilizzare inoltre due primer di sequenziamento interni, come 701F (Tabella 1, 5'-GTGTAGCGGTGAAATGGG-3') e 785R (Tabella 1, 5'-GGATTAGATACCCTGGTAGTCC-3')6.
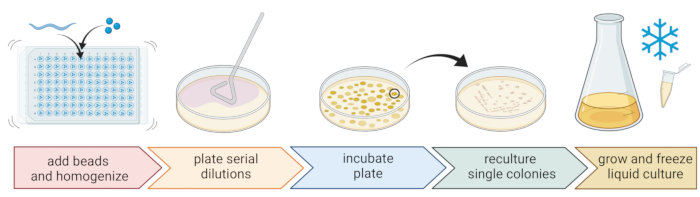
Figura 2: Identificazione delle specie e isolamento dei singoli batteri. I singoli nematodi vengono suddivisi utilizzando un omogeneizzatore di perline e il DNA viene isolato per la determinazione delle specie tramite PCR o sequenziamento. In alternativa, il materiale del nematode rotto viene diluito in serie e placcato su piastre di terreno di crescita. Le piastre vengono incubate fino alla comparsa di colonie batteriche e le singole colonie vengono striate su nuove piastre per ottenere colture pure. Le singole colonie delle colture pure vengono utilizzate per coltivare colture batteriche liquide per la preparazione di scorte batteriche per la conservazione a lungo termine a -80 °C. Figura creata con BioRender.com. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Il nematode C. elegans si trova frequentemente in frutti in decomposizione, come le mele, e anche campioni di compost. Nella Germania settentrionale, C. elegans e specie congenerose (in particolare C. remanei ma anche C. briggsae) si trovano principalmente da settembre al2 novembre. I nematodi si trovano più comunemente nella materia vegetale in decomposizione, in particolare nei frutti in decomposizione come mele o pere, e anche nel compost, in particolare nel...
Access restricted. Please log in or start a trial to view this content.
Discussione
Il nematode Caenorhabditis elegans è uno degli organismi modello più intensamente studiati nella ricerca biologica. È stato introdotto da Sydney Brenner nel 1960, originariamente per comprendere lo sviluppo e la funzione del sistema nervoso29. Da allora, C. elegans è diventato un potente modello per lo studio dei processi fondamentali in tutte le discipline biologiche, tra cui biologia comportamentale, neurobiologia, invecchiamento, biologia evolutiva, biologia cellulare, bio...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Dichiariamo di non avere alcun conflitto di interessi.
Riconoscimenti
Riconosciamo il sostegno finanziario della German Science Foundation (progetti A1.1 e A1.2 del Collaborative Research Center 1182 sull'origine e la funzione dei metaorganismi). Ringraziamo i membri del laboratorio Schulenburg per i loro consigli e supporto.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
Riferimenti
- Schulenburg, H., Félix, M. -A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4(2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19(2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485(2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728(2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604(2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24(2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59(2012).
- Barrière, A., Félix, M. -A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and related nematodes. WormBook. , ed. The C elegans research community 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670(2016).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon