È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un saggio di legame proteico della superficie cellulare basato sulla citometria a flusso per valutare la selettività e la specificità di un aptamero antitumorale
In questo articolo
Riepilogo
Un passo necessario nello sviluppo dell'aptamero antitumorale è testare il suo legame con il bersaglio. Dimostriamo un saggio basato sulla citometria a flusso per studiare questo legame, sottolineando l'importanza di includere un aptamero di controllo negativo e cellule tumorali positive o negative per quella particolare proteina.
Abstract
Una sfida chiave nello sviluppo di un aptamero antitumorale è determinare in modo efficiente la selettività e la specificità dell'aptamero sviluppato rispetto alla proteina bersaglio. Grazie ai suoi numerosi vantaggi rispetto agli anticorpi monoclonali, lo sviluppo dell'aptamero ha guadagnato un'enorme popolarità tra i ricercatori sul cancro. L'evoluzione sistematica dei ligandi mediante arricchimento esponenziale (SELEX) è il metodo più comune per sviluppare aptameri specifici per le proteine di interesse. Dopo SELEX, un test di legame rapido ed efficiente accelera il processo di identificazione, confermando la selettività e la specificità dell'aptamero.
Questo articolo spiega un saggio di legame basato su citometria a flusso passo-passo di un aptamero specifico per la molecola di adesione cellulare epiteliale (EpCAM). La glicoproteina transmembrana EpCAM è sovraespressa nella maggior parte dei carcinomi e svolge un ruolo nell'inizio, nella progressione e nelle metastasi del cancro. Pertanto, è un valido candidato per la somministrazione mirata di farmaci ai tumori. Per valutare la selettività e la specificità dell'aptamero all'EpCAM legato alla membrana, sono necessarie cellule EpCAM-positive e -negative. Inoltre, è richiesto un aptamero EpCAM non vincolante con una lunghezza e una struttura 2D simili all'aptamero con legame EpCAM. Il test di legame include diversi buffer (buffer di blocco, buffer di lavaggio, buffer di incubazione e buffer FACS) e fasi di incubazione.
L'aptamero viene incubato con le linee cellulari. Dopo le fasi di incubazione e lavaggio, le cellule saranno valutate utilizzando un saggio di citometria a flusso sensibile. L'analisi dei risultati mostra il legame dell'aptamero specifico di EpCAM alle cellule EpCAM-positive e non alle cellule EpCAM-negative. Nelle celle EpCAM-positive, questo è rappresentato come uno spostamento di banda nel legame dell'aptamero EpCAM verso destra rispetto al controllo aptamero non vincolante. Nelle cellule EpCAM-negative, le bande corrispondenti di aptameri leganti e non leganti EpCAM si sovrappongono. Ciò dimostra la selettività e la specificità dell'aptamero EpCAM. Mentre questo protocollo è focalizzato sull'aptamero EpCAM, il protocollo è applicabile ad altri aptameri pubblicati.
Introduzione
Il cancro è ancora una delle principali cause di mortalità in tutto il mondo1. Nonostante il significativo miglioramento nel trattamento del cancro negli ultimi decenni, lo sviluppo di farmaci antitumorali è ancora un argomento molto dibattuto. Questo perché la chemioterapia, come cardine del trattamento del cancro, è accompagnata da gravi effetti collaterali che limitano la compliance del paziente al trattamento. Inoltre, la resistenza al trattamento del cancro indotta dalla chemioterapia ha limitato la sua applicazione come unica scelta di intervento medico. L'applicazione di anticorpi monoclonali (mAbs) ha introdotto una migliore risposta ai trattamenti contro il cancro2. Il razionale dell'uso di mAbs era quello di migliorare l'efficacia dei chemioterapici e ridurre al minimo le loro reazioni avverse. Tuttavia, anche la somministrazione di mAbs è diventata una sfida. Ciò non era dovuto solo alle reazioni immunologiche indotte da mAb, ma anche ai costosi costi di produzione dipendenti dagli animali e alle difficili condizionidi conservazione 3. L'introduzione degli aptameri nel 19904 ha sollevato nuove speranze nel trattamento del cancro, poiché l'applicazione degli aptameri potrebbe affrontare le sfide associate ai mAbs.
Gli aptameri sono brevi sequenze di acido nucleico prodotte specificamente per un determinato bersaglio. L'evoluzione sistematica dei ligandi mediante arricchimento esponenziale (SELEX) è un metodo comune nella produzione di aptameri In SELEX, la proteina di interesse viene incubata con una libreria di sequenze nucleotidiche casuali e, attraverso una serie di cicli iterativi, l'aptamero specifico per quella proteina viene purificato. Gli aptameri hanno selettività e specificità target simili ai mAbs, e quindi lo sviluppo di farmaci in questo campo mostra promettenti applicazioni future. Gli aptameri specifici per i biomarcatori tumorali potrebbero essere applicati come singoli farmaci e strumenti diagnostici per il cancro 5,6,7. A causa della loro struttura di dimensioni nanometriche, questi aptameri potrebbero anche agire come vettori di farmaci per fornire agenti citotossici specificamente al tumore8. Ciò aumenterebbe l'efficacia della somministrazione mirata di farmaci e ridurrebbe le reazioni avverse fuori bersaglio associate alla chemioterapia. Inoltre, questi nanofarmaci hanno un'elevata penetrazione tissutale, che li rende un candidato desiderabile per la somministrazione e il trattamento di farmaci per tumori profondi. Gli aptameri possono anche essere progettati per colpire i trasportatori espressi sulla barriera emato-encefalica (BBB) per migliorare la somministrazione di farmaci ai tumori cerebrali9. Un buon esempio di tale aptamero sono gli aptameri bifunzionali, che prendono di mira il recettore della transferrina (TfR)10 per migliorare la somministrazione del farmaco attraverso la BBB e forniscono un carico utile di farmaci citotossici alle cellule tumorali11.
Nonostante tutti i vantaggi degli aptameri, lo sviluppo di farmaci in questo campo non ha ancora prodotto un farmaco antitumorale commercializzato e di successo. Una ragione di ciò potrebbe essere la mancanza di metodi standard e riproducibili che potrebbero essere seguiti a livello globale dai ricercatori del settore. In questo articolo, viene dimostrato un protocollo passo-passo di un aptamero che si lega a una proteina nativa espressa sulla superficie cellulare. Questo protocollo è un passo prerequisito nella valutazione preclinica degli aptameri antitumorali. Il test viene eseguito per mostrare la selettività e la specificità dell'aptamero purificato raccolto da SELEX o una sequenza di aptameri pubblicata per la conferma della selettività e della specificità. Questo test basato sulla citometria a flusso è un test rapido, affidabile e sensibile che mostra accuratamente la selettività e la specificità dell'aptamero, dove l'aptamero viene testato contro le proteine sulla superficie cellulare12,13,14. Questo metodo è dimostrato utilizzando il legame di un aptamero specifico per EpCAM mostrato in questo articolo15. EpCAM, come glicoproteina transmembrana, svolge ruoli nella segnalazione, progressione, migrazione e metastasi delle cellule tumorali16,17. Per mostrare la selettività e la specificità di questo aptamero, sono state utilizzate cellule tumorali EpCAM-positive e -negative. L'aptamero specifico EpCAM precedentemente sviluppato, TEPP (5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′), e un aptamero di controllo negativo, TENN (5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3), sono stati utilizzati rispettivamente come aptameri leganti e non leganti EpCAM10. L'estremità 3' di TEPP e TENN è stata etichettata con un fluoroforo TYE665.
TEPP è un aptamero bifunzionale che ha come target EpCAM da un'estremità e TfR dall'altra. Ciò ha reso TEPP un candidato adatto per la somministrazione di farmaci ai tumori cerebrali EpCAM + . Utilizzando la sua estremità specifica per TfR, TEPP attraversa la barriera emato-encefalica e, utilizzando l'estremità specifica di EpCAM, trova il tumore e consegna il suo carico (ad esempio, farmaci citotossici) al tumore. TENN ha una lunghezza e una struttura 2D simili a TEPP, ma non ha affinità per l'EpCAM o il TfR, e quindi è un aptamero di controllo negativo adatto. Utilizzando TEPP e TENN, testare il legame di un aptamero alla proteina bersaglio usando la citometria a flusso è mostrato in questo articolo. Questo protocollo si applica allo sviluppo di aptameri cellulo-specifici. È applicabile anche a ulteriori analisi complementari e di conferma delle sequenze aptameri disponibili in letteratura. Il protocollo può essere utilizzato anche da coloro che sono nuovi nel campo degli aptameri che stanno cercando di utilizzare un aptamero precedentemente pubblicato per i loro scopi di ricerca e sviluppo (R & S). In questo articolo vengono studiate due sequenze di aptameri disponibili in letteratura.
Protocollo
NOTA: Prima di iniziare l'esperimento, indossare dispositivi di protezione individuale, tra cui camici da laboratorio, guanti e occhiali protettivi. Vedere la tabella dei materiali per informazioni dettagliate su materiali, reagenti, apparecchiature e software utilizzati in questo protocollo.
1. Tamponi necessari per il test
- Preparare i tamponi necessari per questo esperimento - il tampone SELEX necessario per il ripiegamento dell'aptamero, il tampone bloccante (BB), il tampone di lavaggio (WB) e il tampone di legame (BiB) (tabella 1) - il giorno dell'esperimento e tenerli sul ghiaccio o a 4 °C.
NOTA: Ogni aptamero richiede una condizione di piegatura unica. Ciò include il tampone SELEX e le condizioni di temperatura di piegatura. Si deve prestare attenzione a replicare completamente i metodi dell'articolo originale che descrive l'aptamero10. In questo esperimento, tutti i tamponi sono preparati nella soluzione salina tamponata fosfato di Dulbecco (DPBS). Il volume tampone richiesto in ogni esperimento dipende dal numero di linee cellulari, dal numero di repliche e dal numero di concentrazioni di aptameri testate.
| Ingredienti | Volume richiesto | ||
| Articolo | Concentrazione | ||
| Tampone SELEX | MgCl2 | 5 mM | 50 μL per campione + 10% di errore di pipettaggio |
| Blocco del buffer | MgCl2 | 5 mM | 500 μL per linea cellulare |
| BSA a | 1 mg/ml | ||
| tRNA b | 0,1 mg/ml | ||
| FBS c | 10% (v/v) | ||
| Tampone di lavaggio | MgCl2 | 5 mM | 1 mL per il primo lavaggio + 100 μL per campione di prova + 10% di errore di pipettaggio |
| Buffer di associazione | MgCl2 | 5 mM | 50 μL per campione + 10% di errore di pipettaggio |
| BSA | 2 mg/ml | ||
| Trna | 0,2 mg/ml | ||
| FBS | 20% (v/v) | ||
Tabella 1: Buffer necessari per il test di legame. un Albumina sierica bovina, bTransfer Acido Ribonucleico, cSiero Bovino Fetale.
2. Preparazione di aptameri
NOTA: Gli aptameri utilizzati nel test sono etichettati con una molecola reporter di fluorescenza e pertanto è necessario prestare attenzione per proteggerli dalla luce.
- Prima dell'esperimento, preparare uno stock di 100 μM (stock A) di aptameri di prova e controllo utilizzando acqua ultrapura priva di pirogeni e RNasi (Figura 1).
NOTA: Per una conservazione a lungo termine, la scorta A deve essere conservata in congelatore a -20 °C. - Preparare lo stock B come concentrazione operativa di aptameri diluendo il materiale A utilizzando il tampone SELEX (Tabella 1). Per seguire questo protocollo, diluire lo stock A in uno stock da 1.000 nM per preparare lo stock B (Figura 1).
- Per preparare l'aptamero alla formazione della struttura tridimensionale (3D), in un tubo da 250 μL, diluire il brodo B con tampone SELEX per preparare il volume e la concentrazione richiesti dell'aptamero per il folding.
NOTA: Gli aptameri piegati saranno esposti a un volume uguale di cellule. Pertanto, la concentrazione dell'aptamero impostata per il ripiegamento dovrebbe essere 2 volte più concentrata della concentrazione finale desiderata. Utilizzare l'equazione (1) per calcolare i volumi e le concentrazioni richiesti. Ricordarsi di considerare un volume aggiuntivo del 10% per l'errore di pipettaggio.
Scorta di concentrazione A ×Scorta di volume A = scorta di concentrazione B × scorta di volumeB (1)
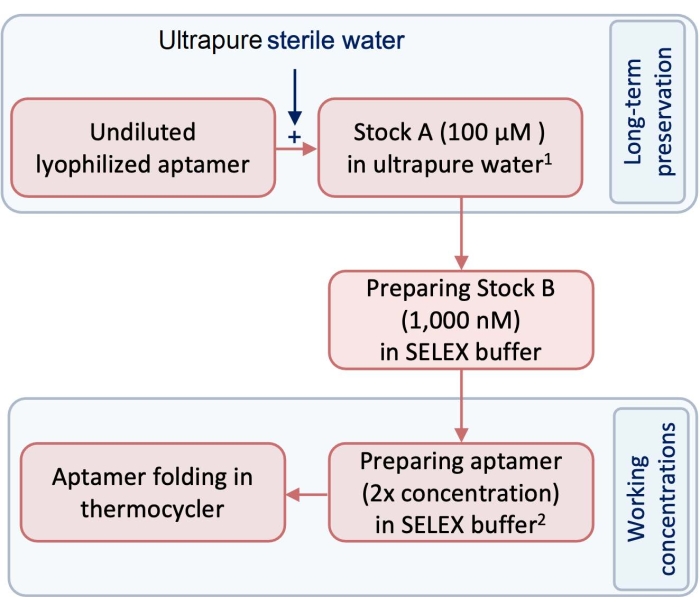
Figura 1: Un diagramma che mostra le fasi della preparazione degli aptameri. 1 Lo stock 1viene conservato a -20 °C per la conservazione a lungo termine. numero arabo Le concentrazioni di lavoro sono preparate nel tampone SELEX e non vengono immagazzinate. Fare clic qui per visualizzare una versione ingrandita di questa figura.
3. Mantenimento delle cellule tumorali
NOTA: Prima di iniziare lo studio, assicurarsi che le cellule siano al loro numero di passaggio precoce, mostrino le loro caratteristiche morfologiche tipiche e siano prive di micoplasma. Per testare la selettività e la specificità dell'aptamero, sono idealmente necessarie linee cellulari che siano espressivi alti, moderati e bassi/negativi della proteina di interesse.
- Seminare le cellule in un matraccio di coltura T75, utilizzando condizioni di coltura appropriate. Coltivarli in un incubatore umidificato al 5% di CO2 , a 37 °C.
NOTA: In questo studio, è stato utilizzato il Modified Eagle Medium (DMEM) di Dulbecco integrato con siero bovino fetale al 10% (terreno completo). - Quando le cellule raggiungono ~ 80% di confluenza, passarle in un nuovo matraccio contenente terreno completo fresco.
NOTA: A seconda della proteina di interesse e della linea cellulare, la confluenza dell'80% potrebbe fornire una popolazione cellulare adatta per il test di legame. Per le linee cellulari in questo esperimento, MDA-MB-231 e HEK 293T, è adatta una confluenza dell'80%. In questa fase, procedere alla sezione 4, il test di legame. Controlla sempre l'espressione della proteina di interesse, utilizzando mAbs specifici per quella proteina.
4. Saggio vincolante
NOTA: la Figura 2 riassume i passaggi necessari nel test di legame nelle celle aderenti.
- In un armadio di biosicurezza di classe II, raccogliere le cellule di ciascun matraccio in provette come segue:
- Raccogliere e scartare il supporto nel pallone, aggiungere 2 ml di PBS, distribuirlo sulle cellule, quindi raccogliere e scartare il PBS. Ripetere questo passaggio altre due volte per rimuovere tutte le tracce di supporto che potrebbero inattivare la tripsina. Aggiungere 1 mL di 0,25% di tripsina/EDTA a ciascun matraccio e incubare per 5-10 minuti a 37 oC. Visualizzare il distacco delle cellule al microscopio.
- Aggiungere 1 mL di mezzo completo alle celle e pipettare le celle su e giù per creare una sospensione a cella singola. Pipettare le cellule in un tubo appropriato e centrifugare a 200 × g per 5 minuti.
NOTA: Per le celle non aderenti, raccogliere le cellule in una provetta, centrifugare (200 × g, 5 min) e procedere al punto 4.1.3. - Eliminare il surnatante e risospendere le cellule in 1 ml di terreno fresco. Contare le cellule usando la colorazione blu tripano, diluendo un certo volume di sospensione cellulare con blu tripano. Distribuire ~ 15 μL della miscela tra un emocitometro e un vetro di copertura. Contare le celle come descritto in precedenza18, usando l'equazione (2):
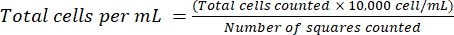 (2)
(2)
NOTA: Utilizzare il volume minimo possibile di sospensione cellulare e prendere nota del fattore di diluizione. Ad esempio, mescolando volumi uguali di sospensione cellulare e 0,04% di blu di tripano si ottiene un fattore di diluizione di 2. Garantire un'elevata vitalità (cellule vive/cellule totali × 100) di ~ 90% per la maggior parte delle linee cellulari aderenti prima di procedere. Le cellule morte assorbono in modo non specifico gli aptameri e alterano i risultati19. È possibile utilizzare altre tecniche di conteggio delle cellule, come l'utilizzo di un contatore di celle. - Raccogliere il numero richiesto di celle, assicurandosi di avere 10 × 104 cellule per campione di prova. Considerare un volume aggiuntivo del 10% per l'errore di pipettaggio.
NOTA: è importante mantenere sempre lo stesso numero di cellule tra esperimenti e repliche. - Incubare le cellule a 37 °C per 2 ore per consentire la stabilizzazione della proteina di interesse sulla membrana cellulare dopo distacco enzimatico.
NOTA: Questo periodo di incubazione potrebbe differire a seconda della proteina di interesse.
- Durante questa incubazione di 2 ore:
- Impostare la temperatura della centrifuga su 4 °C. Lasciare il tRNA e le scorte di aptamero a temperatura ambiente o sul ghiaccio per scongelare. Per proteggere la molecola reporter di fluorescenza, proteggere i tubi aptameri dalla luce.
NOTA: Il ruolo del tRNA è quello di bloccare i siti di legame degli acidi nucleici. - Preparare il tampone SELEX, BB, WB e BiB (vedere paragrafo 1), tenendoli tutti su ghiaccio o a 4 °C. Impostare la termociclatrice su un ciclo vuoto. Posizionare una piastra nera a 96 pozzetti e tubi di citometria a flusso sul ghiaccio.
NOTA: l'impostazione del termociclatore su un ciclo vuoto prepara il sistema di raffreddamento e riscaldamento e aiuta a generare risultati più riproducibili.
- Impostare la temperatura della centrifuga su 4 °C. Lasciare il tRNA e le scorte di aptamero a temperatura ambiente o sul ghiaccio per scongelare. Per proteggere la molecola reporter di fluorescenza, proteggere i tubi aptameri dalla luce.
- Dopo l'incubazione di 2 ore, centrifugare le cellule a 500 × g per 5 minuti. Scartare il surnatante e risospendere le cellule in 500 μL di BB. Incubare le cellule a 4 °C per 30 minuti con miscelazione intermittente.
- Durante questa incubazione di 30 minuti, eseguire il piegamento dell'aptamero come segue:
- Portare le 2 concentrazioni di aptameri (vedere paragrafo 2), quindi mescolare e incubare gli aptameri nella macchina termociclatore, secondo le condizioni di piegatura richieste. Per questo aptamero EpCAM, utilizzare le seguenti condizioni di piegatura di 95 °C, 5 min, seguite da 22 °C, 10 min, e 37 °C, 15 min.
NOTA: Includere sempre un controllo negativo (cioè tampone SELEX senza aptameri).
- Portare le 2 concentrazioni di aptameri (vedere paragrafo 2), quindi mescolare e incubare gli aptameri nella macchina termociclatore, secondo le condizioni di piegatura richieste. Per questo aptamero EpCAM, utilizzare le seguenti condizioni di piegatura di 95 °C, 5 min, seguite da 22 °C, 10 min, e 37 °C, 15 min.
- Dopo l'incubazione di 30 minuti, centrifugare le cellule (500 × g, 5 min, 4 °C), rimuovere il surnatante, aggiungere 1 mL di WB e centrifugare nuovamente le cellule (500 × g, 5 min, 4 °C). Rimuovere il surnatante e risospendere le cellule in un volume adeguato del BiB.
- Pipettare 50 μL delle celle risospese in ciascun pozzetto di una lastra nera ghiacciata a 96 pozzetti. Mantenere le cellule sul ghiaccio per inibire l'internalizzazione della proteina di interesse.
- Pipettare 50 μL degli aptameri su un volume di celle di 50 μL, mescolare e incubare al buio a 4 °C per 30 minuti. Centrifugare la piastra a 500 × g, 5 min, 4 °C e rimuovere con attenzione il surnatante.
- Risospendere con cautela il pellet in WB e centrifugare a 500 × g per 5 minuti. Ripetere la fase di lavaggio (4.7) 2x e risospendere in 100 μL di WB per l'analisi citometrica a flusso.
NOTA: Vedere la Figura 3 per un diagramma delle interazioni tra aptameri e cellule.

Figura 2: Un diagramma che illustra le fasi dell'esecuzione di un test di legame aptamero-proteina. Abbreviazioni: SELEX = Evoluzione sistematica dei ligandi mediante arricchimento esponenziale; BB = buffer di blocco; WB = tampone di lavaggio; BiB = buffer di associazione. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 3: Un diagramma che mostra i diversi tipi di cellule e aptameri necessari per eseguire il test di legame dell'aptamero. Abbreviazione: EpCAM = molecola di adesione cellulare epiteliale. Questa figura è stata creata utilizzando Biorender.com. Fare clic qui per visualizzare una versione ingrandita di questa figura.
5. Citometria a flusso e analisi dei dati
NOTA: Prima di accendere il citometro a flusso, assicurarsi che non vi siano "bolle" nelle unità filtranti a membrana per la soluzione di arresto, la soluzione detergente e il liquido della guaina (0,9% NaCl). Bolle "sanguinare" se ci sono bolle nelle capsule. Assicurati che il contenitore dei rifiuti sia vuoto e che i contenitori di liquido della guaina, acqua e candeggina all'1% in acqua ultrapura siano pieni.
- Accendere il citometro a flusso e quindi il computer.
NOTA: I dettagli di funzionamento del citometro a flusso spiegati qui sono specifici per la macchina e il software dimostrati nel video (vedere la tabella dei materiali). Altri software richiederebbero una formazione adeguata per l'uso. - Aprire il software di analisi della citometria a flusso, accedere al programma e, nella scheda Citometro , eseguire Fluidics Start-up.
- Per creare un nuovo esperimento, nella scheda dell'esperimento, fai clic su Nuova cartella e assegna al nome appropriato/alla cartella/esperimento.
- Fai clic su Nuova cartella per evidenziarla, quindi nella scheda Esperimento , fai di nuovo clic su Nuovo esperimento e assegna un nome appropriato all'esperimento.
- Per aggiungere il primo campione/campione, nella scheda dell'esperimento, fare clic su Nuovo campione e assegnare a questo campione un nome appropriato (nome della linea cellulare/campione di controllo/campione dell'esperimento).
- Per aggiungere un campione di provetta, evidenziare il campione (gruppo) e nella scheda dell'esperimento , fare clic su Nuovo tubo. Aggiungere il numero appropriato di tubi e il nome.
- Per preparare i grafici necessari, nella scheda Foglio di lavoro aprire un nuovo foglio di lavoro . Una volta visualizzata la nuova finestra del foglio di lavoro, aprire quanto segue utilizzando la schermata del foglio di lavoro (passare il mouse sul logo / immagini per trovare i nomi):
- Preparare un grafico a punti macchia di dispersione diretta (FSC) rispetto a dispersione laterale (SCC) per selezionare la popolazione di interesse. Definire la prima porta identificando e selezionando la popolazione di interesse (P1) in un grafico di densità di dispersione diretta e laterale. Escludere i detriti, che costituiscono la popolazione con il segnale di dispersione in avanti più basso.
NOTA: il parametro FSC rileva celle o eventi in base alle loro dimensioni e il SCC li discrimina in base alla loro granularità20. - Preparare un grafico a punti macchia dell'area FSC (FSC-A) rispetto all'altezza FSC (FSC-H) per selezionare la popolazione a cella singola. Definire il secondo gate escludendo le popolazioni di cellule doppiette, poiché le cellule doppiette influenzano considerevolmente i risultati e le conclusioni. Escludere doppietti utilizzando grafici di densità FSC-H rispetto a FSC-A, in cui celle della stessa dimensione mostrano un'area e un'altezza simili. Quindi, i singoletti vengono raggruppati diagonalmente e separati dai doppietti.
NOTA: FSC è approssimativamente proporzionale alla dimensione della cella. Gli impulsi di tensione sono definiti come FSC-H, l'intensità del segnale, FSC-width che riflette la dimensione della cella e la durata del segnale, e FSC-A, che è H × W. I doppietti hanno un doppio valore di larghezza e area; pertanto, il gating per i singoletti si basa sul rilevamento delle sproporzioni tra H, W e A causate da doppietti. - Preparare un istogramma del numero di eventi rispetto al fluoroforo di interesse.
- Preparare un grafico a punti macchia di dispersione diretta (FSC) rispetto a dispersione laterale (SCC) per selezionare la popolazione di interesse. Definire la prima porta identificando e selezionando la popolazione di interesse (P1) in un grafico di densità di dispersione diretta e laterale. Escludere i detriti, che costituiscono la popolazione con il segnale di dispersione in avanti più basso.
- Prima di iniziare la citometria a flusso, assicurarsi che il dashboard di acquisizione per il controllo dell'acquisizione del campione, dell'ispettore e del citometro per regolare i parametri di tensione, nonché il foglio di lavoro con tutti i grafici siano aperti.
NOTA: Per eseguire l'analisi sono necessari almeno 100 μL di sospensione di 10 × 104 cellule in un tubo di citometria a flusso. Soprattutto in caso di vitalità inferiori, la colorazione con ioduro di propidio può essere eseguita per selezionare la popolazione cellulare vitale21,22. - Per eseguire il primo campione, sul lato sinistro dello schermo assicurarsi che la freccia che punta al tubo sia verde. Se questa freccia non è verde, fare clic sulla freccia per renderla verde.
- Utilizzando una pipetta, trasferire ciascun campione dalla piastra nera a 96 pozzetti a un tubo di citometria a flusso. Eseguire il campione di controllo non trattato e non colorato a bassa velocità.
- Nel dashboard di acquisizione, scegliere un numero appropriato di eventi da registrare (30.000), impostare la portata su bassa e fare clic su Acquisisci dati.
- Regolare la tensione per i parametri FSC e SCC. Assicurarsi che la popolazione cellulare sia centralizzata all'interno del dot plot e che nessuna cella tocchi uno degli assi del grafico per evitare di perdere le celle di interesse.
- Aumentare la velocità di acquisizione a media o alta per analizzare i campioni più velocemente ma non superare più di 200 eventi/s. Quindi, fare clic su Registra dati.
- Eseguire il gating per P1 (Figura 4A) e la popolazione a cella singola (Figura 4B). Costruire l'istogramma degli eventi rispetto al fluorocromo utilizzato e selezionare P1 in base ai dati (alloficocianina (APC) in questo caso) (Figura 4C).
- Dopo aver regolato la tensione, il gating e registrato i dati, estrarre il campione e fare clic su Next Tube.
- Inserire il campione successivo e ripetere i dati di registrazione per tutti i campioni di controllo e di prova (Figura 3).
- Una volta raccolti tutti i dati, lavare il citometro a flusso facendo funzionare tre tubi di candeggina al 50%, risciacquo FACS e acqua ultrapura, ciascuno per 5 minuti ad alta portata.
- Quindi, dal menu a discesa Citometro , fare clic su Fluidics Shut Down.
- Prima di chiudere il software e spegnere la macchina e il computer, esportare i risultati come file .fcs su un'unità USB per trasferirli e analizzarli, come segue:
- Nel software di analisi, premere il pulsante NUOVO per creare un nuovo documento e una finestra per gestire l'analisi. Trascinare i file di esempio nella nuova finestra.
- Fare doppio clic per aprire il campione non macchiato. Scegli la popolazione P1, fai doppio clic sulla popolazione P1 per creare un grafico FSC-H rispetto a FSC-A e controlla la popolazione a cella singola.
- Fare doppio clic sulle singole celle gate per creare un istogramma di eventi rispetto al fluorocromo utilizzato.
- Nella finestra originale, selezionare P1 e celle singole e trascinarle in Tutti i campioni in modo che tutti i campioni contengano ora lo stesso gating.
- Fare clic sul pulsante Editor layout per aprire la finestra Layout . Trascinare due campioni (controllo e test) l'uno sull'altro per creare un istogramma sovrapposto.
Risultati
Un aspetto importante della scoperta e dello sviluppo di nuovi farmaci è assicurare la selettività e la specificità del farmaco candidato. Ciò significa che il farmaco candidato dovrebbe essere in grado di discriminare tra cellule diverse e influenzare solo la popolazione cellulare di interesse (selettività). La selettività viene studiata utilizzando linee cellulari che differiscono in termini di espressione della proteina di interesse. In questo studio, le linee cellulari MDA-MB-231 e HEK 293T sono state scelte co...
Discussione
La sfida principale con lo sviluppo di nuovi aptameri è la mancanza di linee guida standard che si applicano alle diverse fasi di questo processo. McKeague et al. hanno recentemente dimostrato alcune delle sfide associate, che portano a presentazioni poco chiare dei dati nelle pubblicazioni e alla mancata replica della ricerca. Hanno proposto linee guida fondamentali necessarie per la caratterizzazione degli aptameri19. Un test di legame dell'aptamero è un passo fondamentale nello screening e / ...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Gli autori riconoscono il finanziamento SEED dell'Institute for Mental and Physical Health and Clinical Translation (IMPACT), il programma "Alfred Deakin Postdoctoral Research Fellowship" presso la Deakin University e la "Australian Government Research Training Program Scholarship".
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes with attached lid | Sigma-Aldrich | T6649 | |
| 15 mL CellStar blue screw cap, conical bottom tube | Greiner Bio One | 188271 | |
| 5 mL serological pipettes | Greiner Bio One | 606180 | |
| BD FACSCanto II Flow Becton Dickinson Cytometer | Becton Dickinson | N/A | |
| BD FACSDiva V9.0 | BD Biosciences | N/A | |
| Bovine Serum Albumin (BSA), Lyophilized powder | Sigma-AldrichTM | A7906-50G | |
| Bright-line Hemocytometer | Sigma-Aldrich | Z359629 | |
| Dulbecco’s Modified Eagle Medium (DMEM) High Glucose Media Powder | Life Technologies | 12100046 | |
| Dulbecco’s Phosphate- Buffered Saline (DPBS) | Life Technologies | 21300025 | |
| FlowJo, LLC 10.8.1 | BD Biosciences | N/A | |
| Foetal Bovine Serum (FBS) | Bovogen | SFBS-F | |
| HEK293T | American Type Culture Collection | ACS-4500 | |
| Heracell 150i CO2 Incubator | Thermo Fisher Scientific | N/A | |
| Heraeus Megafuge 16R Centrifuge | Thermo Fisher Scientific | N/A | |
| Magnesium Chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| MDA-MB-231 | American Type Culture Collection | CRM-HTB-26 | |
| Microplate, PS, 96 well, F-bottom (Chimney well), Black | Greiner Bio One | 655076 | |
| MiniAmp Thermal Cycler | Thermo Fisher Scientific | A37834 | |
| Phosphate-Buffered Saline (PBS) tablets | Life Technologies | 18912014 | |
| Pyrogen- and RNase-free ultrapure water | Milli-Q | ||
| T75 Cell Culture flask | Cellstar | 658170 | |
| TENN | Integrated DNA Technologies | N/A | 5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3 |
| TEPP | Integrated DNA Technologies | N/A | 5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′ |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8508-5X1ML | |
| Trypan Blue Solution | Life Technologies | 15250061 | |
| Trypsin-EDTA | Gibco | 15400054 |
Riferimenti
- Cancer. World Health Organization Available from: https://www.who.int/news-room/fact-sheets/detail/cancer#:~:text=Cancer%20is%20a%20leading%20cause.and%20rectum%20and%20prostate%20cancers (2022)
- Liu, J. K. H. The history of monoclonal antibody development - Progress, remaining challenges and future innovations. Annals of Medicine and Surgery. 3 (4), 113-116 (2014).
- Nakhjavani, M., Shigdar, S. Future of PD-1/PD-L1 axis modulation for the treatment of triple-negative breast cancer. Pharmacological Research. 175, 106019 (2022).
- Bukari, B., Samarasinghe, R. M., Noibanchong, J., Shigdar, S. L. Non-invasive delivery of therapeutics into the brain: the potential of aptamers for targeted delivery. Biomedicines. 8 (5), 120 (2020).
- Wu, X., Chen, J., Wu, M., Zhao, J. X. Aptamers: active targeting ligands for cancer diagnosis and therapy. Theranostics. 5 (4), 322 (2015).
- Feng, X., et al. The aptamer functionalized nanocomposite used for prostate cancer diagnosis and therapy. Journal of Nanomaterials. 2022, (2022).
- Huang, J., et al. Advances in aptamer-based biomarker discovery. Frontiers in Cell and Developmental Biology. 9, 571 (2021).
- Ashrafuzzaman, M. Aptamers as both drugs and drug-carriers. BioMed Research International. 2014, (2014).
- Nakhjavani, M., Samarasinghe, R. M., Shigdar, S. Triple-negative breast cancer brain metastasis: an update on druggable targets, current clinical trials, and future treatment options. Drug Discovery Today. , (2022).
- Macdonald, J., et al. Development of a bifunctional aptamer targeting the transferrin receptor and epithelial cell adhesion molecule (EpCAM) for the treatment of brain cancer metastases. ACS Chemical Neuroscience. 8 (4), 777-784 (2017).
- Macdonald, J., et al. Bifunctional aptamer-doxorubicin conjugate crosses the blood-brain barrier and selectively delivers its payload to EpCAM-positive tumor cells. Nucleic Acid Therapeutics. 30 (2), 117-128 (2020).
- Shigdar, S., Agnello, L., Fedele, M., Camorani, S., Cerchia, L. Profiling cancer cells by cell-SELEX: use of aptamers for discovery of actionable biomarkers and therapeutic applications thereof. Pharmaceutics. 14 (1), 28 (2021).
- Rahimizadeh, K., et al. Development of cell-specific aptamers: recent advances and insight into the selection procedures. Molecules. 22 (12), 2070 (2017).
- Chen, M., et al. Development of cell-SELEX technology and its application in cancer diagnosis and therapy. International Journal of Molecular Sciences. 17 (12), 2079 (2016).
- Shigdar, S., et al. The use of sensitive chemical antibodies for diagnosis: detection of low levels of EpCAM in breast cancer. PLoS One. 8 (2), 57613 (2013).
- Ni, J., et al. Role of the EpCAM (CD326) in prostate cancer metastasis and progression. Cancer and Metastasis Reviews. 31 (3), 779-791 (2012).
- Ni, J., et al. Epithelial cell adhesion molecule (EpCAM) is associated with prostate cancer metastasis and chemo/radioresistance via the PI3K/Akt/mTOR signaling pathway. The International Journal of Biochemistry & Cell Biology. 45 (12), 2736-2748 (2013).
- McKeague, M., Kruse, P. F., Patterson, M. K., et al. . Tissue Culture. , 395-397 (1973).
- McKeague, M., et al. The minimum aptamer publication standards (MAPS guidelines) for de novo aptamer selection. Aptamers. 6, 10-18 (2022).
- Schoofs, G., Van Hout, A., D'huys, T., Schols, D., Van Loy, T. A flow cytometry-based assay to identify compounds that disrupt binding of fluorescently-labeled CXC Chemokine ligand 12 to CXC Chemokine receptor 4. Journal of Visualized Experiments. (133), e57271 (2018).
- McKinnon, K. M. Flow cytometry: an overview. Current Protocols in Immunology. 120 (1), 1-11 (2018).
- Kamiloglu, S., Sari, G., Ozdal, T., Capanoglu, E. Guidelines for cell viability assays. Food Frontiers. 1 (3), 332-349 (2020).
- Ruscito, A., DeRosa, M. C. Small-molecule binding aptamers: Selection strategies, characterization, and applications. Frontiers in Chemistry. 4, 14 (2016).
- McKeague, M., et al. Comprehensive analytical comparison of strategies used for small molecule aptamer evaluation. Analytical Chemistry. 87 (17), 8608-8612 (2015).
- Henri, J., Bayat, N., Macdonald, J., Shigdar, S. A guide to using nucleic acid aptamers in cell based assays. Aptamers. 23, (2019).
- Mao, H., et al. The mechanism and regularity of quenching the effect of bases on fluorophores: the base-quenched probe method. Analyst. 143 (14), 3292-3301 (2018).
- McKeague, M., et al. Analysis of in vitro aptamer selection parameters. Journal of Molecular Evolution. 81 (5), 150-161 (2015).
- Chen, B., et al. Targeting negative surface charges of cancer cells by multifunctional nanoprobes. Theranostics. 6 (11), 1887 (2016).
- Shigdar, S., et al. RNA aptamers targeting cancer stem cell marker CD133. Cancer Letters. 330 (1), 84-95 (2013).
- Amraee, M., Oloomi, M., Yavari, A., Bouzari, S. DNA aptamer identification and characterization for E. coli O157 detection using cell based SELEX method. Analytical Biochemistry. 536, 36-44 (2017).
- Yu, X. -. X., et al. Selection and characterization of a novel DNA aptamer, Apt-07S specific to hepatocellular carcinoma cells. Drug Design, Development and Therapy. 14, 1535 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon