Method Article
Uso di metodi minimamente invasivi per valutare l'utilizzo del carburante e i ritmi circadiani negli anziani
In questo articolo
Riepilogo
Viene presentato un protocollo innovativo e minimamente invasivo per valutare l'impatto sinergico dell'utilizzo del carburante e dei ritmi circadiani sugli individui che invecchiano, utilizzando cellule mononucleate del sangue periferico.
Abstract
L'invecchiamento è associato a molteplici cambiamenti fisiologici che contribuiscono sinergicamente e indipendentemente alla disabilità fisica e al rischio di malattie croniche. Sebbene l'eziologia della disabilità fisica legata all'età sia complessa e multifattoriale, il declino della funzione mitocondriale sembra coincidere con la progressione del declino funzionale in molti anziani. Il motivo per cui c'è una diminuzione della funzione mitocondriale con l'invecchiamento rimane sfuggente, ma la scienza emergente indica che sia il metabolismo del carburante che i ritmi circadiani possono influenzare la funzione mitocondriale.
Studi recenti hanno stabilito che i ritmi circadiani vengono disturbati con l'invecchiamento e che i ritmi circadiani interrotti hanno conseguenze patologiche che influiscono sulla funzione mitocondriale e si sovrappongono a molte malattie croniche associate all'età. Gli attuali metodi quantitativi per la valutazione diretta della funzione mitocondriale sono invasivi e in genere richiedono una biopsia muscolare, che può porre difficoltà con il reclutamento dei partecipanti e l'aderenza allo studio, dati i livelli percepiti di potenziale dolore e rischio. Pertanto, è stato adattato un protocollo innovativo e relativamente non invasivo per valutare i cambiamenti nella funzione mitocondriale a livello cellulare e nei modelli circadiani negli anziani. In particolare, un analizzatore di flusso metabolico in tempo reale viene utilizzato per valutare la funzione bioenergetica mitocondriale dei globuli bianchi in disponibilità differenziale di substrato.
Vengono inoltre analizzate l'espressione dei geni dell'orologio circadiano nei globuli bianchi per correlare in modo incrociato con la bioenergetica mitocondriale e gli esiti del ritmo circadiano. Si ritiene che questi approcci metodologici innovativi aiuteranno i futuri studi clinici fornendo metodi minimamente invasivi per lo studio della preferenza del substrato mitocondriale e dei ritmi circadiani negli anziani.
Introduzione
I progressi dell'ultimo secolo hanno portato a un aumento dell'aspettativa di vita e della popolazione di adulti che invecchiano. Guardando al futuro, si prevede che la percentuale di adulti di età pari o superiore a 65 anni aumenterà del 5% dal 2020 al 2050 negli Stati Uniti1. Questo aumento dell'aspettativa di vita non implica un aumento della durata della salute, il periodo della vita associato al funzionamento indipendente. La realtà è che l'invecchiamento è accompagnato da innumerevoli cambiamenti biologici che influenzano il metabolismo cellulare e la fisiologia, producendo un graduale declino del funzionamento cognitivo e fisico 2,3. Poiché l'aspettativa di vita umana continua ad aumentare, c'è una maggiore necessità di preservare la capacità funzionale e l'indipendenza con l'età di4 anni.
È noto da tempo che il declino della funzione fisica e dell'indipendenza con l'età è multifattoriale, sebbene sia spesso associato all'insorgenza di malattie croniche e di eventi stimolanti acuti5. Al contrario, è stato dimostrato che questi cali delle prestazioni fisiche e delle caratteristiche muscolari sono associati allo sviluppo di disabilità con l'età senza una chiara connessione con una singola malattia6. Con le difficoltà nel conoscere l'esatta eziologia della malattia cronica e della disabilità fisica, si è pensato che le menomazioni della funzione mitocondriale coincidessero con l'insorgenza e la progressione della malattia cronica e la perdita della funzione fisica negli adulti che invecchiano 7,8.
I mitocondri forniscono la maggior parte dell'adenosina trifosfato (ATP), necessaria per molti processi cellulari9. I tessuti altamente ossidativi si affidano ai mitocondri per un'adeguata produzione di energia; con l'invecchiamento, la capacità ossidativa e il declino della sintesi mitocondriale di ATP. Questo declino è dovuto in parte al danno ossidativo al DNA mitocondriale (mtDNA), che si traduce in un accumulo incrementale di mutazioni e delezioni del mtDNA10. L'accumulo di mutazioni e delezioni del mtDNA provoca una diminuzione della formazione di proteine funzionali della catena di trasporto degli elettroni, causando così una ridotta capacità delle cellule di produrre ATP. Il declino della funzione mitocondriale associato all'età è più evidente nei tessuti altamente ossidativi, come il cuore e il muscolo scheletrico11. Gli studi hanno dimostrato che i mitocondri del muscolo gastrocnemio nei campioni di ratto più anziani mostrano una riduzione di circa il 50% della produzione e del contenuto di ATP rispetto ai campioni più giovani12. Inoltre, è stato dimostrato che la capacità di produzione mitocondriale di ATP nel muscolo scheletrico umano diminuisce di circa l'8% per decennio di vita13. Questi risultati suggeriscono che il declino della funzione mitocondriale legato all'età può contribuire a ridurre la produzione di energia negli organismi.
Si ritiene che un regolatore chiave dell'attività mitocondriale sia il coattivatore-1 (PGC-1α)14 del recettore attivato dal proliferatore dei perossisomi γ (PPARγ)14. Il deterioramento dell'attività di PGC-1α o un declino della sua abbondanza porta a una ridotta attività ossidativa mitocondriale e, di conseguenza, a una ridotta produzione di energia. Inoltre, un declino della qualità mitocondriale può influenzare la qualità del muscolo scheletrico e successivamente portare allo sviluppo o all'esacerbazione di sarcopenia, dinapenia e declino della capacità funzionale15,16. L'evidenza del declino concomitante correlato all'età della funzione mitocondriale e della qualità del muscolo scheletrico suggerisce una connessione tra la compromissione mitocondriale e la patogenesi del declino funzionale17. Recentemente, questo è stato confermato negli anziani funzionali che vivono in comunità, dimostrando che le riduzioni del metabolismo dei mitocondri del muscolo scheletrico predicono il declino della mobilità in questa popolazione18. Sebbene l'esatto meccanismo che porta al declino mitocondriale con l'età non sia chiaro, recenti evidenze hanno evidenziato un'interazione reciproca tra l'orologio circadiano e la funzione mitocondriale, con conseguenze per l'utilizzo del carburante mitocondriale e la biogenesi19.
Utilizzo del carburante
La funzione mitocondriale sembra essere influenzata dal metabolismo del carburante e dal tipo di carburante utilizzato a livello cellulare nel tessuto muscolare scheletrico11. Durante i periodi di esaurimento del combustibile, in particolare l'esaurimento dei carboidrati nell'uomo, la preferenza per il carburante per la produzione di energia (mitocondriale) cambia. A bassi livelli di glucosio, la preferenza per il carburante si sposta dal glucosio agli acidi grassi e ai corpi chetonici di derivazione acida. Questo interruttore metabolico è caratterizzato dalla sovraregolazione del metabolismo lipidico negli adipociti, seguita da un aumento del rilascio di chetoni nel sangue4. Il passaggio dall'utilizzo del carburante dal glucosio ai chetoni con una dieta chetogenica sembra avere un effetto benefico sulla produzione di specie reattive dell'ossigeno mitocondriale, sulla difesa antiossidante, sulla sintesi di ATP e sulla biogenesi20.
Il passaggio metabolico dal metabolismo dei carboidrati a quello dei lipidi si verifica in periodi di bassa disponibilità di nutrienti ambientali e quando le riserve di glicogeno sono state esaurite. Quando questo passaggio viene avviato, i trigliceridi immagazzinati vengono scomposti in glicerolo, un substrato per la gluconeogenesi, e acidi grassi liberi, che vengono trasportati al fegato per essere ossidati tramite β-ossidazione in acetil coenzima A (acetil CoA). I corpi chetonici sono sintetizzati, principalmente nel fegato, mediante una condensazione in due fasi di tre molecole di acetil CoA in β-idrossi-β-metilglutaril-CoA, che vengono poi ulteriormente trasformati in corpi chetonici, tra cui acetoacetato e 3-βeta idrossibutirrato21. Questi corpi chetonici sono distribuiti ai tessuti di tutto il corpo, con il consumo più elevato che si verifica nel cuore, nel cervello e nel muscolo scheletrico21. Con l'invecchiamento, l'ossidazione degli acidi grassi mitocondriali viene compromessa, influenzando così l'interruttore metabolico22. È stato proposto che le menomazioni nell'utilizzo del carburante mitocondriale portino a un'ulteriore disfunzione mitocondriale, che a sua volta contribuisce alla malattia legata all'età e al declino funzionale23.
I cambiamenti nel consumo di ossigeno mitocondriale delle cellule mononucleate del sangue periferico (PBMC) sono stati studiati per valutare i modelli associati alla disfunzione e alla vascolarizzazione. Hartman et al. hanno condotto uno studio che mirava a determinare la correlazione tra il consumo di ossigeno e la dilatazione mediata in modo diverso, che è stato trovato per suggerire un legame tra la disfunzione mitocondriale e la disfunzione delle cellule muscolari lisce vascolari24. Per quanto riguarda altri organi, le PBMC sono state correlate con un funzionamento cognitivo e cerebrale più elevato, come determinato dalla respirometria25. Pertanto, la bioenergetica e la capacità respiratoria della PBMC possono fungere da potenziali biomarcatori per valutare la capacità funzionale di organi o tessuti in tutto il corpo.
Ritmo circadiano
Un altro fattore importante che influenza la salute mitocondriale è il ritmo circadiano. I ritmi circadiani sono oscillazioni di ~24 ore nel comportamento e nella fisiologia che si verificano in assenza di segnali ambientali26. Questi ritmi funzionano in modo predittivo per supportare l'omeostasi del sistema e dei tessuti. Il meccanismo che sta alla base dei ritmi circadiani è un ciclo di feedback trascrizione-traduzione chiamato orologio circadiano27. È stato dimostrato negli ultimi 15 anni che il meccanismo dell'orologio circadiano esiste praticamente in tutte le cellule del corpo28. Oltre a tenere il tempo, il meccanismo dell'orologio molecolare contribuisce anche a un programma giornaliero di espressione genica, indicato come output dell'orologio circadiano29. I geni di uscita dell'orologio sono unici per ogni tipo di tessuto e sono funzionalmente associati a percorsi importanti per il metabolismo cellulare, l'autofagia, la riparazione e l'omeostasi. Prove recenti hanno dimostrato che la salute mitocondriale dipende dalla funzione dell'orologio circadiano e influenza la funzione mitocondriale, tra cui la biogenesi mitocondriale, l'utilizzo del carburante e la mitofagia30.
Prove emergenti sia in studi preclinici che clinici hanno dimostrato che durante l'invecchiamento ci sono disturbi nei ritmi circadiani31. Questi includono interruzioni dei normali cicli di sonno e veglia, una diminuzione dell'ampiezza dei ritmi della temperatura corporea interna e una capacità ritardata di adattarsi ai cambiamenti nella fase31. Uno studio, ad esempio, ha sfidato il sistema circadiano di topi adulti e anziani (20+ mesi) spostando il programma di illuminazione di 6 ore. Si è scoperto che i vecchi topi impiegavano più tempo per riportare i loro modelli di attività al nuovo programma di luce32. Coerentemente con i cambiamenti nel comportamento circadiano, l'analisi degli orologi tissutali ha rilevato che sia gli orologi dei tessuti centrali che quelli periferici erano compromessi nella coorte di invecchiamento.
Più recentemente, diversi gruppi hanno eseguito analisi trascrittomiche dell'orologio circadiano e dell'output dell'orologio in diversi tessuti all'età di33 anni. I risultati di questi studi evidenziano che esiste una riprogrammazione su larga scala dell'output dell'orologio circadiano con l'età. Ciò significa che, anche se l'orologio centrale mantiene una funzione di temporizzazione, i geni bersaglio per l'espressione quotidiana sono in gran parte diversi. Ad esempio, due studi hanno raccolto biopsie muscolari da soggetti umani ogni 4 ore per 24 ore, i risultati hanno determinato che il picco e la depressione dell'espressione genica dell'orologio sono invertiti tra i roditori notturni e gli esseri umani diurni 34,35,36. Ciò indica che quando l'espressione del gene dell'orologio viene confrontata basandosi esclusivamente sulla fase attiva rispetto alla fase di riposo (e non alla luce rispetto al buio), i modelli di espressione del gene dell'orologio nei muscoli sono praticamente gli stessi tra le specie. Si propone che questo cambiamento associato all'età nell'output dell'orologio provochi menomazioni nella regolazione dei percorsi che includono i segni distintivi noti dell'invecchiamento, come la funzione mitocondriale, il danno e la riparazione del DNA e l'autofagia37.
Logica dello studio
La connessione tra la funzione mitocondriale e il declino della funzione fisica è ben stabilita. Tuttavia, la causa alla base della disfunzione mitocondriale rimane oggetto di dibattito. Ricerche recenti suggeriscono che l'utilizzo del carburante cellulare e i ritmi circadiani possono svolgere un ruolo in questo processo. I metodi tradizionali per valutare la funzione mitocondriale, come la misurazione del consumo di ossigeno mitocondriale in un campione di biopsia muscolare, sono spesso percepiti come dolorosi e invasivi, il che può scoraggiare la partecipazione, in particolare nelle popolazioni con bassa massa muscolare, come gli adulti fragili e sarcopenici38.
Date queste limitazioni, c'è bisogno di un metodo meno invasivo per valutare i cambiamenti nell'utilizzo del carburante cellulare e nel ritmo circadiano negli anziani. Questo studio mira a valutare un nuovo protocollo minimamente invasivo che può essere utilizzato per valutare il metabolismo del carburante e il ritmo circadiano in questa popolazione. I risultati di questo studio contribuiranno a una migliore comprensione dei cambiamenti legati all'età e della risposta agli interventi medici o comportamentali, fungendo da modello per futuri studi in questo campo.
Protocollo
Le procedure che coinvolgono partecipanti umani sono state approvate dal comitato etico della ricerca (Florida Ethics Policy 1.0104) e dall'Institutional Review Board dell'Università della Florida.
1. Funzione mitocondriale
- Isolamento di cellule mononucleate del sangue periferico (PBMC)
- Raccogliere le PBMC utilizzando speciali provette per la raccolta del sangue da 8 ml (16 mm x 125 mm; contenenti 0,1 M di citrato di sodio anticoagulante o di sodio eparina) (vedere la Tabella dei materiali).
NOTA: Le provette per la raccolta del sangue contengono un mezzo di separazione del sangue composto da un gel di poliestere tissotropico e da un mezzo di gradiente (vedere la Tabella dei materiali) per migliorare la separazione dei globuli bianchi. - Processare le provette di preparazione cellulare da 8 mL entro 2 ore dalla raccolta per ottenere risultati validi.
- Miscelare le provette per il prelievo del sangue dopo averle riempite di sangue (mediante leggera inversione o su un miscelatore) e mantenerle a temperatura ambiente (RT) per un massimo di 2 ore.
- Centrifugare le provette per la raccolta del sangue in un rotore oscillante a 2.000 × g per 15 minuti (o equivalente a 30.000 g/min; non superare i 2.000 × g) a RT.
NOTA: Queste provette sono più alte di una normale provetta da centrifuga da 15 mL; Pertanto, è necessario prestare particolare attenzione all'utilizzo del rotore corretto. - Aspirare e gettare in un contenitore per rifiuti a rischio biologico ~80% dello strato di plasma e raccogliere lo strato cellulare dalla provetta per la raccolta del sangue (utilizzando una pipetta di trasferimento) in una provetta da centrifuga da 15 mL.
NOTA: Quando si utilizza una provetta da 15 mL, aggiungere 7 mL di strato cellulare e riempire fino a 14 mL con soluzione fisiologica tamponata con fosfato sterile (PBS). Il rapporto tra cellule e PBS dovrebbe essere 1:1. Se necessario, utilizzare più tubi. Mescolare delicatamente invertendo. - Centrifugare in un rotore oscillante a 900 × g per 5 min (o equivalente a 4.500 g-min) a RT.
- Senza disturbare il pellet della cella, aspirare la soluzione PBS e gettarla in un contenitore per rifiuti a rischio biologico.
- Risospendere il pellet cellulare in 1 mL di terreno basale (BM; vedere Tabella dei materiali) triturando delicatamente. Aggiungere il BM fino a un totale di 10 ml e mescolare delicatamente capovolgendo. Eseguire un conteggio delle celle.
- Centrifugare la sospensione della cella in un rotore oscillante a 900 × g per 5 minuti a RT.
NOTA: Quando le cellule vengono utilizzate lo stesso giorno per la valutazione bioenergetica, procedere al passaggio 1.1.10; Se le celle sono congelate per la conservazione, procedere al passaggio 1.1.13. - Calcolare il volume del terreno di prova (AM; vedere la tabella dei materiali) per la risospensione del pellet cellulare successivo per ottenere la concentrazione cellulare desiderata.
NOTA: Ad esempio, per una densità di semina di 150.000 cellule/pozzetto in 50 μL, la concentrazione desiderata della sospensione cellulare è di 3 milioni di cellule/mL. - Senza disturbare il pellet cellulare, aspirare il surnatante e gettarlo in un contenitore per rifiuti a rischio biologico.
- Risospendere delicatamente il pellet cellulare in 1 mL o meno di AM. Aggiungere AM al volume totale calcolato per la concentrazione di celle desiderata. Le cellule sono ora pronte per essere seminate in una piastra di coltura cellulare rivestita (vedi Tabella dei materiali).
NOTA: I seguenti passaggi servono per il congelamento delle celle per la conservazione (1.1.13-1.1.16) e non si applicano alla procedura in giornata. - Per il congelamento delle cellule, aspirare il surnatante senza disturbare il pellet cellulare e gettarlo in un contenitore per rifiuti a rischio biologico.
- Risospendere delicatamente le cellule in un terreno di congelamento (vedere la Tabella dei materiali) alla concentrazione desiderata (5-10 milioni di cellule/mL). Aliquotare i volumi desiderati in crioviali etichettati.
- Congelare lentamente la sospensione cellulare utilizzando un contenitore di congelamento (vedere la tabella dei materiali) per almeno 4 ore o durante la notte a -80 °C.
- Dopo 4 ore, o la mattina successiva, trasferire i crioviali in un deposito di azoto liquido, dove dovrebbero risiedere nella fase vapore.
- Il giorno prima della misurazione, rivestire il fondo dei pozzetti di una micropiastra per colture cellulari (vedere Tabella dei materiali) con adesivo cellulare (vedere Tabella dei materiali). Preparare l'adesivo secondo le istruzioni del produttore, con una concentrazione consigliata di 22,4 μg/μL, diluendo il volume appropriato di soluzione madre adesiva in bicarbonato di sodio 0,1 M (pH 8,0). Applicare 25 μl di adesivo su ciascun pozzetto della micropiastra per colture cellulari.
- Dopo 20 minuti, aspirare il liquido rimanente da ciascun pozzetto e lavare i pozzetti due volte con 200 μL di acqua sterile e purificata. Lasciare asciugare la piastra nella cabina di biosicurezza (per ~2 ore).
- Avvolgere il piatto in un foglio di alluminio o metterlo in un contenitore secondario chiuso, e conservarlo in frigorifero a 4 °C.
- Raccogliere le PBMC utilizzando speciali provette per la raccolta del sangue da 8 ml (16 mm x 125 mm; contenenti 0,1 M di citrato di sodio anticoagulante o di sodio eparina) (vedere la Tabella dei materiali).
- Valutazione bioenergetica di PBMC con analizzatore di flusso metabolico a tempo di latenza
NOTA: Utilizzando un analizzatore di flusso e il lettore multimodale (vedere Tabella dei materiali), sono stati determinati una densità di semina ottimale di 150.000 PBMC per pozzetto, una concentrazione finale di cianuro di carbonile-4 (trifluorometossi) fenilidrazone (FCCP) di 2 μM e una concentrazione finale di Hoechst 33342 di 4 μM (con un tempo di incubazione di almeno 20 minuti dopo l'iniezione).- Per idratare le cartucce del sensore con l'hydrobooster, rimuovere la cartuccia del sensore con la piastra di utilità dalla scatola e posizionare la cartuccia "in alto" sul banco. Inserire 200 μL di calibrante (vedere la Tabella dei materiali) in ciascun pozzetto della piastra di servizio e posizionare saldamente l'hydrobooster sulla piastra di servizio, quindi la cartuccia del sensore. Ispezionare e rimuovere eventuali bolle d'aria intrappolate e collocare in un'incubatrice non CO2 a 37 °C per una notte.
- Accendere l'analizzatore di flusso e successivamente aprire il software di analisi del saggio (vedere la tabella dei materiali). Impostare la temperatura a 37 °C. Lasciare lo strumento acceso per una notte per consentire la stabilizzazione della temperatura impostata.
NOTA: I seguenti passaggi di programmazione (1.2.4-1.2.10) possono essere eseguiti il giorno prima o il giorno dell'esperimento - Per programmare il file del software di analisi del saggio (vedere la Tabella dei materiali), aprire il modello per il test di stress ossidativo del substrato. Vai su Definizioni di gruppo, apri la scheda Strategie di iniezione , modifica il test da sforzo Inibitore + Sub Ox in Etomoxir + Test da sforzo Sub Ox e aggiungi altre due strategie di iniezione: UK5099 + Test da sforzo Sub Ox e BTPES + Test da sforzo Sub Ox. All'interno di ciascuna strategia di iniezione, all'interno della finestra Condizione di iniezione , fare clic su A per la porta A e inserire la concentrazione finale del rispettivo inibitore (Etomoxir: 4 μM; UK5099: 2 μM; BTPES: 3 μM).
- Fare clic su Porte B, C e D e inserire le rispettive concentrazioni finali (porta B: concentrazione di oligomicina [1,50 μM], porta C: FCCP [2,0 μM]; porta D: Rotenone + Antimicina A [0,5 μM]). Aggiungere Hoechst 33342, 4 μM alla finestra dei composti per la porta D.
- Apri la scheda Pretrattamenti e scegli i pretrattamenti, se applicabile. Ad esempio: Controllo, Sperimentale o Consuetudinario (ad esempio, Linea di base e Follow-up). Se necessario, aggiungi le descrizioni nella finestra sottostante.
- Aprire la scheda Terreni di prova e dal menu a discesa Terreni scegliere Terreno RPMI, pH 7,4, inserire il numero di lotto, il personale che prepara i terreni, il tempo di preparazione e gli integratori aggiunti ai terreni (10 mM di glucosio, 2 mM di glutammina, 1 mM di piruvato; vedere la Tabella dei materiali).
- Aprire la scheda Tipo di cella e immettere l'ID soggetto PBMC come nome, PBMC come tipo di cella, 150000 come densità di semina, ID soggetto come origine, il personale che ha preparato le cellule, la data di preparazione e il giorno di disgelo delle cellule, se applicabile.
- Fare clic su Genera gruppi, andare su Mappa piastre e assegnare i pozzetti della piastra a 96 pozzetti ai rispettivi gruppi. Mantenere i pozzetti A1, A12, H1 e H12 assegnati come pozzetti di fondo.
- Andare su Protocollo, selezionare la casella Equilibra (impostazione predefinita) e immettere le seguenti informazioni: Linea di base: cinque cicli di misurazione: 3 min mix, 0 min wait e 3 min measure; Fluido o inibitore (porta A): sei cicli di misurazione: 3 min di miscelazione, 0 min di attesa e 3 min di misurazione; Oligomicina (porta B): tre cicli di misurazione: 3 min di miscelazione, 0 min di attesa e 3 min di misurazione; FCCP (Porta C): tre cicli di misurazione: 3 min di miscelazione, 0 min di attesa e 3 min di misurazione; Rotenone + Antimicina A + Hoechst 33342 (Porta D): tre cicli di misurazione: 3 min di miscelazione, 0 min di attesa e 3 min di misurazione.
- Il giorno della valutazione bioenergetica, prelevare la micropiastra di coltura cellulare rivestita dal frigorifero e lasciarla riscaldare a RT nella cappa di biosicurezza.
- Preparare il terreno di coltura (AM) mescolando 97 mL di BM preriscaldato e integratori: 1 mL di piruvato (concentrazione finale: 1 mM), 1 mL di glucosio (concentrazione finale 10 mM) e 1 mL di glutammina (concentrazione finale: 2 mM). Mettere l'AM a 37 °C in un'incubatrice non contenente CO2 fino al momento dell'uso.
- Per seminare le cellule, aggiungere 50 μL di sospensione cellulare (da 1.1.12) contenente 150.000 PBMC a ciascun pozzetto, ad eccezione di A1, H1, A12 e H12 (assegnati come pozzetti di fondo).
CRITICO: La densità di semina ottimale è stata determinata in precedenza. - Coprire la piastra cellulare con il coperchio e lasciare riposare 60 minuti di cellule a RT nella cabina di biosicurezza per facilitare la distribuzione uniforme delle cellule.
CRITICO: Osservare le cellule al microscopio per garantire la distribuzione omogenea delle cellule desiderata in tutto il pozzetto. - Per l'imaging cellulare, accendere il lettore multimodale (vedere la Tabella dei materiali) alcune ore prima della prima incubazione cellulare; attendere il completamento del protocollo di avvio del lettore prima di aprire il software di imaging (vedere la Tabella dei materiali). Aprire il software di imaging e impostare la temperatura di preriscaldamento a 37 °C.
- Per preparare i reagenti del saggio, seguire le istruzioni del kit di analisi per ricostituire i reagenti, con lievi modifiche come indicato nella Tabella 1.
CRITICO: Le concentrazioni finali ottimali di FCCP e Hoechst33342 sono state determinate in precedenza. - Dopo il riposo cellulare di 60 minuti, ispezionare i pozzetti con un microscopio per colture cellulari e prendere nota di eventuali anomalie visive, come le aggregazioni cellulari.
- centrifugare la piastra di coltura cellulare a RT in una centrifuga con rotore oscillante per micropiastre a 60 × g per 1 minuto, con accelerazione minima e rottura minima; Girare il piatto e centrifugare nuovamente a 40 × g per 1 min. Impostare l'accelerazione e la frenata al minimo. Incubare le cellule per 25-30 minuti a 37 °C in un incubatore non CO2 .
NOTA: Quando si esegue l'imaging cellulare, la piastra cellulare viene invece incubata nel lettore multimodale preriscaldato (vedere la Tabella dei materiali) e viene scattata un'immagine in campo chiaro di ciascun pozzetto durante questo periodo di incubazione (prima incubazione cellulare). - Scansionare il codice a barre della piastra per colture cellulari con lo scanner di codici a barre (vedere Tabella dei materiali), selezionare Avvia scansione in campo chiaro, posizionare la piastra per colture cellulari sul vassoio della piastra e avviare la retrazione del vassoio. Selezionare i pozzetti contenenti celle per la scansione e avviare la scansione.
- Durante la prima incubazione cellulare, caricare le porte della cartuccia del sensore con i reagenti, come indicato nella Tabella 1: porta (A): 20 μL di AM (controlli), etomoxir (inibitore del trasporto degli acidi grassi a catena lunga nei mitocondri), UK 5099 (acido 2-ciano-3-(1-fenil-1H-indol-3-il)-2-propenoico; inibitore del vettore mitocondriale del piruvato), o BPTES (Bis-2-(5-fenilacetamido-1, 3,4-tiadiazolo-2-il)etilsolfuro; inibitore della conversione della glutammina in glutammato); porta (B): 22 μL di oligomicina (inibitore dell'ATP sintasi); porta (C): 25 μl di FCCP (disaccoppiatore di cianuro di carbonile-4 (trifluorometossi) fenilidrazone); porta (D): 27 μL di rotenone/antimicina A/Hoechst 33342 (inibitori dei complessi mitocondriali I e III, rispettivamente, e colorante nucleare). Rimettere la cartuccia del sensore nell'incubatore a 37 °C senza CO2 per altri 5 minuti.
NOTA: Seguire le istruzioni del produttore per il caricamento delle porte. - Estrarre la piastra per colture cellulari dall'incubatore o dal lettore di piastre dopo la prima incubazione cellulare e dopo il completamento dell'imaging cellulare (se applicabile) e aggiungere AM calda a ciascun pozzetto fino a un volume finale di 180 μl per pozzetto. Porre la piastra cellulare a 37 °C in un incubatore non CO2 per altri 15-25 minuti (seconda incubazione cellulare).
- Avviare il test metabolico avviando la calibrazione del sensore durante la seconda incubazione cellulare. Fare clic su Esegui test quando il test è pronto per essere avviato con la calibrazione della cartuccia del sensore. Quando richiesto, trasferire la cartuccia del sensore dall'incubatore non CO2 a 37 °C al vassoio dell'analizzatore di flusso e avviare la calibrazione. Seguire le istruzioni del software di analisi del saggio e sostituire la piastra di utilità per la piastra cellulare al termine della calibrazione.
NOTA: La calibrazione dura circa 20 minuti. - Dopo il test metabolico, seguire le istruzioni del software e rimuovere la cartuccia del sensore e la piastra cellulare dallo strumento. Rimuovere la cartuccia del sensore dalla piastra della cella e coprire la piastra della cella con il coperchio.
- Dopo aver determinato il tempo di incubazione del colorante Hoechst negli esperimenti preliminari, scansionare il codice a barre della piastra e seguire le istruzioni del software per acquisire un'immagine fluorescente di ciascun pozzetto contenente cellule con il lettore multimodale.
NOTA: Il software di imaging e analisi dei saggi è interfacciato, consentendo l'importazione dei dati della conta cellulare nel file di dati dell'analisi del saggio per la normalizzazione dei dati alla conta cellulare.
2. Espressione genica dell'orologio circadiano
NOTA: L'espressione dei geni orologio dei partecipanti dalle PBMC sarà esaminata isolando l'RNA utilizzando il kit di sangue RNA (vedi Tabella dei materiali).
- Aspirare 3 mL di sangue del partecipante direttamente in una provetta di RNA (vedere la Tabella dei materiali) con 6 mL di reagenti stabilizzanti. Vortice per 10 s per miscelare completamente.
NOTA: Se i reagenti stabilizzanti non vengono accuratamente miscelati con il campione di sangue del partecipante, aumenta il rischio di errore nello studio. - Trasferire 1,0 mL del campione di sangue stabilizzato in una provetta da 15 mL, con 5 mL di tampone per lisi eritrocitaria. Incubare con ghiaccio per 10-15 minuti. Agitare il tubo 2 volte durante l'incubazione. Centrifugare la provetta a 4 °C a 400 × g per 10 min.
- Identificare il pellet e versare con cura il surnatante contenente globuli rossi lisati. Aggiungere 2 mL di tampone per la lisi degli eritrociti (vedere la Tabella dei materiali), risospendere le cellule, agitare brevemente e centrifugare a 400 × g per 10 minuti a 4 °C.
- Versare il surnatante e lasciare il tubo capovolto su carta assorbente per 1-2 minuti. Tamponare il liquido intorno al bordo del tubo con carta pulita prima del passaggio successivo.
- Aggiungere con cautela 600 μl di tampone di lisi dell'RNA (vedere la tabella dei materiali). Seguendo il protocollo del produttore, aggiungere β-mercaptoetanolo e risospendere il pellet.
- Pipettare il lisato direttamente in un omogeneizzatore monouso di lisato cellulare (vedere la Tabella dei materiali), una colonna di centrifuga posta in una provetta di raccolta da 2 mL e centrifugare per 2 minuti alla massima velocità per omogeneizzare. Eliminare la colonna di centrifuga monouso dell'omogeneizzatore di lisato cellulare e conservare il lisato omogeneizzato.
- Aggiungere 1 volume (600 μl) di etanolo al 70% al lisato omogeneizzato e miscelare mediante pipettaggio. Pipettare con cautela il campione, compreso l'eventuale precipitato formatosi, in una nuova colonna di centrifuga in una provetta di raccolta da 2 mL, ma non inumidire il bordo. Centrifugare per 15 s a >8.000 × g.
- Trasferire la colonna di centrifuga (vedere la Tabella dei materiali) in una nuova provetta di raccolta da 2 mL. Applicare 700 μl di tampone di lavaggio a rigore (vedere la tabella dei materiali) sulla colonna di centrifuga e centrifugare per 15 s a >8.000 × g per il lavaggio. Scartare il flusso.
- Aprire con cautela la colonna di centrifuga e aggiungere 500 μl di tampone di lavaggio delicato (vedere la tabella dei materiali). Chiudere il tappo e centrifugare alla massima velocità (20.000 × g) per 3 min.
- Trasferire la colonna di centrifuga in una provetta da microcentrifuga da 1,5 ml e pipettare 30-50 μl di acqua priva di RNasi direttamente sulla membrana di silice. Centrifugare per 1 minuto a >8.000 × g per eluire l'RNA. Ripeti ancora una volta.
NOTA: L'RNA può essere conservato a -80 °C. - Eseguire il trattamento con DNasi su una colonna utilizzando il set DNasi privo di RNasi (vedere la Tabella dei materiali), secondo il protocollo del produttore.
- Generare cDNA utilizzando 500 ng di RNA totale e un sistema di reazione a catena della polimerasi (PCR) in tempo reale (vedi Tabella dei materiali), secondo il protocollo del produttore. Diluire tutti i campioni di cDNA 1:25 in acqua priva di RNasi e utilizzare 4 mL per eseguire la PCR quantitativa a trascrizione inversa (qRT-PCR).
- Utilizzare un metodo di aggiunta di primer (vedere la Tabella dei materiali) per completare la qRT-PCR con 10 mM di ciascuno dei primer mostrati nella Tabella 2. Completare la qRT-PCR utilizzando un sistema in tempo reale (vedere la Tabella dei materiali).
- Normalizzare i livelli di mRNA dei geni scelti utilizzando i livelli di mRNA di Rpl26 . Utilizzare il metodo 2-DDCt per calcolare la quantificazione relativa.
- Per determinare se l'espressione di un dato mRNA mostrava un'oscillazione circadiana, utilizzare un test basato sulla verosimiglianza LR_rhythmicity39 (utilizzando p≤ 0,01) per rilevare la ritmicità circadiana in una condizione sperimentale40.
3. Piano di analisi dei dati
NOTA: Verrà utilizzato un inventario medico per classificare i partecipanti in base all'uso dei farmaci43.
- Utilizzare un modello lineare a effetti misti, in cui età, sesso, peso, altezza, pressione sanguigna, frequenza cardiaca e altri fattori di rischio dovrebbero essere inclusi come covariate.
- Dovrebbe essere incluso un termine a effetto casuale per spiegare la correlazione dei dati all'interno del soggetto.
- Per l'adattamento del modello, è necessario implementare una selezione delle variabili a ritroso con il principio gerarchico.
- Dal modello adattato, la misura cambia dal punto temporale 1 al punto temporale 2, date tutte le covariate nel modello adattato.
Risultati
Il protocollo proposto include dati preliminari che servono come convalida per la metodologia. Il protocollo incorpora un analizzatore di flusso metabolico in tempo reale per esaminare la funzione mitocondriale e l'utilizzo del carburante cellulare, e l'estrazione dell'RNA e la qRT-PCR per analizzare i geni del ritmo circadiano (ad esempio, BMAL1, CLOCK, Nfil2, Nr1d1, Dbp, Cry1, Per2).
Il tasso di consumo di ossigeno (OCR) di PBMC umane isolate da cinque partecipanti di controllo, 10 giorni dopo un'analisi iniziale, è presentato nella Figura 1. I dati vengono utilizzati per confrontare i valori pre e post e mostrano i valori medi per la respirazione basale, la risposta acuta, la respirazione massima e la capacità di riserva dopo l'iniezione di un controllore, etomoxir, UK5099 e BPTES. In particolare, la Figura 1C mostra una significativa risposta acuta negativa dopo l'iniezione di etomoxir, ma non sono stati osservati effetti significativi per la respirazione basale, la respirazione massima o la capacità di riserva.
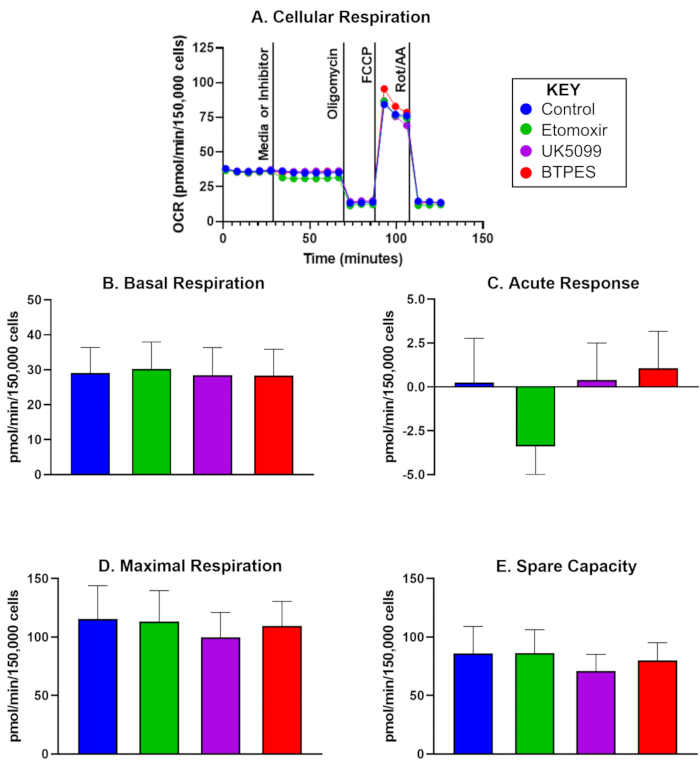
Figura 1: Consumo di ossigeno di cellule mononucleate isolate del sangue periferico umano (PBMC). (A) Tasso di consumo di ossigeno in tempo reale (OCR; pmol/(min∙150.000 cellule) di PBMC isolate da un soggetto di controllo, misurato con un analizzatore di flusso e valutato con il saggio di ossidazione del substrato. Le cellule sono state seminate a una densità di 150.000 cellule/pozzetto. La prima iniezione è stata di tipo mediano (controllo) o inibitore (etomoxir, UK5099 o BPTES; vedere il testo per i dettagli) ed è avvenuta dopo aver misurato la frequenza respiratoria cellulare basale. La risposta acuta alla limitazione del substrato mitocondriale è stata determinata come la differenza dell'OCR basale prima e dopo l'iniezione dell'inibitore. L'oligomicina, l'inibitore dell'ATP sintasi, inibisce la respirazione accoppiata alla produzione di ATP e produce la respirazione con perdita di protoni. FCCP, l'uncoupler, induce la respirazione massima e non accoppiata; Il rotenone e l'antimicina A (inibitori del complesso I e III, rispettivamente) inibiscono tutta la respirazione tranne quella non mitocondriale (vedi testo per i dettagli). (B-E) Quantificazione della respirazione cellulare (n = 5; i dati sono rappresentati come media ± DS). (B) OCR basale prima dell'iniezione dell'inibitore, (C) risposta acuta all'inibitore (variazione dell'OCR rispetto alla velocità basale prima dell'iniezione dell'inibitore), (D) OCR massimale e (E) capacità di riserva (differenza tra OCR massimale e OCR basale dopo la prima iniezione). La risposta acuta (C) all'iniezione di etomoxir potrebbe suggerire una maggiore dipendenza dell'OCR dall'acido grasso come substrato energetico in condizioni basali rispetto agli altri gruppi di substrati, senza un effetto notevole sull'OCR durante un'elevata richiesta di energia (D). Clicca qui per visualizzare una versione più grande di questa figura.
| Composto | AM (μL) aggiunto al composto | Scorte (μM) | stock (μL) per il materiale da lavoro | AM (μL) per materiale da lavoro | Massa lavorativa (μM) | Scorte di lavoro (μL) [porta] | Conc. finale (μM) |
| Etomoxir | 700 | 160 | 500 | 1500 | 40 | 20 [A] | 4 |
| UK5099 | 700 | 80 | 500 | 1500 | 20 | 20 [A] | 2 |
| BPTES | 700 | 120 | 500 | 1500 | 30 | 20 [A] | 3 |
| oligo | 420 | 150 | 300 | 2700 | 15 | 22 [B] | 1.5 |
| FCCP | 720 | 100 | 600 | 2400 | 20 | 25 [C] | 2 |
| Rot/AA/H | 540 | 50 | 300 | 2700 | 5 | 27 [D] | 0.5 |
Tabella 1: Preparazione dei reagenti per il test di ossidazione del substrato e concentrazioni delle soluzioni stock, di lavoro e finali. Tutti i reagenti fanno parte del test da sforzo mitoscopico cellulare o dei kit per il test da stress ossidativo del substrato. Abbreviazioni: oligo = oligomicina; FCCP = disaccoppiatore di cianuro di carbonile-4 (trifluorometossi) fenilidrazone; Rot/AA/H = rotenone/antimicina A/Hoechst 33342. Etomoxir, UK5099, BTPES: inibitori rispettivamente dell'ossidazione degli acidi grassi, del glucosio e della glutammina.
| Bmal1 | Avanti – GCACGACGTTCTTTTTTCTGT |
| Inverso – GCAGAAGCTTTTTCGATCTGCTTTT | |
| Orologio | Avanti – CGTCTCAGACCCTTCCTCAAC |
| Inverso – GTAAATGCTGCCTGGGGA | |
| Piangere 1 | Avanti – ACTGCTATTGCCCTGTTGGT |
| Rovescio – GACAGGCAAATAACGCCTGA | |
| Per1 | Avanti – ATTCGGGTTACGAAGCTCCC |
| Inverso – GGCAGCCCTTTCATCCACAT | |
| Per2 | Attaccante – CATGTGCAGTGGAGCAGATTC |
| Inverso – GGGGTGGTAGCGGATTTCAT | |
| Rev-erb α | Avanti – ACAGATGTCAGCAGATGTCGC |
| Retro – CGACCAAACCGAACAGCATC |
Tabella 2: Primer genici dell'orologio circadiano.
Discussione
Il declino della funzione mitocondriale e la regolazione del ritmo circadiano con l'età sono sempre più visti come fattori che contribuiscono alle malattie legate all'età. L'alterazione dei ritmi circadiani attraverso modifiche dello stile di vita, come la dieta e l'attività fisica, rappresenta una potenziale strategia per promuovere un invecchiamento sano e ridurre il declino della mobilità associato all'invecchiamento. Tuttavia, gli attuali metodi per valutare direttamente la funzione mitocondriale sono invasivi e spesso richiedono una biopsia muscolare, che può porre sfide con il reclutamento e la ritenzione dei partecipanti a causa del dolore e dei rischi percepiti.
La valutazione dei marcatori di salute circadiana e metabolica attraverso metodi meno invasivi, come la raccolta del sangue, fornirebbe risultati preziosi per esplorare e testare bersagli terapeutici in studi futuri. Questi metodi minimamente invasivi hanno il potenziale per far progredire notevolmente il campo, fornendo nuove intuizioni sulla complessa interazione tra ritmo circadiano e salute metabolica e sul loro impatto sulla funzione. L'obiettivo di questo studio è valutare la relazione tra il metabolismo energetico cellulare e il ritmo circadiano. In particolare, l'analisi del flusso bioenergetico viene utilizzata per valutare la funzione mitocondriale in varie condizioni di disponibilità del substrato, insieme al monitoraggio dell'espressione genica di un gruppo di geni circadiani nei globuli bianchi dei partecipanti. Impiegando entrambi i bracci dell'analisi, bioenergetica ed espressione genica, è possibile ottenere una comprensione completa della relazione tra questi due processi fondamentali.
L'analisi statistica di questi dati di serie temporali da una prospettiva circadiana offre informazioni sulla forza, l'intervallo e la tempistica dei ritmi circadiani. In conclusione, l'integrazione dell'analisi dell'espressione genica, della bioenergetica cellulare e delle misure metaboliche a livello dell'organismo costituisce un approccio nuovo e innovativo che farà luce sull'interazione tra metabolismo energetico e ritmi circadiani nell'uomo.
In uno studio pilota, abbiamo rilevato una risposta acuta nell'OCR delle PBMC alla limitazione dell'utilizzo degli acidi grassi (dopo l'iniezione di etomoxir, un inibitore della carnitina palmitoil transferasi 1a). Questa scoperta suggerisce che nelle PBMC di questo particolare gruppo di partecipanti, potrebbe esserci una dipendenza dagli acidi grassi come substrato energetico durante la respirazione basale. Tuttavia, la respirazione massima non è stata influenzata, suggerendo che fonti di energia alternative, come il glucosio e la glutammina, possono compensare il ridotto utilizzo di acidi grassi durante l'elevata domanda di energia. Studi futuri dovrebbero indagare se a) la bioenergetica delle PBMC riflette l'energia di tutto il corpo e b) se interventi come l'alimentazione a tempo limitato potrebbero influenzare le preferenze del substrato energetico.
Ci sono diversi passaggi critici riguardanti l'analisi del flusso delle PBMC. In primo luogo, prima di valutare i campioni sperimentali, la densità di semina delle cellule (cellule per pozzetto) deve essere ottimizzata assicurandosi che vi sia una distribuzione uniforme continua delle cellule all'interno di ciascun pozzetto e su ciascuna piastra, la concentrazione finale di FCCP deve essere ottimizzata eseguendo test di concentrazione utilizzando le concentrazioni 0, 0,125, 0,25, 0,5, 1,0 e 2,0 μM, e, se applicabile, la colorazione Hoechst 33342 deve essere ottimizzata seguendo le istruzioni del produttore. In secondo luogo, la normalizzazione dei dati metabolici ai parametri cellulari è fondamentale per la comparabilità dei dati tra gli esperimenti. Nel presente protocollo, viene descritta la conta cellulare dopo la conclusione del test dell'analizzatore di flusso utilizzando cellule colorate con Hoechst 33342 e un dispositivo di imaging cellulare. Se non è disponibile un dispositivo appropriato, è possibile applicare metodi di normalizzazione alternativi, come il contenuto totale di proteine cellulari o DNA nucleare per pozzetto. C'è una modifica nota che può essere utilizzata all'interno del protocollo, rispetto a quelle che sono state proposte. In particolare, il protocollo può essere completato utilizzando un kit di test individuale per ciascuno dei tre inibitori, rispetto ai soli due kit qui proposti (vedi Tabella dei materiali).
L'uso delle PBMC come surrogato per studiare l'interazione tra metabolismo energetico e ritmi circadiani negli anziani è limitato dal presupposto che la loro risposta al trattamento possa riflettere accuratamente la risposta in altri tessuti e organi. Sebbene questo approccio sia nuovo e minimamente invasivo, è importante riconoscere che diversi tessuti e organi, come il cervello, il fegato e il muscolo scheletrico, possono reagire in modo diverso in varie condizioni. Uno studio preclinico ha dimostrato che l'espressione del gene dell'orologio era alterata nei topi alimentati e a digiuno, portando alla parziale sovraregolazione dei geni bersaglio di BMAL1 nel fegato e nel tessuto muscolare, ma alla sottoregolazione di altri41. Questi tessuti e organi periferici sono altamente rappresentativi dei processi metabolici e possono essere influenzati da segnali ambientali che influenzano i meccanismi di espressione genica dell'orologio42. Sono necessarie ulteriori ricerche per comprendere appieno la relazione tra i tessuti periferici, gli organi e l'orologio circadiano centrale.
Un'altra limitazione è che i partecipanti non sono esclusi dall'assunzione di alcuna prescrizione, il che potrebbe porre limitazioni all'analisi statistica. Per contrastare questa limitazione, nella ricerca futura, verrà utilizzato un inventario medico, che è stato convalidato nelle popolazioni di anziani che assumono farmaci43. I partecipanti saranno classificati in base ai farmaci raccomandati registrati nella sezione di revisione dei dati del protocollo. Ci sono un totale di tre categorie, in termini di farmaci che hanno dimostrato di 1) accelerare il declino funzionale, 2) rallentare il declino funzionale e 3) influenzare la funzione del muscolo scheletrico.
Infine, la capacità ossidativa mitocondriale del muscolo scheletrico umano mostra un ritmo giorno-notte, con un picco tra le 18:00 e le 23:00 e un calo tra le 08:00 e le 11:0044. Non è ancora chiaro se questo valga per la capacità ossidativa mitocondriale delle PBMC. Tuttavia, dati preliminari suggeriscono che le PBMC e il metabolismo mitocondriale sono correlati45. Dato che le informazioni sulle biopsie muscolari e sulle alterazioni nelle PBMC non sono così chiare, è necessario prestare attenzione quando si analizzano i risultati. Data questa limitazione, è importante tenere a mente queste informazioni durante la valutazione e lo sviluppo di un protocollo, in quanto possono fornire un contesto e informazioni preziose che potrebbero aiutare a garantire la validità e l'efficacia del protocollo.
Per quanto ne sappiamo, nessuno studio precedente ha valutato i modelli di utilizzo del carburante o i ritmi circadiani attraverso i metodi proposti in questo progetto. Il nostro obiettivo è esaminare la reattività dei marcatori dell'utilizzo del carburante mitocondriale e della salute circadiana ai cambiamenti. Questo studio presenta un metodo minimamente invasivo per misurare un biomarcatore altamente sensibile, che può servire come alternativa in futuri studi interventistici in cui la biopsia muscolare non è fattibile.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo studio è stato finanziato dall'Older American's Independence Center (NIH/NIA P30AG028740), con l'assistenza del Clinical and Translational Science Institute (NIH/NCRR UL1TR000064).
Materiali
| Name | Company | Catalog Number | Comments |
| Agilent Technologies Cell Imaging | Agilent Technologies | Cell image software | |
| Agilent/Seahorse Wave desktop program | Agilent Technologies | 5994-0039EN | Software used to analyse data from the celluar analyser and stress test assay |
| Agilent/Seahorse XFe96 Flux Analyzer | Agilent Technologies | S7800B | Real-time cellular flux analyzer; flux analyser |
| Bar Code Reader | Agilent Technologies | G2615-90007 | |
| Seahorse Wave Desktop Software | Agilent technologies | Data acquisition software; assay analysis; wave program | |
| Seahorse XF 1.0 M Glucose solution | Agilent Technologies | 103577-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 100 mM Pyruvate solution | Agilent Technologies | 103578-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 200 mM Glutamine solution | Agilent Technologies | 103579-100 | Supplement to basal medium to make assay medium |
| Seahorse XF Cell Mito Stress Test | Agilent Technologies | 103015-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF Mito Fuel Flex Test | Agilent Technologies | 103260-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF RPMI Medium | Agilent Technologies | 103576-100 | Basal medium for PBMCs |
| Seahorse XFe96 FluxPak mini | Agilent Technologies | 102601-100 | Sensor cartridges and cell culture microplates |
| Cytation 1 Cell Imaging Multi-Mode Reader | Agilent/BioTek | Multimode reader to image cells | |
| CPT Sodium Heparin Tube, 16 x 125 mm x 8.0 mL | Becton Dickinson | 362753 | Blood collection tubes for isolation of peripheral blood mononuclear cells |
| CellTak Cell and Tissue Adhesive | Corning | 354240 | Cell adherent to coat cell culture microplate |
| Phosphate Buffered Saline | Corning | 21-040-CV | Buffer to wash blood cells |
| Ficoll Paque Plus | Cytiva | GE17-1440-02 | Gradient medium |
| Lunar Prodigy DXA scanner | General Electric | EN 60601-2-7 5.1 | Whole body lean mass and fat/lean tissue mass ratio |
| Freezing container, Nalgene Mr. Frosty | MilliporeSigma | C1562 | Freezing container used to slow-freeze cell suspension |
| Buffer EL. | Qiagen | 79217 | Erythrocyte lysis buffer |
| Buffer RLT | Qiagen | 79216 | RNA lysis buffer |
| Buffer RPE | Qiagen | 1018013 | Mild washing buffer |
| Buffer RW1 | Qiagen | 1053394 | Stringent washing buffer |
| QIAamp DNA Micro Kit | Qiagen | 56304 | DNA preps: QIAamp MinElute Columns, Proteinase K, Carrier RNA, Buffers, Collection Tubes (2 ml) |
| QIAamp RNA Blood Mini Kit | Qiagen | 52304 | RNA blood kit; Used to isolate RNA |
| QIAshredder | Qiagen | 79656 | disposable cell-lysate homogenizers |
| RNase-Free DNase Set | Qiagen | 79254 | Used to perform DNA digest |
| 2-Mercaptoethanol (Reagent) | Thermo Fisher Scientific | MFCD00004890 | |
| 2-mL collection tubes, 100 count | Thermo Fisher Scientific | AM12480 | |
| Fast SYBR Green Master Mix | Thermo Fisher Scientific | 4385612 | Primers are added to this and used to carry out qRT-PCR |
| Microcentrifuge Tubes, 1.5 mL | Thermo Fisher Scientific | 69715 | Used to hold RNA purification filter during RNA purification |
| Narrow p1000 pipette tips | Thermo Fisher Scientific | 02-707-402 | |
| QuantStudio 3 Real-Time PCR System, MiniAmp Plus Thermal Cycler, and 96-Well Plates Package | Thermo Fisher Scientific | A40393 | |
| Tempus Blood RNA Tube | Thermo Fisher Scientific | 4342792 | RNA Tube |
| Tempus Spin RNA Isolation kit | Thermo Fisher Scientific | 4380204 | RNA extraction and isolation |
Riferimenti
- UN Department of Economic and Social Affairs. Population Division 2019, World Population Prospects. UN Department of Economic and Social Affairs. , (2019).
- Anton, S., Leeuwenburgh, C. Fasting or caloric restriction for healthy aging. Experimental Gerontology. 48 (10), 1003-1005 (2013).
- Dziechciaż, M., Filip, R. Biological psychological and social determinants of old age: Bio-psycho-social aspects of human aging. Annals of Agricultural and Environmental Medicine. 21 (4), 835-838 (2014).
- Anton, S. D., et al. Flipping the metabolic switch: understanding and applying the health benefits of fasting. Obesity. 26 (2), 254-268 (2018).
- Fried, L. P., Guralnik, J. M. Disability in older adults: evidence regarding significance, etiology, and risk. Journal of the American Geriatrics Society. 45 (1), 92-100 (1997).
- Manini, T. Development of physical disability in older adults. Current Aging Science. 4 (3), 184-191 (2011).
- Chung, H. Y., et al. Molecular inflammation: underpinnings of aging and age-related diseases. Ageing Research Reviews. 8 (1), 18-30 (2009).
- Sun, N., Youle, R. J., Finkel, T. The mitochondrial basis of aging. Molecular Cell. 61 (5), 654-666 (2016).
- Tarasov, A. I., Griffiths, E. J., Rutter, G. A. Regulation of ATP production by mitochondrial Ca2. Cell Calcium. 52 (1), 28-35 (2012).
- Chistiakov, D. A., Sobenin, I. A., Revin, V. V., Orekhov, A. N., Bobryshev, Y. V. Mitochondrial aging and age-related dysfunction of mitochondria. Biomed Research International. 2014, 238463 (2014).
- Boengler, K., Kosiol, M., Mayr, M., Schulz, R., Rohrbach, S. Mitochondria and ageing: role in heart, skeletal muscle and adipose tissue. Journal of Cachexia, Sarcopenia, and Muscle. 8 (3), 349-369 (2017).
- Drew, B., et al. Effects of aging and caloric restriction on mitochondrial energy production in gastrocnemius muscle and heart. American Journal of Physiology Regulatory, Integrative and Comparative Physiology. 284 (2), R474-R480 (2003).
- Short, K. R., et al. Decline in skeletal muscle mitochondrial function with aging in humans. Proceedings of the National Academy of Sciences. 102 (15), 5618-5623 (2005).
- Musci, R. V., Hamilton, K. L., Miller, B. F. Targeting mitochondrial function and proteostasis to mitigate dynapenia. European Journal of Applied Physiology. 118 (1), 1-9 (2018).
- Picca, A., et al. Targeting mitochondrial quality control for treating sarcopenia: lessons from physical exercise. Expert Opinion on Therapeutic Targets. 23 (2), 153-160 (2019).
- Fernandez-Marcos, P. J., Auwerx, J. Regulation of PGC-1α, a nodal regulator of mitochondrial biogenesis. The American Journal of Clinical Nutrition. 93 (4), 884-890 (2011).
- Kim, Y., Triolo, M., Hood, D. A. Impact of aging and exercise on mitochondrial quality control in skeletal muscle. Oxidative Medicine and Cellular Longevity. 2017, 3165396 (2017).
- Wang, H., Hiatt, W. R., Barstow, T. J., Brass, E. P. Relationships between muscle mitochondrial DNA content, mitochondrial enzyme activity and oxidative capacity in man: alterations with disease. European Journal of Applied Physiology and Occupational Physiology. 80 (1), 22-27 (1999).
- Tian, Q., et al. Muscle mitochondrial energetics predicts mobility decline in well-functioning older adults: The baltimore longitudinal study of aging. Aging Cell. 21 (2), e13552 (2022).
- Sardon Puig, L., Valera-Alberni, M., Cantó, C., Pillon, N. J. Circadian rhythms and mitochondria: connecting the dots. Frontiers in Genetics. 9, 452 (2018).
- Gano, L. B., Patel, M., Rho, J. M. Ketogenic diets, mitochondria, and neurological diseases. Journal of Lipid Research. 55 (11), 2211-2228 (2014).
- Liesa, M., Shirihai, O. S. Mitochondrial dynamics in the regulation of nutrient utilization and energy expenditure. Cell Metabolism. 17 (4), 491-506 (2013).
- Lesnefsky, E. J., Chen, Q., Hoppel, C. L. Mitochondrial metabolism in aging heart. Circulation Research. 118 (10), 1593-1611 (2016).
- Hartman, M. L., et al. Relation of mitochondrial oxygen consumption in peripheral blood mononuclear cells to vascular function in type 2 diabetes mellitus. Vascular Medicine. 19 (1), 67-74 (2014).
- Mahapatra, G., et al. Blood-based bioenergetic profiling is related to differences in brain morphology in African Americans with Type 2 diabetes. Clinical Science. 132 (23), 2509-2518 (2018).
- Moore-Ede, M. C. Physiology of the circadian timing system: predictive versus reactive homeostasis. The American Journal of Physiology. 250 (5), R737-R752 (1986).
- Young, M. W. Life's 24-hour clock: molecular control of circadian rhythms in animal cells. Trends in Biochemical Sciences. 25 (12), 601-606 (2000).
- Yoo, S. H., et al. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proceedings of the National Academy of Sciences. 101 (15), 5339-5346 (2004).
- Zhang, R., Lahens, N. F., Ballance, H. I., Hughes, M. E., Hogenesch, J. B. A circadian gene expression atlas in mammals: implications for biology and medicine. Proceedings of the National Academy of Sciences. 111 (45), 16219-16224 (2014).
- de Goede, P., Wefers, J., Brombacher, E. C., Schrauwen, P., Kalsbeek, A. Circadian rhythms in mitochondrial respiration. Journal of Molecular Endocrinology. 60 (3), R115-R130 (2018).
- Hood, S., Amir, S. The aging clock: circadian rhythms and later life. The Journal of Clinical Investigation. 127 (2), 437-446 (2017).
- Sellix, M. T., et al. Aging differentially affects the re-entrainment response of central and peripheral circadian oscillators. The Journal of Neuroscience. 32 (46), 16193-16202 (2012).
- Sato, S., et al. Circadian reprogramming in the liver identifies metabolic pathways of aging. Cell. 170 (4), 664-677 (2017).
- Lundell, L. S., et al. et al. feeding alters lipid and amino acid metabolite rhythmicity without perturbing clock gene expression. Nature Communications. 11 (1), 4643 (2020).
- Perrin, L., et al. Transcriptomic analyses reveal rhythmic and CLOCK-driven pathways in human skeletal muscle. eLife. 7, e34114 (2018).
- Gutierrez-Monreal, M. A., Harmsen, J. -. F., Schrauwen, P., Esser, K. A. Ticking for metabolic health: the skeletal-muscle clocks. Obesity. 28, S46-S54 (2020).
- Wolff, C. A., et al. Defining the age-dependent and tissue-specific circadian transcriptome in male mice. bioRxiv. 42 (1), 111982 (2023).
- Wilson, D., Breen, L., Lord, J. M., Sapey, E. The challenges of muscle biopsy in a community based geriatric population. BMC Research Notes. 11 (1), 830 (2018).
- Ding, H., et al. Likelihood-based tests for detecting circadian rhythmicity and differential circadian patterns in transcriptomic applications. Briefings in Bioinformatics. 22 (6), 224 (2021).
- Ding, Z., Lamb, T. M., Boukhris, A., Porter, R., Bell-Pedersen, D. Circadian clock control of translation initiation factor eIF2α activity requires eIF2γ-dependent recruitment of rhythmic PPP-1 phosphatase in Neurospora crassa. mBio. 12 (3), e00871 (2021).
- Di Francesco, A., Di Germanio, C., Bernier, M., de Cabo, R. A time to fast. Science. 362 (6416), 770-775 (2018).
- Kalfalah, F., et al. Crosstalk of clock gene expression and autophagy in aging. Aging. 8 (9), 1876-1895 (2016).
- Psaty, B. M., et al. Assessing the use of medications in the elderly: methods and initial experience in the cardiovascular health study. Journal of Clinical Epidemiology. 45 (6), 683-692 (1992).
- van Moorsel, D., et al. Demonstration of a day-night rhythm in human skeletal muscle oxidative capacity. Molecular Metabolism. 5 (8), 635-645 (2016).
- Janssen, J. J. E., et al. Extracellular flux analyses reveal differences in mitochondrial PBMC metabolism between high-fit and low-fit females. American Journal of Physiology. Endocrinology and Metabolism. 322 (2), E141-E153 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon