È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Applicazione di AlDeSense per stratificare cellule tumorali ovariche basate sull'attività dell'aldeide deidrogenasi 1A1
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
I metodi per misurare l'attività di ALDH1A1 nelle cellule vive sono fondamentali nella ricerca sul cancro a causa del suo status di biomarcatore della staminalità. In questo studio, abbiamo impiegato una sonda fluorogenica isoform-selettiva per determinare i livelli relativi di attività di ALDH1A1 in un pannello di cinque linee cellulari di cancro ovarico.
Abstract
La recidiva dopo il trattamento del cancro è spesso attribuita alla persistenza di una sottopopolazione di cellule tumorali note come cellule staminali tumorali (CSC), che sono caratterizzate dalla loro notevole capacità di iniziare il tumore e di auto-rinnovarsi. A seconda dell'origine del tumore (ad esempio, ovaie), il profilo del biomarcatore di superficie CSC può variare notevolmente, rendendo l'identificazione di tali cellule tramite colorazione immunoistochimica uno sforzo impegnativo. Al contrario, l'aldeide deidrogenasi 1A1 (ALDH1A1) è emersa come un eccellente marcatore per identificare le CSC, grazie al suo profilo di espressione conservato in quasi tutte le cellule progenitrici, comprese le CSC. L'isoforma ALDH1A1 appartiene a una superfamiglia di 19 enzimi responsabili dell'ossidazione di varie aldeidi endogene e xenobiotiche ai corrispondenti prodotti di acido carbossilico. Chan et al. hanno recentemente sviluppato AlDeSense, una sonda "turn-on" selettiva per l'isoforma per il rilevamento dell'attività di ALDH1A1, nonché un reagente di controllo di corrispondenza non reattivo (Ctrl-AlDeSense) per tenere conto della colorazione fuori bersaglio. Questo strumento selettivo per le isoforme ha già dimostrato di essere uno strumento chimico versatile attraverso la rilevazione dell'attività di ALDH1A1 nelle cellule di leucemia mieloide K562, nelle mammosfere e negli xenotrapianti CSC derivati dal melanoma. In questo articolo, l'utilità della sonda è stata mostrata attraverso ulteriori esperimenti di fluorimetria, microscopia confocale e citometria a flusso in cui la relativa attività di ALDH1A1 è stata determinata in un pannello di cinque linee cellulari di cancro ovarico.
Introduzione
Le cellule staminali tumorali (CSC) sono una sottopopolazione di cellule tumorali che presentano proprietà simili alle cellule staminali1. Simili alle loro controparti non cancerose, le CSC possiedono la straordinaria capacità di auto-rinnovarsi e proliferare. Insieme ad altri meccanismi incorporati, come la sovraregolazione dei trasportatori di cassette che legano ATP, le CSC sono spesso risparmiate dagli sforzi iniziali di debulking chirurgico, nonché dalla successiva terapia adiuvante2. A causa del loro ruolo critico nella resistenza al trattamento3, recidiva4 e metastasi5, le CSC sono diventate una priorità nella ricerca sul cancro. Sebbene ci sia una varietà di antigeni di superficie cellulare (ad esempio, CD133) che possono essere utilizzati per identificare le CSCs6, sfruttare l'attività enzimatica delle aldeidi deidrogenasi (ALDH) trovate nel citoplasma è emerso come un'alternativa attraente7. Gli ALDH sono una superfamiglia di 19 enzimi responsabili della catalizzazione dell'ossidazione delle aldeidi reattive endogene e xenobiotiche nei corrispondenti prodotti di acido carbossilico8.
In generale, la disintossicazione da aldeide è fondamentale per proteggere le cellule da eventi di reticolazione indesiderati e stress ossidativo che possono danneggiare l'integrità delle cellule staminali9. Inoltre, l'isoforma 1A1 controlla il metabolismo dell'acido retinoico, che a sua volta influenza la staminalità attraverso la segnalazione10 della retinaldeide. AlDeSense 11,12, una sonda di rilevamento basato sull'attività (ABS) a piccole molecole per rilevare selettivamente l'attività di ALDH1A1, è stata recentemente sviluppata. I progetti ABS ottengono il rilevamento dell'analita attraverso un cambiamento chimico piuttosto che un evento di legame, consentendo un'elevata selettività e una riduzione delle risposte fuori bersaglio13,14,15,16. Il principio di progettazione della sonda fluorogenica selettiva per isoforme si basa su un meccanismo di spegnimento17 del trasferimento elettronico fotoindotto da donatore (d-PeT), originato dal gruppo funzionale aldeidico, che serve a sopprimere la firma fluorescente della sonda18. Dopo la conversione mediata da ALDH1A1 nell'acido carbossilico, il rilassamento radiativo viene sbloccato per produrre un prodotto altamente fluorescente. Poiché la tempra d-PeT non è mai efficiente al 100%, la fluorescenza residua che può portare a possibili risultati falsi positivi è stata considerata quando si stabilisce questo test attraverso lo sviluppo di Ctrl-AlDeSense, un reagente non reattivo con caratteristiche fotofisiche corrispondenti (ad esempio, resa quantica) e un identico modello di colorazione citoplasmatica nelle cellule. Se utilizzato in tandem, questo accoppiamento unico può distinguere in modo affidabile le cellule con elevata attività di ALDH1A1 da quelle che presentano bassi livelli tramite fluorimetria, imaging molecolare e citometria a flusso. Diversi vantaggi chiave sono associati all'uso di coloranti attivabili isoform-selettivi rispetto ai metodi immunoistochimici tradizionali. Ad esempio, si ipotizza che le CSC siano sepolte in profondità all'interno di un tumore, e quindi sono più accessibili a una piccola molecola rispetto ai grandi anticorpi19. Inoltre, il prodotto fluorescente capovolto non modifica covalentemente alcun componente cellulare, il che significa che può essere facilmente rimosso tramite cicli di lavaggio per lasciare un CSC in uno stato non modificato. Infine, la risposta di accensione identifica solo cellule e funzioni vitali, proprio come il test MTT, a causa della sua dipendenza dal cofattore NAD +.
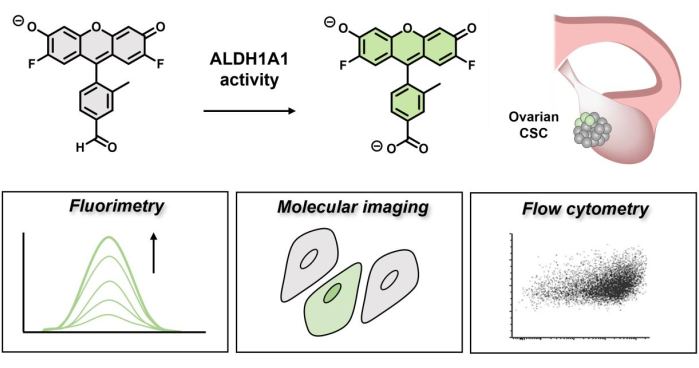
Figura 1: Schema che dimostra l'accensione fluorescente di AlDeSense. Il colorante isoform-selettivo è attivato da ALDH1A1 e può essere utilizzato per identificare un'elevata attività di ALDH1A1 nelle cellule tumorali ovariche tramite fluorimetria, imaging molecolare e citometria a flusso. Fare clic qui per visualizzare una versione ingrandita di questa figura.
In lavori precedenti, il test della sonda fluorogenica selettiva per isoforme ha stratificato con successo cellule ALDH alte (ALDH +) da cellule ALDH basse (ALDH-) in cellule di leucemia cronica umana K562, cellule di cancro al seno umano MDA-MB-231 e cellule di melanoma murino B16F0. Questo è importante perché, per molti tipi di cancro, un'elevata espressione della proteina ALDH1A1 significa una prognosi clinica peggiore20. Ciò presuppone che livelli elevati di ALDH1A1 siano indicativi di CSC che possono eludere il trattamento, sviluppare resistenza e diffondersi in tutto il corpo. Tuttavia, nel caso del carcinoma ovarico, ci sono studi che riportano il risultato opposto (un'elevata espressione di ALDH1A1 è legata a una migliore sopravvivenza della paziente)21,22,23,24. Sebbene ciò possa sembrare contraddittorio a prima vista, l'espressione non è necessariamente correlata all'attività enzimatica, che può essere influenzata da cambiamenti nel microambiente tumorale (ad esempio, flusso di pH, gradienti di ossigeno), disponibilità del cofattore NAD+ o substrati aldeidici, livelli di acidi carbossilici (inibizione del prodotto) e modifiche post-traduzionali che possono alterare l'attività enzimatica25 . Inoltre, il carcinoma ovarico è diviso in cinque tipi istologici principali (sieroso di alto grado, sieroso di basso grado, endometrioide, a cellule chiare e mucinoso), che ipotizziamo saranno caratterizzati da livelli variabili di attività ALDH1A126. Con l'obiettivo di studiare l'attività di ALDH1A1 nei tumori ovarici, è stato utilizzato un saggio di sonda fluorogenica selettiva per identificare le popolazioni di ALDH1A1+ in un pannello di cinque linee cellulari di cancro ovarico appartenenti ai diversi tipi istologici sopra menzionati. Le linee cellulari testate in questo studio includono cellule BG-1, Caov-3, IGROV-1, OVCAR-3 e PEO4, che coprono istotipi a cellule chiare e sierosi. Qui, la versatilità e la generalizzabilità della sonda è stata evidenziata per identificare le CSC per i ricercatori che cercano di eseguire studi simili in altre linee cellulari tumorali immortalizzate e campioni di pazienti. L'uso di AlDeSense farà luce sui percorsi biochimici coinvolti nel mantenimento delle CSC in microambienti tissutali complessi e potenzialmente servirà come strumento clinico per determinare la prognosi e misurare l'aggressività del cancro.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Misurare l'attività totale di ALDH1A1 negli omogenati delle cellule tumorali ovariche tramite fluorimetria
- Scongelare 1 × 106 cellule in un matraccio di coltura cellulare T25 in 5 mL dei seguenti terreni di coltura cellulare:
- IGROV-1 e PEO4: Roswell Park Memorial Institute (RPMI) 1640 terreno con 10% siero bovino fetale (FBS) e 1% penicillina/streptomicina (P/S).
- BG-1 e Caov-3: Modified Eagle's Medium (DMEM) di Dulbecco con 10% FBS e 1% P/S.
- OVCAR-3: RPMI 1640 con 20% FBS, 1% P/S e 0,01 mg/mL di insulina.
- Mantenere le cellule in un incubatore a 37 °C e al 5% di CO2 per due o tre passaggi. Assicurarsi che le celle non superino l'80% -90% di confluenza prima di passare.
- Tripsinizzare le cellule in tripsina allo 0,25% per 10 minuti, contarle utilizzando un contatore automatico di celle e pellettare 1 × 107 cellule mediante centrifugazione (180 × g) a 25 °C per 5 minuti.
- Rimuovere accuratamente il surnatante tramite aspirazione, lavare il pellet risospendendo le cellule in 1 mL di 1x PBS e ripellettare le cellule tramite centrifugazione nelle stesse condizioni descritte al punto 4.
- Risospendere le cellule in una soluzione di 1x inibitore della proteasi in 1x PBS. Utilizzare 1 mL di questa soluzione per 2,5 × 106 cellule.
- Sonicare la sospensione cellulare sul ghiaccio per 2 minuti (impulso 1 s, ampiezza 40%) con una sonda omogeneizzatore cellulare.
- Frazioni insolubili/membrana di pellet tramite centrifugazione (3.200 × g) a 25 °C per 15 min. Rimuovere e mantenere il surnatante, in quanto questo è l'omogenato che verrà utilizzato nei passaggi successivi. Separare gli omogenati in tre cuvette per eseguire l'esperimento in triplice copia.
- Aggiungere la sonda all'omogenato per ottenere una concentrazione finale della sonda di 4 μM. Pipettare su e giù tre volte per miscelare bene la soluzione.
- Misurare il segnale fluorescente immediatamente dopo l'aggiunta e dopo i tempi di incubazione desiderati su un fluorimetro. Il tempo di incubazione può essere ottimizzato per vedere la massima accensione della piega (1 ora in questo esperimento). Il tempo di incubazione deve essere costante su tutte le linee cellulari confrontate.
- Impostare la lunghezza d'onda di eccitazione su 496 nm.
- Impostare l'emissione su 510-600 nm.
- Impostare la larghezza della fessura su 0,5 mm.
- Pipetta la soluzione in una cuvetta al quarzo da 1 ml, inserirla nel fluorimetro e premere la scansione.
- Dividere l'intensità finale di fluorescenza a 516 nm (lunghezza d'onda fluorescente massima) per l'intensità alla stessa lunghezza d'onda dalla lettura iniziale per determinare l'attivazione della piega del colorante selettivo per isoforme.
2. Uso della microscopia a fluorescenza per l'immagine di cellule con elevata attività di ALDH1A1
- Scongelare 1 × 106 cellule in un matraccio di coltura cellulare T25 in 5 mL del terreno di coltura cellulare appropriato.
- Mantenere le cellule in un incubatore a 37 °C e al 5% di CO2 per due o tre passaggi. Assicurarsi che le celle non superino l'80% -90% di confluenza prima di passare.
- Il giorno prima dell'imaging confocale, la piastra 4 × 105 celle in un vetrino a 8 pozzetti.
- Rivestire il fondo di ciascun pozzetto con poli-L-lisina (0,1 mg/ml, 100 μL per pozzetto) per 10 minuti, aspirando successivamente.
- Lavare ogni pozzetto 3 volte aggiungendo acqua per coltura cellulare (100 μL per pozzetto) e aspirando.
- Tripsinizzare le cellule in tripsina allo 0,25% per 10 minuti, contarle usando un contatore automatico delle cellule e placcare le cellule a 4 × 105 cellule per pozzetto.
- Lasciare che le cellule si depositino e si attacchino durante la notte (12-16 h).
- Aspirare il terreno di coltura e aggiungere mezzi privi di siero (500 μL per pozzetto), integrati con 2 μM della sonda o della sonda di controllo.
- Incubare con la sonda a temperatura ambiente per 30 minuti e visualizzare immediatamente le cellule.
- Accendere il microscopio confocale e regolare il campione.
- Caricare il campione con attenzione al minor ingrandimento; Trova le celle per garantire il corretto posizionamento.
- Assicurarsi che l'obiettivo abbia un ingrandimento 10x e individuare le celle.
- Per questo esperimento sono necessari il canale FITC (eccitazione: laser 488 nm; emissione: 516-521 nm) e il canale T-PMT (luce trasmessa). Individuare e concentrarsi sulle cellule utilizzando T-PMT per concentrarsi sullo stesso piano z per l'intero esperimento per rimuovere la distorsione.
- Regolare la potenza del laser e il guadagno FITC sull'impostazione appropriata, in cui il segnale proveniente dai campioni AM Ctrl-AlDeSense è minimamente rilevabile, pur continuando a vedere il segnale nei campioni AM AlDeSense. Ogni parametro può essere regolato facendo scorrere la barra corrispondente. Potrebbe essere necessario regolare alcune volte le impostazioni per identificare i parametri corretti. Una volta ottimizzato, completa il resto dell'esperimento all'interno di quella linea cellulare usando parametri identici.
- Scatta tre immagini per pozzetto per un totale di tre pozzetti per condizione di trattamento (nove immagini in totale). Concentrati sul piano corretto usando T-PMT per evitare distorsioni piuttosto che utilizzare il canale di fluorescenza o l'immagine unita.
- Elaborare le immagini per determinare la percentuale di cellule ALDH1A1+.
- Utilizzando il software di elaborazione delle immagini, dividere il file czi in diversi canali.
- Contare il numero totale di celle e il numero totale di celle fluorescenti.
- Per determinare la percentuale di celle ALDH1A1+, dividere il numero di celle fluorescenti per il numero totale di celle in ciascuna immagine. È imperativo contare allo stesso modo per ogni immagine senza manipolare le immagini, poiché, ad esempio, la regolazione della luminosità può aggiungere una variabile confondente.
3. Applicazione della citometria a flusso per identificare cellule con elevata attività di ALDH1A1
- Scongelare 1 × 106 cellule in un matraccio di coltura cellulare T25 in 5 mL del terreno di coltura cellulare appropriato.
- Mantenere le cellule in un incubatore a 37 °C e al 5% di CO2 per due o tre passaggi. Assicurarsi che le celle non superino l'80% -90% di confluenza prima di passare.
- Tripsinizzare, contare e pellettare le celle in una provetta da centrifuga da 15 mL mediante centrifugazione (180 × g) a 25 °C per 5 minuti.
- Risospendere le cellule in 1 mL di soluzione sonda da 2 μM/sonda di controllo in PBS. Scuotere le cellule a temperatura ambiente per 60 minuti per garantire che l'esposizione al colorante sia uniforme.
- Dopo il periodo di incubazione, pellettare le cellule mediante centrifugazione (180 × g) a 25 °C per 5 minuti. Risospendere le cellule in 0,5 ml di PBS. Far passare le cellule attraverso un filtro cellulare (rete di nylon da 35 μm) per rimuovere i grumi di cellule che potrebbero ostruire il citometro a flusso. Posizionare immediatamente le celle sul ghiaccio.
- Accendere lo strumento ed eseguire il protocollo di avvio.
- Verificare la presenza di liquidi per guaine e rifiuti vuoti.
- Fai funzionare le linee con candeggina al 10% e acqua per 5 minuti ciascuna.
- Eseguire perline di controllo qualità per garantire il corretto funzionamento.
- Nella scheda delle impostazioni, selezionare FSC (forward scatter), SSC (side scatter) e FITC (fluorescein isothiocyanate) per il filtro a fluorescenza.
- Disegna i seguenti grafici per rilevare vitalità, canottiere e fluorescenza. Ottimizzare la potenza del laser (specifica per lo strumento dell'utente) in modo che le popolazioni cellulari rientrino nei parametri indicati per ulteriori analisi.
- Grafico a dispersione FSC-A contro SSC-A: la popolazione cellulare principale è vicina al centro del grafico.
- Grafico a dispersione FSC-A contro FSC-W: banda orizzontale stretta indicativa di singoletti (piuttosto che grumi di cellule).
- Grafico a dispersione FITC-A versus FSC-A: osservare la distribuzione delle cellule ordinate dalla sonda fluorogenica selettiva per isoforme.
- Istogramma FITC-A: osservare lo spostamento della popolazione basato su FITC per determinare la percentuale di cellule ALDH1A1+.
- Per ottimizzare la potenza del laser FITC, eseguire un campione con la sonda in modo che la coda destra della curva dell'istogramma sia vicina al segnale FITC-A massimo. Successivamente, eseguire un campione con la sonda di controllo. Uno spostamento della popolazione dovrebbe essere osservabile per rivelare la gamma dinamica massima. La fase di ottimizzazione della potenza del laser potrebbe dover essere ripetuta più volte, ma la potenza del laser non dovrebbe essere alterata tra i campioni una volta che un'impostazione è stata designata per un esperimento.
- Eseguire ogni campione per 10.000 conteggi (operazione eseguita in triplice copia).
- Ripetere il passaggio 3.12 per ciascuna linea cellulare, poiché ci sarà variabilità nell'assorbimento e nell'attività di ALDH1A1.
- Dopo aver completato la raccolta dei campioni, eseguire le linee con candeggina al 10% e acqua per 5 minuti ciascuna, quindi avviare lo spegnimento dello strumento.
- Elaborare i dati utilizzando il software di citometria a flusso e controllare la popolazione cellulare desiderata. Impostare le porte in modo che tutti gli eventi rientrino nella porta ALDH1A1- o ALDH1A1+. Utilizzando la selezione del gate rettangolare, impostare il gate ALDH1A1- in modo che il >99,5% degli eventi nei campioni della sonda di controllo si verifichi all'interno di questo gate. Le celle rimanenti saranno considerate ALDH1A1+. Queste stesse porte possono quindi essere applicate al campione di sonda per quantificare il numero di eventi considerati ALDH1A1- e ALDH1A1+.
Access restricted. Please log in or start a trial to view this content.
Risultati
Attività totale di ALDH1A1 degli omogenati delle cellule tumorali ovariche
I turn-on di piegatura medi per ciascuna linea cellulare ottenuta da questo test sono: BG-1 (1,12 ± 0,01); IGROV-1 (1,30 ± 0,03); Caov-3 (1,72 ± 0,06); PEO4 (2,51 ± 0,29); e OVCAR-3 (10,25 ± 1,46) (Figura 2).
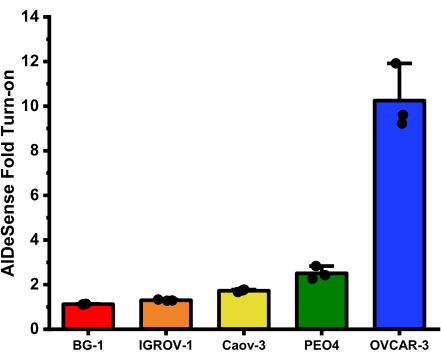
Figura 2<...
Access restricted. Please log in or start a trial to view this content.
Discussione
La panselettività è una delle principali limitazioni di molte sonde ALDH; Tuttavia, sono stati recentemente riportati diversi esempi selettivi per isoforme 32,33,34,35,36,37,38,39,40,41.
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Divulghiamo un brevetto in corso (US20200199092A1) per la tecnologia AlDeSense.
Riconoscimenti
Questo lavoro è stato sostenuto dal National Institutes of Health (R35GM133581 a JC) e dal Cancer Center at Illinois Graduate Scholarship (assegnato a SG). JC ringrazia la Fondazione Camille e Henry Dreyfus per il supporto. Gli autori ringraziano il Dr. Thomas E. Bearrood per il suo contributo iniziale alla preparazione delle scorte di AlDeSense e AlDeSense AM. Ringraziamo Oliver D. Pichardo Peguero e Joseph A. Forzano per la loro assistenza nella preparazione di vari precursori sintetici. Ringraziamo il Prof. Erik Nelson (Dipartimento di Fisiologia Molecolare e Integrativa, UIUC) per le cellule Caov-3, IGROV-1 e PEO4. Ringraziamo il Prof. Paul Hergenrother (Dipartimento di Chimica, UIUC) per le cellule BG-1. Ringraziamo le strutture principali del Carl R. Woese Institute for Genomic Biology per l'accesso al microscopio confocale Zeiss LSM 700 e al software corrispondente. Si ringrazia la Flow Cytometry Facility per l'accesso all'analizzatore BD LSR II CMtO. Ringraziamo la dott.ssa Sandra McMasters e la Cell Media Facility per l'assistenza nella preparazione dei terreni di coltura cellulare.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin, 0.1% EDTA in HBSS w/o Calcium, Magnesium and Sodium Bicarbonate | Corning | 25-050-CI | |
| 1x Phosphate Buffer Saline | Corning | 21-040-CMX12 | |
| AccuSpin Micro 17R | Fisher Scientific | 13-100-675 | |
| AlDeSense | Synthesized in-house | ||
| BG-1 | A gift provided by the Prof. Paul Hergenrother Lab, University of Illinois Urbana-Champaign | ||
| BioLite 25cm2 Flask | Thermo Fisher Scientific | 130189 | |
| Biosafety Cabinet 1300 series A2 | Thermo Fisher Scientific | ||
| Caov-3 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| Cell homogenizer | Fisher Scientific | ||
| Centrifuge 5180R | Eppendorf | 22627040 | |
| Contrl-AlDeSense | Synthesized in-house | ||
| DMEM, 10% FBS, 1% P/S | Prepared by UIUC cell media facility | ||
| Falcon Round-Bottom Polystyrene Test Tubes with Cell Strainer Snap Cap, 5mL | Corning | 352003 | |
| FCS Express 6 | Provided by UIUC CMtO | ||
| FL microscope | EVOS | ||
| Fluorometer | Photon Technology International | ||
| Forma Series II Water-Jacketed CO2 Incubator | Fisher Scientific | 3110 | |
| IGROV-1 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| ImageJ | NIH | ||
| Innova 42R Incubated Shaker | |||
| LSM 700 | Zeiss | ||
| LSR II | BD | ||
| Nunc Lab-Tek Chambered #1.0 Borosicilate Coverglass System | Thermo Fisher Scientific | 155383 | |
| OVCAR-3 | ATCC | HTB-161 | |
| PEO4 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| Pierce Protease Inhibitor Tablets | Thermo Scientific | A32963 | |
| Poly-L-Lysine | Cultrex | 3438-100-01 | |
| Rocker | VWR | ||
| RPMI, 10% FBS, 1% P/S | Prepared by UIUC cell media facility | ||
| RPMI, 20% FBS, 1% P/S, 0.01 mg/mL Insulin | Prepared by UIUC cell media facility |
Riferimenti
- Bonnet, D., Dick, J. E. Human acute myeloid leukaemia is organised as a heirarchy that originates from a primitive haematopoetic cell. Nature Medicine. 3 (7), 730-737 (1997).
- Begicevic, R. R., Falasca, M. ABC transporters in cancer stem cells: Beyond chemoresistance. International Journal of Molecular Sciences. 18 (11), 2362(2017).
- Cojoc, M., Mäbert, K., Muders, M. H., Dubrovska, A. A role for cancer stem cells in therapy resistance: Cellular and molecular mechanisms. Seminars in Cancer Biology. 31, 16-27 (2015).
- Islam, F., Gopalan, V., Smith, R. A., Lam, A. K. Y. Translational potential of cancer stem cells: A review of the detection of cancer stem cells and their roles in cancer recurrence and cancer treatment. Experimental Cell Research. 335 (1), 135-147 (2015).
- Li, F., Tiede, B., Massagué, J., Kang, Y. Beyond tumorigenesis: Cancer stem cells in metastasis. Cell Research. 17 (1), 3-14 (2007).
- Kim, W. T., Ryu, C. J. Cancer stem cell surface markers on normal stem cells. BMB Reports. 50 (6), 285-298 (2017).
- Pors, K., Moreb, J. S. Aldehyde dehydrogenases in cancer: An opportunity for biomarker and drug development. Drug Discovery Today. 19 (12), 1953-1963 (2014).
- Jackson, B., et al. Update on the aldehyde dehydrogenase gene (ALDH) superfamily. Human Genomics. 5 (4), 283-303 (2011).
- Vasiliou, V., Pappa, A., Petersen, D. R. Role of aldehyde dehydrogenases in endogenous and xenobiotic metabolism. Chemico-Biological Interactions. 129 (1-2), 1-19 (2000).
- Tomita, H., Tanaka, K., Tanaka, T., Hara, A. Aldehyde dehydrogenase 1A1 in stem cells and cancer. Oncotarget. 7 (10), 11018-11032 (2016).
- Anorma, C., et al. Surveillance of cancer stem cell plasticity using an isoform-selective fluorescent probe for aldehyde dehydrogenase 1A1. ACS Central Science. 4 (8), 1045-1055 (2018).
- Bearrood, T. E., Aguirre-Figueroa, G., Chan, J. Rational design of a red fluorescent sensor for ALDH1A1 displaying enhanced cellular uptake and reactivity. Bioconjugate Chemistry. 31 (2), 224-228 (2020).
- Chan, J., Dodani, S. C., Chang, C. J. Reaction-based small-molecule fluorescent probes for chemoselective bioimaging. Nature Chemistry. 4 (12), 973-984 (2012).
- East, A. K., Lucero, M. Y., Chan, J. New directions of activity-based sensing for in vivo NIR imaging. Chemical Science. 12 (10), 3393-3405 (2021).
- Yadav, A. K., et al. Activity-based NIR bioluminescence probe enables discovery of diet-induced modulation of the tumor microenvironment via nitric oxide. ACS Central Science. 8 (4), 461-472 (2022).
- Yadav, A. K., et al. An activity-based sensing approach for the detection of cyclooxygenase-2 in Live Cells. Angewandte Chemie. 59 (8), 3307-3314 (2020).
- Ueno, T., et al. Rational principles for modulating fluorescence properties of fluorescein. Journal of the American Chemical Society. 126 (43), 14079-14085 (2004).
- Tanaka, F., Mase, N., Barbas 3rd, C. F. Design and use of fluorogenic aldehydes for monitoring the progress of aldehyde transformations. Journal of the American Chemical Society. 126 (12), 3692-3693 (2004).
- Thurber, G. M., Schmidt, M. M., Wittrup, K. D. Antibody tumor penetration: transport opposed by systemic and antigen-mediated clearance. Advanced Drug Delivery Reviews. 60 (12), 1421-1434 (2008).
- Marcato, P., Dean, C. A., Giacomantonio, C. A., Lee, P. W. K. Aldehyde dehydrogenase its role as a cancer stem cell marker comes down to the specific isoform. Cell Cycle. 10 (9), 1378-1384 (2011).
- Meng, E., et al. ALDH1A1 maintains ovarian cancer stem cell-like properties by altered regulation of cell cycle checkpoint and DNA repair network signaling. PLoS One. 9 (9), e107142(2014).
- Kaipio, K., et al. ALDH1A1-related stemness in high-grade serous ovarian cancer is a negative prognostic indicator but potentially targetable by EGFR/mTOR-PI3K/aurora kinase inhibitors. The Journal of Pathology. 250 (2), 159-169 (2020).
- Deng, S., et al. Distinct expression levels and patterns of stem cell marker, aldehyde dehydrogenase isoform 1 (ALDH1), in human epithelial cancers. PLoS One. 5 (4), e10277(2010).
- Chang, B., et al. ALDH1 expression correlates with favorable prognosis in ovarian cancers. Modern Pathology. 22 (6), 817-823 (2009).
- Gardner, S. H., Reinhardt, C. J., Chan, J. Advances in activity-based sensing probes for isoform-selective imaging of enzymatic activity. Angewandte Chemie. 60 (10), 5000-5009 (2021).
- Reid, B. M., Permuth, J. B., Sellers, T. A. Epidemiology of ovarian cancer: a review. Cancer Biology and Medicine. 14 (1), 9-32 (2017).
- Tulake, W., et al. Upregulation of stem cell markers ALDH1A1 and OCT4 as potential biomarkers for the early detection of cervical carcinoma. Oncology Letters. 16 (5), 5525-5534 (2018).
- Nwani, N. G., et al. A novel ALDH1A1 inhibitor targets cells with stem cell characteristics in ovarian cancer. Cancers. 11 (4), 502(2019).
- Roy, M., Connor, J., Al-Niaimi, A., Rose, S. L., Mahajan, A. Aldehyde dehydrogenase 1A1 (ALDH1A1) expression by immunohistochemistry is associated with chemo-refractoriness in patients with high-grade ovarian serous carcinoma. Human Pathology. 73, 1-6 (2018).
- Landen Jr, C. N., et al. Targeting aldehyde dehydrogenase cancer stem cells in ovarian cancer. Molecular Cancer Therapeutics. 9 (12), 3186-3199 (2010).
- Condello, S., et al. β-catenin-regulated ALDH1A1 is a target in ovarian cancer spheroids. Oncogene. 34 (18), 2297-2308 (2015).
- Storms, R. W., et al. Isolation of primitive human hematopoietic progenitors on the basis of aldehyde dehydrogenase activity. Proceedings of the National Academy of Sciences. 96 (16), 9118-9123 (1999).
- Duellman, S. J., et al. A bioluminescence assay for aldehyde dehydrogenase activity. Analytical Biochemistry. 434 (2), 226-232 (2013).
- Minn, I., et al. A red-shifted fluorescent substrate for aldehyde dehydrogenase. Nature Communications. 5, 3662(2014).
- Dollé, L., Boulter, L., Leclercq, I. A., van Grunsven, L. A. Next generation of ALDH substrates and their potential to study maturational lineage biology in stem and progenitor cells. American Journal of Physiology. Gastrointestinal and Liver Physiology. 308 (7), 573-578 (2015).
- Yagishita, A., et al. Development of highly selective fluorescent probe enabling flow-cytometric isolation of ALDH3A1-positive viable cells. Bioconjugate Chemistry. 28 (2), 302-306 (2017).
- Maity, S., et al. Thiophene bridged aldehydes (TBAs) image ALDH activity in cells: Via modulation of intramolecular charge transfer. Chemical Science. 8 (10), 7143-7151 (2017).
- Koenders, S. T. A., et al. Development of a retinal-based probe for the profiling of retinaldehyde dehydrogenases in cancer cells. ACS Central Science. 5 (12), 1965-1974 (2019).
- Oe, M., et al. Deep-red/near-infrared turn-on fluorescence probes for aldehyde dehydrogenase 1A1 in cancer stem cells. ACS Sensors. 6 (9), 3320-3329 (2021).
- Yagishita, A., Ueno, T., Tsuchihara, K., Urano, Y. Amino BODIPY-based blue fluorescent probes for aldehyde dehydrogenase 1-expressing cells. Bioconjugate Chemistry. 32 (2), 234-238 (2021).
- Okamoto, A., et al. Identification of breast cancer stem cells using a newly developed long-acting fluorescence probe, C5S-A, targeting ALDH1A1. Anticancer Research. 42 (3), 1199-1205 (2022).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon