È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Metodi ottimizzati per l'immobilizzazione superficiale di saggi di collagene e leganti il collagene
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo lavoro presenta un protocollo ottimizzato per immobilizzare e quantificare in modo riproducibile il collagene di tipo I e III su micropiastre, seguito da un protocollo migliorato di saggio di legame in vitro per studiare le interazioni collagene-composto utilizzando un metodo di fluorescenza risolto nel tempo. Vengono fornite le successive analisi e interpretazioni dei dati passo dopo passo.
Abstract
La fibrosi si verifica in vari tessuti come risposta riparativa a lesioni o danni. Se eccessiva, tuttavia, la fibrosi può portare a cicatrici tissutali e insufficienza d'organo, che è associata a un'elevata morbilità e mortalità. Il collagene è un fattore chiave della fibrosi, con il collagene di tipo I e di tipo III che sono i tipi principali coinvolti in molte malattie fibrotiche. A differenza dei protocolli convenzionali utilizzati per immobilizzare altre proteine (ad esempio, elastina, albumina, fibronectina, ecc.), non sono prontamente disponibili protocolli completi per immobilizzare in modo riproducibile diversi tipi di collagene al fine di produrre rivestimenti stabili. L'immobilizzazione del collagene è sorprendentemente impegnativa perché molteplici condizioni sperimentali possono influenzare l'efficienza dell'immobilizzazione, tra cui il tipo di collagene, il pH, la temperatura e il tipo di micropiastra utilizzata. Qui, viene fornito un protocollo dettagliato per immobilizzare e quantificare in modo riproducibile i collageni di tipo I e III con conseguente gel/film stabili e riproducibili. Inoltre, questo lavoro dimostra come eseguire, analizzare e interpretare studi in vitro di legame della fluorescenza risolti nel tempo per studiare le interazioni tra collageni e composti candidati leganti il collagene (ad esempio, un peptide coniugato a un chelato metallico che trasporta, ad esempio, l'europio [Eu(III)]). Tale approccio può essere universalmente applicato a varie applicazioni biomediche, tra cui il campo dell'imaging molecolare per sviluppare sonde di imaging mirate, lo sviluppo di farmaci, studi di tossicità cellulare, studi di proliferazione cellulare e saggi immunologici.
Introduzione
L'accumulo di tessuto connettivo fibroso come parte del naturale processo di guarigione delle ferite a seguito di una lesione tissutale è noto come fibrosi. Tuttavia, se la deposizione di tessuto fibroso non termina e continua oltre quanto necessario per la riparazione dei tessuti, la fibrosi diventa eccessiva 1,2. Un'eccessiva fibrosi compromette la fisiologia e la funzione degli organi e potrebbe portare a danni agli organi e potenzialmente insufficienza d'organo 3,4,5. Due fattori principali della fibrosi sono le proteine della matrice extracellulare (ECM), il collagene di tipo I e di tipo III6. Il collagene è una proteina strutturale presente in vari organi che costituisce circa un terzo del contenuto proteico totale del corpo umano1. Esistono 28 diversi tipi di collagene identificati dal sequenziamento del genoma umano e i più abbondanti sono i collageni fibrillari7. Il collagene fibrillare primario è il collagene di tipo I, che fornisce alla ECM resistenza alla trazione e alla deformazione8. Il collagene di tipo III è un componente strutturale che fornisce elasticità e si colocalizza con il collagene di tipo I. È espresso durante l'embriogenesi e si trova naturalmente in piccole quantità nella pelle, nei muscoli e nei vasi sanguigni degli adulti9.
In vivo la sintesi del collagene inizia con un processo intracellulare in cui l'mRNA viene trascritto nel nucleo e poi si sposta nel citoplasma, dove viene tradotto. Dopo la traduzione, la catena formata subisce una modificazione post-traduzionale nel reticolo endoplasmatico, dove si forma il pro-collagene (il precursore del collagene). Il pro-collagene viaggia quindi verso l'apparato di Golgi per la modifica finale prima di essere escreto nello spazio extracellulare10. Attraverso la scissione proteolitica, il pro-collagene viene trasformato in tropocollagene. Questo viene quindi reticolato tramite una via di cross-linking enzimatica mediata catalizzata dall'enzima lisil ossidasi (LOX) o tramite una via di cross-linking non enzimaticamente mediata che coinvolge la reazione di Maillard11. I protocolli in vitro per immobilizzare il collagene si basano principalmente sulla capacità del collagene di autoassemblarsi. Il collagene viene estratto dai tessuti in base alla sua solubilità, che dipende in gran parte dall'entità della reticolazione delle singole fibrille di collagene7. Il collagene fibrillare è disciolto in acido acetico e le fibrille possono riformarsi quando il pH e la temperatura vengono regolati12. In vitro, la fibrillogenesi del collagene può essere vista come un processo in due fasi7. La prima fase è la fase di nucleazione, in cui le fibre di collagene formano dimeri e fibrille trimeri prima di essere riarrangiate per formare una struttura a tripla elica. La seconda fase è la fase di crescita, in cui le fibrille iniziano a crescere lateralmente e provocano la caratteristica formazione della banda D, che è generalmente osservata da cambiamenti nella torbidità7. Gli studi di microscopia a forza atomica (AFM) hanno anche rivelato che il collagene di tipo I e di tipo III hanno caratteristiche diverse (Tabella 1)13.
Per studiare le interazioni di legame tra il collagene e altri composti, il collagene deve essere immobilizzato in modo riproducibile nei pozzetti delle micropiastre. Esistono vari protocolli per immobilizzare il collagene solubile 14,15,16. Le micropiastre disponibili in commercio pre-rivestite con collagene sono tipicamente utilizzate per la coltura cellulare. Tuttavia, le micropiastre pre-rivestite hanno uno strato molto sottile di una quantità sconosciuta di collagene rivestita sui pozzetti, il che le rende inadatte per i saggi di legame in vitro. Ci sono diverse sfide quando si immobilizza il collagene sui pozzetti della piastra. Una delle sfide principali è la scelta di un tipo di micropiastra adatto, perché diversi tipi di collagene (ad esempio, tipo I e III) hanno proprietà chimiche diverse e, quindi, immobilizzano in modo più stabile ed efficace a seconda del materiale della micropiastra. Un'altra sfida sono le condizioni sperimentali del protocollo di immobilizzazione, poiché il processo di fibrillogenesi dipende da molteplici fattori, tra cui la temperatura, il pH, la concentrazione di stock di collagene e la concentrazione ionica del tampone7.
Per studiare le interazioni tra il collagene (il bersaglio) e altri composti (ad esempio, un peptide bersaglio), è anche necessario sviluppare un robusto saggio di screening per indagare la specificità e la selettività del composto verso il bersaglio misurando la costante di dissociazione, Kd. La posizione dell'equilibrio di formazione di un complesso bimolecolare tra una proteina (collagene) e un ligando è espressa in termini della costante di associazione Ka, la cui grandezza è proporzionale all'affinità di legame. Tuttavia, più comunemente, i biochimici esprimono relazioni di affinità in termini della costante di dissociazione di equilibrio, Kd, del complesso bimolecolare, che è definito come Kd = 1/Ka (Kd ed è l'inverso di Ka). Più basso è il valore di Kd, più forte è la forza di legame tra la proteina e il ligando. Il vantaggio di utilizzare Kd per confrontare l'affinità di legame di diversi ligandi per la stessa proteina (e viceversa) è legato al fatto che le unità di Kd per un complesso bimolecolare sono mol/L (cioè unità di concentrazione). Nella maggior parte delle condizioni sperimentali, il valore di Kd corrisponde alla concentrazione del ligando che porta alla saturazione del 50% dei siti di legame disponibili sul bersaglio all'equilibrio17,18. La costante di dissociazione viene tipicamente estratta analizzando l'occupazione frazionaria del recettore (FO), che è definita come il rapporto tra i siti di legame occupati e i siti di legame totali disponibili, in funzione della concentrazione del ligando. Ciò può essere fatto a condizione che sia disponibile un test analitico in grado di distinguere e misurare la quantità di ligando legato.
I saggi di legame del ligando in vitro possono essere eseguiti utilizzando vari metodi bioanalitici, tra cui la fotometria ottica, i metodi del radioligando, la spettrometria di massa al plasma accoppiato induttivamente (ICP-MS) e la risonanza plasmonica di superficie (SPR). Tra i metodi fotometrici, quelli basati sull'emissione di fluorescenza richiedono tipicamente la marcatura di ligandi o proteine con fluorofori per aumentare la sensibilità e migliorare il limite di rilevabilità del saggio. I chelati di alcuni ioni lantanide (III), come l'Eu (III), sono molto attraenti come fluorofori in quanto hanno grandi spostamenti di Stokes, bande di emissione strette (che forniscono un buon rapporto segnale/rumore), fotosbiancamento limitato e lunga durata di emissione. È importante sottolineare che quest'ultima proprietà consente l'uso della fluorescenza risolta nel tempo (TRF) dai fluorofori Eu(III) per abolire l'autofluorescenza di fondo19. Nella versione DELFIA (Dissociation-Enhancement Lanthanide fluorescent immunoassay) del test TRF basato su Eu(III), i ligandi marcati con un chelato di Eu(III) non luminescente vengono incubati con il recettore immobilizzato su micropiastre. Il complesso ligando/recettore marcato viene separato dal ligando non legato e la fluorescenza Eu(III) viene attivata dalla dissociazione del complesso Eu(III) a pH acido, seguita dalla ricomplessazione con un chelante che migliora la fluorescenza per formare un complesso Eu(III) altamente fluorescente20 incorporato nella micella.
La fase di decomplessazione può essere ragionevolmente ottenuta con chelanti, come il dietilentriammina pentaacetato (DTPA), che mostrano una cinetica di decomplessazione rapida. Tuttavia, i complessi Eu(III) con alcuni chelanti macrociclici, come DOTA (acido 1,4,7,10-tetraazaciclododecano1,4,7,10-tetraacetico) e i suoi derivati monoammidici (DO3AAm), mostrano un'elevata stabilità termodinamica e un'elevata inerzia cinetica. In questo caso, le fasi di decomplessazione devono essere accuratamente ottimizzate per ottenere un'attivazione sufficiente e riproducibile del TRF21 a base di Eu(III). Vale la pena notare che i complessi lantanidi (Ln(III))-DOTA e Ln(III)-DO3AAm sono quelli più comunemente impiegati come agenti di contrasto per l'imaging molecolare in vivo mediante tecniche di risonanza magnetica (MRI)22. Pertanto, il test TRF basato su Ln(III) è lo strumento di scelta per studiare in vitro l'affinità di legame delle sonde molecolari MRI con i bersagli biologici previsti. Attualmente, mancano protocolli completi e riproducibili per immobilizzare il collagene di tipo I e di tipo III e una pipeline riproducibile per l'esecuzione di esperimenti di TRF di legame in vitro con Eu(III). Per superare queste limitazioni, sono stati sviluppati metodi riproducibili per autoassemblare e immobilizzare il collagene di tipo I e di tipo III e generare gel e film stabili, rispettivamente, con la concentrazione sufficiente di collagene richiesta per i saggi di legame in vitro . Viene presentato un protocollo ottimizzato per Eu(III) TRF di complessi altamente inerti basati su Eu(III)-DO3Aam. Infine, è stato dimostrato un test TRF Eu(III) in micropiastra ottimizzato per misurare il Kd dei ligandi marcati con Eu(III) verso il collagene immobilizzato di tipo I e di tipo III (Figura 1).
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Tutte le informazioni sul prodotto utilizzate per questo lavoro sono presentate nella Tabella dei materiali.
1. Immobilizzazione del collagene
NOTA: Assicurarsi che ogni pozzetto nella micropiastra utilizzata durante il saggio di legame abbia pozzetti adiacenti liberi per evitare la fluorescenza incrociata. Esegui questa parte del protocollo sul ghiaccio perché il collagene si autoassembla all'aumentare delle temperature e dei livelli di pH. Eseguire questa procedura in una cappa per colture tissutali e in condizioni sterili poiché le micropiastre vengono successivamente incubate in un incubatore per colture tissutali (TC).
- Immobilizzazione del collagene di tipo I sulle micropiastre a 96 pozzetti (Figura 2)
Giorno 1- Preparare una vaschetta in silicone con ghiaccio. Metti la fiala contenente collagene di tipo I, la soluzione salina fredda tamponata con fosfato (PBS) 10x e le micropiastre sul ghiaccio e spruzza tutto con etanolo al 70%. Posizionare il materiale sotto il cofano TC.
- Neutralizzare il collagene utilizzando volumi uguali di collagene di tipo I e 10x PBS (pH 7,4).
- Capovolgere la soluzione alcune volte, assicurandosi che non si formino bolle.
- Aggiungere 100 μL di collagene neutralizzato a ogni altro pozzetto e a ogni altra fila della micropiastra e incubare a 37 °C per 18-20 ore per far evaporare il collagene fino a quando non sarà asciutto.
Giorno 2 - Lavare le micropiastre con 100 μl di 1x PBS, pH 7,4, due volte per rimuovere il collagene non legato.
- Trasferire le micropiastre nell'incubatore a 37 °C per altre 2 ore ad asciugare prima di utilizzarle per ulteriori esperimenti di legame.
- Immobilizzazione del collagene di tipo III sulle micropiastre a 96 pozzetti (Figura 3)
Giorno 1- Preparare una vaschetta in silicone con ghiaccio. Metti la fiala contenente il collagene di tipo III, il PBS freddo 10x e le micropiastre sul ghiaccio e spruzza il tutto con etanolo al 70%. Posizionare il materiale sotto il cofano TC.
- Neutralizzare il collagene utilizzando volumi uguali di collagene di tipo III e 10x PBS (pH 7,4).
- Aggiungere 70 μl di collagene neutralizzato a ogni altro pozzetto e a ogni altra fila della micropiastra e incubare a 37 °C per 2 ore posizionando la micropiastra sotto la cappa di coltura tissutale per far evaporare il collagene fino a renderlo asciutto.
Giorno 2 - Lavare le micropiastre con 70 μL di 1x PBS, pH 7,4, due volte per rimuovere il collagene non legato.
- Trasferire le micropiastre nell'incubatore per 1 ora a 37 °C, quindi trasferire le micropiastre sul banco e lasciare asciugare 1 ora prima di utilizzarle in ulteriori esperimenti di legame.
2. Valutazione della stabilità dei gel/film di collagene immobilizzati
- Incubazione con PBS per 1 ora
NOTA: Durante l'esperimento di legame, incubare il collagene immobilizzato con il composto di interesse. È importante studiare la stabilità del gel o del film di collagene risultante. Per fare ciò, misurare la stabilità di tre condizioni: no wash = misura il collagene immobilizzato subito dopo l'incubazione; lavaggio = misura il collagene immobilizzato dopo aver lavato la piastra due volte con 100 μL di PBS; e 1 ora PBS mimic & wash = misura il collagene immobilizzato dopo l'incubazione per 1 ora con PBS seguito da due lavaggi con PBS. Di seguito, viene spiegato il metodo di incubazione PBS.- Aggiungere 70 μl di PBS (1x) a ciascuno dei pozzetti rivestiti di collagene e incubare la micropiastra a temperatura ambiente per 1 ora.
- Aspirare il liquido in eccesso da ciascun pozzetto utilizzando una pipetta e lavare con PBS (1x) due volte prima di eseguire il test di quantificazione delle proteine descritto di seguito.
- Quantificazione della quantità di collagene immobilizzato mediante un saggio dell'acido bicinconninico (BCA)
NOTA: Utilizzare il kit per il dosaggio delle proteine Pierce BCA (tabella dei materiali) seguendo le istruzioni del produttore. Stabilire i rispettivi standard di collagene per questo test. L'intervallo di concentrazione per il collagene I è compreso tra 0 e 3.000 μg/mL e per il collagene III tra 0 e 750 μg/mL. In totale, crea 11 standard per collagene.- Preparare il volume totale di reagente di lavoro (WR) necessario seguendo le istruzioni del produttore.
- Aggiungere 25 μl di ciascuno standard di collagene nei pozzetti della micropiastra (in duplicato). Queste soluzioni vengono utilizzate per disegnare la curva standard.
- Aggiungere 200 μl di soluzione reagente di lavoro a ciascuno dei pozzetti contenenti gli standard e i pozzetti rivestiti con concentrazioni sconosciute di collagene.
- Posizionare la micropiastra su uno shaker per 30 s. Coprire le micropiastre e incubare a 37 °C per 30 minuti.
- Togliete le micropiastre e lasciatele raffreddare a temperatura ambiente. Misurare l'assorbanza a 560 nm utilizzando un lettore di piastre.
- Tracciare una curva di calibrazione tracciando l'A560 (AU) rispetto alla concentrazione (μg/mL) delle 11 soluzioni standard e utilizzare la curva di calibrazione per calcolare la quantità di collagene.
3. Saggio di legame del ligando TRF dell'europio (III) (Figura 1)
NOTA: Il composto utilizzato è un peptide candidato legante il collagene (CBP) marcato con un singolo complesso Eu(III)-DO3AAm, denominato Eu(III)-DO3AAm-CBP (Figura 4).
- Incubazione delle piastre rivestite di collagene con il composto Eu(III)-DO3AAm-CBP
- Preparare soluzioni del composto Eu(III)-DO3AAm-CBP con concentrazioni comprese tra 0,1 e 15 μM (0,1 μM, 0,5 μM, 1 μM, 3 μM, 5 μM, 7 μM, 10 μM e 15 μM) in 1x PBS.
- Aggiungere 75 μl di ciascuna concentrazione di composto nei pozzetti rivestiti di collagene (Piastra A). Esegui l'esperimento in triplicato per calcolare la quantità di composto che si lega al collagene.
- Utilizzare una seconda piastra non rivestita (piastra B) e aggiungere 75 μl di ciascun composto ai pozzetti vuoti per calcolare il legame non specifico del composto alla piastra. Utilizzare triplicati per ogni concentrazione.
- Incubare le micropiastre per 1 ora a temperatura ambiente.
- Utilizzando una pipetta, aspirare ed eliminare la soluzione in eccesso da ciascun pozzetto e lavare i pozzetti con 1x PBS due volte per rimuovere il composto in eccesso e non legato. Eseguire questo passaggio utilizzando sia le micropiastre rivestite di collagene che quelle non rivestite.
- A una terza piastra non rivestita (piastra C), aggiungere 10 μl dello stesso intervallo di concentrazioni di Eu(III)-DO3AAm-CBP (in duplicato). Utilizzare la lettura della fluorescenza dell'Eu(III)-DO3AAm-CBP in soluzione per creare una curva di calibrazione.
NOTA: Non lavare o aspirare la soluzione da questa piastra.
- Estrazione acida di europio (III) e letture di fluorescenza risolta nel tempo (TRF)
NOTA: Fare riferimento alle informazioni supplementari sulla preparazione e la calibrazione dei volumi della soluzione acida (AS) e della soluzione tampone (BS). I volumi di AS e BS necessari per raggiungere in modo riproducibile un pH ottimale erano rispettivamente di 54 μl e 46 μl in questo lavoro. Eseguire le seguenti operazioni sulla piastra A, sulla piastra B e sulla piastra C.- Aggiungere 54 μL di soluzione acida (AS) a ciascun pozzetto e posizionare la piastra nell'incubatore a 37 °C per 90 minuti, coprendo le micropiastre con un foglio per evitare l'evaporazione. La temperatura e il tempo di incubazione devono essere attentamente controllati per ottenere una decomplessazione riproducibile.
- Aggiungere 46 μL di soluzione tampone (BS) a ciascun pozzetto e agitare delicatamente la piastra per 30 s.
- Aggiungere 100 μl di soluzione di potenziamento (ES) e agitare la piastra per 30 s.
- Attendere 30 minuti prima di leggere la targa utilizzando un lettore di piastre TRF. Utilizzare i parametri elencati nella Tabella 2.
4. Analisi dei dati
- Quantificazione della concentrazione di collagene immobilizzato sui pozzetti
- Ottenere l'equazione della curva di calibrazione dell'A560 (AU) rispetto alla concentrazione (μg/mL) delle 11 soluzioni standard.
- Utilizzare le letture dell'assorbanza acquisite dai pozzetti contenenti gli standard di collagene.
- Tabulare i valori medi dei pozzetti duplicati e tracciare l'assorbanza media rispetto alle concentrazioni note di proteine (collagene) (μg/mL) per ottenere l'equazione per la curva standard.
- Utilizzare i valori di assorbanza per calcolare la massa (μg) e la concentrazione (M) del collagene immobilizzato.
- Calcola i valori medi di assorbanza nei tre pozzetti che contenevano collagene immobilizzato e registra la deviazione standard.
- Utilizzare l'equazione della curva standard ottenuta dalla curva standard del collagene (passaggio 2.2.6) per convertire l'assorbanza misurata dai pozzetti rivestiti di collagene in concertazione. Da questo, calcolare la concentrazione di collagene che è stato immobilizzato all'interno dei pozzetti sperimentali in μg/mL.
- Convertire la concentrazione calcolata al punto 4.1.2.2 (μg/mL) prima in grammi/litro e poi, in base al peso molecolare del collagene, in molare (M).
- Infine, calcolare la massa del collagene immobilizzato in ciascun pozzetto dividendo la concentrazione per il volume di collagene aggiunto al pozzetto (100 μL per il collagene di tipo I e 70 μL per il collagene di tipo III).
- Ottenere l'equazione della curva di calibrazione dell'A560 (AU) rispetto alla concentrazione (μg/mL) delle 11 soluzioni standard.
- Calcolo della costante di dissociazione (Kd) (Figura 4)
- Estrarre le letture della fluorescenza.
- Esporta le letture della fluorescenza dal lettore di piastre in un foglio di calcolo.
NOTA: Nei saggi di legame, è importante tenere conto del potenziale legame non specifico di un composto alla superficie plastica delle piastre. - Calcolare i valori medi delle misurazioni in triplicato da ciascuna concentrazione di composti per le tre diverse piastre: le letture di legame specifico dai pozzetti rivestiti (Piastra A), il legame non specifico dai pozzetti non rivestiti (Piastra B) e l'Eu(III)-DO3AAm-CBP totale in soluzione nei pozzetti non rivestiti (Piastra C).
- Determinare i valori di fluorescenza per il composto legato sottraendo le letture di fluorescenza dei pozzetti non rivestiti (piastra B) da quelle dei pozzetti rivestiti (piastra A).
Equazione 1: Determinazione della fluorescenza legata17:
Fluorescenza legata = Specifico (pozzetti rivestiti) - Non specifico (pozzetti non rivestiti) - Generare una curva di calibrazione utilizzando le letture del composto marcato con Eu(III) in soluzione (Piastra C). Tracciare le letture di fluorescenza ottenute rispetto alla concentrazione del composto marcato con Eu(III). Eseguire un adattamento di regressione lineare.
- Esporta le letture della fluorescenza dal lettore di piastre in un foglio di calcolo.
- Convertire le letture della fluorescenza in concentrazioni.
- Convertire le letture della fluorescenza legata (passaggio 4.2.3) in concentrazione utilizzando la curva di fluorescenza standard dai dati generati utilizzando le concentrazioni di composti in soluzione (passaggio 4.2.1.4).
NOTA: Quando si confrontano le proprietà di legame di un composto verso diverse proteine bersaglio che immobilizzano a diverse concentrazioni, quest'ultima dovrà essere considerata quando si calcola la quantità di composto legato al bersaglio (cioè, composto/proteina legata). - Dividi la concentrazione del composto legato per la concentrazione della proteina immobilizzata nel pozzetto.
NOTA: Per questo calcolo, utilizzare la concentrazione di collagene immobilizzato che è stata calcolata dopo che i pozzetti sono stati incubati con PBS per 1 ora (il cosiddetto esperimento mimico PBS; sezione 2.1 sopra). Questo per tenere conto delle potenziali perdite di collagene durante la fase di incubazione e la fase di lavaggio che non contribuiranno al segnale di fluorescenza finale. - Traccia i dati utilizzando un grafico a dispersione che ha le concentrazioni del composto sull'asse x (μM) e il composto/proteina legato sull'asse y.
- Convertire le letture della fluorescenza legata (passaggio 4.2.3) in concentrazione utilizzando la curva di fluorescenza standard dai dati generati utilizzando le concentrazioni di composti in soluzione (passaggio 4.2.1.4).
- Ottenere i valoriK d .
- Adattare i dati acquisiti nel passaggio 4.2.2.3 utilizzando due possibili modelli cinetici di legame: legame in un sito e legame in un sito con una pendenza collinare. Le equazioni per ciascun modello sono mostrate nella Figura 6.
- Scegliere il modello che fornisce un adattamento non ambiguo con il valore R al quadrato più alto durante l'adattamento dei dati.
- Escludere i valori anomali per ciascuna serie di letture di fluorescenza per concentrazione per piastra.
- Calcola il valore finaledi K d e presenta i dati come media ± deviazione standard di esperimenti indipendenti.
NOTA: Per ottenere risultati affidabili, eseguire misurazioni in triplice copia all'interno di ciascuna piastra e almeno tre esperimenti indipendenti con diverse micropiastre.
- Calcola l'occupazione frazionaria (FO).
NOTA: Dall'equazione 2, la concentrazione dell'obiettivo è sconosciuta, e, quindi, usando l'algebra e il Kd, dall'equazione 3, si ottiene un'equazione praticabile per calcolare l'occupazione frazionaria sotto forma di equazione 4.
Equazione 2: Definizione di occupazione frazionaria17:
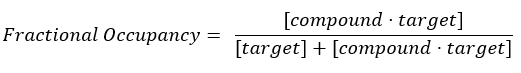
Equazione 3: La costante di dissociazione, Kd, che è la concentrazione alla quale il composto occupa il 50% del bersaglio all'equilibrio17:
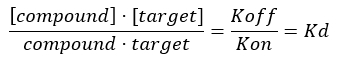
Equazione 4: Equazione riarrangiata per calcolare l'equazione FO17:
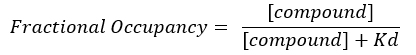
- Calcolare il FO utilizzando i valori indipendenti di Kd ottenuti per ogni singola piastra. Tracciare i risultati, la media e le deviazioni standard del FO rispetto alla concentrazione del composto.
- Riportare il FO con valori compresi tra 0 e 1 o in percentuale con valori compresi tra 0% e 100%.
- Estrarre le letture della fluorescenza.
Access restricted. Please log in or start a trial to view this content.
Risultati
Valutazione della stabilità e della concentrazione del collagene di tipo I e III immobilizzato in gel/film
La quantificazione della concentrazione di collagene immobilizzato per pozzetto è stata effettuata utilizzando tre diverse condizioni: a) in pozzetti senza lavaggio con PBS dopo immobilizzazione delle proteine (no wash); b) in pozzetti con una fase di lavaggio (due volte con PBS) dopo l'immobilizzazione per rimuovere qualsiasi proteina non rivestita; c) in pozz...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo lavoro presenta un metodo riproducibile per immobilizzare il collagene di tipo I e di tipo III. Dimostra inoltre un protocollo per l'acquisizione, l'analisi e l'interpretazione dei dati di legame TRF Eu(III) in vitro per caratterizzare le proprietà di legame di un ligando candidato verso il collagene di tipo I e III. I protocolli per immobilizzare il collagene di tipo I e di tipo III qui presentati sono stati sviluppati e ottimizzati considerando il lavoro precedentement...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Siamo grati ai seguenti finanziatori per aver sostenuto questo lavoro: (1) il Medical Research Council del Regno Unito (MR/N013700/1) e il King's College di Londra, membro della MRC Doctoral Training Partnership in Biomedical Sciences; (2) Sovvenzione del programma BHF RG/20/1/34802; (3) Sovvenzione per il progetto BHF PG/2019/34897; (4) Sovvenzione del King's BHF Centre for Research Excellence RE/18/2/34213; (5) il programma ANID Millennium Science Initiative - ICN2021_004; e (6) FB210024 di sovvenzione basale ANID.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| Ice | |||
| Ice | |||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH meter (sevenCompact) | Mettler Toledo | ||
| pH meter (sevenCompact) | Mettler Toledo | ||
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Tissue culture incubator (37 °C, 5% CO2) | |||
| Type I bovine collagen, 3 mg/mL | Corning | 354231 | |
| Type III human placenta collagen, 0.99 mg/mL | Advanced Biomatrix | 5021 |
Riferimenti
- Distler, J. H. W., et al. Review: Frontiers of antifibrotic therapy in systemic sclerosis. Arthritis and Rheumatology. 69 (2), 257-267 (2017).
- Wynn, T. A. Fibrotic disease and the TH1/TH2 paradigm. Nature Reviews Immunology. 4 (8), 583-594 (2004).
- Saha, P., et al. Magnetic resonance T1 relaxation time of venous thrombus is determined by iron processing and predicts susceptibility to lysis. Circulation. 128 (7), 729-736 (2013).
- Mirshahi, M., et al. Defective thrombolysis due to collagen incorporation in fibrin clots. Thrombosis Research. 8, 73-80 (1988).
- Comerota, A. J. The ATTRACT trial: Rationale for early intervention for iliofemoral DVT. Perspectives in Vascular Surgery and Endovascular Therapy. 21 (4), 221-225 (2009).
- Bateman, E. D., Turner-Warwick, M., Adelmann-Grill, B. C. Immunohistochemical study of collagen types in human foetal lung and fibrotic lung disease. Thorax. 36 (9), 645-653 (1981).
- Pawelec, K. M., Best, S. M., Cameron, R. E. Collagen: A network for regenerative medicine. Journal of Materials Chemistry B. 4 (40), 6484-6496 (2016).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Copes, F., Pien, N., Van Vlierberghe, S., Boccafoschi, F., Mantovani, D. Collagen-based tissue engineering strategies for vascular medicine. Frontiers in Bioengineering and Biotechnology. 7, 166(2019).
- Veis, A. The biochemistry of collagen. Annals of Clinical and Laboratory Science. 5 (2), 123-131 (1975).
- Bielajew, B. K., Hu, J. C., Athanasiou, K. A. Collagen: Quantification, biomechanics, and role of minor subtypes in cartilage. Nature Reviews Materials. 5, 730-747 (2020).
- Zhao, Z., et al. Structural and functional plasticity of collagen fibrils. DNA and Cell Biology. 38 (4), 367-373 (2019).
- Eryilmaz, E., Teizer, W., Hwang, W. In vitro analysis of the co-assembly of type-I and type-III collagen. Cellular and Molecular Bioengineering. 10 (1), 41-53 (2017).
- Jagnow, J., Clegg, S. Klebsiella pneumoniae MrkD-mediated biofilm formation on extracellular matrix- and collagen-coated surfaces. Microbiology. 149 (9), 2397-2405 (2003).
- O'Sullivan, D., O'Neill, L., Bourke, P. Direct plasma deposition of collagen on 96-well polystyrene plates for cell culture. ACS Omega. 5 (39), 25069-25076 (2020).
- Caravan, P., et al. Collagen-targeted MRI contrast agent for molecular imaging of fibrosis. Angewandte Chemie - International Edition. 46 (43), 8171-8173 (2007).
- Copeland, R. A. Enzymes: A Practical Introduction to Structure, Mechanism, and Data Analysis. , John Wiley & Sons, Ltd. Hoboken, NJ. (2000).
- Salahudeen, M. S., Nishtala, P. S. An overview of pharmacodynamic modelling, ligand-binding approach and its application in clinical practice. Saudi Pharmaceutical Journal. 25 (2), 165-175 (2017).
- Bünzli, J. C. G., Piguet, C. Taking advantage of luminescent lanthanide ions. Chemical Society Reviews. 34 (12), 1048-1077 (2005).
- Hemmilii, I. Luminescent lanthanide chelates - A way to more sensitive diagnostic methods. Journal of Alloys and Compounds. 225 (1-2), 480-485 (1995).
- De Silva, C. R., Vagner, J., Lynch, R., Gillies, R. J., Hruby, V. J. Optimization of time-resolved fluorescence assay for detection of europium-tetraazacyclododecyltetraacetic acid-labeled ligand-receptor interactions. Analytical Biochemistry. 398 (1), 15-23 (2010).
- Digilio, G., Lacerda, S., Lavin Plaza, B., Phinikaridou, A. Extracellular matrix targeted MRI probes. Analysis & Sensing. 3 (1), (2022).
- Phinikaridou, A., et al. Tropoelastin: A novel marker for plaque progression and instability. Circulation. Cardiovascular imaging. 11 (8), 007303(2018).
- Guzaeva, T. V., et al. Protein A used in DELFIA for the determination of specific antibodies. Immunology Letters. 35 (3), 285-289 (1993).
- Nasiri, A. H., Nasiri, H. R. Polymerase assays for lead discovery: An overall review of methodologies and approaches. Analytical Biochemistry. 563, 40-50 (2018).
- Capuana, F., et al. Imaging of dysfunctional elastogenesis in atherosclerosis using an improved gadolinium-based tetrameric MRI probe targeted to tropoelastin. Journal of Medicinal Chemistry. 64 (20), 15250-15261 (2021).
- Drescher, D. G., Drescher, M. J., Ramakrishnan, N. A. Surface plasmon resonance (SPR) analysis of binding interactions of proteins in inner-ear sensory epithelia. Methods in Molecular Biology. 493, 323-343 (2009).
- Murali, S., Rustandi, R. R., Zheng, X., Payne, A., Shang, L. Applications of surface plasmon resonance and biolayer interferometry for virus-ligand binding. Viruses. 14 (4), 717(2022).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon