Method Article
Editing genetico delle cellule B del macaco rhesus primario
In questo articolo
Riepilogo
Presentiamo un metodo per la coltura e l'editing genetico delle cellule B primarie del macaco rhesus utilizzando CRISPR / Cas9 e il sierotipo 6 del virus adeno-associato ricombinante per lo studio delle terapie con cellule B.
Abstract
Le cellule B e la loro progenie sono le fonti di anticorpi altamente espressi. Le loro elevate capacità di espressione proteica insieme alla loro abbondanza, alla facile accessibilità tramite sangue periferico e alla possibilità di semplici trasferimenti adottivi li hanno resi un bersaglio attraente per gli approcci di editing genico per esprimere anticorpi ricombinanti o altre proteine terapeutiche. L'editing genetico delle cellule B primarie di topo e umane è efficiente e i modelli murini per gli studi in vivo hanno mostrato risultati promettenti, ma la fattibilità e la scalabilità per modelli animali più grandi non sono state finora dimostrate. Pertanto, abbiamo sviluppato un protocollo per modificare le cellule B primarie del macaco rhesus in vitro per consentire tali studi. Riportiamo le condizioni per la coltura in vitro e l'editing genetico di cellule B primarie di macaco rhesus da cellule mononucleate del sangue periferico o splenociti utilizzando CRISPR / Cas9. Per ottenere l'integrazione mirata di cassette di grandi dimensioni (<4,5 kb), è stato incluso un protocollo rapido ed efficiente per la preparazione del sierotipo 6 del virus adeno-associato ricombinante come modello di riparazione diretto dall'omologia utilizzando un vettore di supporto adenovirale autosilenziante abilitato alla tetraciclina. Questi protocolli consentono lo studio di terapie prospettiche a cellule B nei macachi rhesus.
Introduzione
Le cellule B sono il fondamento dell'immunità umorale. Dopo l'attivazione da parte dell'antigene affine e dei segnali secondari, le cellule B naïve danno origine alle cellule B del centro germinale, alle cellule B di memoria e alle plasmacellule1. Quest'ultimo è la fonte degli anticorpi secreti che mediano le funzioni protettive della maggior parte dei vaccini attualmente disponibili2. Le plasmacellule sono state descritte come fabbriche di anticorpi in quanto secernono grandi quantità di anticorpi nel siero, circa 2 ng / giorno / cellula3, pari a 7-16 g / L siero, rendendo gli anticorpi una delle tre proteine più abbondanti nel siero4. Le cellule B sono abbondanti nel sangue e possono, quindi, essere facilmente ottenute e reinfuse in un individuo.
Questi tratti hanno reso le cellule B un bersaglio degli sforzi della terapia cellulare per modificare geneticamente il recettore delle cellule B (BCR) ed esprimere anticorpi ampiamente neutralizzanti (bNAbs) al virus dell'immunodeficienza umana (HIV)5,6,7,8,9,10,11,12,13,14,15 e altre proteine 16, 17,18,19,20,21. Tali approcci hanno mostrato il potenziale in numerosi studi sui topi in vivo 7,8,10,11,16,22. Tuttavia, diversi ostacoli devono ancora essere superati per la traduzione clinica 9,15,23, tra cui la sicurezza, la durata e l'entità dell'efficacia terapeutica, nonché il ridimensionamento ad animali più grandi come i primati non umani (NHP). Infatti, gli NHP, e in particolare i macachi rhesus, che hanno una lunga storia nella ricerca sugli anticorpi e sull'HIV24,25, sono il modello più adatto per testare questi parametri.
Qui, abbiamo sviluppato protocolli che consentono di affrontare questi problemi. Ad oggi, pochi studi hanno tentato di coltivare cellule B di macaco rhesus ex vivo, e solo una selezione positiva utilizzando CD20 è stata riportata per la purificazione delle cellule B del macaco rhesus26,27,28. Abbiamo stabilito un protocollo per l'isolamento delle cellule B del macaco rhesus intatte mediante deplezione negativa di altri tipi di cellule. Inoltre, sono definite condizioni di coltura per l'editing genetico mirato delle cellule B del macaco rhesus. Questo protocollo delinea l'uso delle ribonucleoproteine (RNP) CRISPR / Cas9 e del sierotipo 6 del virus adeno-associato ricombinante (rAAV6) come modello di riparazione diretto dall'omologia (HDRT) per modificare le cellule B del macaco rhesus in coltura di genetica. Utilizzando questo protocollo, sono state raggiunte efficienze di editing fino al 40% con inserti di grandi dimensioni (~ 1,5 kb). Presentiamo anche un metodo rapido ed economico per produrre rAAV6 utilizzando un helper adenovirale29 abilitato alla tetraciclina e autosilenziante per consentire il test rapido degli HDRT in questo formato. Combinati, questi protocolli descrivono un flusso di lavoro efficiente per l'editing genetico delle cellule B del macaco rhesus (Figura 1), consentendo la valutazione delle terapie con cellule B in un modello NHP.
Per iniziare gli esperimenti, il materiale donatore può essere ordinato da fonti commerciali o ottenuto mediante flebotomie o splenectomia. In questo studio, le flebotomie e le raccolte di sangue sono state eseguite come descritto in precedenza30 utilizzando l'anticoagulante EDTA. Per ottenere cellule B di macaco rhesus primitivo splenico, sono state eseguite splenectomie parziali (25% -50%) o totali utilizzando tecniche riportate in precedenza31. Gli animali sono stati sottoposti a digiuno durante la notte prima dell'intervento. In breve, durante l'intervento, l'addome è stato tagliato e preparato con scrub alternati di clorexidina e alcool isopropilico al 70% tre volte. Un'incisione (5-10 cm) è stata fatta nell'addome per identificare e isolare la milza. La vascolarizzazione della milza era legata con punti di sutura o morsetti vascolari. L'incisione è stata chiusa in due strati con suture di polidiossanone 4-0 PDS. La splenectomia è stata eseguita una sola volta per un singolo animale. Le sospensioni monocellulari sono state preparate dalla milza del macaco mediante macerazione attraverso filtri cellulari. Le cellule mononucleate del sangue e delle sospensioni di cellule spleniche sono state preparate utilizzando la centrifugazione a gradiente di densità e conservate in azoto liquido.
Protocollo
Tutte le procedure e gli esperimenti sugli animali sono stati eseguiti secondo i protocolli approvati dal Comitato istituzionale per la cura e l'uso degli animali dell'Istituto nazionale di allergie e malattie infettive, National Institutes of Health. Un riepilogo dei seguenti protocolli è presentato nella Figura 1. Macachi rhesus maschi e femmine (Macaca mulatta) di origine genetica indiana di età compresa tra 2 e 8 anni sono stati ospitati e curati in conformità con le linee guida del Comitato per la cura e l'uso degli animali da laboratorio in una struttura di livello di biosicurezza 2.
ATTENZIONE: Tutti gli esperimenti sono stati eseguiti in conformità con le precauzioni universali per i patogeni trasmessi per via ematica, con tecniche sterili / asettiche e adeguate apparecchiature di livello di biosicurezza 2 in cappe a flusso laminare.
1. Produzione di rAAV6
- Preparare i reagenti per la produzione di rAAV6.
- Progettare e clonare il modello di riparazione diretto dall'omologia tra le ripetizioni terminali invertite (ITR) di AAV2 nel vettore pAAV utilizzando tecniche standard. Assicurarsi che i bracci di omologia siano di almeno ~ 250 bp su entrambi i lati, ma un minimo di 60 bp può essere sufficiente, anche se i bracci di omologia più lunghi sono preferiti se il design del costrutto lo consente. Se le sequenze bersaglio di uno qualsiasi degli sgRNA utilizzati sono presenti nell'HDRT, rimuoverle usando mutazioni silenti, che sono più efficaci nel motivo adiacente del protospaziatore o nella regione del seme del sito bersaglio.
NOTA: È possibile eseguire la sintesi genica combinata con l'assemblaggio Gibson per una clonazione efficiente32. Preparare un Maxiprep di un clone corretto per la trasfezione. Per la progettazione di sgRNA, CHOPCHOP33 è raccomandato e un elenco di altri strumenti può essere trovato su https://zlab.bio/guide-design-resources. La capacità massima di packaging per AAV inclusi ITR è ~4.7 kb. AAV6 è il sierotipo più comunemente usato per l'editing delle cellule ematopoietiche, in particolare le cellule B9. Altri sierotipi di AAV per l'editing genico delle cellule B del macaco rhesus non sono stati testati, ma AAV28 e AAV-DJ10,11 sono stati utilizzati negli studi sui topi. - Preparare il terreno di coltura 293AAV e il terreno di produzione secondo la Tabella 1 e la Tabella 2. Filtro sterile attraverso un'unità filtrante a membrana in polietersulfone (PES) da 0,2 μm. Conservare a 4 °C.
- Preparare 1x soluzione di polietilenimina (PEI) (1 mg/ml, 100 ml).
- In un becher di vetro da 250 ml, riscaldare ~ 70 mL di H2O in un forno a microonde per ~30 s, quindi aggiungere 100 mg di PEI. Aggiungere un agitatore magnetico e mescolare fino a quando il PEI è in gran parte sciolto.
- Regolare il pH a 7 con 1 M HCl, quindi rabboccare fino a 100 ml con H2O, attendere 10 minuti, controllare nuovamente il pH e regolare se necessario.
- Filtrare sterile la soluzione PEI attraverso un'unità filtrante a membrana PES da 0,2 μm, aliquota e conservare -20 °C. Dopo lo scongelamento, la soluzione può essere conservata a 4 °C per un massimo di 2 mesi.
- Preparare una soluzione 5x di polietilenglicole (PEG)/NaCl.
- Pesare 400 g di PEG 8.000 e 24 g di NaCl.
- Aggiungere un agitatore magnetico a un becher di vetro da 2 litri, aggiungere il PEG 8.000 pesato e NaCl e risciacquare con ~ 550 ml di acqua deionizzata.
- Mescolare con il riscaldamento e portare a ebollizione o 80-90 °C fino a completa dissoluzione.
- Regolare il pH a ~ 7,4 con 1 M NaOH, quindi regolare il volume a 1 L utilizzando un cilindro graduato e trasferirlo in una bottiglia di vetro da 2 L con l'agitatore magnetico.
- Autoclavare il flacone, l'agitatore magnetico e la soluzione a bagnomaria per 30 minuti a 121 °C.
- Dopo l'autoclave, raffreddare la soluzione in una cella frigorifera mescolando utilizzando l'agitatore magnetico per evitare la separazione in diverse fasi. Aliquot se necessario, e conservare a 4 °C.
- Preparare il buffer di formulazione.
- Mescolare 500 mL di DPBS con 50 μL di 10% Pluronic F-68. Filtrare sterile attraverso un'unità filtrante a membrana PES da 0,2 μm e conservare a temperatura ambiente (RT).
- Progettare e clonare il modello di riparazione diretto dall'omologia tra le ripetizioni terminali invertite (ITR) di AAV2 nel vettore pAAV utilizzando tecniche standard. Assicurarsi che i bracci di omologia siano di almeno ~ 250 bp su entrambi i lati, ma un minimo di 60 bp può essere sufficiente, anche se i bracci di omologia più lunghi sono preferiti se il design del costrutto lo consente. Se le sequenze bersaglio di uno qualsiasi degli sgRNA utilizzati sono presenti nell'HDRT, rimuoverle usando mutazioni silenti, che sono più efficaci nel motivo adiacente del protospaziatore o nella regione del seme del sito bersaglio.
- Coltura cellulare, trasfezione e trasduzione per la produzione di rAAV6
- Scongelare, coltivare e congelare le cellule 293AAV come descritto dal produttore utilizzando il terreno di coltura 293AAV sopra riportato e Trypsin-EDTA per la scissione. Si raccomanda di congelare alcuni passaggi precoci e utilizzare le cellule per la produzione di AAV prima che raggiungano il passaggio 40.
- Per la produzione di rAAV6, seminare quattro piatti di coltura cellulare da 15 cm con 5 x 106 cellule in 30 ml ciascuno. Le cellule sono pronte per la trasfezione di solito 1-2 giorni dopo la semina quando raggiungono l'80% -90% di confluenza.
- Scongelare un Maxiprep di plasmide pAAV contenente l'HDRT da confezionare in AAV6. Risospendere 85,6 μg del plasmide pAAV in 3 ml di terreno DMEM puro.
- Sciogliere 342 μL di soluzione PEI da 1 mg/mL in 3 mL di mezzo DMEM puro. Incubare entrambe le soluzioni per 10 minuti a RT.
- Mescolare entrambi i tubi da 3 ml in un tubo di ~ 6,4 ml di miscela di trasfezione e incubare per 20 minuti a RT.
- Nel frattempo, scongelare il vettore helper autosilenziante abilitato alla tetraciclina RepCap6 dal congelatore a -80 °C in un bagno d'acqua a 37 °C. Per trasdurre le cellule 293AAV, aggiungere il vettore helper ad una molteplicità di infezione (MOI) di 25 utilizzando la dose infettiva mediana di coltura tissutale (TCID 50) e assumendo 1,15 x10 7 cellule/piatto; tipicamente, vengono utilizzati 2-10 μL per piatto di 15 cm. Oscillare e far roteare delicatamente i piatti per distribuire.
- Dopo l'incubazione della miscela di trasfezione, aggiungerne 1,6 ml a goccia su ciascuno dei quattro piatti da 15 cm. Incubare a 37 °C e 5% di CO2 durante la notte.
NOTA: In alternativa, se i vettori rAAV6 di interesse sono già disponibili, questi vettori possono essere utilizzati per fornire il genoma virale da impacchettare, il che annulla la necessità di plasmidi con questo sistema e produce titoli rAAV6 comparabili. Per questo approccio, le cellule 293AAV sono co-trasdotte con il desiderato rAAV6 ad un MOI di 50 (basato su copie del genoma rAAV6 [GC]/mL) insieme al vettore helper. - Il giorno successivo, aspirare e scartare con cura il terreno di coltura e sostituirlo con 30 ml di terreno di produzione preriscaldato. Incubare per altre 96 ore prima della raccolta. Non si raccomanda alcun ulteriore cambiamento del mezzo per massimizzare i rendimenti.
- Raccolta e purificazione dell'AAV6 ricombinante dal substrato
- Senza rimuovere le cellule dal piatto, raccogliere tutto il surnatante cellulare in un'unità filtrante con una membrana PES di 0,2 μm superiore di almeno il 50% al volume di terreno da filtrare. Quindi, filtrare il surnatante.
NOTA: Se si desidera una maggiore resa di rAAV6, le cellule possono essere raccolte e l'rAAV estratto dal pellet cellulare utilizzando kit commerciali o protocolli stabiliti34,35. Poiché AAV6 è per lo più secreto nel mezzo36, è stato utilizzato solo il surnatante che riduce il lavoro, i costi e il tempo. - Aggiungere 5x soluzione PEG/NaCl al surnatante filtrato al 25% del volume raccolto; questo è tipicamente 30 ml se vengono utilizzati quattro piatti da 15 cm da 30 ml.
- Mescolare bene invertendo e quindi incubare per una notte a 4 ° C per precipitare le particelle virali.
NOTA: le particelle AAV sono stabili fino a 2 giorni in questa soluzione. - Preraffreddare una centrifuga a secchio oscillante con 250 ml di inserti per tubi a 4 °C. Preparare un'unità filtrante centrifuga da 4 mL con un cutoff di 100 kDa e un filtro a siringa in PES idrofilo da 0,22 μm pretrattando ogni membrana con 2 mL di F-68 ploronico al 10% per almeno 1 ora a RT.
- Trasferire la miscela AAV-PEG/NaCl in una provetta da 250 ml, centrifugare a 2.500 x g per 1 ora a 4 °C, quindi rimuovere accuratamente l'intero surnatante mediante aspirazione.
- Risospendere il pellet virale da beige a bianco vorticando in 4 ml di HEPES 1 M fino a completa risospensione. Se necessario, lasciare riposare per 5 minuti e vortice di nuovo. Risospendere utilizzando una pipetta sierologica da 5 mL e trasferire il volume totale in una provetta da 15 ml.
- In una cappa aspirante, aggiungere un volume uguale di cloroformio alla sospensione del virus, in genere 4 ml.
- Vortice vigoroso per 2 minuti, quindi centrifugare a 1.000 x g per 5 minuti a RT.
- Raccogliere lo strato superiore (supernatante contenente AAV) in un nuovo tubo da 50 ml ed eliminare lo strato inferiore (cloroformio).
ATTENZIONE: Le soluzioni contenenti cloroformio sono rifiuti pericolosi. Seguire le linee guida istituzionali per il suo smaltimento. - Posizionare il surnatante contenente AAV sotto una cappa aspirante e lasciare evaporare il cloroformio rimanente per 30 minuti.
- Nel frattempo, lavare l'unità filtrante centrifuga pretrattata e il filtro a siringa.
- Aggiungere 1,5 mL di tampone di formulazione all'unità filtrante centrifuga pretrattata. Centrifugare a 3.500 x g per 10 minuti a 15 °C in un rotore a benna oscillante. Ripetere questo passaggio con 4 ml di tampone di formulazione per lavare la membrana.
- Risciacquare il filtro della siringa due volte con 5 mL di tampone di formulazione utilizzando una siringa da 5 ml.
- Caricare ~4 mL di surnatante contenente AAV dall'estrazione del cloroformio in una siringa da 5 ml, collegare il filtro della siringa lavata e filtrare direttamente nell'unità filtrante centrifuga.
- Centrifugare a 3.500 x g per 25 minuti a 15 °C, quindi confermare che la soluzione AAV nel filtro è compresa tra 50-100 μL. Se il volume della soluzione è >100 μL, continuare a centrifugare.
- Dopo aver rimosso il filtrato, aggiungere 4 mL di tampone di formulazione all'interno della tazza dell'unità filtrante centrifuga e miscelare uniformemente la soluzione mediante pipettaggio. Centrifugare a 3.500 x g per 25 minuti a 15 °C, quindi confermare che la soluzione AAV nel filtro è compresa tra 50-100 μL. Se il volume della soluzione è >100 μL, continuare a centrifugare. Ripetere questo passaggio per un altro lavaggio.
- Dopo la centrifugazione finale, confermare che il volume della soluzione è 50-70 μL; In caso contrario, continuare a centrifugare. Trasferire il preparato in una provetta da 1,5 ml. Aliquot se lo si desidera, e conservare a -80 °C.
- Senza rimuovere le cellule dal piatto, raccogliere tutto il surnatante cellulare in un'unità filtrante con una membrana PES di 0,2 μm superiore di almeno il 50% al volume di terreno da filtrare. Quindi, filtrare il surnatante.
- Determinazione del titolo AAV6 ricombinante mediante qPCR
NOTA: I primer qPCR ricottura nella regione ITR e dovrebbero, quindi, essere adatti a tutti i costrutti clonati in pAAV.- Scongelare un'aliquota del vaa6 da titolare e un'aliquota del materiale di riferimento AAV6. Il materiale di riferimento AAV6 deve essere vicino a 4 x 1011 GC/ml; In caso contrario, regolare le diluizioni di conseguenza.
- Eseguire un digesto DNasi I per rimuovere qualsiasi DNA plasmidico libero residuo nella preparazione di rAAV6 combinando 2,0 μL del campione o del materiale di riferimento AAV6 con 15,6 μL di H 2 O privo di nucleasi,2,0μL di tampone 10x DNasi I e 0,4 μL di DNasi I.
- Mescolare delicatamente e incubare per 30 minuti a 37 °C, quindi trasferire sul ghiaccio. Si tratta della diluizione 1 (cfr. tabella 3).
- Preparare con acqua diluizioni seriali quintuplicate di tutti i campioni e del materiale di riferimento AAV6 come indicato nella tabella 3 .
- Preparare un master mix SYBR Green qPCR. Per pozzetto, miscelare 4,7 μL di acqua priva di nucleasi con 10 μL di master mix SYBR Green, 0,15 μL di primer ITR in avanti a 100 μM e 0,15 μL di primer ITR reverse a 100 μM.
NOTA: Ogni campione viene misurato in duplicato, con 16 pozzetti per lo standard di riferimento, 8 pozzi per campione e 2 pozzetti per un controllo senza modello. Preparare il 10% in più di master mix per tenere conto dell'errore di pipettaggio. - In una piastra di reazione ottica a 96 pozzetti o 384 pozzetti, caricare 15 μL/pozzetto della miscela master qPCR SYBR Green.
- Quindi, caricare 5 μL di campioni e materiale di riferimento AAV6 o acqua priva di nucleasi per il controllo senza modello. Per lo standard di riferimento AAV6, diluizione del carico da 2 a diluizione 9. Per i campioni, caricare la diluizione da 5 a 8. Misurare ogni diluizione in duplice copia. Evita le bolle.
- Sigillare la piastra caricata con pellicola ottica trasparente, centrifugare a 800 x g per 1 minuto a RT e caricare la piastra nello strumento qPCR con la configurazione appropriata a 96 pozzetti o 384 pozzetti.
- Impostare ed eseguire lo strumento qPCR utilizzando il rilevamento SYBR con le seguenti condizioni di ciclo: 98 °C per 3 minuti, poi 40 cicli di 98 °C per 15 s e 58 °C per 30 s, seguiti da una curva di fusione.
- Analizzare i dati con il software dello strumento utilizzando la concentrazione del materiale di riferimento AAV6 in copie del genoma per millimetro (GC/mL) come curva standard (vedere Tabella 3). Calcolare la concentrazione finale del campione moltiplicando per il fattore di diluizione.
- Assicurarsi che la curva standard R2 sia vicina a 1,0, che l'efficienza della PCR sia del 90%-110%, che la linea di base sia stata rimossa, che la curva di fusione mostri un singolo picco, che i valori di C t cambino in base alle diluizioni e che i duplicati siano entro 0,5 Ct; In caso contrario, escludere i valori anomali. Aspettatevi rendimenti come nella Figura 2.
2. Preparazione dei mezzi e degli stimoli delle cellule B
- Preparare il mezzo di scongelamento: combinare RPMI-1640 con FCS al 20%. Filtro sterile attraverso un'unità filtrante a membrana PES da 0,2 μm. Conservare a 4 °C.
- Preparare il terreno di coltura delle cellule B: combinare i reagenti nella Tabella 4 e quindi filtrare sterile attraverso un'unità filtrante a membrana PES da 0,2 μm. Conservare a 4 °C.
- Risospendere ciascuno degli stimolanti delle cellule B nella Tabella 5 alle concentrazioni di stock nel terreno di coltura di cellule B, ad eccezione di CpG ODN che deve essere risospeso in acqua priva di nucleasi. Conservare a -80 °C.
- Se si esegue deplezione negativa delle cellule non B (fase 4 facoltativa), preparare DPBS (senza calcio, senza magnesio) con FCS al 2% (DPBS al 2% FCS). Filtro sterile attraverso un'unità filtrante a membrana PES da 0,2 μm. Conservare a 4 °C.
3. Preparazione e coltura di cellule B di macaco rhesus
NOTA: Le PBMC o gli splenociti del macaco rhesus crioconservati vengono utilizzati per impostare la coltura cellulare30,31.
- Terreno di scongelamento precaldo e terreno di coltura a cellule B in bagnomaria a 37 °C. Scongelare gli stimolanti delle cellule B dalla Tabella 5 sul ghiaccio.
- Preparare un tubo di dimensioni appropriate contenente un mezzo di scongelamento preriscaldato. Questo dovrebbe idealmente essere più di 10 volte il volume delle cellule scongelate.
- Scongelare uno o due crioviali di PBMC o splenociti alla volta in bagnomaria a 37 °C e decantare nel tubo preparato con mezzo preriscaldato. Risciacquare i criotubi per raccogliere tutte le cellule.
- Centrifugare le cellule a 200 x g per 10 minuti a RT.
NOTA: Queste impostazioni di centrifugazione riducono la contaminazione piastrinica preservando le rese di PBMC. È possibile utilizzare velocità più elevate, ad esempio 350 x g per 5 minuti. - Risospendere le cellule in 10 ml di terreno di scongelamento per il lavaggio.
- Ripetere il punto 3.4 e il passaggio 3.5 per un totale di tre centrifugazioni per rimuovere il mezzo di congelamento. Dopo l'ultima centrifugazione, risospendere le cellule a una stima di ~ 5 x 106 cellule / ml in mezzo di coltura cellulare B.
NOTA: Il protocollo di cui sopra coltiva PBMC intere o preparati di splenociti con contaminazione da altre cellule. Se sono necessarie colture di cellule B più pure, anche se con rese totali di cellule B significativamente ridotte, continuare con la fase 4. Non sono state osservate differenze nell'efficienza di editing tra i due metodi. - Diluire un'aliquota di 10 μL di cellule secondo necessità con terreno di coltura di cellule B per il conteggio. Contare utilizzando un emocitometro e la colorazione blu tripano, combinando volumi uguali di cellule risospese e soluzione di tripano blu allo 0,4%.
- Regolare la concentrazione cellulare a 3 x 106 cellule / ml con terreno di coltura di cellule B in base alla conta cellulare. Quindi, aggiungere gli stimolanti delle cellule B alle loro concentrazioni finali secondo la Tabella 5 e mescolare.
- Trasferire le cellule in un piatto di coltura cellulare appropriato. Nel complesso, si raccomanda 0,6 x 10 6-0,7 x 106 celle / cm2. Incubare le cellule a 37 °C con il 5% di CO 2 per 48 ore ±2 ore.
4. Deplezione negativa facoltativa delle cellule non-B
NOTA: La resa e la purezza dipendono dalla percentuale di input delle cellule B tra le PBMC, che può differire drasticamente tra i singoli macachi rhesus27. Aspettatevi purezza dell'80%-95%, efficienza del 60% e 1 x 10 6-1,5 x 106 celle da 1 x 107 PBMC.
- Dopo l'ultimo lavaggio (fase 3.6), risospendere le cellule a 1 x 108 cellule/ml in DPBS 2% FCS e blocco Fc-umano diluito 1:200. Il conteggio delle cellule si basa sul numero di cellule scongelate.
- Incubare per 15 minuti su ghiaccio per bloccare i recettori Fc e quindi aggiungere gli anticorpi biotinilati nella Tabella 6. Incubare per altri 20 minuti sul ghiaccio.
- Rabbboccare il tubo con DPBS 2% FCS e centrifugare a 200 x g per 10 minuti a 4 °C.
- Risospendere le celle in DPBS 2% FCS all'80% del volume dal punto 4.1 (cioè 80 μL per 1 x 107 celle).
- Aggiungere sfere magnetiche di streptavidina alla sospensione cellulare al 20% del volume dal punto 4.1 (cioè 20 μL di perline per 1 x 107 celle).
- Incubare le cellule per 15 minuti sul ghiaccio e agitare di tanto in tanto.
- Nel frattempo, per 1 x 108 celle, preparare un separatore magnetico con una grande colonna di esaurimento magnetico e un filtro di pre-separazione. Risciacquare il filtro e la colonna di pre-separazione con 2 ml di DPBS 2% FCS per gravità e scartare il flusso passante. Installare una provetta di raccolta da 15 ml.
NOTA: L'uso di altre colonne come colonne di selezione positiva o altri sistemi di purificazione magnetica delle perline può ridurre drasticamente la purezza. - Dopo l'incubazione, rabboccare le cellule a 0,5 ml con DPBS 2% FCS se il volume è <0,5 mL. Se il volume è ≥0,5 ml, è sufficiente procedere.
- Caricare la sospensione della cella nel filtro di pre-separazione sulla colonna preparata e raccogliere il flusso nel tubo da 15 ml.
- Eluire due volte le cellule B arricchite non legate aggiungendo 1 mL di DPBS 2% FCS nel filtro di pre-separazione. Raccogliere le celle non legate nello stesso tubo per flusso di gravità.
NOTA: Un'ulteriore eluizione può aumentare marginalmente la resa. La purezza e l'efficienza possono essere valutate dalla citometria a flusso delle cellule di input, delle cellule arricchite e delle cellule trattenute sulla colonna. Per ottenere le celle trattenute sulla colonna, rimuovere la colonna dal magnete e sciacquare con 3 mL di DPBS 2% FCS utilizzando lo stantuffo fornito. Se lo si desidera, valutare la purezza mediante citometria a flusso come indicato nella Figura 3 utilizzando i reagenti nella Tabella 7. - Centrifugare le cellule B arricchite a 200 x g per 10 minuti a 4 °C.
- Risospendere le cellule a una stima di ~ 5 x 106 cellule / ml in terreno di coltura cellulare B e continuare al punto 3.7.
5. Editing genetico primario delle cellule B del macaco rhesus
- Dopo aver attivato le cellule B del macaco rhesus per 48 ore ± 2 ore, preparare i reagenti per l'elettroporazione e la trasduzione.
- DMSO preriscaldato, tampone duplex privo di nucleasi, buffer T e buffer E (kit di elettroporazione da 10 μL) o E2 (kit di elettroporazione da 100 μL) dal kit di elettroporazione a RT.
- Scongelare gli stimolanti delle cellule rAAV6 HDRT e B dalla Tabella 5 sul ghiaccio.
- Risospendere gli sgRNA di CRISPR-Cas9 a 100 μM in tampone duplex. Ricostituire per 10 minuti a RT e mescolare vorticando e sfarfallando. Mantenere gli sgRNA ricostituiti su ghiaccio fino all'uso. Conservare a -80 °C.
NOTA: Gli sgRNA CRISPR-Cas9 possono essere progettati con vari strumenti online (vedi 1.1.1) e possono variare drasticamente nella loro efficienza di taglio. Si consiglia di testare empiricamente l'efficienza di taglio utilizzando saggi come TIDE37 o ICE38. - Per elettroporazione da 10 μL, preparare 550 μL di terreno di coltura di cellule B con tutti gli stimolanti della Tabella 5 e aggiungere l'1% di DMSO. Scala i volumi di 10 volte per elettroporazioni da 100 μL. Opzionalmente, il 10% di questo mezzo può essere preparato senza antibiotico-antimicotico, che aumenta leggermente la vitalità cellulare dopo la trasfezione.
- Per elettroporazione da 10 μL, preparare un pozzetto di una piastra di coltura cellulare a 48 pozzetti con 50 μL di terreno di coltura cellulare B con stimolanti e senza antibiotico-antimicotico, se lo si utilizza. Per elettroporazioni da 100 μL, pipettare 500 μL nei pozzetti di una piastra a 6 pozzetti.
- Aggiungi rAAV6 HDRT al mezzo nei pozzetti, fino al 20% del volume nel pozzo. Puntare a MOI che vanno da 1 x 10 5-1 x 10 6 in base al numero di cellule per trasfezione (elettroporazione 10 μL: 5 x 10 5 cellule; 100 μL elettroporazione:5 x 106 cellule) e il GC nella preparazione rAAV6. Si raccomandano elevate concentrazioni di stock di rAAV6 da 5 x 1013 GC/ml a 5 x 1014 GC/ml per ottenere MOI elevati con volumi ridotti.
NOTA: MOI inferiori possono portare a una riduzione dell'efficienza di editing e MOI di 5 x 105 sono generalmente vicini alle massime efficienze di editing che abbiamo visto. Non è stata osservata un'influenza di MOI variabili sulla vitalità delle cellule B. Si consiglia di includere controlli senza rAAV6 HDRT, senza trasfezione RNP e senza entrambi. - Preriscaldare i piatti preparati e il terreno rimanente trasferendoli in un incubatore a 37 °C con il 5% di CO2.
- Per elettroporazione da 10 μL, preparare 1,15 μL di ribonucleoproteina (RNP): mescolare 0,4 μL di 61 μM Cas9 con 0,75 μL di 100 μM di sgRNA in tampone duplex. Preparare extra (si consiglia il 30% in più per una singola elettroporazione) a causa di errori di pipettaggio e per evitare bolle durante il caricamento delle punte di elettroporazione. Scala 10 volte per punte da 100 μL.
- Incubare l'RNP per almeno 15 minuti a RT prima di mescolare con le cellule. Dopo l'incubazione, più RNP possono essere combinati se più di un locus deve essere preso di mira contemporaneamente. Non sono state osservate differenze significative nell'efficienza con un massimo di tre loci contemporaneamente.
- Nel frattempo, preparare le cellule per l'elettroporazione. Mantenere le celle a RT in ogni momento per evitare shock termici. Raccogliere le cellule dopo 48 ore ± 2 ore di coltura in un recipiente appropriato. Risciacquare i piatti con DPBS per raccogliere il numero massimo di celle.
- Centrifugare le cellule a 200 x g per 10 minuti a RT. Scartare il surnatante e risospendere le cellule in DPBS a ~ 2 x 106 cellule / ml.
- Combinare 10 μL di soluzione di tripano blu allo 0,4% con 10 μL della sospensione cellulare e contare utilizzando un emocitometro.
NOTA: A questo punto, a causa della perdita durante la raccolta e il lavaggio, aspettatevi circa il 60% delle cellule che sono state messe in coltura 48 h ± 2h prima. - Nel frattempo, centrifugare le cellule a 200 x g per 10 minuti a RT. Scartare il surnatante, assicurandosi di ridurre al minimo eventuali DPBS rimanenti. Risospendere le cellule nel tampone T preriscaldato (RT) a 5,55 x 107 cellule/ml in base al conteggio delle cellule di cui sopra.
- Impostare il sistema di trasfezione accendendo la macchina e impostandola su 1.350 V, 15 ms e 1 impulso. Posizionare la stazione della pipetta all'interno della cappa a flusso laminare
- Per ogni set di 10 elettroporazioni, preparare un tubo di trasfezione con 3 ml di tampone E (per trasfezioni da 10 μL) o E2 (per trasfezioni da 100 μL). Inserire il tubo nella stazione delle pipette.
- Per elettroporazione da 10 μL, combinare 1,15 μL di RNP con 9 μL di cellule. Assicurarsi di avere un volume sufficiente (+30%) per evitare di aspirare aria nella punta dell'elettroporazione. Incubare a RT per 1-2 minuti prima dell'elettroporazione.
- Aspirare 10 μL o 100 μL di RNP e miscela cellulare nella punta di elettroporazione di dimensioni appropriate su una pipetta per elettroporazione, inserire la pipetta caricata nella stazione della pipetta e avviare l'elettroporazione. Assicurati che le punte siano completamente prive di bolle d'aria per evitare l'arco. Osservare durante l'elettroporazione per verificare che non si verifichi un arco elettrico.
- Espellere immediatamente le cellule elettroporate nel piccolo volume di mezzo preparato, preriscaldato, con o senza rAAV6 all'interno della piastra da 48 pozzetti (trasfezioni da 10 μL) o 6 pozzetti (trasfezioni da 100 μL). Ripetere i passaggi 5.15-5.17 con i campioni rimanenti. Aggiungi campioni di controllo senza trasfezione ai pozzetti di coltura.
- Incubare le cellule a 37 °C con il 5% di CO 2 per 4 ore ±2 ore, quindi aggiungere il terreno di coltura cellulare B preparato e preriscaldato contenente stimolanti, DMSO e antibiotici/antimicotici: 450 μL per trasfezioni da 10 μL o 4,5 ml per trasfezioni da 100 μL.
- Continuare l'incubazione a 37 °C con 5% di CO2 per 12-24 ore. Quindi, cambiare il terreno in terreno di coltura di cellule B contenente stimolanti e antibiotici / antimicotici senza DMSO se si desidera una coltura estesa. L'analisi del DNA genomico può essere effettuata dopo 24 ore. La PCR digitale a goccia utilizzando un primer all'esterno del braccio di omologia e un primer all'interno dell'inserto può essere utilizzato per quantificare l'efficienza di editing39. Eseguire PCR per amplificare il sito di inserimento e sequenziamento Sanger per verificare la corretta modifica.
- Per l'analisi dei livelli proteici, coltivare le cellule per 40-48 ore dopo l'elettroporazione per consentire cambiamenti di espressione proteica ed eseguire un'analisi mediante citometria a flusso utilizzando i reagenti nella Tabella 7.
Risultati
La produzione di rAAV6 con l'uso dell'helper adenovirale autosilenziante abilitato alla tetraciclina ha portato alla produzione di 4 x 1010 GC / ml di terreno di coltura cellulare in media, superando così la produzione utilizzando una tripla trasfezione standard senza helper di 30-40 volte (Figura 2).
La purificazione opzionale delle cellule B del macaco rhesus ha portato all'eliminazione della stragrande maggioranza delle cellule T CD3+ e delle cellule mieloidi CD14+ e/o CD16+, con purezze dell'80%-95% delle cellule B CD20+ ottenute di routine (Figura 3). Sulla base dei nostri precedenti progetti nelle cellule B murine7, abbiamo sviluppato un metodo per modificare la specificità del recettore delle cellule B delle cellule B del macaco rhesus, mantenendo contemporaneamente l'esclusione allelica nella stragrande maggioranza delle cellule B eliminando le catene leggere degli anticorpi endogeni attraverso la rottura della loro regione costante. Abbiamo costruito un HDRT senza promotore da inserire nel locus IGH tra l'ultimo gene IGHJ e l'enhancer Eμ delle cellule B del macaco rhesus (Figura 4). Questo costrutto utilizza il promotore endogeno VH della regione VDJ a monte naturalmente riorganizzata nelle cellule B mature e non è, quindi, espresso dai genomi episomiali AAV. Inoltre, questo costrutto richiede lo splicing nelle regioni costanti della catena pesante degli anticorpi a valle da esprimere sulla superficie cellulare. Pertanto, il legame specifico dell'antigene sulla superficie cellulare mostrato dalla citometria a flusso indica la corretta integrazione del locus bersaglio e che la sequenza inserita è funzionale.
Abbiamo impacchettato un tale costrutto codificante l'anticorpo Ab1485, un anti-HIV bNAb40 derivato dal macaco rhesus, in rAAV6 e lo abbiamo usato per modificare colture di splenociti di macaco rhesus primario attivato o PBMC, come descritto sopra (Figura 5A). Il protocollo ha mantenuto un'elevata vitalità cellulare (~ 90%) eliminando contemporaneamente l'espressione della catena leggera in ~ 80% delle cellule B. La maggior parte delle cellule B esprimeva ancora l'isotipo IgM (Figura 5B). L'aggiunta dell'rAAV6 che codifica per l'Ab1485 HDRT ha portato all'editing genico e all'espressione superficiale di Ab1485 nel 16%-21% delle cellule B (Figura 5A), sebbene a un'intensità di fluorescenza inferiore per le catene anticorpali rispetto alle cellule B non modificate (Figura 5A pannello di destra, Figura 5C). Ciò può essere il risultato della competizione epitopica tra la colorazione dell'antigene e i monoclonali utilizzati per rilevare il BCR superficiale nella citometria a flusso, nonché dell'effettiva ridotta espressione proteica dovuta alla natura policistronica dell'HDRT e allo splicing meno efficiente. L'aggiunta dell'1% di DMSO e incubazioni estese e concentrate con l'HDRT rAAV6 hanno generalmente aumentato l'efficienza di editing (Figura 6A-C). Utilizzando questo metodo specifico, in genere 5% -20% e fino al 40%, l'efficienza di editing viene raggiunta a seconda del singolo macaco rhesus (Figura 5A, Figura 6A-E) e della qualità del lotto HDRT rAAV6 (Figura 6E). Nel complesso, presentiamo protocolli per una produzione efficiente di rAAV6, nonché la coltura, la purificazione e l'editing genetico delle cellule B del macaco rhesus.
| Reagenti | Volume | Ceppo | Concentrazione finale |
| DMEM, Alto Glucosio | 500 ml | 1 x | ~ 88,5% |
| FCS, inattivato termicamente | 50 ml | 1 x | ~ 8,85% |
| Antibiotico/Antimicotico | 5 ml | 100 x | 1 x |
| Glutammina | 5 ml | 200 mM | 2 mM |
| Piruvato di sodio | 5 ml | 100 mM | 1 mM |
Tabella 1: Il terreno di coltura cellulare 293AAV.
| Reagenti | Volume | Ceppo | Concentrazione finale |
| DMEM, Alto Glucosio | 500 ml | 1 x | ~ 95,2% |
| FCS, inattivato termicamente | 10 ml | 1 x | ~ 1,9% |
| Antibiotico/Antimicotico | 5 ml | 100 x | 1 x |
| Glutammina | 5 ml | 200 mM | 2 mM |
| Piruvato di sodio | 5 ml | 100 mM | 1 mM |
Tabella 2: Il mezzo di produzione delle celle 293AAV.
| Serie di diluizione | Volume del campione (μL) | Diluente e volume | Fattore di diluizione | Diluizione totale | Riferimento AAV6 |
| GC/mL | |||||
| Diluizione 1 | Campione da 2 μL o standard di riferimento AAV a 4,1 x 1011 GC/mL | 18 μL di tampone DNAseI ed enzima | 10 x | 10 x | 4,1 x 1010 |
| Diluizione 2 | 15 μL Dil. 1 | 60 μL H2O | 5 x | 50 x | 8,2 x 109 |
| Diluizione 3 | 20 μL Dil. 2 | 80 μL H2O | 5 x | 250 x | 1,6 x 109 |
| Diluizione 4 | 20 μL Dil. 3 | 80 μL H2O | 5 x | 1250 x | 3,3 x 108 |
| Diluizione 5 | 20 μL Dil. 4 | 80 μL H2O | 5 x | 6250x | 6,6 x 107 |
| Diluizione 6 | 20 μL Dil. 5 | 80 μL H2O | 5 x | 31250 x | 1,3 x 107 |
| Diluizione 7 | 20 μL Dil. 6 | 80 μL H2O | 5 x | 156250 x | 2,6 x 106 |
| Diluizione 8 | 20 μL Dil. 6 | 80 μL H2O | 5 x | 781250 x | 5,24 x 105 |
| Diluizione 9 | 20 μL Dil. 7 | 80 μL H2O | 5 x | 3906250 x | 1,05 x 105 |
Tabella 3: tabella di diluizione qPCR.
| Reagente | Volume | Ceppo | Concentrazione finale |
| RPMI-1640 | 420 ml | 1 x | 84% |
| FCS, inattivato termicamente | 50 ml | 1 x | 10% |
| Antibiotico/Antimicotico | 5 ml | 100 x | 1 x |
| Glutammina | 5 ml | 200 mM | 2 mM |
| Piruvato di sodio | 5 ml | 100 mM | 1 mM |
| HEPES | 5 ml | 1 m | 10 mM |
| 2-B-mercapto-etanolo | 550 μL | 55 mM | 55 μM |
| Aminoacidi non essenziali | 5 ml | 100 x | 1 x |
| Insulina-transferina-selenio | 5 ml | 100 x | 1 x |
Tabella 4: Terreno di coltura di cellule B.
| Reagente | Diluizione | Ceppo | Concentrazione finale |
| MegaCD40L | 1:1000 | 100 μg/mL | 100 ng/mL |
| CpG ODN | 1:300 | 1 mg/ml | 3,33 μg/mL |
| BAFF umano | 1:1000 | 40 μg/mL | 40 ng/mL |
| IL-2 umano | 1:1000 | 50 μg/mL | 50 ng/mL |
| IL-10 umano | 1:1000 | 50 μg/mL | 50 ng/mL |
Tabella 5: Stimolanti delle cellule B.
| Anticorpo | Clone | Diluizione | Conc. finale |
| CD3 anti-umano | FN-18 · | 1:40 | 2,5 μg/mL |
| CD8a anti-umano | RPA-T8 | 1:200 | 2,5 μg/mL |
| CD14 anti-umano | M5E2 | 1:200 | 2,5 μg/mL |
| CD16 anti-umano | 3G8 | 1:200 | 2,5 μg/mL |
| CD33 anti-umano | AC104.3E3 | 1:50 | 1 prova |
| CD64 anti-umano | 10.1 | 1:800 | 0,625 μg/mL |
| CD66 anti-umano | TET2 | 1:11 | 1 prova |
| CD89 anti-umano | A59 | 1:800 | 0,625 μg/mL |
Tabella 6: Anticorpi per la deplezione facoltativa delle cellule non-B.
| Reagente | Tipo/clone | Diluizione/concentrazione di lavoro |
| CD14 anti-umano AlexaFluor647 | M5E2 | 1:50 |
| CD16 anti-umano AlexaFluor700 | 3G8 | 1:50 |
| CD20 PECy7 anti-umano | 2H7 | 1:50 |
| CD3 PE anti-umano | SP34-2 | 1:50 |
| Zombie-NIR | - | 1:500 |
| anti-umano HLA-DR BV605 | L243 | 1:200 |
| catena leggera Ig anti-umana lambda APC | MHL-38 · | 1:50 |
| catena leggera Kappa anti-umana FITC | policlonale | 1:500 |
| anti-umano IgM BV421 | MHM-88 · | 1:50 |
| Antigene RC1, biotinilato casualmente | - | 5 μg/mL |
| Streptavidina-PE | - | 1:500 |
Tabella 7: Reagenti citometrici a flusso per analisi.
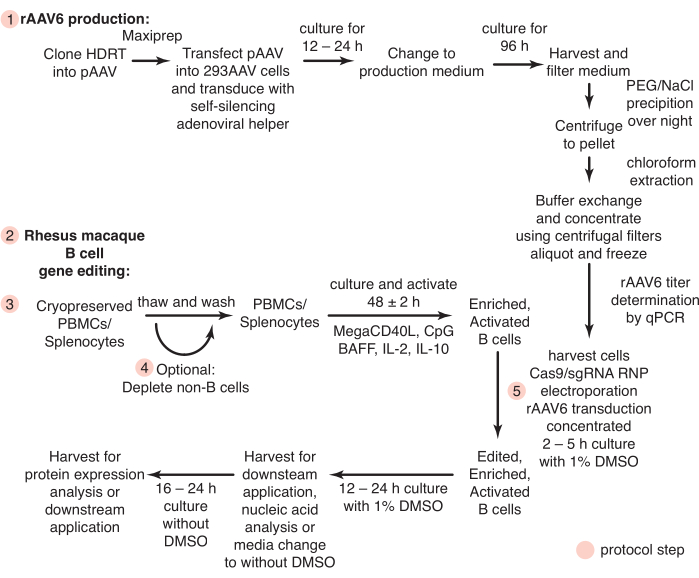
Figura 1: Panoramica schematica della produzione di rAAV6 e dell'editing genetico delle cellule B primarie di macaco rhesus. I protocolli sono suddivisi in produzione di rAAV6 (fase 1) e editing genetico delle cellule B del macaco rhesus (fasi 2-5), inclusa una fase opzionale per l'esaurimento delle cellule non B (fase 4). I passaggi nei protocolli sono indicati con cerchi rossi. Fare clic qui per visualizzare una versione ingrandita di questa figura.
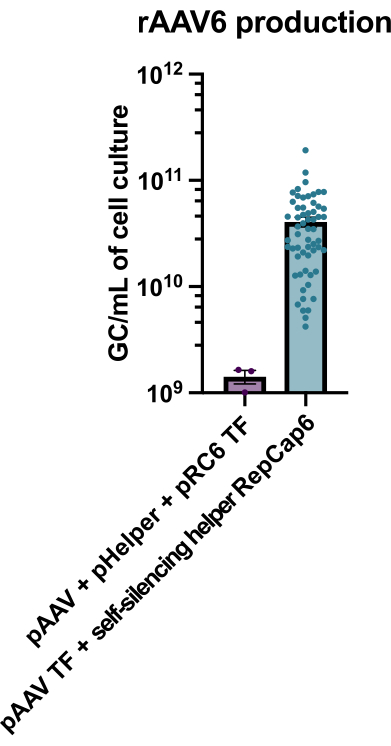
Figura 2: Elevate rese di rAAV6 utilizzando un aiutante adenovirale autosilenziante. rAAV6 è stato prodotto utilizzando i metodi qui descritti (trasfezione pAAV [TF] + helper autosilenziante RepCap6, aiutante adenovirale autosilenziante) o tipica tripla trasfezione senza aiutanti di pAAV, pHelper e pRepCap6 (pRC6). rAAV6 è stato purificato solo dal surnatante cellulare. I metodi che utilizzano i vettori helper adenovirali autosilenzianti hanno prodotto 30-40 volte più rAAV titolato mediante qPCR, come descritto sopra. Ogni punto rappresenta una singola produzione di rAAV utilizzando vari costrutti pAAV da 2 a 20 esperimenti indipendenti. Viene tracciata la media ± SEM. Fare clic qui per visualizzare una versione ingrandita di questa figura.
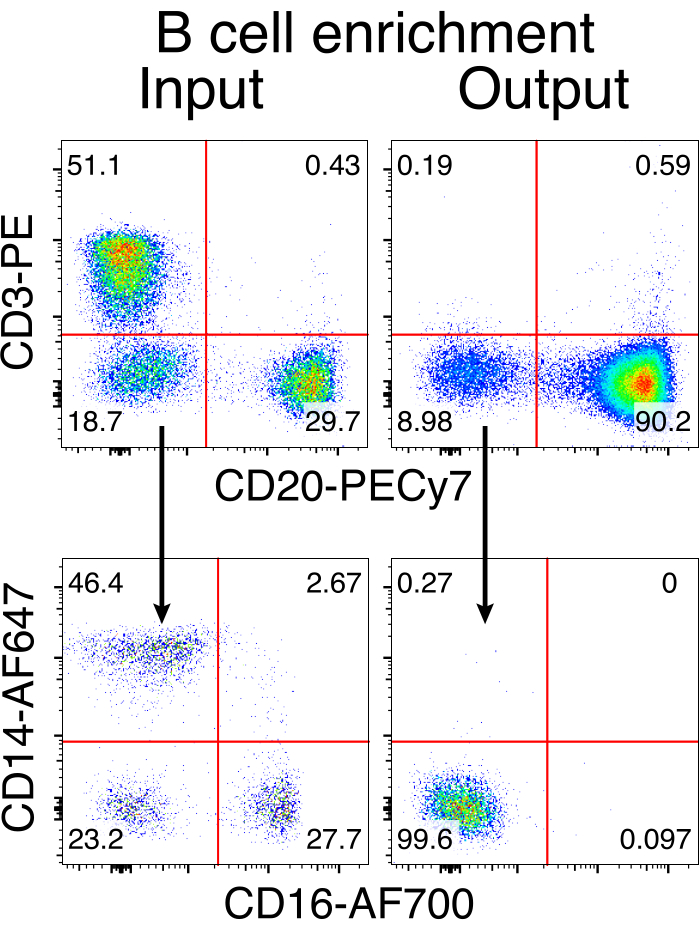
Figura 3: Arricchimento delle cellule B mediante deplezione negativa di cellule non-B. Le cellule B del macaco Rhesus sono state arricchite da PBMC utilizzando il protocollo descritto e arricchite al 90% di purezza. Vengono mostrati l'input e l'output di pre-arricchimento dopo l'arricchimento. Gated su PBMC dal vivo, singoletto. Rappresentativo di cinque esperimenti indipendenti. Fare clic qui per visualizzare una versione ingrandita di questa figura.
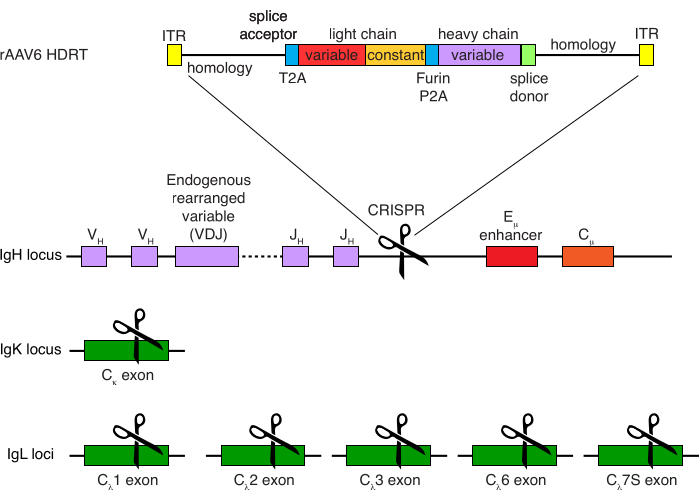
Figura 4: Strategia di targeting utilizzata per modificare la specificità del recettore delle cellule B del macaco rhesus. rAAV6 è stato prodotto contenente l'HDRT raffigurato. L'HDRT è costituito da un braccio di omologia di 266 bp 5', seguito da 111 bp dell'accettore di giunzione dell'esone 1 del macaco rhesus IGHM, quindi da un GSG-linker con una sequenza peptidica 2A auto-scissione del virus Thosea asigna (T2A), seguita da una sequenza leader e dalla catena leggera completa dell'anticorpo Ab1485 del macaco rhesus come macaco rhesus IGLC1. Questo è seguito da un sito di scissione della furina, un linker GSG e una sequenza peptidica 2A auto-scissione del teschovirus suino (Furin-P2A), seguita da un'altra sequenza leader e dalla variabile della catena pesante Ab1485, seguita da 52 bp della sequenza donatrice di giunzione IGHJ4 del macaco rhesus, per consentire lo splicing nelle regioni costanti della catena pesante degli anticorpi a valle, e un braccio di omologia di 514 bp. Questo costrutto è stato mirato nel locus IGH tra l'ultimo gene IGHJ e il potenziatore Eμ utilizzando la sequenza bersaglio sgRNA GAGATGCCAGAGCAAACCAG. Entrambi i bracci di omologia sono stati progettati per terminare nel sito di taglio di questo sgRNA, rimuovendo così la sequenza bersaglio e consentendo efficienze di integrazione ottimali. Allo stesso tempo, per mantenere l'esclusione allelica e l'espressione di un singolo recettore delle cellule B, abbiamo eliminato le catene leggere endogene usando sgRNA che colpiscono il macaco rhesus IGKC con la sequenza target GGCGGGAAGATGAAGACAGA e IGLC1, IGLC2, IGLC3, IGLC6 e IGLC7S utilizzando la sequenza target CTGATCAGTGACTTCTACCC. L'HDRT includeva mutazioni silenti che impedivano la scissione della sequenza IGLC1 da parte di questo sgRNA. Fare clic qui per visualizzare una versione ingrandita di questa figura.
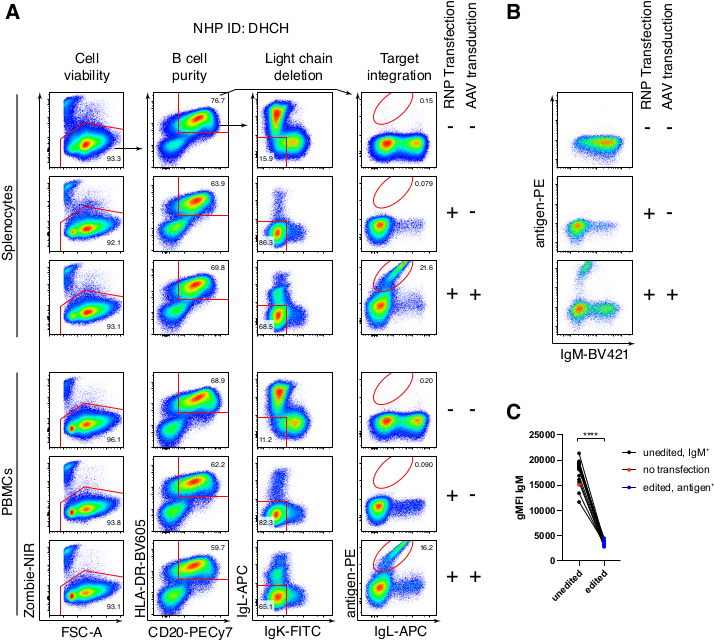
Figura 5: Editing genetico delle cellule B del macaco rhesus primario. (A) Gli splenociti primari (pannello superiore) o PBMC (pannello inferiore) dello stesso macaco rhesus sono stati coltivati senza l'esaurimento delle cellule non B e modificati come descritto sopra. La strategia di targeting era come mostrato nella Figura 4. Due giorni dopo l'elettroporazione, le cellule sono state raccolte e colorate in superficie per l'analisi citometrica a flusso. La colonna di sinistra è stata recintata su celle singolette e le altre colonne sono state quindi gate, come indicato nella riga superiore. La vitalità delle cellule, la purezza delle cellule B, l'efficienza di delezione delle catene leggere e l'efficienza knock-in di Ab1485 mediante colorazione con l'antigene specifico RC141 sono indicate in campioni non trattati, RNP trasfettati o RNP trasfettati + rAAV6 trasdotti (MOI = 5 x 105). Rappresentativo di sei esperimenti indipendenti con cellule di diversi macachi rhesus. (B) Espressione di IgM su controlli colturali di cellule B di macaco rhesus o dopo editing e (C) intensità geometrica media di fluorescenza (gMFI) di IgM su cellule B che non hanno perso l'espressione di Ig a causa del targeting di IgLC e IgKC (non modificato) o cellule B che legano l'antigene atteso (modificato). Il punto rosso indica la gMFI delle cellule B di controllo non trasfettate in coltura. Indica p < 0,0001 in un test t associato. Fare clic qui per visualizzare una versione ingrandita di questa figura.
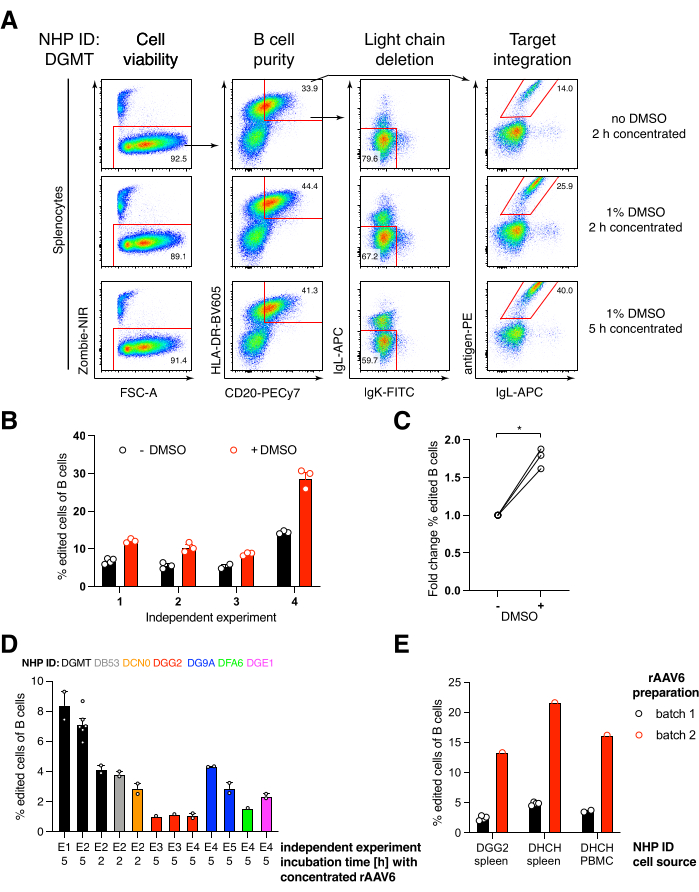
Figura 6: Effetti del DMSO, incubazione concentrata prolungata con rAAV6 HDRT, qualità del lotto di rAAV e riproducibilità tra diversi NHP donatori sull'efficienza dell'editing genico nelle cellule B del macaco rhesus primario. (A) Gli splenociti sono stati coltivati e modificati come descritto. Dopo l'elettroporazione, 5 x 10 5 cellule sono state coltivate in terreno con o senza DMSO all'1% e incubate in 50 μL di terreno contenente HDRT rAAV6 con un MOI di 5 x 10 5 per 2 ore o5 ore prima dell'aggiunta di altri 450 μL di terreno. Le cellule sono state analizzate 2 giorni dopo l'elettroporazione mediante citometria a flusso, come in Figura 5. Rappresentante di quattro esperimenti indipendenti. (B) Quantificazione di (A) su quattro esperimenti indipendenti. I punti indicano repliche tecniche con impostazioni di trasfezione di 1.350 V, 10-20 ms e 1 durata dell'elettroporazione a impulsi e concentrazioni di DMSO comprese tra 0,75% e 1,25%. (C) Variazione media della piegatura nell'efficienza di editing da (B). * p > 0,05 nel test U di Mann-Whitney. (D) Efficienza di modifica rispetto a esperimenti indipendenti con diversi macachi utilizzando un lotto commerciale rAAV6 a bassa efficienza. (E) Efficienza di editing utilizzando due diversi lotti commerciali di rAAV6 in cui lo stesso costrutto è stato confezionato nelle cellule B di due diversi NHP nello stesso esperimento. I punti indicano repliche tecniche con impostazioni di trasfezione di 1.350 V, 10-20 ms e 1 elettroporazione a impulsi. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
I protocolli qui presentati forniscono un metodo rapido ed efficiente per generare alti rendimenti e titoli di rAAV6 come HDRT e nuovi metodi per modificare in modo efficiente le cellule B del macaco rhesus primario in vitro.
Il protocollo di produzione rAAV6 è relativamente semplice e veloce, consentendo la produzione e il collaudo di molti costrutti diversi contemporaneamente senza lavoro eccessivo. Se lo si desidera, rAAV6 può essere ulteriormente purificato utilizzando protocolli consolidati come l'ultracentrifugazione a gradiente di iodixanolo34 o il partizionamento acquoso a due fasi35 prima dello scambio tampone e della concentrazione.
Sebbene abbia ridotto la resa complessiva, abbiamo optato per utilizzare solo il terreno di coltura cellulare ridotto dal siero per la purificazione di rAAV6 invece della purificazione dal pellet cellulare, poiché la maggior parte di rAAV6 viene rilasciata nel mezzo36 e la purificazione dal pellet cellulare aggiunge più costi e manodopera. L'uso dell'helper adenovirale autoinattivante ha aumentato le rese in media di 30-40 volte, consentendo di testare costrutti confezionati in AAV6 in un unico piatto da 15 cm. Sebbene il nostro metodo di purificazione sia di base, utilizzando questo metodo, otteniamo relativamente poca variazione da lotto a lotto nell'efficienza di editing genico o nella vitalità cellulare dopo la trasduzione utilizzando varie linee cellulari o altre cellule primarie (dati non mostrati).
Abbiamo sviluppato un protocollo di purificazione delle cellule B del macaco rhesus per ottenere cellule B primarie intatte utilizzando l'esaurimento negativo delle popolazioni indesiderate. Sebbene non sia necessario per l'editing genetico di queste cellule, fornisce un modo per ottenere una popolazione relativamente pura di cellule B primarie di macaco rhesus per questa o altre applicazioni nel caso in cui altri tipi di cellule interferiscano con gli obiettivi sperimentali. Tuttavia, la purezza arriva al costo di una riduzione delle rese complessive delle cellule B. In particolare, sia per le colture di cellule B arricchite che non arricchite, la frazione di cellule B nelle preparazioni iniziali di PBMC o splenociti è cruciale. Per le PBMC in particolare, raccomandiamo lo screening di diversi macachi per individui con un'alta percentuale di cellule B nel sangue periferico per ottenere un numero elevato di cellule B per gli esperimenti, poiché questo valore può differire notevolmente tra gli individui27. Le PBMC possono essere ottenute mediante sanguinamento regolare o leucaferesi42.
Il protocollo di editing genetico porta a un editing genetico efficiente, in genere tra il 60% -80% delle cellule knock-out e il 5% -20% delle cellule B knock-in, sebbene abbiamo raggiunto fino al 90% di knock-out BCR e il 40% di cellule B knock-in BCR (Figura 5 e Figura 6).
I parametri principali per l'editing efficiente delle cellule B del macaco rhesus sono l'efficienza di taglio dell'sgRNA, i parametri di elettroporazione, il MOI e la qualità della preparazione di rAAV6. Le efficienze di taglio degli sgRNA candidati dovrebbero essere determinate empiricamente per consentire l'editing e la progettazione ottimali dell'HDRT. I parametri di elettroporazione qui presentati bilanciano l'efficienza con la fattibilità per ottenere il numero totale massimo di cellule B modificate piuttosto che la più alta percentuale di cellule B modificate. Se è necessaria una percentuale maggiore di celle modificate, si raccomanda un aumento delle tensioni (fino a 1.750 V) o lunghezze di impulso alterate (10-30 ms), sebbene si possa osservare una maggiore morte cellulare. Abbiamo anche notato efficienze di editing leggermente superiori nelle cellule B spleniche rispetto alle cellule B delle PBMC dello stesso individuo (Figura 5); Tuttavia, la ragione di fondo di ciò è attualmente sconosciuta.
Abbiamo scoperto che l'aggiunta dell'1% di DMSO dopo l'elettroporazione ha aumentato significativamente l'efficienza di editing genico di ~ 40% nelle cellule B del macaco rhesus senza influenzare la vitalità cellulare (Figura 6A-C), in linea con i rapporti in altre cellule43. Tuttavia, la coltura estesa in DMSO all'1% deve essere evitata e può influire sulla vitalità cellulare. DMSO può essere completamente omesso se lo si desidera.
La coltura delle cellule in un piccolo volume dopo l'elettroporazione per diverse ore insieme a rAAV6 porta a maggiori efficienze di editing, probabilmente a causa della migliore trasduzione dell'HDRT da parte dell'rAAV6 e, quindi, della maggiore concentrazione intracellulare di HDRT nel momento in cui Cas9 è attivo. Abbiamo scoperto che coltivare le cellule in questo modo per un massimo di 8 ore non ha influenzato la vitalità cellulare, ma l'efficienza di editing non è aumentata drasticamente oltre le 5 ore (Figura 6). Se è necessario solo foratura anziché knock-in, questo passaggio può essere omesso.
In conclusione, presentiamo protocolli completi per l'editing genetico delle cellule B del macaco rhesus in vitro e la produzione di rAAV6 HDRT necessari per l'efficiente knock-in dei costrutti desiderati. Questi protocolli consentono la sperimentazione rapida ed economica di molti costrutti confezionati come rAAV6 e consentono la sperimentazione preclinica della fattibilità e della scalabilità delle terapie con cellule B in un modello di primate non umano più rilevante.
Divulgazioni
Non vengono dichiarati interessi concorrenti.
Riconoscimenti
Vorremmo ringraziare Harry B. Gristick e Pamela Bjorkman per aver fornito l'antigene RC1 e tutti i laboratori Nussenzweig e Martin per una discussione critica. Questo lavoro è stato sostenuto dalla sovvenzione della Fondazione Bill e Melinda Gates INV-002777 (a MCN) e dal Programma di ricerca intramurale del National Institute of Allergy and Infectious Diseases, National Institutes of Health. (R.G. e M.A.M). M.C.N. è un investigatore HHMI.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL tube sterile, Dnase, Rnase and purogen free | Stellar Scientific | T17-125 | or similar |
| 10 mL serological pipette, polystyrene, sterile, nonpyrogenic, DNase-/RNase-free, and Human DNA-free | Corning | 4488 | or similar |
| 15 cm tissue culture dish | Falcon | 353025 | or similar |
| 15 mL polypropylene conical tybe | Falcon | 352097 | or similar |
| 25 mL serological pipette, polystyrene, sterile, nonpyrogenic, DNase-/RNase-free, and Human DNA-free | Corning | 4489 | or similar |
| 250 mL polypropylene conical tybe | Corning | 430776 | or similar |
| 293AAV cell line | Cell Biolabs | AAV-100 | |
| 2-B-mercapto-ethanol, 55mM (1000x) | Gibco | 21985-023 | |
| 48-well tissue culture plate | Corning | 3548 | or similar |
| 5 mL serological pipette, polystyrene, sterile, nonpyrogenic, DNase-/RNase-free, and Human DNA-free | Corning | 4487 | or similar |
| 5 mL syringes with Luer-Lok Tip | BD | 309646 | or similar |
| 50 mL polypropylene conical tybe | Falcon | 352070 | or similar |
| 50 mL serological pipette, polystyrene, sterile, nonpyrogenic, DNase-/RNase-free, and Human DNA-free | Corning | 4490 | or similar |
| 6-well tissue culture plate | Falcon | 353046 | or similar |
| AAV-6 Packaging System (plasmids) | Cell Biolabs | VPK-406 | |
| AAV6 Reference Materials (full capsids) | Charles River | RS-AAV6-FL | |
| Accu-jet S Pipette Controller | Brand | 26350 | or similar pipette controller |
| Antibiotic/Antimycotic 100x | Gibco | 15260-062 | |
| anti-human CD14 AlexaFluor647 | Biolegend | 301812 | |
| anti-human CD14 biotin | BioLegend | 301826 | |
| anti-human CD16 AlexaFluor700 | BD Biosciences | 557920 | |
| anti-human CD16 biotin | BioLegend | 302004 | |
| anti-human CD20 PECy7 | Biolegend | 302312 | |
| anti-human CD3 biotin | Thermo Fisher | APS0309 | |
| anti-human CD3 PE | BD Biosciences | 552127 | |
| anti-human CD33 biotin | Miltenyi | 130-113-347 | |
| anti-human CD64 biotin | BioLegend | 305004 | |
| anti-human CD66 biotin | Miltenyi | 130-100-143 | |
| anti-human CD89 biotin | BioLegend | 354112 | |
| anti-human CD8a biotin | BioLegend | 301004 | |
| anti-human HLA-DR BV605 | Biolegend | 307640 | |
| anti-human Ig light chain lambda APC | Biolegend | 316610 | |
| anti-human IgM BV421 | Biolegend | 314516 | |
| anti-Human Kappa Light Chain FITC | Fisher Scientific | A18854 | |
| Autoclave | Steris | Amsco Lab 250 | or similar |
| Cell culture CO2 incubator | Fisher Scientific | 51026331 | or similar |
| Centrifugal Filter Unit (Amicon Ultra - 4, 100 kDa) | Millipore | UFC810024 | |
| Centrifuge 5920 R | Eppendorf | EP022628188 | or any other, coolable swinging bucket centrifuge with inserts for 96-well plates, 15, 50 and 250 mL size tubes |
| Chloroform | Fisher Scientific | C298SK-4 | |
| Cpg ODN | Invivogen | tlrl-2395 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | 34869-500ML | |
| DMEM, High Glucose | Gibco | 11965092 | |
| DNaseI (RNase-free) | New England Biolabs | M0303L | |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | |
| Electroporation kit (Neon Transfection System 10 µL) | Fisher Scientific | MPK1096 | or other sizes or 100 uL transfection kit MPK 10096 |
| Electroporation system (Neon Transfection System) | Fisher Scientific | MPK5000 | |
| FCS | Hyclone | SH30910.03* | |
| Ficoll-PM400 (Ficoll-Paque PLUS) | Cytiva | 17144002 | or similar |
| Fume Hood | Fisher Scientific | FH3943810244 | or similar |
| Glutamine 200 mM | Gibco | 25030-081 | |
| Graduated Cylinder 1L | Corning | 3022-1L | or similar |
| Hemocytometer | Sigma-Aldrich | Z375357-1EA | or similar |
| HEPES 1M | Gibco | 15630-080 | |
| HEPES 1M | Gibco | 15630-080 | |
| Hot Plate Magnetic Stirrer | Fisher Scientific | SP88857200 | or similar |
| Human BAFF | Peprotech | 310-13 | |
| Human BD Fc Block | BD | 564220 | |
| Human IL-10 | Peprotech | 200-10 | |
| Human IL-2 | Peprotech | 200-02 | |
| Hydrochloric acid | Fisher Scientific | A144S-500 | |
| Hydrophilic Polyethersulfone Syringe Filters, (Supor membrane), Sterile - 0.2 µm, 25 mm | Pall | 4612 | |
| Insulin-Transferin-Selenium, 100x | Gibco | 41400-045 | |
| ITR primer forward: GGAACCCCTAGTGATGGAGTT | Integrated DNA Technologies | custom | |
| ITR primer reverse: CGGCCTCAGTGAGCGA | Integrated DNA Technologies | custom | |
| Laminar flow biosafety cabinet | The Baker Company | SG403A | or similar |
| Large magnetic depletion (LD) Column | Miltenyi Biotec | 130-042-901 | |
| Magentic seperator (MidiMACS separator and multistand) | Miltenyi Biotec | 130-090-329 | |
| Magnetic stir bar | Fisher Scientific | 14-512-127 | or similar |
| Magnetic streptavidin beads (Streptavidin MicroBeads) | Miltenyi Biotec | 130-048-101 | |
| Maxiprep kit | Machery-Nagel | 740414.5 | or similar |
| Media Bottles 2L with cap | Cole-Parmer | UX-34514-26 | or similar |
| MegaCD40L | Enzo | ALX-522-110-C010 | |
| MicroAmp Optical 384-well Reaction Plate | Fisher Scientific | 4309849 | |
| MicroAmp Optical Adhesive Film | Fisher Scientific | 4311971 | |
| Microcentrifuge 5424 R | Eppendorf | 5404000014 | or any other table top centrifuge for 1.5 mL tubes |
| Microwave oven | Panasonic | NN-SD987SA | or similar |
| Nikon TMS Inverted Phase Contrast Microscope | Nikon | TMS | or any other Inverted phase-contrast microscope for cell culture |
| Non-essential amino acids, 100x | Gibco | 11140-050 | |
| Nuclease-free Duplex buffer | Integrated DNA Technologies | 11-01-03-01 | |
| Nuclease-free Water | Qiagen | 129115 | |
| pH meter | Mettler Toledo | 30019028 | or similar |
| Pipetman Classic Starter Kit, 4 Pipette Kit, P2, P20, P200, P1000 and tips | Gilson | F167380 | or similar set of pipettes and tips |
| Pluronic F-68 10 % | Gibco | 24040-032 | |
| Polyethylene Glycol 8000 | Fisher Scientific | BP233-1 | |
| Polyethylenimine, Linear, MW 25000, Transfection Grade (PEI 25K | Polysciences | 23966-100 | |
| Precision Balance | Mettler Toledo | ME4001TE | or similar |
| Pre-Separation Filters (30 µm) | Miltenyi Biotec | 130-041-407 | |
| Pyrex glass beaker 2 L | Cole-Parmer | UX-34502-13 | or similar |
| Pyrex glass beaker 250 mL | Millipore Sigma | CLS1000250 | or similar |
| qPCR Instrument | Fisher Scientific | 4485691 | or similar |
| RC1 antigen randomly biotinylated | Bjorkman lab, CalTech | in house | |
| RPMI-1640 | Gibco | 11875-093 | |
| S.p. Cas9 Nuclease | Integrated DNA Technologies | 1081059 | |
| Scientific 1203 Water Bath | VWR | 24118 | or any water bath set to 37 °C |
| Sodium chloride | Sigma-Aldrich | S7653-5KG | |
| Sodium hydroxide | Sigma-Aldrich | S8045-500G | |
| Sodium Pyruvate 100 mM | Gibco | 11360-070 | |
| Sterile Disposable Filter Units with PES Membranes | Thermo Scientific Nalgene | 567-0020 | |
| Streptavidin-PE | BD Biosciences | 554061 | |
| SYBR Green Master Mix | Fisher Scientific | A25742 | |
| Tetracycline-enabled, self-silencing adenoviral vector RepCap6 | Oxgene | TESSA-RepCap6 | |
| Trypan Blue Solution, 0.4% | Gibco | 15250061 | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300054 | |
| Water Purification System | Millipore Sigma | ZEQ7000TR | or similar |
| Zombie-NIR | Biolegend | 423106 |
Riferimenti
- Victora, G. D., Nussenzweig, M. C. Germinal centers. Annual Review of Immunology. 40, 413-442 (2022).
- Plotkin, S. A. Correlates of protection induced by vaccination. Clinical and Vaccine Immunology. 17 (7), 1055-1065 (2010).
- Brinkmann, V., Heusser, C. H. T cell-dependent differentiation of human B cells into IgM, IgG, IgA, or IgE plasma cells: High rate of antibody production by IgE plasma cells, but limited clonal expansion of IgE precursors. Cellular Immunology. 152 (2), 323-332 (1993).
- Chernecky, C. C., Berger, B. J. . Protein Electrophoresis - Serum., 6th edition. , 917-920 (2013).
- Balazs, A. B., et al. Antibody-based protection against HIV infection by vectored immunoprophylaxis. Nature. 481 (7379), 81-84 (2011).
- Greiner, V., et al. CRISPR-mediated editing of the B cell receptor in primary human B cells. iScience. 12, 369-378 (2019).
- Hartweger, H., et al. HIV-specific humoral immune responses by CRISPR/Cas9-edited B cells. Journal of Experimental Medicine. 216 (6), 1301-1310 (2019).
- Huang, D., et al. Vaccine elicitation of HIV broadly neutralizing antibodies from engineered B cells. Nature Communications. 11, 5850 (2020).
- Jeske, A. M., Boucher, P., Curiel, D. T., Voss, J. E. Vector strategies to actualize B cell-based gene therapies. Journal of Immunology. 207 (3), 755-764 (2021).
- Nahmad, A. D., et al. In vivo engineered B cells secrete high titers of broadly neutralizing anti-HIV antibodies in mice. Nature Biotechnology. 40 (8), 1241-1249 (2022).
- Nahmad, A. D., et al. Engineered B cells expressing an anti-HIV antibody enable memory retention, isotype switching and clonal expansion. Nature Communications. 11, 5851 (2020).
- Voss, J. E., et al. Reprogramming the antigen specificity of B cells using genome-editing technologies. eLife. 8, 42995 (2019).
- Pesch, T., et al. Molecular design, optimization, and genomic integration of chimeric B cell receptors in murine B cells. Frontiers in Immunology. 10, 2630 (2019).
- Cheong, T. C., Compagno, M., Chiarle, R. Editing of mouse and human immunoglobulin genes by CRISPR-Cas9 system. Nature Communications. 7, 10934 (2016).
- Rogers, G. L., Cannon, P. M. Genome edited B cells: A new frontier in immune cell therapies. Molecular Therapy. 29 (11), 3192-3204 (2021).
- Hung, K. L., et al. Engineering protein-secreting plasma cells by homology-directed repair in primary human B cells. Molecular Therapy. 26 (2), 456-467 (2018).
- Johnson, M. J., Laoharawee, K., Lahr, W. S., Webber, B. R., Moriarity, B. S. Engineering of primary human B cells with CRISPR/Cas9 targeted nuclease. Scientific Reports. 8, 12144 (2018).
- Wu, C. M., et al. Genetic engineering in primary human B cells with CRISPR-Cas9 ribonucleoproteins. Journal of Immunological Methods. 457, 33-40 (2018).
- Luo, B., et al. Engineering of alpha-PD-1 antibody-expressing long-lived plasma cells by CRISPR/Cas9-mediated targeted gene integration. Cell Death and Disease. 11 (11), 973 (2020).
- Laoharawee, K., et al. Genome engineering of primary human B cells using CRISPR/Cas9. Journal of Visualized Experiments. (165), e61855 (2020).
- Laoharawee, K., Johnson, M. J., Moriarity, B. S. CRISPR/Cas9-mediated genome engineering of primary human B cells. Methods in Molecular Biology. 2115, 435-444 (2020).
- Moffett, H. F., et al. B cells engineered to express pathogen-specific antibodies protect against infection. Science Immunology. 4 (35), (2019).
- Hartweger, H., Nussenzweig, M. C. CRISPR comes a-knock-in to reprogram antibodies in vivo. Nature Biotechnology. 40 (8), 1183-1184 (2022).
- Nishimura, Y., Martin, M. A. Of mice, macaques, and men: Broadly neutralizing antibody immunotherapy for HIV-1. Cell Host & Microbe. 22 (2), 207-216 (2017).
- Shedlock, D. J., Silvestri, G., Weiner, D. B. Monkeying around with HIV vaccines: Using rhesus macaques to define 'gatekeepers' for clinical trials. Nature Reviews Immunology. 9 (10), 717-728 (2009).
- Kreuser, S., et al. Efficient methods for generation and expansion of, and gene delivery to rhesus macaque plasma B cells. bioRxiv. , (2021).
- Gujer, C., Sundling, C., Seder, R. A., Karlsson Hedestam, G. B., Lore, K. Human and rhesus plasmacytoid dendritic cell and B-cell responses to Toll-like receptor stimulation. Immunology. 134 (3), 257-269 (2011).
- Kim, J. S., et al. Cell enrichment-free massive ex-vivo expansion of peripheral CD20(+) B cells via CD40-CD40L signals in non-human primates. Biochemical and Biophysical Research Communications. 473 (1), 92-98 (2016).
- Su, W., et al. Self-attenuating adenovirus enables production of recombinant adeno-associated virus for high manufacturing yield without contamination. Nature Communications. 13, 1182 (2022).
- Endo, Y., et al. Short- and long-term clinical outcomes in rhesus monkeys inoculated with a highly pathogenic chimeric simian/human immunodeficiency virus. Journal of Virology. 74 (15), 6935-6945 (2000).
- Balaphas, A., Buchs, N. C., Meyer, J., Hagen, M. E., Morel, P. Partial splenectomy in the era of minimally invasive surgery: The current laparoscopic and robotic experiences. Surgical Endoscopy. 29 (12), 3618-3627 (2015).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47, 171-174 (2019).
- Strobel, B., Miller, F. D., Rist, W., Lamla, T. Comparative analysis of cesium chloride- and iodixanol-based purification of recombinant adeno-associated viral vectors for preclinical applications. Human Gene Therapy Methods. 26 (4), 147-157 (2015).
- Guo, P., et al. Rapid and simplified purification of recombinant adeno-associated virus. Journal of Virological Methods. 183 (2), 139-146 (2012).
- Vandenberghe, L. H., et al. Efficient serotype-dependent release of functional vector into the culture medium during adeno-associated virus manufacturing. Human Gene Therapy. 21 (10), 1251-1257 (2010).
- Brinkman, E. K., Chen, T., Amendola, M., van Steensel, B. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Research. 42 (22), 168 (2014).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. The CRISPR Journal. 5 (1), 123-130 (2022).
- Wilkinson, A. C., et al. Cas9-AAV6 gene correction of beta-globin in autologous HSCs improves sickle cell disease erythropoiesis in mice. Nature Communications. 12, 686 (2021).
- Wang, Z., et al. A broadly neutralizing macaque monoclonal antibody against the HIV-1 V3-Glycan patch. eLife. 9, 61991 (2020).
- Escolano, A., et al. Immunization expands B cells specific to HIV-1 V3 glycan in mice and macaques. Nature. 570 (7762), 468-473 (2019).
- Pathiraja, V., Matar, A. J., Gusha, A., Huang, C. A., Duran-Struuck, R. Leukapheresis protocol for nonhuman primates weighing less than 10 kg. Journal of the American Association for Laboratory Animal Science. 52 (1), 70-77 (2013).
- Stratigopoulos, G., De Rosa, M. C., LeDuc, C. A., Leibel, R. L., Doege, C. A. DMSO increases efficiency of genome editing at two non-coding loci. PLoS One. 13 (6), 0198637 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon