È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutazione dei livelli di autofagia in due diversi modelli di cellule pancreatiche utilizzando l'immunofluorescenza LC3
In questo articolo
Riepilogo
L'obiettivo di questo protocollo è determinare i livelli autofagici nel cancro del pancreas e nelle cellule acinose pancreatiche attraverso l'immunofluorescenza LC3 e la quantificazione dei punti LC3.
Abstract
L'autofagia è un processo catabolico specializzato che degrada selettivamente i componenti citoplasmatici, comprese le proteine e gli organelli danneggiati. L'autofagia consente alle cellule di rispondere fisiologicamente agli stimoli di stress e, quindi, mantenere l'omeostasi cellulare. Le cellule tumorali potrebbero modulare i loro livelli di autofagia per adattarsi a condizioni avverse come ipossia, carenza di nutrienti o danni causati dalla chemioterapia. L'adenocarcinoma pancreatico duttale è uno dei tipi più mortali di cancro. Le cellule tumorali pancreatiche hanno un'elevata attività autofagica a causa della sovraregolazione trascrizionale e dell'attivazione post-traduzionale delle proteine dell'autofagia.
Qui, la linea cellulare PANC-1 è stata utilizzata come modello di cellule tumorali umane pancreatiche e la linea cellulare acinosa pancreatica AR42J è stata utilizzata come modello fisiologico di cellule di mammifero altamente differenziate. Questo studio ha utilizzato l'immunofluorescenza della catena leggera 3 (LC3) della proteina associata ai microtubuli come indicatore dello stato di attivazione dell'autofagia. LC3 è una proteina autofagica che, in condizioni basali, mostra un modello diffuso di distribuzione nel citoplasma (noto come LC3-I in questa condizione). L'induzione dell'autofagia innesca la coniugazione di LC3 alla fosfatidiletanolammina sulla superficie degli autofagosomi appena formati per formare LC3-II, una proteina legata alla membrana che aiuta nella formazione e nell'espansione degli autofagosomi. Per quantificare il numero di strutture autofagiche etichettate, il software open source FIJI è stato utilizzato con l'aiuto dello strumento "3D Objects Counter".
La misura dei livelli autofagici sia in condizioni fisiologiche che nelle cellule tumorali ci permette di studiare la modulazione dell'autofagia in diverse condizioni come l'ipossia, il trattamento chemioterapico o l'abbattimento di alcune proteine.
Introduzione
La macroautofagia (comunemente indicata come autofagia) è un processo catabolico specializzato che degrada selettivamente i componenti citoplasmatici, comprese le proteine e gli organelli danneggiati 1,2. L'autofagia consente alle cellule di rispondere fisiologicamente agli stimoli di stress e, quindi, mantenere l'omeostasi cellulare3. Durante l'autofagia, si forma una doppia vescicola di membrana: l'autofagosoma. L'autofagosoma contiene le molecole di carico e le guida al lisosoma per la degradazione 1,4.
Gli autofagosomi sono decorati dalla proteina autofagica microtubule-associated protein microtubule-associated protein light chain (LC3)5. Quando l'autofagia non è indotta, la CL3 è diffusa nel citoplasma e nel nucleo nella conformazione LC3-I. D'altra parte, quando viene indotta l'autofagia, LC3 è coniugata con una fosfatidiletanolammina nella membrana delle strutture autofagiche6. Questa nuova conformazione LC3 è nota come LC3-II1. Lo spostamento della conformazione di LC3 provoca cambiamenti nella sua localizzazione cellulare e nella sua migrazione di elettroforesi su gel di sodio solfato-poliacrilammide (SDS-PAGE), che può essere rilevata con tecniche come l'immunofluorescenza e il western blot 5,7. In questo modo, la coniugazione LC3 è un evento chiave nel processo autofagico che può essere utilizzato per misurare l'attività autofagica.
La cellula acinosa pancreatica è una cellula altamente differenziata che, in condizioni di salute, ha un basso tasso di autofagia. Tuttavia, in diverse condizioni fisiologiche o sotto stimolazione farmacologica, possono attivare l'autofagia. Pertanto, la determinazione dei livelli autofagici in questa linea cellulare è utile per studiare i potenziali effetti diretti o indiretti di diversi agenti farmacologici o biologici sull'autofagia 8,9.
L'adenocarcinoma pancreatico duttale è uno dei tipi più letali di cancro, data la sua diagnosi tardiva e la sua elevata resistenza alla chemioterapia10. Le cellule tumorali pancreatiche hanno un'elevata attività autofagica a causa della sovraregolazione trascrizionale e dell'attivazione post-traduzionale delle proteine correlate all'autofagia11. Le cellule tumorali pancreatiche possono regolare i loro livelli di autofagia in risposta a condizioni sfavorevoli come ipossia, privazione di nutrienti o danni indotti dalla chemioterapia11. Quindi, l'analisi dei livelli di autofagia nelle cellule tumorali pancreatiche può aiutare a capire come si adattano a diversi ambienti e valutare l'efficacia dei trattamenti che modulano l'autofagia.
Questo studio mostra un metodo per eseguire l'immunofluorescenza LC3 in due distinti modelli cellulari pancreatici. Il primo modello, le cellule PANC-1, è servito come modello per l'adenocarcinoma duttale pancreatico. Queste cellule sono state trattate con gemcitabina, un agente chemioterapico che ha precedentemente dimostrato di indurre l'autofagia, in particolare nelle cellule tumorali pancreatiche portatrici del gene oncogeno del virus del sarcoma di ratto Kirsten (KRAS)12,13. Il secondo modello, le cellule AR42J, è servito come modello più fisiologico delle cellule pancreatiche esocrine. Queste cellule sono state differenziate con desametasone per diventare più simili alle cellule pancreatiche acinose14. In queste cellule, l'autofagia è stata farmacologicamente indotta attraverso l'uso di PP242, che è un potente inibitore mTOR15. In questo studio, dimostriamo l'applicabilità del protocollo descritto con due diversi modelli pancreatici e la sua capacità di discriminare tra stati di autofagia bassa e alta.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparazione delle cellule
- Immergere i coperchi rotondi da 12 mm in etanolo assoluto e posizionarli verticalmente nei pozzetti di una piastra a 24 pozzetti.
- Rimuovere il coperchio ed esporre la piastra multipozzetto alle radiazioni ultraviolette per 15 minuti.
- Posizionare i coprivetrini orizzontalmente e lavarli con Modified Eagle Medium (DMEM) di Dulbecco.
- Semina un basso numero di cellule pancreatiche. L'importo deve essere adeguato per ottenere una confluenza del 50% -75% il giorno della fissazione16.
NOTA: Si consiglia di seminare 2,5 × 10 4 PANC-1 o 4 × 104 cellule AR42J per pozzetto per fissare le cellule dopo 3 giorni. - Coltura delle cellule in DMEM contenente il 10% di siero bovino fetale, 100 U/mL di penicillina e 100 μg/mL di streptomicina in un incubatore a 37 °C in atmosfera umidificata con il 5% di anidride carbonica (CO2).
NOTA: Per le cellule PANC-1, si consiglia di incubare le cellule per 2 giorni tra la semina cellulare e i seguenti passaggi. Dopo questo tempo, le cellule possono essere trasfettate, trattate o fissate. Questo protocollo esemplifica il trattamento con gemcitabina nelle cellule PANC-1 non trasfettate e il differenziamento e il trattamento PP242 per le cellule AR42J non trasfettate.
2. Trattare le cellule
- Trattamento con gemcitabina per cellule PANC-1
- Preparare una soluzione di 1 μg/μL di gemcitabina in DMEM 2 giorni dopo la semina. Trattare ciascun pozzetto con 2,6 μL della soluzione di gemcitabina da 1 μg/μL per ottenere una diluizione finale di 20 μM.
- Incubare le cellule per 24 ore nell'incubatore.
- Differenziazione AR42J e trattamento PP242
- Preparare una soluzione di 4 μg/mL di desametasone in DMEM.
- Trattare ciascun pozzetto con 4,9 μL di soluzione di desametasone da 4 μg/mL per ottenere una diluizione finale di 100 nM.
- Incubare le cellule per 48 ore nell'incubatore.
- Rimuovere il mezzo e trattare ogni pozzetto con 0,5 μL di 1 mM PP242 per ottenere una diluizione finale di 1 μM.
- Incubare le cellule per 2 ore nell'incubatore.
3. Fissare e permeabilizzare le cellule
- Preparare una piastra a 24 pozzetti con metanolo freddo e una piastra a 6 pozzetti con soluzione salina tamponata fosfato freddo (PBS; 137 mM NaCl, 2,7 mM KCl, 8 mM Na 2 HPO4, 2 mM KH2PO4). Mantenerli sul ghiaccio.
- Prendere ogni coprislip con una pinzetta, lavarlo due volte in PBS e incubare per 6 minuti in metanolo.
4. Blocco delle celle
- Lavare ogni foglietto due volte in PBS e incubare per 1 ora in siero bovino fetale al 10% in PBS (soluzione bloccante).
Nota : in questo passaggio, il protocollo potrebbe essere sospeso. I coprivetrini possono essere conservati durante la notte in frigorifero nella soluzione bloccante e il protocollo può essere continuato il giorno successivo.
5. Incubazione dei vetrini con l'anticorpo primario
- Preparare una soluzione 1:1.000 di anti-LC3 nella soluzione bloccante e mantenerla su ghiaccio.
- Posizionare un pezzo di pellicola sigillante da laboratorio sul coperchio multi-pozzetto.
- Porre una goccia (25 μL) per coprivetrino di soluzione anti-LC3 sulla pellicola sigillante.
- Prendere ogni foglietto con una pinzetta e posizionarlo sopra la goccia di anticorpi primari, avendo cura che il lato cellulare sia in contatto con la soluzione.
- Preparare una camera umida mettendo un pezzo di carta umido in una scatola di plastica a fondo piatto.
- Posizionare la piastra multipozzetto nella camera di umidità, coprirla con un foglio e incubare per una notte in frigorifero.
6. Incubazione dei vetrini di copertura con l'anticorpo secondario
- Rimuovere la piastra multipozzetto dalla camera di umidità e riposizionare i coperchi nella piastra multipozzetto.
- Eseguire tre lavaggi con PBS.
- Preparare una soluzione di anti-coniglio marcata con fluorescenza con una diluizione di 1:800 nella soluzione bloccante e mantenerla su ghiaccio al riparo dalla luce.
- Posizionare un pezzo di pellicola sigillante sul coperchio multi-pozzetto.
- Introdurre una goccia (25 μL) per coprivetrino di soluzione anti-coniglio sulla pellicola sigillante.
- Prendere ogni foglietto con una pinzetta e posizionarlo sopra la goccia di anticorpi primari, avendo cura che il lato cellulare sia in contatto con la soluzione.
- Incubare la piastra multipozzetto nella camera di umidità per 2 ore a temperatura ambiente (RT) al riparo dalla luce.
7. Colorazione delle cellule con 4′ ,6-diamidino-2-fenilindolo (DAPI)
- Rimuovere la piastra multipozzetto dalla camera di umidità e riposizionare i coperchi nella piastra multipozzetto.
- Eseguire tre lavaggi con PBS.
- Preparare una soluzione 300 nM di DAPI in PBS (protetto dalla luce).
- Incubare ogni coprivetrino con la soluzione DAPI per 10 minuti.
- Eseguire tre lavaggi con PBS. Mantenere la piastra multi-pozzetto al riparo dalla luce.
8. Montaggio
- Preparare due bicchieri con acqua e un pezzo di carta.
- Porre una goccia (10 μL) per coprivetrino di una soluzione di addotto di alcool polivinilico Bis(trimetilalluminio)-1,4-diazabiciclo[2.2.2]ottano (PVA-DABCO) su un vetrino.
NOTA: PVA-DABCO viene preparato combinando 0,25 M DABCO, 10% W/V PVA, 20% glicerolo e 50% Tris HCl (1,5 M, pH 8,8) in acqua ultrapura. - Prendere ogni foglietto con una pinzetta, lavarlo in ogni bicchiere d'acqua, asciugarlo sulla carta e posizionarlo sopra la goccia PVA-DABCO (con le cellule a contatto con la soluzione).
- Lasciare asciugare per una notte, al riparo dalla luce.
9. Visualizzazione al microscopio confocale e acquisizione di immagini
- Visualizza i vetrini in un microscopio confocale invertito usando un obiettivo di circa 63x17.
- Acquisire immagini rappresentative delle celle etichettate.
10. Quantificare i punti LC3
- Trascinare e rilasciare ogni file di immagine contenente i canali acquisiti, ad esempio ".czi", nella schermata ImageJ (FIJI) per aprire. Fare clic su OK nella finestra di dialogo e chiudere la finestra della console .
- Dalla scheda Immagine , selezionare Colore > Dividi canali.
- Chiudere le immagini corrispondenti ai canali diversi dall'immagine LC3.
- Dalla scheda Immagine , selezionare Regola > bilanciamento colori
- Spostare il dispositivo di scorrimento Massimo verso sinistra fino a saturare l'immagine per visualizzare i contorni delle celle.
- Disegnate il contorno della cella con lo strumento selezione a mano libera .
- Fare clic sul pulsante Ripristina per ripristinare la regolazione del colore.
- Nella scheda Modifica selezionare Taglia per tagliare l'elemento selezionato.
- Chiudere l'immagine senza salvarla.
- Nella scheda Modifica selezionare Incolla.
- Nel menu Analizza , scegliete lo strumento Contatore oggetti 3D.
- Impostare la soglia. Nell'esempio fornito in questo studio, la soglia è fissata a 2.000.
- Impostare il filtro delle dimensioni. In questo studio, è impostato tra 50 e 500.
- Assicurarsi che le caselle Oggetti e Riepilogo siano contrassegnate.
- Fare clic su OK. Il numero di punti verrà descritto come Oggetti rilevati nel Riepilogo.
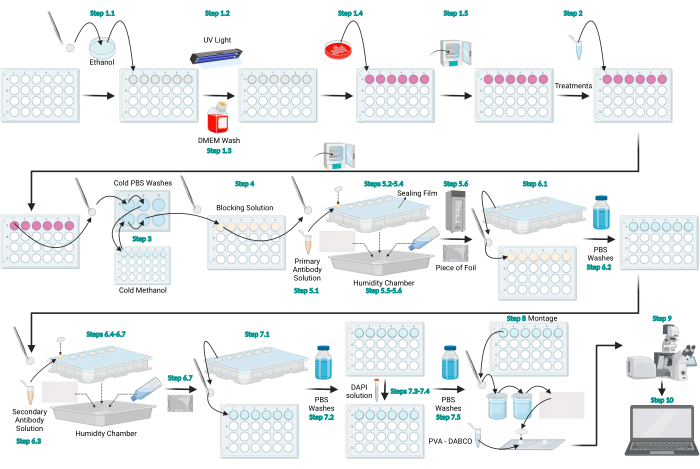
Figura 1: Schema del protocollo di immunofluorescenza CL3. Schema che rappresenta il protocollo generale previsto per l'immunofluorescenza della CL3. Figura creata con BioRender.com. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Questo protocollo esegue l'immunofluorescenza di CL3 in linee cellulari pancreatiche per determinare i livelli di autofagia in diverse condizioni. Il risultato di questo esperimento è stato l'ottenimento di immagini cellulari dai canali rosso e blu, corrispondenti a LC3 e DAPI. Le immagini LC3 indicano la distribuzione cellulare di questa proteina, mentre il DAPI mostra la localizzazione nucleare. La Figura 2A mostra un'immagine rappresentativa dell'immunofluorescenza di LC3 e della sua fus...
Access restricted. Please log in or start a trial to view this content.
Discussione
Il metodo descritto in questo protocollo consente di visualizzare la distribuzione endogena di LC3 nella cellula e quantificare i livelli autofagici in diverse condizioni. Un altro metodo simile utilizzato per analizzare la distribuzione di LC3 e determinare l'attivazione dell'autofagia coinvolge la trasfezione LC3 marcata con fluorescenza (come RFP-LC3)19. La trasfezione RFP-LC3 ha il vantaggio di non aver bisogno di fissazione (che consente di applicare questo metodo nell'imaging di cellule vive...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Non sono stati dichiarati conflitti di interesse.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni dell'Università di Buenos Aires (UBACyT 2018-2020 20020170100082BA), del Consiglio nazionale per la ricerca scientifica e la tecnologia (CONICET) (PIP 2021-2023 GI− 11220200101549CO; e PUE 22920170100033) e dell'Agenzia nazionale per la promozione scientifica e tecnologica (PICT 2019-01664).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 10x Phosphate-Buffered Saline (PBS) | Corning | 46-013-CM | |
| 12 mm round coverslips | HDA | CBR_OBJ_6467 | |
| 24 Well- Cell Culture Plate | Sorfa | 220300 | |
| Absolute ethanol | Biopack | 2207.10.00 | |
| Alexa Fluor 594 Donkey anti-rabbit IgG (H+L) | Invitrogen | R37119 | |
| Confocal Laser Scanning Microscope | Zeiss | LSM 800 | |
| Dexamethasone | Sigma Aldrich | D4902 | |
| DMEN | Sartorius | 01-052-1A | |
| Fetal Bovine Serum | NATOCOR | Lintc-634 | |
| Gemcitabina | Eli Lilly | VL7502 | |
| LC3B (D11) XP Rabbit mAb | Cell Signaling Technology | 3868S | |
| Methanol | Anedra | 6197 | |
| Parafilm "M" (Laboratory Sealing Film) | Bemis/Curwood | PM-996 | |
| Pen-Strep Solution | Sartorius | 03-031-1B | |
| PP242 | Santa Cruz Biotechnology | SC-301606 | |
| Trypsin EDTA | Gibco | 11570626 |
Riferimenti
- Grasso, D., Renna, F. J., Vaccaro, M. I. Initial steps in mammalian autophagosome biogenesis. Frontiers in Cell and Developmental Biology. 6, 146(2018).
- Galluzzi, L., et al. Molecular definitions of autophagy and related processes. EMBO Journal. 36 (13), 1811-1836 (2017).
- Kitada, M., Koya, D. Autophagy in metabolic disease and ageing. Nature Reviews Endocrinology. 17 (11), 647-661 (2021).
- Ktistakis, N. T., Tooze, S. A. Digesting the expanding mechanisms of autophagy. Trends in Cell Biology. 26 (8), 624-635 (2016).
- Tanida, I., Ueno, T., Kominami, E. LC3 and autophagy. Methods in Molecular Biology. 445, 77-88 (2008).
- Kabeya, Y., et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO Journal. 19 (21), 5720-5728 (2000).
- Mizushima, N., Yoshimori, T. How to interpret LC3 immunoblotting. Autophagy. 3 (6), 542-545 (2007).
- Vanasco, V., et al. Mitochondrial dynamics and VMP1-related selective mitophagy in experimental acute pancreatitis. Frontiers in Cell and Developmental Biology. 9, 640094(2021).
- Williams, J. A. Regulatory mechanisms in pancreas and salivary acini. Annual Review of Physiology. 46, 361-375 (1984).
- Mizrahi, J. D., Surana, R., Valle, J. W., Shroff, R. T. Pancreatic cancer. Lancet. 395 (10242), 2008-2020 (2020).
- Li, J., et al. Regulation and function of autophagy in pancreatic cancer. Autophagy. 17 (11), 3275-3296 (2021).
- Ropolo, A., et al. A novel E2F1-EP300-VMP1 pathway mediates gemcitabine-induced autophagy in pancreatic cancer cells carrying oncogenic KRAS. Frontiers in Endocrinology. 11, 411(2020).
- Pardo, R., et al. Gemcitabine induces the VMP1-mediated autophagy pathway to promote apoptotic death in human pancreatic cancer cells. Pancreatology. 10 (1), 19-26 (2010).
- Logsdon, C. D., Moessner, J., Williams, J. A., Goldfine, I. D. Glucocorticoids increase amylase mRNA levels, secretory organelles, and secretion in pancreatic acinar AR42J cells. Journal of Cell Biology. 100 (4), 1200-1208 (1985).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy (4th edition). Autophagy. 17 (1), 1(2021).
- Segeritz, C. -P., Vallier, L. Chapter 9 - Cell culture: Growing cells as model systems in vitro. Basic Science Methods for Clinical Researchers. Jalali, M., Saldanha, F. Y. L., Jalali, M. , Academic Press. Cambridge, MA. 151-172 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Current Protocols in Cytometry. 92 (1), 68(2020).
- Grasso, D., et al. a novel selective autophagy pathway mediated by VMP1-USP9x-p62, prevents pancreatic cell death. Journal of Biological Chemistry. 286 (10), 8308-8324 (2011).
- Ropolo, A., et al. The pancreatitis-induced vacuole membrane protein 1 triggers autophagy in mammalian cells. Journal of Biological Chemistry. 282 (51), 37124-37133 (2007).
- Karanasios, E., Stapleton, E., Walker, S. A., Manifava, M., Ktistakis, N. T. Live cell imaging of early autophagy events: Omegasomes and beyond. Journal of Visualized Experiments. (77), e50484(2013).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Zhang, Z., Singh, R., Aschner, M. Methods for the detection of autophagy in mammalian cells. Current Protocols in Toxicology. 69, 1-26 (2016).
- Yoshii, S. R., Mizushima, N. Monitoring and measuring autophagy. International Journal of Molecular Sciences. 18 (9), 1865(2017).
- Betriu, N., Andreeva, A., Semino, C. E. Erlotinib promotes ligand-induced EGFR degradation in 3D but not 2D cultures of pancreatic ductal adenocarcinoma cells. Cancers. 13 (18), 4504(2021).
- Wang, W., Dong, L., Zhao, B., Lu, J., Zhao, Y. E-cadherin is downregulated by microenvironmental changes in pancreatic cancer and induces EMT. Oncology Reports. 40 (3), 1641-1649 (2018).
- Kim, S. K., et al. Phenotypic heterogeneity and plasticity of cancer cell migration in a pancreatic tumor three-dimensional culture model. Cancers. 12 (5), 1305(2020).
- Meng, Y., et al. Cytoplasmic EpCAM over-expression is associated with favorable clinical outcomes in pancreatic cancer patients with Hepatitis B virus negative infection. International Journal of Clinical and Experimental Medicine. 8 (12), 22204-22216 (2015).
- Brock, R., Hamelers, I. H., Jovin, T. M. Comparison of fixation protocols for adherent cultured cells applied to a GFP fusion protein of the epidermal growth factor receptor. Cytometry. 35 (4), 353-362 (1999).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon