È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Una serie di tecniche di screening per una rapida panoramica della funzione dei neutrofili
In questo articolo
Riepilogo
Questo protocollo presenta una serie di saggi funzionali dei neutrofili da utilizzare come metodo di screening per coprire le funzioni di diverse vie di segnalazione. Il protocollo include una valutazione iniziale e semplice della vitalità cellulare, della purezza, della produzione di specie reattive dell'ossigeno, della migrazione in tempo reale, della fagocitosi e di un suggerimento preliminare di trappole extracellulari per neutrofili.
Abstract
I neutrofili sono noti come una delle prime linee di difesa nella risposta immunitaria innata e possono svolgere molte funzioni cellulari particolari, come la chemiotassi, la migrazione inversa, la fagocitosi, la degranulazione di enzimi e metaboliti citotossici e il rilascio di DNA come trappole extracellulari per neutrofili (NET). I neutrofili non solo hanno una segnalazione strettamente regolata, ma partecipano anche alla regolazione di altri componenti del sistema immunitario. Poiché i neutrofili freschi sono terminalmente differenziati, di breve durata e altamente variabili tra gli individui, è importante sfruttare al meglio i campioni raccolti. I ricercatori hanno spesso bisogno di eseguire saggi di screening per valutare una panoramica delle numerose funzioni dei neutrofili che possono essere influenzate da condizioni specifiche in fase di valutazione. Per rispondere a questa esigenza è stata sviluppata una serie di test che seguono un singolo processo di isolamento di neutrofili a densità normale, cercando un equilibrio tra velocità, completezza, costi e precisione. I risultati possono essere utilizzati per ragionare e guidare studi di follow-up approfonditi. Questa procedura può essere eseguita in un tempo medio di 4 ore e include la valutazione della vitalità cellulare, la produzione di specie reattive dell'ossigeno (ROS), la migrazione in tempo reale e la fagocitosi del lievito su vetrini, lasciando un numero sufficiente di cellule per approcci più dettagliati come gli studi omici. Inoltre, la procedura include un modo per osservare facilmente un suggerimento preliminare di NET dopo una rapida colorazione panottica osservata dalla microscopia ottica, con una mancanza di marcatori specifici, anche se sufficienti per indicare se ulteriori sforzi in questo modo varrebbero la pena. La diversità delle funzioni testate combina punti comuni tra i test, riducendo i tempi e i costi di analisi. La procedura è stata chiamata NeutroFun Screen e, pur avendo delle limitazioni, bilancia i fattori sopra citati. Inoltre, lo scopo di questo lavoro non è un set di test definito, ma piuttosto una linea guida che può essere facilmente adattata alle risorse e alle esigenze di ciascun laboratorio.
Introduzione
I neutrofili sono le cellule immunitarie innate più abbondanti nel sangue umano e sono noti per svolgere un ruolo importante nelle infezioni e nell'infiammazione, essendo i primi soccorritori ad arrivare al sitodel danno tissutale. Negli ultimi anni, c'è stato un crescente riconoscimento del ruolo cruciale che i neutrofili svolgono in una varietà di malattie e nel sostenere l'omeostasi2. I neutrofili non solo hanno una segnalazione strettamente regolata, ma partecipano anche alla regolazione di altri componenti del sistema immunitario 3,4,5. Pertanto, lo studio dei neutrofili e delle loro numerose funzioni cellulari insolite, come la chemiotassi, la migrazione inversa6, la fagocitosi7, il burst respiratorio8 e il rilascio di trappole extracellulari per neutrofili (NET)7, è fondamentale in numerosi contesti di ricerca in cui è necessario valutare i potenziali cambiamenti funzionali, morfologici o molecolari dei neutrofili innescati da specifiche condizioni in analisi.
I neutrofili appena isolati sono terminalmente differenziati, di breve durata, altamente dinamici e facilmente attivabili9. Tuttavia, non è stato ancora raggiunto un metodo di conservazione efficiente che non influisca sulle risposte dei neutrofili, rendendo difficile l'esecuzione di più saggi che devono essere ininterrotti. Inoltre, le analisi funzionali10,11 precedentemente descritte, basate su saggi che richiedono citometria e/o colorazione fluorescente, potrebbero non essere una scelta praticabile quando è necessaria un'ampia e iniziale valutazione del neutrofilo.
Per affrontare questi problemi, questo protocollo descrive una serie di test che possono essere eseguiti a seguito di un singolo processo di isolamento, tra cui la valutazione della vitalità cellulare, la produzione di specie reattive dell'ossigeno (ROS), la migrazione in tempo reale e la fagocitosi di Saccharomyces cerevisiae, i cui risultati possono essere utilizzati per ragionare studi di follow-up approfonditi. Questa procedura, denominata NeutroFun Screen, è stata progettata per comprendere le principali attività effettrici, ad eccezione della degranulazione, e può essere completata in un tempo medio di 4 ore, inclusa 1 ora di attivazione. Inoltre, le cellule rimanenti possono essere utilizzate per approcci più dettagliati come gli studi omici. Il vantaggio di questo metodo risiede nel suo equilibrio tra velocità, completezza, costo e precisione.
Inoltre, c'è un modo per osservare facilmente un suggerimento preliminare di NET, senza marcatori specifici, ma sufficiente per indicare se ulteriori sforzi in quella direzione varrebbero la pena. La diversità delle funzioni testate ha lo scopo di combinare punti comuni tra i test, riducendo i tempi e i costi di analisi. L'obiettivo principale di questo metodo è quello di fornire un'analisi funzionale bilanciata per quanto riguarda la velocità, la completezza, il costo e l'accuratezza che consenta una panoramica della risposta del neutrofilo, rendendolo un utile passo iniziale nello studio degli effetti di nuovi stimoli sui neutrofili a densità normale.
Protocollo
Tutti gli esperimenti hanno seguito rigorosamente le linee guida etiche stabilite dal comitato di revisione istituzionale dell'Università di Brasilia (processo 13364819.0.0000.5558) e i campioni sono stati identificati da codici per garantire l'anonimato del donatore. Le cellule sono state ottenute da donatori maschi sani normali di età compresa tra 18 e 35 anni, che hanno firmato il consenso informato e hanno soddisfatto i seguenti criteri di idoneità: non fumatori/vapers, nessuna condizione di salute cronica e nessuna storia di condizioni infiammatorie negli ultimi 14 giorni.
1. Prelievo di sangue
- Mettere asetticamente 0,3 mL di eparina da 5.000 UI/mL (vedere Tabella dei materiali) in una siringa sterile da 20 mL per eparinizzarla.
- Applicare un laccio emostatico venoso a circa 4 pollici sopra il sito di puntura e identificare la vena cubitale o cefalica mediana per la venipuntura.
NOTA: Assicurarsi che il tempo totale del laccio emostatico non superi 1 minuto. - Pulire il sito della puntura con alcol al 70% ed eseguire la puntura venosa.
- Capovolgere delicatamente la siringa tre o quattro volte dopo aver raccolto il sangue per mescolare correttamente il sangue e l'eparina.
2. Isolamento dei neutrofili
NOTA: I leucociti polimorfonucleati (PMN) vengono isolati mediante centrifugazione in gradiente di densità seguita da lisi ipotonica dei restanti globuli rossi (RBC), come descritto in precedenza11 con alcune modifiche. Questo metodo non è obbligatorio per eseguire i saggi di screening e può essere sostituito a condizione che il metodo scelto si traduca in una vitalità del >97%, nell'innesco o nell'attivazione del <3% dei PMN e produca cellule sufficienti per tutti i saggi, le repliche e le condizioni. L'esecuzione di questi passaggi in condizioni asettiche e l'utilizzo di soluzioni prive di endotossine sono obbligatorie per evitare l'attivazione cellulare.
- Effettuare diluizioni da 12 mL di terreni di separazione al 60% e al 70% (disponibili in commercio; vedere la tabella dei materiali) in provette coniche da 50 mL.
- Preparare il gradiente dal basso verso l'alto aggiungendo 4 mL alla volta della diluizione del 60% rispetto alla diluizione del 70%, utilizzando una pipetta da 5 mL. Fallo delicatamente per evitare di confondere l'interfaccia.
- Stratificare accuratamente 12 mL di sangue eparinizzato sopra il gradiente di densità. Centrifugare a 200 x g per 15 min a temperatura ambiente.
NOTA: Da questa fase in poi, fino all'attivazione del PMN, tutti i reagenti e le provette utilizzate devono essere conservati in un refrigeratore riempito di ghiaccio. - Scartare lo strato plasma/cellula mononucleata, quindi trasferire delicatamente lo strato sopra il pellet eritrocitario in due provette coniche da 15 mL con circa 7,5 mL ciascuna. Aumentare il volume della provetta con la soluzione salina bilanciata di Hank (HBSS; vedere la tabella dei materiali).
- Centrifugare a 300 x g per 5 min a 19 °C.
- Lavare il pellet cellulare con HBSS.
- Scartare il surnatante versando la provetta e risospendere delicatamente il pellet in 7 mL di HBSS.
- Centrifugare a 300 x g per 5 minuti a 19 °C per rimuovere tutti i mezzi di separazione.
- Eseguire la lisi ipotonica dei restanti globuli rossi.
- Scartare il surnatante e unire i pellet in un unico tubo.
- Risospendere il pellet RBC/PMN in 3 mL di H2O sterile e aggiungere 3 mL di HBSS (2x) entro 25 s per ripristinare l'osmolarità. Quindi, centrifugare a 300 x g per 5 minuti a 19 °C.
- Ripetere i passaggi 2.8.1 e 2.8.2 per un pellet bianco privo di eritrociti.
NOTA: Il surnatante deve essere rimosso il prima possibile per ridurre al minimo il contatto dei neutrofili con i prodotti di degradazione dei globuli rossi. In alternativa, la seconda lisi ipotonica può essere sostituita risospendendo delicatamente lo strato di globuli rossi residui e rimuovendo tutto il surnatante, poiché i restanti globuli rossi sedimenteranno sopra il pellet PMN.
- Scartare il surnatante versando la provetta, risospendere delicatamente i PMN nel tampone rimanente e trasferirli in una microprovetta ghiacciata.
NOTA: Assicurarsi di annotare il volume durante il trasferimento delle cellule risospese con una micropipetta. - Trasferire 3 x 1 μL della sospensione cellulare su un vetrino pulito (tre pozzetti da 1 μL ciascuno) e colorare con un panottico veloce (vedere la tabella dei materiali) per la valutazione della morfologia e della purezza12.
- Per colorare con il panottico veloce, immergere il vetrino cinque volte nel fissativo panottico n° 1, sei volte nell'eosina n° 2 e due volte nell'ematossilina n° 3, con ogni immersione della durata di 1 s.
- Lavare delicatamente il vetrino con acqua distillata.
- Lasciar scolare e asciugare all'aria.
- Osservate al microscopio e contate 300 cellule casuali in ogni pozzetto, differenziando così i neutrofili dagli altri granulociti.
- Trasferire 1 μL della sospensione cellulare a 49 μL di colorante blu tripano13 allo 0,2% e contare le cellule utilizzando una camera di Neubauer, distinguendo tra cellule morte e vitali.
- Regolare la concentrazione cellulare a 6.667 cellule/μL utilizzando una soluzione di plasma autologo al 50% e HBSS al 50% integrata con calcio e magnesio. Dividere equamente le 6.667 cellule/μL di sospensione tra le microprovette corrispondenti alle condizioni da testare, compreso il controllo negativo.
NOTA: È possibile utilizzare qualsiasi concentrazione cellulare simile ai neutrofili circolanti nell'organismo modello, ma è importante utilizzare la stessa concentrazione cellulare in tutti gli esperimenti per la riproducibilità.
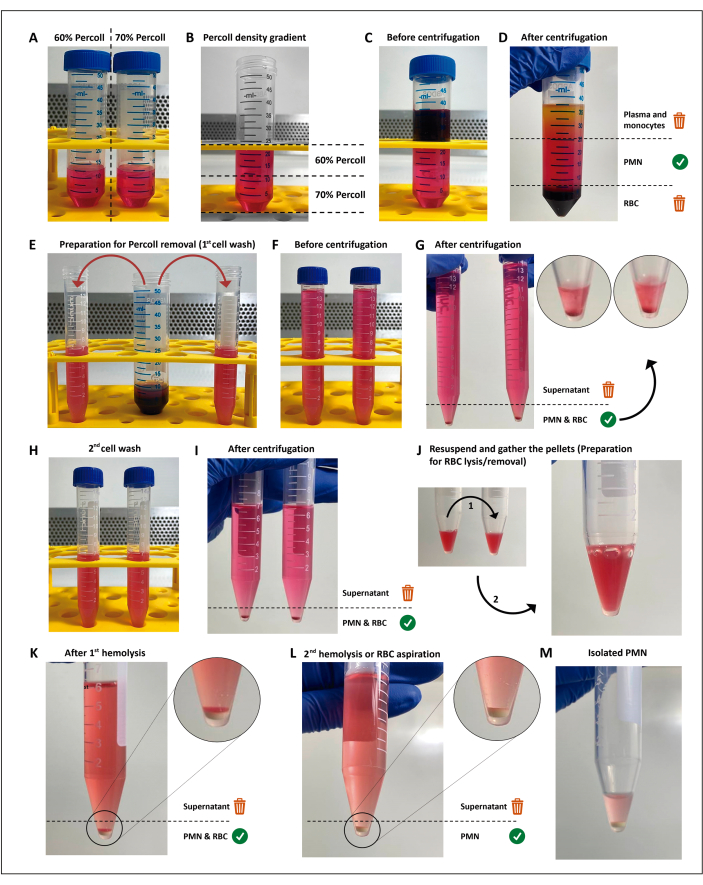
Figura 1: Il protocollo di isolamento dei neutrofili. Due concentrazioni del mezzo di separazione (percoll) (A) vengono impilate (B), quindi il sangue viene stratificato sopra il gradiente di separazione (C). Dopo la centrifugazione, il PMN si trova nello strato centrale (D), che è diviso in due provette da 15 mL (E). La sospensione cellulare viene lavata due volte in HBSS e centrifugata (G-I) per rimuovere il terreno, quindi le cellule vengono risospese e i globuli rossi residui vengono sottoposti a due cicli di lisi ipotonica (J-M). Fare clic qui per visualizzare una versione più grande di questa figura.
3. Preparazione per l'attivazione dei neutrofili
- In microprovette da 1,5 mL, preparare un sistema di attivazione per ogni condizione in modo che la concentrazione cellulare finale sia di 6.600 cellule/μL. Ad esempio, per testare gli effetti della fMLP 100 nM (N-formil-metionil-leucil-fenilalanina; vedere la tabella dei materiali), aggiungere 5 μL di 10 μM fMLP a 495 μL della sospensione di 6.667 cellule/μL. Per il controllo negativo (non stimolato), aggiungere HBSS contenente Ca2+ e Mg2+.
NOTA: Per dimostrare questa metodologia, sono state utilizzate le seguenti concentrazioni finali degli stimoli: 100 nM fMLP, 16 μM di fallaxina, un peptide antimicrobico14 presente in natura, e 100 nM PMA (forbolo 12-miristato 13-acetato) (vedi Tabella dei materiali). - Incubare a 37 °C senza rotazione.
NOTA: Tutte le aliquote per i saggi funzionali sono prelevate da questa sospensione cellulare, di seguito denominata sistema di attivazione.
4. Saggio del cloruro blu di nitrotetrazolio (NBT) per la valutazione della produzione di ROS
- Preparazione della soluzione di lavoro NBT: Per ogni condizione sperimentale, preparare una soluzione di lavoro NBT (vedi Tabella dei materiali) di 6 mM seguendo le seguenti fasi:
- Sciogliere 0,0005 g di NBT in 10 μL di dimetilsolfossido (DMSO) e vortice per almeno 15 min.
- Aggiungere 90 μL di HBSS Ca2+Mg2+ e vorticare fino a 2 min.
NOTA: Tutti i passaggi che coinvolgono NBT devono essere eseguiti al buio.
- Eseguire il test del vetrino NBT.
- Dopo 20 minuti di attivazione cellulare, mescolare delicatamente la sospensione cellulare e trasferire con cautela 2 μL di PMN su un vetrino pulito. Incubare per 20 minuti in camera umidificata a 37 °C.
NOTA: Non stendere troppo la sospensione cellulare sul vetrino; in caso contrario, potrebbe seccarsi prima dell'incubazione. - Aggiungere 1 μL della soluzione di lavoro NBT sulle cellule e incubare ulteriormente per 20 minuti al riparo dalla luce.
- Asciugare il vetrino con aria calda e fissarlo con una goccia di metanolo in ogni pozzetto per 1 min. Colorare con safranina allo 0,03% (vedi Tabella dei Materiali) per 1 min.
- Lavare delicatamente il vetrino con acqua distillata.
- Lasciare asciugare il vetrino all'aria e osservarlo al microscopio.
- Contare 100 cellule casuali in ogni pozzetto, differenziando i neutrofili con e senza depositi di formazano.
- Dopo 20 minuti di attivazione cellulare, mescolare delicatamente la sospensione cellulare e trasferire con cautela 2 μL di PMN su un vetrino pulito. Incubare per 20 minuti in camera umidificata a 37 °C.
- Eseguire il test di spettrofotometria NBT.
- Dopo 40 minuti di attivazione cellulare, mescolare delicatamente la sospensione cellulare e trasferire 90 μL di PMN dal sistema di attivazione a un microtubo pulito. Quindi, aggiungere con cautela 20 μL della soluzione NBT 6 mM. Incubare al buio per 20 minuti a 37 °C.
- Aggiungere 100 μL di sodio dodecil solfato al 10% (SDS; vedere la tabella dei materiali) e vortice.
- Sonicare utilizzando un sonicatore a punta con un'ampiezza del 60%, cinque cicli di 15 s ciascuno con intervalli di 15 s. Centrifugare a 12.000 x g per 5 min.
- Trasferire 60 μL del surnatante su una piastra a 96 pozzetti a fondo trasparente e misurare l'assorbanza del prodotto formazan a 570 nm.
5. Saggio di fagocitosi
- Preparare una sospensione di 33.000 lieviti/μL per ogni condizione, come descritto di seguito:
- Aggiungere circa 0,75 mg di lievito secco (Saccharomyces cerevisiae; vedi tabella dei materiali) a 200 μL di HBSS Ca2+Mg2+ e incubare in un termomiscelatore a 100 °C a 500 giri/min per almeno 15 minuti.
- Omogeneizzare la miscela mediante vortex e trasferire 5 μL di sospensione di lievito in 45 μL di colorante blu tripano allo 0,2%. Contare i lieviti con una camera di Neubauer.
- Regolare la concentrazione della sospensione iniziale a 33.000 cellule di lievito/μL utilizzando HBSS Ca2+Mg2+. Tenere la sospensione sul ghiaccio fino all'uso.
- Dopo 20 minuti di attivazione cellulare, mescolare delicatamente la sospensione cellulare e trasferire 5 μL del sistema di attivazione a 5 μL dei 33.000 lieviti/μL di sospensione di Saccharomyces cerevisiae in una nuova microprovetta sterile.
NOTA: Il rapporto neutrofili/lieviti è 1:5 (PMN:lievito). - Trasferire immediatamente 6 μL di sospensione di PMN/lievito in tre pozzetti di un vetrino pulito (2 μL ciascuno) e incubare il vetrino in una camera umidificata per 40 minuti.
NOTA: Non stendere troppo la sospensione cellulare sul vetrino; in caso contrario, potrebbe seccarsi prima dell'incubazione. - Asciugare il vetrino sotto l'aria calda e macchiare con un panottico rapido, come descritto al punto 2.9 sopra.
NOTA: La terza fase della colorazione rapida del panottico è fondamentale per l'analisi microscopica del vetrino. Colorare il vetrino per ≥3 s in questa fase può renderlo inadatto all'analisi, poiché sarà difficile differenziare il lievito dai lobi nucleari dei neutrofili. - Osservare i vetrini al microscopio, contando 100 neutrofili casuali di ciascun pozzetto e discriminando tra PMN positivi e negativi per la fagocitosi.
NOTA: Almeno una particella di lievito all'interno o a diretto contatto con la membrana cellulare PMN indica un PMN positivo per la fagocitosi. Se sei interessato al rapporto lievito/neutrofili, conta anche il numero di particelle di lievito inghiottite.
6. Saggio di chemiotassi PMN in tempo reale
NOTA: Il test di migrazione viene eseguito in modo simile al protocollo descritto in precedenza15, con i seguenti adattamenti:
- Preparare il gradiente chemiotattico aggiungendo 160 μL di chemioattrattivo (ad es. fMLP, IL-8, C5 o LTB4; vedere la tabella dei materiali) alla camera inferiore di una piastra RTCA (Real Time Cell Analyzer) basata sull'impedenza. Per i controlli negativi e i bianchi, aggiungere 160 μL di HBSS Ca2+Mg2+.
- Collegare la camera superiore e aggiungere 25 μL di HBSS Ca2+Mg2+. Incubare a temperatura ambiente per almeno 1 ora per formare il gradiente chemiotattico.
- Dopo 60 minuti di attivazione cellulare, mescolare delicatamente la sospensione cellulare e posizionare 60 μL di sospensione cellulare nella camera superiore. Aggiungere 60 μL di HBSS Ca2+Mg2+ al bianco.
- Posizionare la piastra RTCA e programmare il software RTCA per misurare l'indice di cella (CI) ogni 60 s per 2 ore.
NOTA: Le piastre RTCA possono essere lavate per il riutilizzo come descritto in precedenza16. In sintesi, lavare le camere RTCA e gli elettrodi con soluzione salina fosfatata (PBS) tre volte, quindi con acqua ultrapura di tipo I due volte. Incubare la camera inferiore e superiore con tripsina 0,25% 0,53 mM di acido etilendiamminotetraacetico (EDTA) per 40 minuti. Lavare con acqua ultrapura tre volte.
7. Saggio suggestivo NET
- Dopo 10 minuti di attivazione cellulare, miscelare delicatamente la sospensione cellulare e trasferire 4 μL di PMN da ciascun sistema di attivazione in esame, diviso in due pozzetti di un vetrino pulito. Incubare in camera umidificata a 37 °C per 30 min.
- Aggiungere 1 μL di DNAsi I in uno dei pozzetti e incubare per 20 minuti a 37 °C (in camera umida).
- Asciugare il vetrino e la macchia con un panottico veloce, come descritto in precedenza nel passaggio 2.9 della sezione "Isolamento dei neutrofili".
- Valutare i vetrini al microscopio.
NOTA: Cercare qualsiasi indicazione di rilascio di NET, caratterizzata dalla presenza di strutture simili a Web. Una volta identificato, verificare se il trattamento con DNAsi I è stato in grado di rimuovere tali strutture. Questo test è indicativo della formazione di NET, poiché sono necessari ulteriori test per confermarne la presenza.
Risultati
Il metodo di isolamento basato sulla densità utilizzato in questo studio (Figura 1) ha soddisfatto i criteri per gli esperimenti proposti. I parametri dei neutrofili ottenuti con questo metodo includevano vitalità ≥98%, purezza ≥94% e resa cellulare ≥1,5 x 107, senza alcuna attivazione rilevabile dai test di screening. Due fasi rilevanti nell'isolamento dei PMN sono l'anticoagulazione e la rimozione dei globuli rossi. Mantenere la provetta o la siringa anticoagulata a una ...
Discussione
I neutrofili sono cellule altamente dinamiche e reattive che hanno vita breve e non possono ancora essere crioconservate19, rendendo difficili le indagini sulla loro biologia. Pertanto, è essenziale seguire attente misure per ottenere neutrofili vitali, arricchiti e a riposo11,20. Questo studio ha impiegato una tecnica di isolamento basata sulla densità che enfatizza la manipolazione delicata e minima, nonché l'uso di basse temperature ...
Divulgazioni
Gli autori dichiarano di non avere alcun conflitto di interessi.
Riconoscimenti
Gli autori ringraziano le seguenti agenzie di finanziamento: FAPDF, CNPq, CAPES, UnB, FINEP e FINATEC.
Materiali
| Name | Company | Catalog Number | Comments |
| CIM-Plate 16 | Agilent | 5665825001 | |
| CLARIOstar Plate Reader | BMG LABTECH | US Patent Number 9,733,124 Product details: MARS Data Analysis Software | |
| Dimethyl sulfoxide | Dinâmica | 1582 | |
| DNAse I | Sigma - Aldrich | DN 25 | |
| Ethylenediaminetetraacetic acid disodium salt dihydrate | Sigma - Aldrich | E5134 | |
| Fast panoptic stain | Laborclin | 620529 | |
| Glass slide | Exacta | 7102 | |
| Hank’s Balanced Salt Solution with calcium, with magnesium, without phenol red. | Sigma - Aldrich | 55037C | |
| Hank’s Balanced Salt Solution without calcium chloride, magnesium sulfate and sodium bicarbonate. | Sigma - Aldrich | H4641 | |
| Heparin | Blau | 7896014655229 | |
| Laminar flow cabinet | Veco | VLFS-12 | |
| Microscope | Zeiss | 415501-0101-002 | Product details: Primostar 1 |
| Mixing Block | BIOER | MB-102 | |
| Neubauer improved bright-lined | New Optik | 1110000 | |
| N-formyl-methionyl-leucyl-phenylalanine | Sigma - Aldrich | F3506 | |
| Nitroblue tetrazolium | Neon | CAS 298-83-9 | |
| Percoll | Cytiva | 17089101 | separation media |
| Phorbol 12-myristate 13-acetate | Sigma - Aldrich | P8139 | |
| Phosphate buffered saline tablet | Sigma - Aldrich | P4417 | |
| ROTOFIX 32 A | Hettich | 1206 | |
| Saccharomyces cerevisiae | Fleischmann | ||
| Safranin | Sigma - Aldrich | 50240 | |
| Sodium dodecyl sulfate | Cytiva | 17-1313-01 | |
| Sonicator | Qsonica | Q125 | |
| Trypan blue solution | Vetec | C.I. 23850 | |
| Vortex Genie 2 | Scientific Industries, Inc. | 0K-0500-902 | |
| xCELLigence Real-Time Cell Analysis (RTCA) DP (dual purpose) | Agilent | 380601050 | Product details: RTCA system composed of detection hardware, cell plates and software |
Riferimenti
- Nauseef, W. M., Borregaard, N. Neutrophils at work. Nature Immunology. 15 (7), 602-611 (2014).
- Groeneweg, L., Hidalgo, A. Emerging roles of infiltrating granulocytes and monocytes in homeostasis. Cellular and Molecular Life Sciences. 77 (19), 3823-3830 (2020).
- Rosales, C., Lowell, C. A., Schnoor, M., Uribe-Querol, E. Neutrophils: their role in innate and adaptive immunity 2017. Journal of Immunology Research. 2017, 9748345 (2017).
- Castro, M., et al. Proteome analysis of resting human neutrophils. Protein & Peptide Letters. 13 (5), 481-487 (2006).
- Li, Y., et al. The regulatory roles of neutrophils in adaptive immunity. Cell Communication and Signaling. 17, 147 (2019).
- de Oliveira, S., Rosowski, E. E., Huttenlocher, A. Neutrophil migration in infection and wound repair: going forward in reverse. Nature Reviews Immunology. 16 (6), 378-391 (2016).
- Burn, G. L., Foti, A., Marsman, G., Patel, D. F., Zychlinsky, A. The neutrophil. Immunity. 54 (7), 1377-1391 (2021).
- El-Benna, J., et al. Priming of the neutrophil respiratory burst: role in host defense and inflammation. Immunological Reviews. 273 (1), 180-193 (2016).
- Castro, M. S., Cilli, E. M., Fontes, W. Combinatorial synthesis and directed evolution applied to the production of alpha-helix forming antimicrobial peptides analogues. Current Protein & Peptide Science. 7 (6), 473-478 (2006).
- Mihaila, A. C., et al. Transcriptional profiling and functional analysis of N1/N2 neutrophils reveal an immunomodulatory effect of S100A9-blockade on the pro-inflammatory N1 subpopulation. Frontiers in Immunology. 12, 708770 (2021).
- Kuhns, D. B., Priel, D. A. L., Chu, J., Zarember, K. A. Isolation and functional analysis of human neutrophils. Current Protocols in Immunology. 111 (1), 7-23 (2015).
- Paulíková, E., Kociková, A., Sabol, M. Modification of a panoptic method of staining isolated cells. Bratislavske Lekarske Listy. 94 (12), 638-640 (1993).
- Strober, W. Trypan blue exclusion test of cell viability. Current Protocols in Immunology. 111 (1), 1-3 (2015).
- Libério, M. S., et al. Anti-proliferative and cytotoxic activity of pentadactylin isolated from Leptodactylus labyrinthicus on melanoma cells. Amino Acids. 40 (1), 51-59 (2011).
- Cano, P. M., Vargas, A., Lavoie, J. P. A real-time assay for neutrophil chemotaxis. BioTechniques. 60 (5), 245-251 (2016).
- Stefanowicz-Hajduk, J., Adamska, A., Bartoszewski, R., Ochocka, J. R. Reuse of E-plate cell sensor arrays in the xCELLigence Real-Time Cell Analyzer. BioTechniques. 61 (3), 117-122 (2016).
- Björkstén, B., Nyström, K., Lindqvist, B. The nitroblue tetrazolium (NBT) test in endemic benign (epidemic) nephropathy. Acta Medica Scandinavica. 199 (1-6), 147-150 (1976).
- Aquino, E., et al. Proteomic analysis of neutrophil priming by PAF. Protein & Peptide Letters. 23 (2), 142-151 (2016).
- Blanter, M., Gouwy, M., Struyf, S. Studying neutrophil function in vitro: cell models and environmental factors. Journal of Inflammation Research. 14, 141-162 (2021).
- Hsu, A. Y., Peng, Z., Luo, H., Loison, F. Isolation of human neutrophils from whole blood and buffy coats. Journal of Visualized Experiments. (175), e62837 (2021).
- Moghadam, Z. M., Henneke, P., Kolter, J. From flies to men: ROS and the NADPH oxidase in phagocytes. Frontiers in Cell and Developmental Biology. 9, 628991 (2021).
- Pattan, S. S., Bhat, K. G., Pattar, G. D., Kuntagi, M. Comparison of three different techniques for isolation of neutrophils from blood and their utility in performing nitroblue tetrazolium test. International Journal of Basic and Applied Physiology. 8 (1), 41 (2019).
- Gooty, J. R., Shashirekha, A., Guntakala, V. R., Palaparthi, R. Estimation of phagocytic activity of polymorphonuclear leukocytes in chronic and aggressive periodontitis patients with nitroblue tetrazolium test. Journal of Indian Society of Periodontology. 23 (4), 316 (2019).
- Langer, S., et al. Clinical and laboratory profiles of 17 cases of chronic granulomatous disease in north India. Indian Journal of Hematology and Blood Transfusion. 37 (1), 45-51 (2021).
- Oualha, R., et al. Infection of human neutrophils with Leishmania infantum or Leishmania major strains triggers activation and differential cytokines release. Frontiers in Cellular and Infection Microbiology. 9, 153 (2019).
- Zilinskas, J., Zekonis, J., Zekonis, G., Valantiejiene, A., Periokaite, R. The reduction of nitroblue tetrazolium by total blood in periodontitis patients and the aged. Stomatologijal. 9 (4), 105-108 (2007).
- Benov, L. Improved formazan dissolution for bacterial MTT assay. Microbiology Spectrum. 9 (3), e01637 (2021).
- Chen, Y., Junger, W. G. Measurement of oxidative burst in neutrophils. Methods in Molecular Biology. 844, 115-124 (2012).
- Richardson, M. P., Ayliffe, M. J., Helbert, M., Davies, E. G. A simple flow cytometry assay using dihydrorhodamine for the measurement of the neutrophil respiratory burst in whole blood: comparison with the quantitative nitrobluetetrazolium test. Journal of Immunological Methods. 219 (1-2), 187-193 (1998).
- Jancinová, V., et al. The combined luminol/isoluminol chemiluminescence method for differentiating between extracellular and intracellular oxidant production by neutrophils. Redox Report. 11 (3), 110-116 (2006).
- Nosál, R., et al. Pharmacological intervention with oxidative burst in human neutrophils. Interdisciplinary Toxicology. 10 (2), 56-60 (2017).
- Mol, S., et al. Efficient neutrophil activation requires two simultaneous activating stimuli. International Journal of Molecular Sciences. 22 (18), 10106 (2021).
- Schneider, L., et al. Flow cytometry evaluation of CD14/CD16 monocyte subpopulations in systemic sclerosis patients: a cross sectional controlled study. Advances in Rheumatology. 61 (1), 27 (2021).
- Akin, E., Pelen, N. N., Tiryaki, I. U., Yalcin, F. Parameter identification for gompertz and logistic dynamic equations. PLoS One. 15 (4), e0230582 (2020).
- Guy, J. B., et al. Evaluation of the cell invasion and migration process: A comparison of the video microscope-based scratch wound assay and the boyden chamber assay. Journal of Visualized Experiments. (129), e56337 (2017).
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- de Bont, C. M., Koopman, W. J. H., Boelens, W. C., Pruijn, G. J. M. Stimulus-dependent chromatin dynamics, citrullination, calcium signalling and ROS production during NET formation. Biochimica et Biophysica Acta. Molecular Cell Research. 1865, 1621-1629 (2018).
- Masuda, S., et al. Measurement of NET formation in vitro and in vivo by flow cytometry. Cytometry Part A. 91 (8), 822-829 (2017).
- Zharkova, O., et al. A flow cytometry-based assay for high-throughput detection and quantification of neutrophil extracellular traps in mixed cell populations. Cytometry Part A. 95 (3), 268-278 (2019).
- Hosseinnejad, A., et al. DNase I functional microgels for neutrophil extracellular trap disruption. Biomaterials Science. 10 (1), 85-99 (2022).
- Chrysanthopoulou, A., et al. Neutrophil extracellular traps promote differentiation and function of fibroblasts. The Journal of Pathology. 233 (3), 294-307 (2014).
- Tong, M., Abrahams, V. M. Visualization and quantification of neutrophil extracellular traps. Methods in Molecular Biology. 2255, 87-95 (2021).
- Santana, C. J. C., et al. Biological properties of a novel multifunctional host defense peptide from the skin secretion of the chaco tree frog, boana raniceps. Biomolecules. 10 (5), 790 (2020).
- Murphy, M. P., et al. Guidelines for measuring reactive oxygen species and oxidative damage in cells and in vivo. Nature Metabolism. 4 (6), 651-662 (2022).
- Boero, E., et al. Use of flow cytometry to evaluate phagocytosis of staphylococcus aureus by human neutrophils. Frontiers in Immunology. 12, 635825 (2021).
- Karsten, C. B., et al. A versatile high-throughput assay to characterize antibody-mediated neutrophil phagocytosis. Journal of Immunological Methods. 471, 46-56 (2019).
- Smirnov, A., Solga, M. D., Lannigan, J., Criss, A. K. Using imaging flow cytometry to quantify neutrophil phagocytosis. Methods in Molecular Biology. 2087, 127-140 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon