È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rilevamento del virus SARS-CoV-2 mediante amplificazione isotermica mediata da loop di trascrizione inversa
In questo articolo
Riepilogo
Qui forniamo un protocollo completo per standardizzare e implementare il metodo per rilevare il virus SARS-CoV-2 in campioni umani mediante amplificazione isotermica mediata da loop di trascrizione inversa (RT-LAMP). Questo metodo, eseguito entro 60 minuti, potrebbe essere adattato a qualsiasi laboratorio o punto di cura a basso costo e utilizzando attrezzature poco costose.
Abstract
Il virus della sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2) ha avuto un impatto drammatico sulla salute umana. Continua a essere una minaccia per la società moderna perché molte persone muoiono a causa dell'infezione. La malattia viene diagnosticata utilizzando test sierologici e molecolari, come la reazione a catena della polimerasi in tempo reale (RT-PCR). Quest'ultimo presenta diversi svantaggi perché richiede infrastrutture specializzate, attrezzature costose e personale qualificato. Qui, presentiamo un protocollo che delinea i passaggi necessari per rilevare il virus SARS-CoV-2 utilizzando l'amplificazione isotermica mediata da loop di trascrizione inversa (RT-LAMP) in campioni umani. Il protocollo include istruzioni per la progettazione di primer in silico, la preparazione dei reagenti, l'amplificazione e la visualizzazione. Una volta standardizzato, questo metodo può essere facilmente implementato e adattato a qualsiasi laboratorio o punto di cura entro 60 minuti a basso costo e utilizzando attrezzature poco costose. È adattabile al rilevamento di diversi agenti patogeni. Pertanto, può potenzialmente essere utilizzato sul campo e nei centri sanitari per effettuare una sorveglianza epidemiologica tempestiva.
Introduzione
La sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2) causa la malattia da coronavirus 2019 (COVID-19). Il 30 gennaio 2020 l'Organizzazione mondiale della sanità ha dichiarato un'emergenza sanitaria pubblica di rilevanza internazionale e l'11 marzo 2020 una pandemia. La pandemia ha provocato oltre 760 milioni di casi e 6,87 milioni di decessi alla data di stesura di questo articolo1.
L'impatto di questo virus ha evidenziato la necessità di strumenti di sorveglianza migliori, più accurati, più veloci e più ampiamente disponibili per migliorare l'individuazione e il controllo delle malattie infettive 2,3. Durante la pandemia, i test diagnostici SARS-CoV-2 si basavano sul rilevamento di acido nucleico, anticorpi e proteine, ma il rilevamento RT-PCR dell'acido nucleico è il gold standard4. Tuttavia, la RT-PCR presenta alcune limitazioni; Richiede attrezzature specializzate, infrastrutture e personale addestrato in biologia molecolare, limitando la sua applicazione a laboratori specializzati. Inoltre, richiede molto tempo (4-6 ore), escluso il tempo per trasportare i campioni in laboratorio, che può richiedere giorni5. Questi vincoli impediscono un'elaborazione efficiente dei campioni e l'ottenimento delle informazioni necessarie per la pianificazione di emergenza e la gestione epidemiologica.
L'amplificazione isotermica mediata da loop di trascrizione inversa (RT-LAMP) presenta diversi vantaggi rispetto alla RT-PCR, rendendola una strategia interessante per la progettazione di futuri test diagnostici point-of-care (POCT), in particolare in contesti con risorse limitate6. In primo luogo, è molto specifico perché utilizza tra i quattro e i sei primer che riconoscono da sei a otto aree nella sequenza bersaglio, sia che si tratti di DNA o RNA 7,8. In secondo luogo, poiché funziona a temperatura costante, non richiede apparecchiature sofisticate come termociclatori in tempo reale per generare l'amplificazione, né richiede personale altamente qualificato per farla funzionare. In terzo luogo, il tempo di reazione è molto breve (~60 min) e vengono impiegati reagenti non molto specializzati, il che lo rende uno strumento conveniente6. Alla luce di quanto sopra e dell'emergenza sanitaria causata dalla pandemia di COVID-19, questa tecnica può essere vista come un metodo diagnostico alternativo rapido, economico e semplice da implementare in qualsiasi laboratorio di ricerca9.
Il protocollo per la standardizzazione e l'implementazione di una RT-LAMP per rilevare SARS-CoV-2 con metodi colorimetrici utilizzando un termociclatore e un bagno d'acqua è descritto in questo articolo (Figura 1). Vengono discussi i punti critici, i loro limiti e le alternative per farli avanzare.
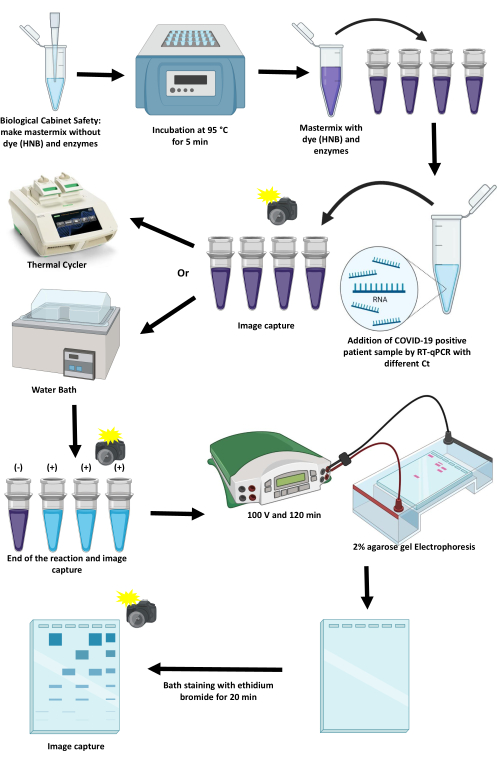
Figura 1: Schema del protocollo per l'amplificazione di SARS-CoV-2 utilizzando la tecnica RT-LAMP. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
I campioni utilizzati sono stati forniti dal laboratorio clinico dell'ospedale universitario Fundación Valle del Lili e corrispondevano all'RNA purificato di pazienti risultati positivi al COVID-19 utilizzando la tecnica RT-qPCR. Tutti i pazienti hanno fornito il consenso informato per la ricerca e questo studio è stato approvato dal comitato bioetico per gli studi sull'uomo dell'ospedale universitario Fundación Valle del Lili.
1. Progettazione e preparazione del primer RT-LAMP
NOTA: I primer LAMP possono essere utilizzati con una varietà di piattaforme, tra cui New England BioLabs (NEB) LAMP, Primer Explorer e LAMP assay versatile analysis (LAVA). Tuttavia, per questo protocollo, è stato utilizzato lo strumento NEB LAMP. La progettazione del primer può essere eseguita utilizzando i genomi SARS-CoV-2 ottenuti dal database NextStrain10. La tabella 1 mostra il set di primer utilizzato in questo protocollo.
- Primer design per LAMP
- Ottenere sequenze del genoma virale.
- Eseguire allineamenti di sequenze per ottenere la sequenza di consenso.
- Passare alla piattaforma11 dello strumento di progettazione del primer NEB LAMP e seguire le istruzioni nella guida rapida. Questo strumento produce gli stessi risultati di Primer Explorer V5, ma è molto più facile da usare nel suo output. Utilizzare i manuali utente di Primer Explorer come guida per la progettazione del primer.
- Valutazione termodinamica del set di primer
- Utilizzare lo strumento Primer-Dimer12 per eseguire un'analisi termodinamica sui primer ottenuti.
- Metti le sequenze di primer nello strumento. Quindi, selezionare l'opzione Analisi multiplex e rapporto sulla struttura del dimero.
- Selezionare i set di primer che hanno ΔG non inferiore a -5.
- Valutazione della specificità dei primer progettati
- Utilizzare il database di raccolta nucleotidica (nt/nt) in BLAST13 per analizzare ogni primer.
- Per eseguire la prima analisi BLAST, selezionare il Refseq_rna Data Base e filtrare la ricerca con il gruppo di generi che appartengono alla sottofamiglia Orthocoronavirinae. Si tratta di Alphacoronavirus (taxid:693996), Gammacoronavirus (taxid:694013) e Deltacoronavirus (taxid:1159901). Inoltre, valutare la sequenza rispetto ad altri virus che co-circolano come sottotipo H1N1 (taxid:114727), virus dell'influenza A (taxid:11320) e virus dell'influenza B (taxid:11520).
- Per eseguire la seconda analisi BLAST, selezionare Betacoronavirus GenBank e filtrare la ricerca con Coronaviridae (taxid:11118) e SARS (taxid:694009). Questi gruppi contengono sequenze di tutti i genomi identificati del SARS Coronavirus, compresi i genomi trovati nei pipistrelli, Betacoronavirus (taxid:694002).
- Per questo protocollo, assicurarsi che i primer non si allineino con genomi diversi dal genoma bersaglio, SARS-CoV-2.
- Preparazione del primer
- Centrifugare i flaconcini contenenti i primer iiofilizzati con una microcentrifuga (10.000 x g, 1 min a temperatura ambiente [RT]) per evitare perdite durante l'apertura della provetta.
- Reidratare la polvere iiofilizzata in acqua di dietil pirocarbonato (DEPC) allo 0,1% o in acqua priva di nucleasi fino a una concentrazione finale di 100 μM (Tabella 2) e scioglierla accuratamente pipettando su e giù. Quindi, centrifugare alla massima velocità (10.000 x g, 1 min a RT) in una microcentrifuga per raccogliere tutte le soluzioni di primer sul fondo della provetta.
- Preparare la miscela di primer 10x sotto una cabina di biosicurezza con il primer interno in avanti (FIP), il primer interno all'indietro (BIP), il primer esterno in avanti (F3), il primer esterno all'indietro (B3), il loop backward (LB) e il loop forward (LF), come riportato nella Tabella 2. Per evitare perdite, pipettare o agitare delicatamente la soluzione di primer prima di eseguire una centrifuga rapida (10.000 x g, 1 min a RT) con una microcentrifuga.
- Conservare la miscela di primer 10x a -20 °C per una conservazione a lungo termine; Tuttavia, preparatevi a sufficienza per un massimo di cinque esperimenti, indipendentemente da diversi campioni, per evitare troppi cicli di gelo-scongelamento.
NOTA Se è necessario un volume minore della miscela di primer, regolare i valori calcolando i nuovi volumi (Tabella 2). Inoltre, i set RdRp e RdRp/Hel non includono il primer LF perché i primer loop non sono necessari per le reazioni RT-LAMP. Di conseguenza, sostituire il volume del primer LF con acqua priva di nucleasi o acqua DEPC allo 0,1%.
2. Reazione RT-LAMP
- Accendere la cabina a flusso laminare secondo le istruzioni del produttore e attendere almeno 3 minuti affinché il flusso d'aria si stabilizzi.
- Una volta che il flusso d'aria è stabile, pulire e sanificare le superfici interne dell'armadio utilizzando una tecnica asettica. Per fare ciò, utilizzare i seguenti disinfettanti in questo ordine: 1000 ppm di ammonio quaternario (benzalconio cloruro), 2% di ipoclorito, 3% di perossido di idrogeno ed etanolo al 70%.
NOTA: In questo caso, la tecnica asettica prevede l'applicazione del disinfettante e la rimozione con tovaglioli dall'interno della cabina verso l'esterno senza passare sopra le superfici precedentemente pulite. - Utilizzando i disinfettanti del passaggio 2.2, pulire i materiali che entreranno nella cabina nello stesso ordine.
NOTA: Nell'armadio devono essere portate micropipette, scatole di puntali filtranti, matracci con provette da 1,5 ml e 0,6 ml, provette per PCR da 0,2 ml, rack e un becher da 400 ml. - Porta dei tovaglioli e dei guanti in nitrile in cabina. Successivamente, spegnere l'armadio ed esporlo alla luce ultravioletta (UV) per 15 minuti.
ATTENZIONE: Per evitare danni ai tessuti e al DNA dovuti all'esposizione prolungata alle radiazioni, evitare i raggi UV fino allo scadere del tempo impostato al punto 2.4.
NOTA Eseguire l'assemblaggio mostrato nella Figura 2 prima di iniziare il protocollo e iniziare il bagno d'acqua dopo aver completato il passaggio 2.4. È fondamentale riempire il contenitore di metallo quasi fino all'orlo con acqua potabile e impostare la temperatura della piastra riscaldante da laboratorio in ferro a 90 °C, poiché ciò si tradurrà in una temperatura di ~66,3 °C nel sistema, che viene monitorato con il termometro a mercurio. - Al termine del periodo di irradiazione, riavviare l'armadio e seguire le raccomandazioni del passaggio 1.1.
- Metti i reagenti (Tabella 3, Tabella 4 e Tabella 5) in un frigorifero pieno di ghiaccio o in un piccolo frigorifero di polistirolo. Mettere il contenitore all'interno dell'armadio dopo averlo pulito con etanolo al 70%.
- In una provetta da microcentrifuga da 0,6 mL, preparare la miscela LAMP del gene da amplificare (RdRp, N-A e RdRp/Hel), aggiungendo solo i seguenti componenti: 10x tampone, MgSO4, dNTP, 1x miscela di primer e acqua priva di nucleasi o 0,1% di acqua DEPC; Mescolare bene pipettando per omogeneizzare.
ATTENZIONE: A causa di una manipolazione e di un comportamento impropri all'interno dell'armadio, esiste un alto rischio di contaminazione dei reagenti. Per mitigare questo problema, è necessario seguire le seguenti regole: (i) utilizzare puntali sterili e filtranti; ii) utilizzare una punta per ciascun reagente; (iii) muoversi lentamente e con attenzione per evitare di interrompere il flusso laminare; (iv) mantenere l'ordine e utilizzare il minor numero possibile di materiali; e (v) utilizzare guanti diversi per preparare la miscela e aggiungere il materiale genetico.
NOTA: Conservare tutti i reagenti, in particolare gli enzimi, sul ghiaccio perché gli sbalzi di temperatura possono denaturarli e alterare l'attività della polimerasi. - Posizionare la/e provetta/e da 0,6 mL con il tappo chiuso in un blocco riscaldante e incubare a 95 °C per 5 minuti.
NOTA: Accendere il blocco riscaldante per provette da 1,5-2,0 mL situato all'esterno dell'armadio per almeno 30 minuti prima di iniziare la preparazione della miscela LAMP e monitorare la temperatura (95 °C) con un termometro a mercurio o ad alcol. - Al termine dell'incubazione, immergere le provette in un refrigeratore di polistirene riempito di ghiaccio per 5 minuti.
- Rimettere le provette nella camera a flusso laminare e completare la preparazione della miscela LAMP aggiungendo gli enzimi DNA polimerasi (Bst 3.0), trascrittasi inversa e DNA polimerasi ad alta fedeltà (Tabella 3, Tabella 4 e Tabella 5). Nel caso di utilizzo del rilevamento colorimetrico, aggiungere il colorante blu idrossinaftolo (HNB).
- Dopo aver aggiunto questi reagenti, mescolare molto bene i reagenti LAMP pipettandoli per solubilizzare gli enzimi e colorare.
- Riempire ogni provetta per PCR con 22,0 μL di miscela, facendo attenzione a non creare bolle. Quindi, aggiungere 3,0 μl di acqua DEPC allo 0,1% o acqua priva di nucleasi al controllo negativo o al tubo senza controllo modello (NTC) e mettere da parte le provette rimanenti per l'aggiunta (materiale genetico).
NOTA: Conservare le provette PCR in un frigorifero pieno di ghiaccio fino all'aggiunta del campione per evitare di attivare l'enzima Bst 3.0 e iniziare la reazione prematuramente. - Rimuovere tutti i materiali dall'armadio e utilizzare etanolo al 70% per pulire le superfici. Quindi spegnerlo seguendo le istruzioni del produttore.
- In un'area separata, aggiungere 3 μl di campione a ciascuna provetta per PCR e omogeneizzarla accuratamente. A tale scopo, utilizzare una micropipetta da 20 μl e puntali con filtro.
ATTENZIONE: La micropipetta utilizzata per aggiungere il materiale genetico deve essere utilizzata esclusivamente per questo scopo e non può essere utilizzata per preparare la miscela. In questo modo si evita la contaminazione dei reagenti. Inoltre, tieni sempre i campioni di RNA sul ghiaccio per ridurre la possibilità di degradazione dell'RNA. Utilizzare i seguenti dispositivi di protezione individuale (DPI) per l'aggiunta del campione: camice monouso, berretto, maschera N95, leggings, occhiali da laboratorio e guanti in nitrile. - Prima di eseguire la reazione colorimetrica, scattare fotografie delle provette PCR con una fotocamera di alta qualità. Il colore di partenza con HNB è il viola.
- Eseguire la reazione nel seguente sistema o apparecchiatura: (i) termociclatore e (ii) bagno d'acqua.
- Termociclatore: Depositare i tubi nel blocco di reazione e posizionare il termoprofilo (vedi Tabella 6) sull'apparecchiatura.
- Bagnomaria: Depositare i tubi in contenitori circolari e regolarli molto bene per evitare che fuoriescano. Successivamente, posizionare i contenitori nel bagnomaria (Figura 2A, B) alla temperatura indicata nella Tabella 6.
- Nel caso del bagnomaria, una volta che i tubi sono all'interno dell'impianto, avviare il timer per 60 min (Tabella 6).
- Al termine del tempo di reazione, rimuovere i tubi dal termociclatore o dal bagnomaria e conservarli a 4 °C per il ciclo elettroforetico o a -20 °C fino all'uso.
- Se è stata eseguita una reazione colorimetrica, scattare fotografie delle provette PCR utilizzando una fotocamera di alta qualità. Il colore finale con HNB è l'azzurro cielo.
3. Analisi dei prodotti di amplificazione in gel di agarosio
NOTA: Questi passaggi sono suggeriti come controlli aggiuntivi alla reazione colorimetrica o al controllo delle prestazioni durante la fase di standardizzazione. Questo perché la tecnica potrebbe presentare un enorme rischio di contaminazione per il laboratorio che esegue questi test.
- Posizionare il letto all'interno della camera di elettroforesi in modo che le gomme del bordo tocchino le pareti, creando uno spazio sigillato per l'aggiunta di agarosio (camera interna) (Figura 3A, B).
- Dopo aver completato il passaggio 3.1, pesare la quantità necessaria di agarosio in un becher da 500 ml per ottenere un gel al 2%. Successivamente, aggiungere il volume richiesto di 0,5 volte il tampone Tris-acetato EDTA (TAE) e cuocere nel microonde per 1-2 minuti.
NOTA: L'agarosio è completamente sciolto quando è traslucido e senza grumi quando viene tolto dal forno. Se ciò non viene confermato, possono rimanere regioni scarsamente gelificate, causando l'alterazione del funzionamento elettroforetico e della visualizzazione dei prodotti di amplificazione. - Sfornare il bicchiere e versare l'agarosio nella camera interna creata al punto 3.1 (Figura 3C). Successivamente, controlla che non ci siano bolle e, se ci sono, rimuovile utilizzando una punta per micropipetta.
- Disporre il favo in modo da formare i pozzetti e lasciare gelificare l'agarosio per circa 30 min a temperatura ambiente (RT).
- Trascorso questo tempo, aggiungere 5 ml di tampone TAE 0,5x per facilitare la rimozione dei pettini e del letto contenente il gel. Quindi posizionare il gel in modo tale che i pozzetti si trovino nell'anodo (Figura 3D).
- Riempire la camera di elettroforesi con tampone TAE 0,5x fino alla capacità specificata dal produttore, assicurandosi che gli elettrodi siano a contatto con il tampone.
- Aggiungere 3 μL di marcatore di peso molecolare al primo pozzetto del gel e aggiungere 9 μL di NTC e ciascun campione ai pozzetti successivi. Realizzarli combinando 7 μl di prodotto di amplificazione con 3 μl di tampone di caricamento; quindi caricare 9 μL di questa miscela nei pozzetti del gel.
- Coprire la camera di elettroforesi con il coperchio e collegare i cavi alle porte di alimentazione nel modello di colore. Impostare il generatore sui seguenti parametri: 100 V e amperaggio costante per 120 min.
- Al termine della corsa elettroforetica, posizionare il gel nel contenitore con la soluzione colorante (bromuro di etidio) e incubare per 30 minuti.
- Dopo l'incubazione, rimuovere il gel dalla soluzione colorante e metterlo in un sacchetto con chiusura a zip. Ciò impedisce la contaminazione dell'apparecchiatura che verrà utilizzata per visualizzare gli ampliconi.
- Visualizza il gel su un transilluminatore o un imager come l'Amersham Imager 600.
Risultati
L'implementazione del protocollo inizia con la progettazione del set di primer per ciascun gene bersaglio seguendo il protocollo sopra descritto. Nel giugno 2020, 5.000 genomi di SARS-CoV-2 sono stati ottenuti dal database NextStrain, con una rappresentatività del 10% dei genomi colombiani. Queste sequenze sono state allineate per ottenere la sequenza di consenso utilizzata nel processo di progettazione del primer. La tabella 1 mostra il set di primer scelto per i primer RdRp/Hel e RdRp. Il set di prime...
Discussione
Sebbene l'RT-LAMP sia considerato una metodologia complementare per l'esecuzione della diagnostica molecolare, presenta anche alcune limitazioni e passaggi critici che devono essere considerati quando il protocollo viene standardizzato e implementato.
La standardizzazione LAMP per la rilevazione di SARS-CoV-2 ha valutato i seguenti parametri e componenti nella miscela master: (a) concentrazione e temperatura di allineamento dei primer; b) concentrazione degli enzimi; c) concentrazione di magne...
Divulgazioni
Natalia Campillo-Pedroza è CEO della società BioDx: Diagnóstico y Soluciones Biotecnológicas S.A.S. Il resto degli autori dichiara di non avere conflitti di interesse.
Riconoscimenti
Questo lavoro è stato finanziato dal Sistema General de Regalías della Colombia, sovvenzione numero BPIN 2020000100092, e dall'Universidad Icesi - Convocatoria Interna, sovvenzione numero CA0413119. MFVT è stato finanziato anche dai fondi per le cattedre assistenziali dell'Universidad de los Andes. Gli enti finanziatori non hanno partecipato alla progettazione, all'esecuzione delle attività, alla raccolta dei dati, all'analisi dei dati e alla preparazione del manoscritto. Ringraziamo l'Ospedale Universitario Fundación Valle del Lili per l'RNA virale da campioni di Sars-CoV-2 e il Dr. Alvaro Barrera-Ocampo per i commenti sul manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 kb DNA Ladder | SOLIS BIODYNE | 07-12-00050 | Store at -20 °C |
| 50x TAE Electrophoresis Buffer | ThermoScientific | B49 | Store at roome temperature |
| Accuris High Fidelity Polymerase | ACCURIS LIFE SCIENCE REAGENTS | PR1000-HF-200 | It can be used in case Q5 High-Fidelity DNA polymerase cannot be purchased. For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Agarose | PanReacAppliChem | A8963,0100 | N/A |
| Bst 3.0 DNA Polymerase 8000 IU/mL | New England BioLabs | M0374S/M0374L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | Store at -20 °C |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220-25G | Handle it with caution under an extraction cabinet |
| GeneRuler 100 bp Plus DNA Ladder, ready-to-use | ThermoScientific | SM0322 | Store at -20 °C |
| Hydroxy naphthol blue disodium salt | Santa Cruz Biotechnology | sc-215156B | N/A |
| Q5 High-Fidelity DNA polymerase 2000 IU/mL | New England BioLabs | M0491S/M0491L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| WarmStart RTx Reverse Transcriptase 15000 IU/mL | New England BioLabs | M0380S/M0380L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
Riferimenti
- World Health Organization. . Who coronavirus (COVID-19) dashboard (no date). , (2023).
- Ibrahim, N. K. Epidemiologic surveillance for controlling Covid-19 pandemic: types, challenges and implications. Journal of Infection and Public Health. 13 (11), 1630-1638 (2020).
- Rojas-Gallardo, D. M., et al. COVID-19 in Latin America: Contrasting phylodynamic inference with epidemiological surveillance. (Molecular epidemiology of COVID-19 in Latin America). medRxiv. , (2020).
- Liu, R., et al. Positive rate of RT-PCR detection of SARS-CoV-2 infection in 4880 cases from one hospital in Wuhan, China, from Jan to Feb 2020. Clinica Chimica Acta. 505, 172-175 (2020).
- Kevadiya, B. D., et al. Diagnostics for SARS-CoV-2 infections. Nature Materials. 20 (5), 593-605 (2021).
- Tomita, N., Mori, Y., Kanda, H., Notomi, T. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nature Protocols. 3 (5), 877-882 (2008).
- Li, Y., Fan, P., Zhou, S., Zhang, L. Loop-mediated isothermal amplification (LAMP): A novel rapid detection platform for pathogens. Microbial Pathogenesis. 107, 54-61 (2017).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. Journal of Microbiology. 53 (1), 1-5 (2015).
- Augustine, R., et al. Loop-mediated isothermal amplification (LAMP): A rapid, sensitive, specific, and cost-effective point-of-care test for coronaviruses in the context of COVID-19 pandemic. Biology (Basel). 9 (8), 182 (2020).
- . Nextstrain Available from: https://nextstrain.org/ (2023)
- . Neb Lamp, NEB LAMP Available from: https://lamp.neb.com/ (2023)
- . Blast: Basic local alignment search tool (no date) Available from: https://blast.ncbi.nlm.nih.gov/ (2023)
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Lu, R., et al. Development of a novel reverse transcription loop-mediated isothermal amplification method for rapid detection of SARS-CoV-2. Virologica Sinica. 35 (3), 344-347 (2020).
- Najafov, A., Hoxhaj, G. . PCR Guru. , (2017).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. Biotechniques. 69 (3), 178-185 (2020).
- Ramírez-Chavarría, R. G., et al. Automatic analysis of isothermal amplification via impedance time-constant-domain spectroscopy: A SARS-CoV-2 case study. Chemosensors. 11 (4), 230 (2023).
- Haque, M. F. U., et al. A novel RdRp-based colorimetric RT-LAMP assay for rapid and sensitive detection of SARS-CoV-2 in clinical and sewage samples from Pakistan. Virus Research. 302, 198484 (2021).
- Donia, A., et al. Integration of RT-LAMP and microfluidic technology for detection of SARS-CoV-2 in wastewater as an advanced point-of-care platform. Food and Environmental Virology. 14, 364-373 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon