Method Article
PyDesigner v1.0: un'implementazione pythonica della pipeline DESIGNER per la risonanza magnetica a diffusione
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui presentiamo PyDesigner, una pipeline di elaborazione della risonanza magnetica a diffusione (dMRI) basata su Python in grado di correggere i tipici artefatti dMRI e produrre imaging del tensore di diffusione (DTI), imaging della curtosi di diffusione (DKI), imaging della sfera in fibra (FBI), modellazione della microstruttura (integrità della sostanza bianca [WMTI] e materia bianca della sfera in fibra [FBWM]) e output di trattografia.
Abstract
PyDesigner è un pacchetto software basato su Python basato sul parametro di diffusione originale EStImation with Gibbs and NoisE Removal (DESIGNER) pipeline (Dv1) per la pre-elaborazione dMRI e la stima del tensore. Questo software è fornito apertamente per la ricerca non commerciale e non può essere utilizzato per l'assistenza clinica. PyDesigner combina gli strumenti di FSL e MRtrix3 per eseguire il denoising, la correzione del ringing di Gibbs, la correzione del movimento a correnti parassite, il mascheramento del cervello, il livellamento dell'immagine e la correzione del bias di Rician per ottimizzare la stima di più misure di diffusione. Può essere utilizzato su più piattaforme su Windows, Mac e Linux per derivare con precisione le metriche di uso comune dai set di dati DKI, DTI, WMTI, FBI e FBWM, nonché dai file ODF e .fib della trattografia. È anche indipendente dal formato di file e accetta input sotto forma di .nii, .nii.gz, .mif e dicom. Facile da usare e da installare, questo software emette anche metriche di controllo della qualità che illustrano grafici del rapporto segnale/rumore, voxel anomali e movimento della testa per valutare l'integrità dei dati. Inoltre, questa pipeline di elaborazione dMRI supporta l'elaborazione di più set di dati echo-time e offre la personalizzazione della pipeline, consentendo all'utente di specificare quali processi vengono utilizzati e quali output vengono prodotti per soddisfare una varietà di esigenze dell'utente.
Introduzione
La risonanza magnetica a diffusione (dMRI) è ampiamente applicata per lo studio non invasivo delle proprietà microstrutturali nel cervello. Sebbene siano stati proposti molti metodi dMRI, due comunemente usati sono l'imaging del tensore di diffusione (DTI) e l'imaging della curtosi diffusionale (DKI). Queste tecniche sono strettamente correlate, con la DKI che è un'estensione della DTI che include la quantificazione della non-gaussianità diffusionale1. Entrambi forniscono una varietà di misure di diffusione scalare e consentono la costruzione di trattografie in fibra di sostanza bianca. Un importante vantaggio di DTI e DKI è che hanno una solida base nella fisica della diffusione, in modo che la loro validità non si basi su ipotesi dettagliate riguardanti la microstruttura tissutale 2,3. Ciò consente di applicare DTI e DKI in tutto il cervello e il corpo per partecipanti di qualsiasi età e stato patologico.
Poiché le immagini grezze pesate in diffusione (DWI) sono degradate da molteplici fattori, tra cui il rumore del segnale, il movimento, il ringing di Gibbs e la distorsione delle correnti parassite, è necessario utilizzare la pre-elaborazione prima del calcolo di qualsiasi quantità di diffusione4. La pre-elaborazione dei DWI è un campo di ricerca attivo che è ora molto sviluppato. Tuttavia, la combinazione delle fasi di elaborazione necessarie in un'unica pipeline che fornisce risultati coerenti è impegnativa perché è necessario regolare diverse impostazioni definite dall'utente a seconda dei dettagli dell'acquisizione della dMRI e perché l'ordine in cui vengono eseguite le fasi di pre-elaborazione influisce sul risultato. Per questo motivo, la pipeline EStImation with Gibbs and NoisE Removal (DESIGNER, GitHub: NYU-DiffusionMRI/DESIGNER) è stata inizialmente proposta nel 2016 per ottimizzare, standardizzare e semplificare la pre-elaborazione per DWI5. DESIGNER Dv1 si basa su strumenti software incorporati in FSL, MRtrix3, MATLAB e Python per creare un processo DWI completo e senza soluzione di continuità, che comprende la correzione dell'immagine attraverso la pre-elaborazione e la stima del tensore di diffusione/curtosi5. Con i flag di controllo per attivare o disattivare le fasi di pre-elaborazione, le correzioni DWI possono essere eseguite in modo selettivo. DESIGNER pre-elabora in un ordine specifico: (i) analisi delle componenti principali di Marchenko-Pastur (MP-PCA) denoising6, (ii) correzione del ringing di Gibbs7, (iii) correzione della distorsione dell'imaging ecoplanare (EPI)8, correzione delle correnti parassite9, correzione del movimento10 e sostituzione dei valori anomali11, (iv) correzione del campo di polarizzazione B1, (v) generazione della maschera cerebrale, (vi) smoothing, (vii) correzione della distorsione del rumore Rician e (viii) normalizzazione b0. La pre-elaborazione in questo ordine specifico migliora sia la precisione che l'effettivo rapporto segnale/rumore (SNR)5. Vale la pena notare che ogni passaggio di PyDesigner è facoltativo e può essere utilizzato o saltato in base alle preferenze dell'utente. Lo smoothing, ad esempio, potrebbe non essere una fase di pre-elaborazione necessaria per alcuni set di dati. Sebbene aiuti a mitigare gli errori di filtraggio, potrebbe non essere necessario per set di dati di altissima qualità. Pertanto, gli utenti possono scegliere di utilizzare solo i passaggi necessari per i propri dati.
L'implementazione di DESIGNER su più piattaforme è impegnativa a causa delle differenze nei sistemi operativi e nelle impostazioni dell'ambiente. In particolare, il fatto che DESIGNER Dv1 sia scritto principalmente in MATLAB crea significativi problemi di portabilità derivanti da complicati requisiti di configurazione necessari per abilitare l'interfaccia Python-MATLAB. Inoltre, diverse combinazioni di MATLAB, Python e versioni di dipendenza compromettono la riproducibilità di questa pipeline. Per questo motivo, abbiamo sviluppato PyDesigner, che è interamente basato su Python. Questa pipeline di elaborazione dMRI non solo consente una pre-elaborazione senza interruzioni, ma consente anche di incorporare la pipeline in un contenitore Docker, migliorando notevolmente la portabilità e la riproducibilità. Inoltre, sostituendo il codice MATLAB, PyDesigner evita tutti i costi di licenza e migliora l'accessibilità.
Questa pipeline di elaborazione dMRI aumenta l'approccio a mani libere introdotto da DESIGNER, aggiunge diverse nuove funzionalità e incorpora strumenti di FSL e MRtrix3 per eseguire la preelaborazione. Le librerie matematiche Python standard come Numpy12, SciPy13 e CVXPY14,15 sono state utilizzate per sostituire le parti MATLAB di DESIGNER con codice Python. Questo software è fornito apertamente e disponibile su Github16. L'obiettivo di questo documento non è quello di convalidare o confrontare il nostro software con software simili, ma piuttosto di fornire agli utenti una guida passo passo per l'elaborazione dei loro dati con PyDesigner, qualora scegliessero di farlo.
Protocollo
Tutti i dati utilizzati per sviluppare e testare il software sono stati raccolti nell'ambito di un protocollo di studio approvato dal comitato di revisione istituzionale.
NOTA: PyDesigner è stato compilato su MacOS 10.14 e richiede un sottosistema Windows per Linux (WSL) per l'esecuzione su Windows. Gli stessi comandi possono essere utilizzati per i sistemi Linux/Mac.
1. Compatibilità del sistema operativo
- Per i sistemi Windows, seguire il collegamento per installare WSL17 e quindi procedere al passaggio 2.6.
- Per i sistemi basati su Unix (come Linux e Mac) procedere alla sezione 2.
NOTA: i sistemi basati su Unix possono installare in modo nativo PyDesigner e le sue dipendenze.
2. Installa FSL
NOTA: Tutti i test di PyDesigner sono stati eseguiti con FSL versione 6.0.2 e si consiglia di utilizzare v6.0.2 o versioni successive.
- Vai al wiki FSL18 e seleziona Scarica/Installa. Per gli utenti Windows, andare al passaggio 2.6.
- Scegliere Scarica FSL sotto l'intestazione Installazione di FSL. Selezionare quindi la licenza software di installazione FSL, leggere il contratto e selezionare Accetto.
- Compila i campi obbligatori e seleziona Registrati.
- Scegli il link Scarica FSL (primo link nella pagina). Quindi selezionare il collegamento alle istruzioni di installazione (secondo collegamento nella pagina). Segui queste istruzioni per scaricare FSL per ogni sistema operativo.
NOTA: L'installazione di FSL richiede circa 10 minuti. - Per verificare la corretta installazione, immettere quanto segue in una nuova finestra del terminale:
$ flirt -version. Questo dovrebbe restituire il numero di versione. Procedere al passaggio 3. - Per gli utenti Windows, seguire la guida all'installazione di WSL per FSL19.
3. Creare un ambiente Conda
- Installare un pacchetto conda per gestire gli ambienti Python. Gli utenti possono scegliere tra Miniconda o Anaconda.
- Scarica Miniconda/Anaconda20. Gli utenti Mac e Linux procedono al passaggio 3.2.
- Per Windows, configurare conda all'interno di WSL21.
NOTA: Le seguenti istruzioni sono per Miniconda, ma gli stessi comandi funzionano per Anaconda. Sebbene conda non supporti più Python 3.7, è ancora possibile creare un ambiente conda con Python 3.7, consentendo di implementare questo protocollo esattamente come descritto.
- Immettere quanto segue nel terminale per creare l'ambiente conda:
$ conda create -n mri python=3.7. Utilizzare questo ambiente conda per tutte le attività correlate a PyDesigner.- Quando viene visualizzato il messaggio
Proceed ([y]/n)?Invioy.
NOTA: flag -n: Utilizzare questo per indicare il nome dell'ambiente conda. Nel video, l'ambiente conda è chiamato mri.
NOTA:python=: Si consiglia la versione 3.7, poiché PyDesigner è stato creato e testato su questa versione.
- Quando viene visualizzato il messaggio
4. Installare MRtrix3
- Attivare l'ambiente conda inserendo il seguente comando:
$ conda activate mri - Aprire la pagina di download di MRtrix322 e seguire le istruzioni per il download del sistema operativo appropriato. Il codice di installazione chiederà la conferma di alcune dipendenze. Invio
yper continuare il processo di download.- Stampare la documentazione di MRtrix3 per confermare l'installazione inserendo quanto segue nel terminale:
$ mrinfo -h
- Stampare la documentazione di MRtrix3 per confermare l'installazione inserendo quanto segue nel terminale:
5. Installa PyDesigner
- Apri una nuova finestra del terminale e attiva l'ambiente conda inserendo quanto segue nel terminale:
$ conda activate mri - Esegui il comando:
$ pip install PyDesigner-DWI - Per verificare la corretta installazione, stampare il numero di versione inserendo quanto segue nel terminale:
$ pydesigner -v- Se viene visualizzato l'errore "modulo non trovato", consultare la sezione Risoluzione dei problemi alla fine del protocollo (sezione 10)
NOTA: $ pydesigner -h stamperà la documentazione di aiuto che spiega i comandi e la sintassi del software. PyDesigner può anche essere scaricato tramite Github. Vedere le istruzioni di installazione sul sito web23.
- Se viene visualizzato l'errore "modulo non trovato", consultare la sezione Risoluzione dei problemi alla fine del protocollo (sezione 10)
6. Preparazione dei dati
NOTA: PyDesigner funziona con più tipi di file come .nii, .nii.gz, .mif e DICOM. Nel video, i DICOM vengono ordinati e convertiti in NIfTI prima di utilizzare questa pipeline di elaborazione dMRI. I file NIfTI sono più facili da lavorare perché sono anonimizzati e c'è un file per sequenza.
- Per ordinare i DICOM, utilizzare lo strumento DicomSort24.
NOTA: È consentito qualsiasi altro strumento di smistamento DICOM disponibile che svolga la stessa attività. - Dopo aver ordinato i DICOM, convertili in file NIfTI utilizzando dcm2niix25.
- Installa dcm2niix inserendo il seguente comando nel terminale:
$ sudo apt installdcm2niix - Una volta scaricato, digita quanto segue nel terminale per verificare l'installazione stampando la documentazione per dcm2nii:
$ dcm2niix -h
- Installa dcm2niix inserendo il seguente comando nel terminale:
- Per eseguire dcm2niix, inserisci il seguente comando nel terminale:
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
NOTA: -f: Per specificare la struttura del nome del file per il file di output, far seguire a questo tag la struttura del nome file desiderata. Il video utilizza: -f %s_%p_%d . %s, %p e %d sono notazioni rispettivamente per la serie, il numero di protocollo e la descrizione. Qualsiasi struttura di file funzionerà. Gli output attesi da dcm2niix sono i file .bval, .bvec, .json e .nii. Se i dati TOPUP sono stati acquisiti, saranno presenti file TOPUP .json e .nii.
7. Utilizzo di base di PyDesigner
NOTA: PyDesigner può richiedere fino a 1,5 ore per un singolo set di dati a seconda dei parametri di elaborazione e delle capacità del computer.
- Per eseguire l'elaborazione standard, utilizzare il flag
-so-standard. Questo comando eseguirà la pipeline di pre-elaborazione consigliata nel seguente ordine: Denoising6, Correzione del ringing di Gibbs7, Correzione della distorsione EPI8, Correzione del movimento delle correnti parassite9, Creazione di maschere cerebrali e del liquido cerebrospinale (CSF), smoothing, Correzione del bias Rician, Rilevamento dei valori anomali11, Stima del tensore e Creazione di mappe parametriche. - Per eseguire il comando di base di PyDesigner, attivare prima l'ambiente conda inserendo il seguente comando nel terminale:
$ conda activate mri - Stampare la documentazione di aiuto per il software inserendo il seguente comando:
$ pydesigner -h. Questa documentazione illustra tutti i possibili comandi e la sintassi per PyDesigner. - Utilizzare l'esempio seguente per strutturare il comando.
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - Si notino i seguenti flag importanti:
--verbose: utilizzare questa opzione per visualizzare la console PyDesigner durante l'elaborazione dei dati (non obbligatorio).--rpe pairs #: Utilizzare questa opzione per consentire agli utenti con una sequenza TOPUP di accelerare il processo di correzione della distorsione EPI8 utilizzando TOPUP. Il simbolo # si riferisce al numero di volumi B0 della sequenza TOPUP che verranno utilizzati. L'uso di #=1 impedisce la sovrastima del campo di distorsione e riduce il tempo necessario per creare questo campo. Gli utenti hanno utilizzato abitualmente #=1 con risultati affidabili. Quando si utilizza #=1, PyDesigner prenderà per impostazione predefinita il primo volume.
8. Utilizzo avanzato di PyDesigner: adattamento del software alle esigenze individuali degli utenti
NOTA: Quando si personalizza PyDesigner, omettere il -s flag; questo flag è il comando per la pre-elaborazione automatica.
- Attiva l'ambiente conda inserendo il seguente comando nel terminale:
$ conda activate mri - Consultare il sito web del software23 per un elenco delle bandiere; ogni flag consente all'utente di eseguire ogni parte di PyDesigner individualmente.
- Strutturare il comando. Iniziare con i flag per ogni sequenza che l'utente desidera eseguire. Quindi, indirizzare PyDesigner ai file di input e output. Per esempio:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - Esempio 1: eseguire il solo raccordo del tensore (nessuna pre-elaborazione aggiuntiva).
- Attiva l'ambiente conda inserendo il seguente comando nel terminale:
$ conda activate mri - Consulta il sito web di PyDesigner23 per un elenco di bandiere. In questo esempio vengono utilizzati
-mi file . -m: Aggiungi questo flag per limitare l'adattamento del tensore solo all'interno dei voxel cerebrali. Ciò accelera l'adattamento tensoriale + il rilevamento dei valori anomali.- Utilizzare l'esempio seguente per strutturare il comando:
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
NOTA: L'input per l'adattamento tensoriale è un file .nii pre-elaborato.
- Attiva l'ambiente conda inserendo il seguente comando nel terminale:
- Esempio 2: Eseguire il denoising da solo (nessuna pre-elaborazione aggiuntiva).
- Attiva l'ambiente conda inserendo quanto segue nel terminale:
$ conda activate mri - Consulta il sito web di PyDesigner23 per un elenco di bandiere. In questo esempio vengono utilizzati
-ni file . - -n: Aggiungi questo flag per completare il passaggio di riduzione del rumore. Inserire il file DWI precedentemente pre-elaborato etichettato DWI_preproccessed.nii, che si trova nella cartella di output di ciascun soggetto.
- Utilizzare l'esempio seguente per strutturare il comando:
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- Attiva l'ambiente conda inserendo quanto segue nel terminale:
9. Futuri aggiornamenti di PyDesigner
NOTA: Gli aggiornamenti sono disponibili sul sito Web23.
- Per aggiornare PyDesigner, inserisci quanto segue in una nuova finestra del terminale:
$ pip install --upgrade PyDesigner-DWI
NOTA: Se PyDesigner è stato installato tramite Github16, deve essere aggiornato anche utilizzando git.
10. Risoluzione dei problemi di installazione
- Quando si scarica PyDesigner, verrà visualizzato il codice di errore "modulo non trovato" se manca un modulo Python.
- Correggi un modulo mancante inserendo quanto segue nel terminale:
$ pip install nameofmodule - Quindi ripetere il primo comando di installazione inserendo quanto segue nel terminale :
$ pip install PyDesigner-DWI - PyDesigner dovrebbe ora essere completamente installato. Tornare al passaggio 5.3. Se si verifica lo stesso errore, ripetere questo processo fino a quando non vengono installati tutti i moduli Python mancanti.
- Correggi un modulo mancante inserendo quanto segue nel terminale:
11. Risoluzione degli errori di output
- In caso di problemi, esaminare il file log_command.json per identificare eventuali errori di pre-elaborazione. Esaminare i file intermedi per determinare l'errore specifico.
- Per assistenza nella risoluzione dei problemi, inviare una richiesta nella pagina16 di GitHub.
12. Esecuzione di PyDesigner con un set di dati di esempio
- Scarica il set di dati di esempio da OpenNeuro qui26.
NOTA: questo set di dati di esempio contiene sia dati grezzi che pre-elaborati. I dati non elaborati sono intesi come input per l'elaborazione dell'esempio PyDesigner. I dati pre-elaborati sono costituiti dagli output generati dopo l'elaborazione dei dati grezzi utilizzando la pipeline di elaborazione dMRI su un iMac OS v12.4. Gli utenti possono scaricare i dati non elaborati, elaborarli utilizzando PyDesigner e confrontare i propri output con gli output di esempio forniti per assicurarsi che PyDesigner venga eseguito correttamente. I dati non elaborati sono contenuti nella cartella principale (/PyDesigner Example Dataset/sub-01) e i dati pre-elaborati sono contenuti nella cartella derivatives (/PyDesigner Example Dataset/derivatives/sub-01). Se non diversamente specificato, il set di dati OpenNeuro verrà scaricato nella cartella utente predefinita. - Apri un nuovo terminale su Mac/Linux o WSL su Windows.
NOTA: per eseguire la pipeline di elaborazione standard di PyDesigner, seguire la struttura dei comandi di esempio riportata di seguito. Immettere i comandi seguenti nel terminale/WSL. In questo esempio, dove " ./user_download_folder/" indica il percorso della cartella di dati di esempio. Questo percorso sarà specifico per la posizione in cui vengono archiviati i dati di esempio nel computer dei singoli utenti. - Per creare una nuova cartella per le uscite, inserisci quanto segue nel terminale:
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - Per attivare l'ambiente conda, inserisci quanto segue nel terminale:
$ conda activate mri - Per eseguire PyDesigner sul set di dati di esempio, immettere quanto segue nel terminale:
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
NOTA: In questo esempio, ./user_download_folder/ indica il percorso della cartella di dati di esempio. Questo percorso sarà specifico per il computer di ogni utente. I comandi nei passaggi da 12.3 a 12.5 seguono la struttura del passaggio 8.3. Questo comando utilizza il flag -s, il flag di elaborazione standard di PyDesigner, che esegue i seguenti passaggi di elaborazione in questo ordine: denoising6, correzione del ringing di Gibbs7, undistortion 8,9, brain masking, smoothing e correzione del bias rician. - Se il software è installato correttamente, tutti gli output verranno inseriti nella cartella PyDesigner_Outputs appena creata. Confrontare i file di pre-elaborazione prodotti da PyDesigner con la cartella di pre-elaborazione del set di dati di esempio che si trova nella cartella "derivati" scaricata da OpenNeuro.
- Consulta la sezione dei risultati rappresentativi per confrontare le loro mappe metriche con quelle nei risultati del set di dati di esempio.
Risultati
Il software PyDesigner applica più passaggi di correzione dell'immagine ai dati di diffusione grezzi e genera output utilizzati per migliorare la precisione dei file grezzi durante l'esecuzione dell'analisi. Ogni fase disponibile nella pipeline è stata precedentemente convalidata attraverso pubblicazioni peer-reviewed 5,6,7,8,9,10,11 come discusso nell'introduzione. I risultati del software possono essere utilizzati in analisi quali profili trattografici, matrici di connettività, analisi per voxel, analisi ROI, analisi fODF, TBSS e analisi basate su fixel.
Il sito Webdel software 23 elenca tutti i file di output generati durante la pipeline di pre-elaborazione. Dopo l'esecuzione di ogni prompt, la console emetterà una descrizione di tutti i processi completati. Esistono 3 tipi di file di output: file di elaborazione, metriche e controllo qualità. La struttura della directory di output è illustrata nella Figura 1. Questi file sono disponibili quando si utilizza la pre-elaborazione standard (fare riferimento alla sezione 7 del protocollo). Se l'utente richiede un utilizzo più avanzato (fare riferimento alla sezione 8 del protocollo), i file di output disponibili dipenderanno da quali processi sono stati completati.
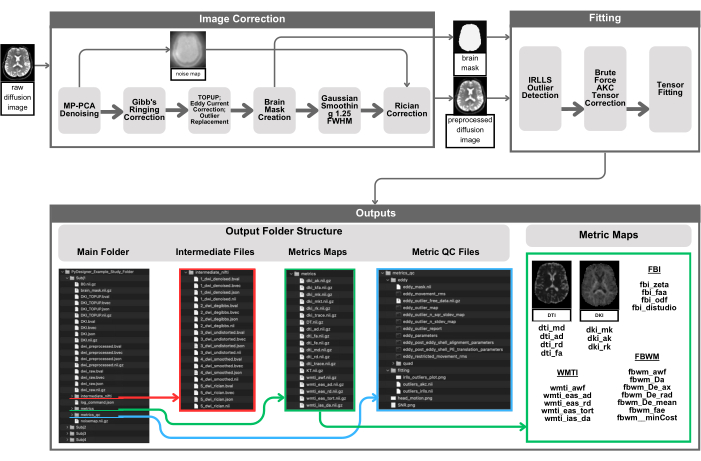
Figura 1: Rappresentazione visiva della pipeline PyDesigner. La pre-elaborazione inizia fornendo un DWI 4D di input a PyDesigner (in alto a sinistra), che quindi viene sottoposto a denoising MP-PCA per produrre un DWI 4D denoised e una mappa del rumore 3D. Il DWI 4D denoised viene quindi sottoposto a correzione del ringing di Gibbs, correzione del bias riciano, TOPUP, correzione delle correnti parassite e correzione dei valori anomali. Una maschera cerebrale viene quindi calcolata per i passaggi successivi, il rilevamento dei valori anomali e l'adattamento del tensore per accelerare i calcoli eseguendoli solo all'interno della maschera cerebrale. Gli output possono essere trovati nelle sottodirectory nella cartella principale di elaborazione del soggetto: intermediate_nifti, metriche e metrics_qc. Si noti che gli screenshot degli output di PyDesigner non sono pensati per essere un elenco esaustivo di tutti i possibili output, ma piuttosto per fornire un esempio visivo schematico di ciò che gli utenti possono aspettarsi. Gli output, sia quelli finali che quelli intermedi, variano in base ai dati di input dell'utente e ai flag di elaborazione utilizzati. Clicca qui per visualizzare una versione più grande di questa figura.
Elaborazione
I file di elaborazione vengono utilizzati durante la pipeline PyDesigner e vengono archiviati nella directory di output radice. Per ogni fase di pre-elaborazione, i file DWI intermedi vengono salvati nella cartella di output "intermediate_nifti", come mostrato nella Figura 1. È necessario fare riferimento a questi file quando si affrontano eventuali problemi con l'elaborazione o gli output per valutare separatamente ogni passaggio della pipeline.
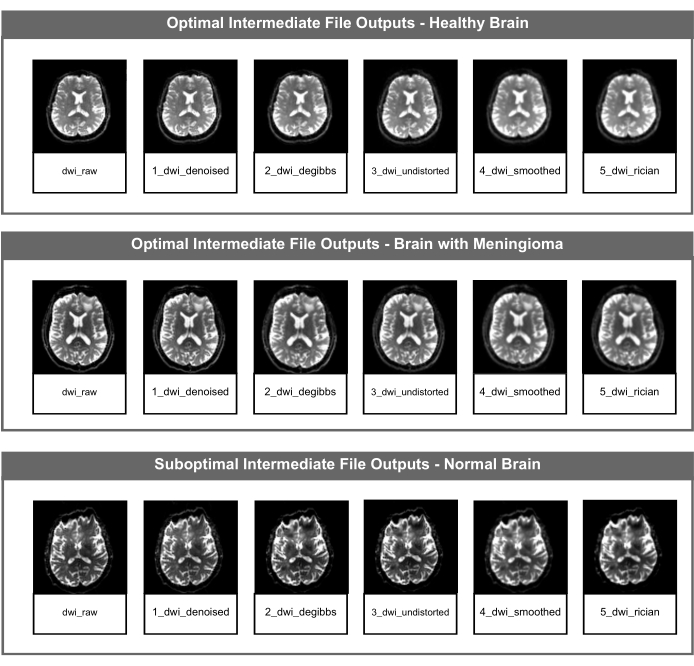
Figura 2: File NifTI DWI intermedi ottimali e subottimali. La figura mostra il file NIfTI intermedio per ogni passaggio di correzione dell'immagine della pipeline di PyDesigner. La riga superiore è un esempio di output di file intermedi ottimali che utilizzano dati provenienti da un cervello adulto sano, la riga centrale è un esempio di output di file intermedi ottimali che utilizzano dati provenienti da un cervello patologico (meningioma) e la riga inferiore mostra output di file intermedi non ottimali utilizzando dati provenienti da un cervello adulto sano con un artefatto di suscettibilità non correlato alla struttura o alla salute del cervello. Clicca qui per visualizzare una versione più grande di questa figura.
Metriche
Questa cartella contiene tutte le mappe parametriche calcolate da PyDesigner (vedere la Figura 1). Ciò include mappe parametriche per DTI/DKI, imaging a sfera in fibra (FBI)/materia bianca a sfera in fibra (FBWM) e metriche di integrità del tratto della sostanza bianca (WMTI) (Tabella 1)16.

Tabella 1: Intervallo di valori previsto per le metriche DTI/DKI e FBI/FBWM. La tabella include un elenco di metriche DTI, DKI, FBI e FBWM affidabili generate da PyDesigner e i relativi intervalli di valori previsti. Vengono inoltre elencate le sequenze e i valori b necessari (s/mm2) per derivare ciascuna metrica. Le metriche DTI elencate sono FA, MD, AD e RD. Le metriche DKI elencate sono MK, AK, RK e KFA. La metrica dell'FBI elencata è la FAA. Le metriche FBWM elencate sono AWF, DA, DE_AX, DE_RAD e FAE.
Gli utenti possono condurre un controllo visivo e di qualità del valore (QC) delle metriche di diffusività media (MD), anisotropia frazionaria (FA) e curtosi media (MK) per identificare risultati non ottimali. Se queste metriche non sono ottimali rispetto agli standard descritti di seguito, gli utenti devono esaminare ogni file intermedio descritto nella Figura 2 per determinare quale passaggio di pre-elaborazione non è andato a buon fine.
Il controllo qualità visivo viene utilizzato per identificare risultati non ottimali (ad esempio, problemi di adattamento tensoriale e artefatti). Si consiglia di utilizzare ImageJ per il controllo qualità visivo per garantire che non venga eseguita alcuna manipolazione dell'immagine tramite le impostazioni predefinite del software. La riga superiore della Figura 3 mostra le tipiche mappe metriche MD, FA e MK utilizzando una soglia biologicamente plausibile rispettivamente di 0-3 μm2/ms, 0-1 μm2/ms e 0-2 μm2/ms (Figura 3 [riga superiore]). La mappa MD dovrebbe avere i valori più alti nei ventricoli e valori alti nella sostanza grigia corticale (Figura 3A [riga superiore]). La mappa FA dovrebbe avere tratti di sostanza bianca evidenti che sono chiari in tutto il cervello (Figura 3B [riga in alto]). La mappa MK dovrebbe avere valori alti in WM e valori più bassi nella materia grigia e CSF (Figura 3C [riga in alto]). La Figura 3D [riga superiore] è un esempio di mappa metrica con problemi di adattamento tensoriale, che si traduce in cluster di voxel a valore zero. In caso di problemi, esaminare il file log_command.json per individuare eventuali errori di pre-elaborazione. Esaminare i file intermedi per determinare l'errore specifico. Per assistenza nella risoluzione dei problemi, inviare una richiesta nella pagina GitHub di PyDesigner.
Il controllo della qualità del valore viene utilizzato per identificare se i voxel di una determinata mappa metrica sono relativamente uniformi tra i soggetti per ciascun set di dati. L'intervallo di valori previsto per ogni mappa e set di dati dipende dai dati e dai parametri di PyDesigner. Nel nostro set di dati di test, abbiamo avuto picchi coerenti negli intervalli da 8000 a 10.000, da 2500 a 4000 e da 5000 a 13.000 rispettivamente per MD, FA e MK, utilizzando il binning dell'istogramma predefinito di FSLeyes. La riga inferiore della Figura 3 fornisce esempi di variabilità dell'istogramma. La tabella 1 contiene i valori previsti dell'asse x per queste metriche. I voxel più alti o più bassi sono caratteristici del set di dati o indicano un artefatto o un problema di pre-elaborazione (Figura 3D [riga inferiore]).
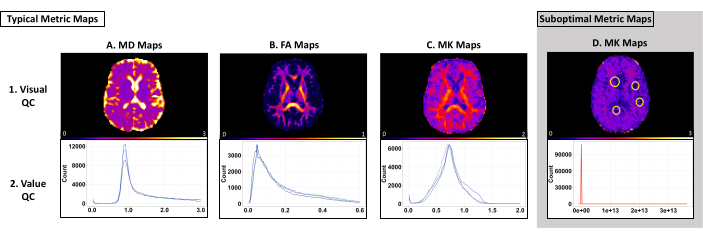
Figura 3: Esempi di mappe metriche e istogrammi da PyDesigner con risultati ottimali e non ottimali. La riga superiore mostra esempi di mappe MD, FA e MK a soggetto singolo utilizzate per il controllo qualità visivo. La riga inferiore mostra gli istogrammi multisoggetto utilizzati per il valore QC. (A-C) Esempi tipici di mappe metriche e istogrammi che superano il controllo visivo e di qualità del valore. Ogni linea blu sugli istogrammi per ogni tipo di metrica rappresenta un singolo set di dati. Si noti che ogni set di dati segue una curva simile e rientra in un intervallo simile. (D) Un esempio di mappa metrica che non supera il controllo visivo o di qualità del valore. Si noti come la linea rossa sull'istogramma mostri una curva che differisce da quella di A-C. Gli zero voxel cerchiati su questa mappa metrica sono dovuti a problemi di adattamento del tensore durante la pre-elaborazione (pannello D, riga superiore). Questo istogramma è un esempio di voxel generalmente più alti o più bassi del previsto in un set di dati (Pannello D, riga inferiore). Clicca qui per visualizzare una versione più grande di questa figura.
Controllo qualità
Una volta che PyDesigner ha elaborato i dati, la cartella metrics_qc (vedere la Figura 1) deve essere utilizzata per identificare i set di dati non ottimali. Per ogni set di dati, PyDesigner restituisce tre grafici utilizzati per il controllo di qualità.
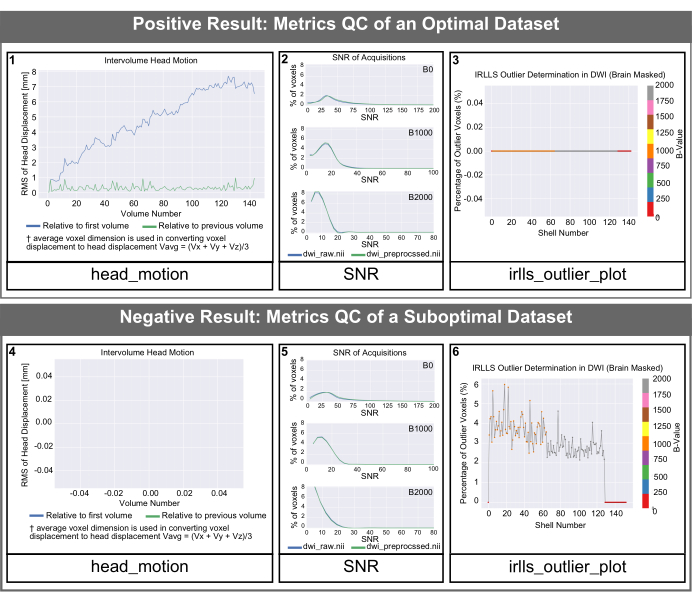
Figura 4: Istogrammi QC generati da PyDesigner per set di dati ottimali e non ottimali. SNR, movimento della testina tra volumi e istogrammi anomali generati da PyDesigner. Entrambe le righe rappresentano i dati di un cervello adulto sano. La riga superiore è un esempio di istogrammi di controllo qualità per un set di dati ottimale. La riga inferiore mostra gli output del controllo di qualità di un set di dati non ottimale con un artefatto di suscettibilità non correlato alla struttura o alla salute del cervello. Si noti che la dimensione del testo delle etichette negli output predefiniti di PyDesigner è inferiore a quella che apparirà in questa figura. Abbiamo aumentato la dimensione del testo in questa figura per la leggibilità. Clicca qui per visualizzare una versione più grande di questa figura.
I grafici head_motion nella Figura 4 mostrano lo spostamento della testa rispetto al primo volume e al volume precedente. Come illustrato nella Figura 4 (pannello 1), lo spostamento della testina è in genere ridotto e PyDesigner regola questi artefatti di movimento nella pipeline di elaborazione standard utilizzando il programma FSL Eddy insieme a TOPUP per la correzione del movimento e delle correnti parassite9. Per set di dati non ottimali, il grafico head_motion può apparire vuoto, come mostrato nella Figura 4 (pannello 4). Ciò indica che la correzione del movimento a correnti parassite non è riuscita, pertanto PyDesigner non è stato in grado di generare un grafico. I file di registro della correzione delle correnti parassite si trovano nella sottocartella delle correnti parassite all'interno della cartella metrics_qc (vedere la Figura 1). Il grafico del rapporto segnale/rumore (SNR) mostra 3 grafici. Ogni grafico è per un valore b diverso e mostra sia i dati pre-elaborati che quelli grezzi. Per un set di dati ottimale, il picco SNR dei dati grezzi dovrebbe essere ≥5 (Figura 4 [pannello 2]). I set di dati non ottimali avranno un picco di SNR dei dati grezzi di ≤3 (Figura 4 [pannello 5]). Idealmente, gli utenti dovrebbero vedere che il picco SNR per tutti i valori b aumenta leggermente, ma non in modo drammatico. Il grafico dei valori anomali si trova nella cartella di adattamento all'interno metrics_qc e mostra la percentuale di valori anomali nel set di dati (Figura 4 [pannello 3 e 6]). Un buon set di dati dovrebbe avere una bassa percentuale di valori anomali, in genere inferiore al 5% (Figura 4 [pannello 3]). Un set di dati non ottimale avrà un'ampia percentuale di valori anomali, come mostrato nella Figura 4 (pannello 6).
Esempi di risultati del set di dati
Una volta che PyDesigner ha terminato l'elaborazione del set di dati di esempio, tutti gli output devono essere contenuti all'interno della cartella "PyDesigner_Outputs". Questi output possono essere confrontati con quelli trovati nella cartella "derivatives" impacchettata con il set di dati di esempio scaricato da OpenNeuro (elaborato su MacOS 12.4). Se il software funziona correttamente, la struttura dei file di "PyDesigner_Outputs" e "derivati" sarà esattamente la stessa. Allo stesso modo, i grafici SNR, movimento della testa e valori anomali trovati nella sottocartella "metrics_qc" dovrebbero corrispondere a quelli nella Figura 5A. Le mappe metriche (che si trovano nella sottocartella "metriche") possono essere confrontate tramite software di imaging come FSLeyes, MRIcron, ImageJ, ecc. Gli istogrammi dei valori FA, MD e MK da metriche pre-elaborate possono essere visti nella Figura 5B. Si noti che tutti gli istogrammi metrici mostrati nella Figura 5B sono scalati in base alle scale dei valori metrici suggerite nella Tabella 1.
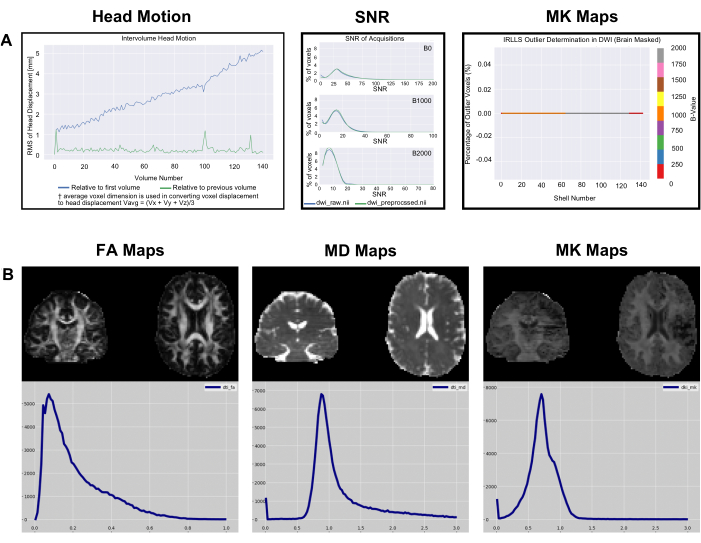
Figura 5: Esempio di istogrammi metrici e metrici QC. (A) SNR, movimento della testa intervolume e istogrammi anomali generati da PyDesigner per i dati di esempio scaricati da OpenNeuro. Si noti che la dimensione del testo è stata aumentata sui grafici in questa figura per garantire la leggibilità. (B) La metrica di un singolo soggetto mappa gli istogrammi dei conteggi dei valori voxel per FA, MD e MK per lo stesso set di dati, visualizzati tramite FSLeyes v6.0. Clicca qui per visualizzare una versione più grande di questa figura.
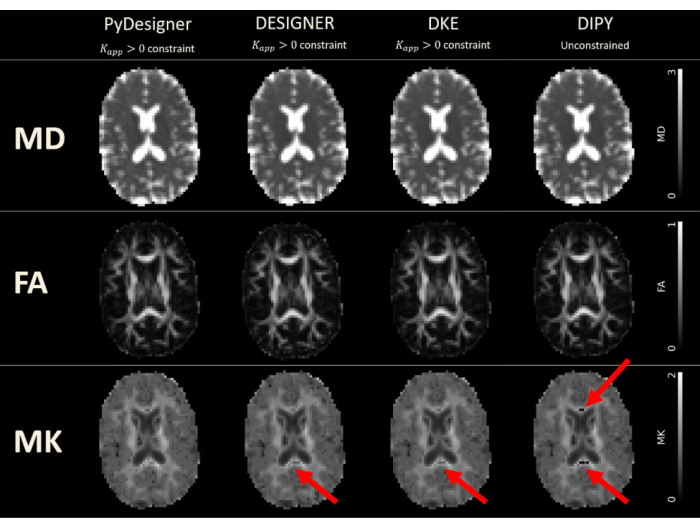
Figura 6: Mappe DTI e DKI derivate da PyDesigner, DESIGNER, DKE e DIPY. L'adattamento tensoriale è stato eseguito con il vincolo Kapp > 0 in PyDesigner, DESIGNER5 e Diffusional Kurtosis Estimator (DKE)27, mentre l'adattamento non vincolato è stato utilizzato in Diffusion Imaging in Python (DIPY)28 a causa di limitazioni software. Le unità per MD sono micrometri quadrati per millisecondo (μm2/ms), mentre le altre metriche sono adimensionali. Le interruzioni dovute a un problema generico dovuto alla correzione dell'anello di Gibbs possono essere viste nelle mappe MK prodotte da DESIGNER, DKE e DIPY. Questa figura è stata riprodotta con il permesso di Dhiman et al.29. Clicca qui per visualizzare una versione più grande di questa figura.
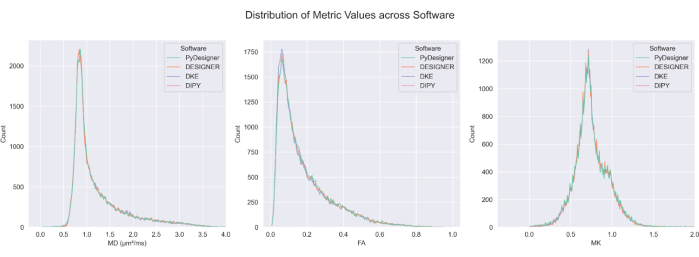
Figura 7: Confronto tra FA, MD e MK tra le pipeline. La distribuzione dei valori calcolati per FA, MD e MK da PyDesigner, DESIGNER5, DKE27 e DIPY28 nei cervelli esclusi dal liquido cerebrospinale è simile nella maggior parte dei voxel. Il calcolo della mappa metrica tra i metodi è confrontabile. Questa figura è stata riprodotta con il permesso di Dhiman et al.29. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
La motivazione principale per lo sviluppo di PyDesigner è stata quella di implementare gli elementi chiave di DESIGNER sostituendo tutto il codice MATLAB con Python, consentendo così una maggiore portabilità e accessibilità. PyDesigner e DESIGNER Dv1 producono output quasi identici29. Tuttavia, ci sono alcune opzioni aggiuntive, impostazioni predefinite e correzioni di bug minori inclusi in PyDesigner. La documentazione online di PyDesigner16 li descrive in dettaglio.
PyDesigner produce risultati simili anche agli strumenti di analisi DKI Diffusional Kurtosis Estimator (DKE)27 e DIPY28 (Diffusion Imaging in Python)28 (vedi Figura 6 e Figura 7), ma le mappe generate sulla base della dMRI pre-elaborata da DESIGNER sono considerate più accurate grazie al loro algoritmo di adattamento, come dimostrato da Ades-Aron et al.5. Vedere la Figura 6 per un confronto delle mappe metriche MD, FA e MK di PyDesigner, DESIGNER5, DKE27 e DIPY28. La Figura 7 mostra il confronto degli istogrammi MD, FA e MK di ciascuna pipeline. La combinazione di adattamento tensoriale vincolato, rilevamento dei valori anomali e correzione del coefficiente di curtosi apparente produce un adattamento tensoriale più robusto e accurato, come mostrato in PyDesigner e DESIGNER5.
Un vantaggio di PyDesigner rispetto a DESIGNER Dv1 è che è disponibile tramite il contenitore DockerNeuroDock 30, che migliora notevolmente la portabilità e semplifica l'installazione. Questo contenitore viene eseguito su tutte le principali piattaforme OS compatibili con Docker, tra cui Windows, Mac OS e varie distribuzioni Linux. In base al feedback degli utenti, PyDesigner v2.0 includerà aggiornamenti al contenitore Docker PyDesigner. I miglioramenti introdotti dalla nuova versione di PyDesigner e l'introduzione di un Dockerfile a più fasi risolveranno tutti i problemi esistenti che gli utenti stanno affrontando. Se gli utenti hanno problemi con l'installazione, consigliamo di inviare domande alla pagina di discussione16 di PyDesigner. La tecnologia dei container di Docker consente inoltre l'implementazione semplice su cluster ad alte prestazioni (HPC) per l'elaborazione rapida di DWI in batch su cluster locali compatibili con Docker.
PyDesigner include anche calcoli di modellazione microstrutturale che vanno oltre DKI, tra cui WMTI3, FBI e FBWM. Per WMTI, un set di dati DKI standard è adeguato e i parametri microstrutturali associati vengono calcolati per impostazione predefinita. Tuttavia, va sottolineato che la validità della WMTI è limitata alle regioni della sostanza bianca con FA elevata (cioè FA ≥ 0,4). Alcune metriche WMTI hanno un'accuratezza limitata a causa dell'assunzione dell'allineamento parallelo degli assoni in un dato voxel31. FBI 32,33,34 è un metodo dMRI distinto applicabile in tutta la sostanza bianca cerebrale, che richiede un valore b elevato (cioè b ≥ 4000 s/mm 2) e dati dMRI campionati con un minimo di 64 direzioni di codifica della diffusione (insieme ai dati per b = 0). I principali output di FBI sono la funzione di densità di orientamento delle fibre (fODF) per ogni voxel della sostanza bianca, che può essere utilizzata per la trattografia della sostanza bianca e funge da input per FBWM, nonché l'anisotropia frazionaria intra-assonale (FAA). FBWM utilizza i dati dMRI sia di DKI che di FBI per stimare gli stessi parametri di WMTI ma con una maggiore precisione, e può essere applicato in tutta la sostanza bianca indipendentemente dal valore FA. Pertanto, se questi dati aggiuntivi sono disponibili, le stime FBWM sono preferite a quelle di WMTI35. Come con l'FBI, FBWM è stato convalidato solo nella sostanza bianca cerebrale adulta.
Oltre alle invarianti rotazionali fornite da PyDesigner (orientamento RAS), i file .fib specifici di DSIstudio (orientamento LPS) vengono prodotti separatamente per DTI, DKI e FBI. I file .fib contengono informazioni direzionali ODF per la generazione di profili trattografici per ciascun metodo. I profili trattografici risultanti possono essere utilizzati per generare matrici di connettività a livello di soggetto e di gruppo. I file .fib DKI e FBI contengono informazioni multidirezionali che trasmettono informazioni sulle fibre incrociate che non sono disponibili con la trattografia DTI. Inoltre, all'interno di ogni file .fib, sono stati inclusi i vari invarianti rotazionali di ciascun metodo, che possono essere utilizzati in varie combinazioni come criteri per la semina, l'esecuzione e l'arresto della trattografia. Ulteriori dettagli su DSIstudio sono disponibili sul loro sito web36.
Un'altra caratteristica degna di nota di PyDesigner è l'input multi-file, che gli consente di gestire vari input di file: NifTi (.nii), NifTi compresso (.nii.gz), DICOM (.dcm) e MRtrix (.mif). PyDesigner è in grado di identificare automaticamente le informazioni di acquisizione dai metadati dell'intestazione indipendentemente dal formato di input ed eseguire le correzioni di conseguenza, supportando così un approccio pratico. Indipendentemente dalle differenze nei protocolli, lo stesso comando (vedi sopra) può essere utilizzato per elaborare un'ampia varietà di DWI. Questo software consente quindi di risparmiare tempo e fatica riducendo al minimo le fasi e i comandi di pre-elaborazione manuale. In un aggiornamento rilasciato di recente (v1.0-RC10), questo è stato migliorato introducendo la compatibilità per più set di dati echo-time (multi-TE). Ciò consente a PyDesigner di eseguire fasi di pre-elaborazione delle immagini, che sono in gran parte indipendenti da TE, su un DWI multi-TE per produrre un'immagine con rumore e artefatti minimi. I calcoli del tensore dipendenti dal TE vengono quindi eseguiti separatamente su ciascun TE per produrre metriche di diffusione o curtosi.
Si noti che PyDesigner v1.0 non rappresenta un endpoint nella progettazione delle pipeline di pre-elaborazione delle immagini. Lo sviluppo e la convalida di strumenti di elaborazione delle immagini è un'area di ricerca attiva. In particolare, durante la preparazione di questo manoscritto, sono stati proposti nuovi sviluppi nel denoising delle immagini e nella rimozione del ringing di Gibbs, con conseguente rilascio di una pipeline di Designer rivista, Designer Dv237, che include una migliore riduzione del rumore e correzione degli artefatti di Gibbs per la dMRI acquisita con acquisizioni parziali di Fourier35, ed è disponibile su DESIGNER Github38. Allo stesso modo, la conformità organizzativa BIDS39 è un aspetto importante di qualsiasi pipeline di analisi MRI. Il formato BIDS consente una condivisione dei dati e un'implementazione della pipeline più efficienti stabilendo una struttura di set specifica per tutti i tipi di dati MRI. Sebbene PyDesigner non sia attualmente conforme a BIDS, una prossima versione di PyDesigner includerà output conformi a BIDS. Inoltre, PyDesigner è stato originariamente scritto utilizzando Python versione 3.7. Al momento di questa pubblicazione, Python 3.7 è ora considerato obsoleto. Tuttavia, le informazioni contenute in questo manoscritto sono indipendenti dalla versione Python. In altre parole, le procedure descritte nel presente documento che sono importanti funzioni di pre-elaborazione (ad esempio, denoising, correzione del ringing di Gibbs, ecc.) seguiranno lo stesso flusso di lavoro operativo e concettuale indipendentemente dalle modifiche apportate alle procedure di installazione. È importante sottolineare che, come per tutti i software basati su Python, le future versioni di PyDesigner verranno aggiornate a una nuova versione di Python. Ci sforziamo di convalidare e integrare ulteriormente tali sviluppi nel tempo.
Per la versione più aggiornata di PyDesigner, inclusa qualsiasi nuova documentazione relativa agli aggiornamenti della versione di Python, i lettori sono incoraggiati a consultare il sito Web prima di iniziare una nuova analisi e a partecipare al forum di discussione in cui è possibile inviare domande relative a PyDesigner16. L'implementazione di Docker per la portabilità si chiama NeuroDock30, che contiene PyDesigner e le sue dipendenze per consentire l'elaborazione su un'ampia gamma di piattaforme.
Divulgazioni
Nessuno.
Riconoscimenti
Siamo grati per le utili discussioni con Olivia Horn, Daniel Lench e Graham Warner.
La ricerca riportata in questa pubblicazione è stata supportata, in parte, dalle sovvenzioni del National Institutes of Health R01AG054159, R01AG057602, R01AG055132, R01DC014021, R01NS110347, R21DA050085, F31NS108623, P20GM109040, P50DC000422, T32GM008716 e T32DC014435. Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali del National Institutes of Health. Ulteriori finanziamenti sono stati forniti dalla Litwin Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
Riferimenti
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon