È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi delle infezioni delle cellule epiteliali con Shigella
In questo articolo
Riepilogo
Il presente protocollo descrive saggi di infezione per interrogare l'aderenza, l'invasione e la replicazione intracellulare di Shigella utilizzando linee cellulari epiteliali in vitro .
Abstract
Il patogeno batterico enterico adattato all'uomo Shigella causa milioni di infezioni ogni anno, crea effetti di crescita a lungo termine tra i pazienti pediatrici ed è una delle principali cause di decessi per diarrea in tutto il mondo. L'infezione induce diarrea acquosa o sanguinolenta a causa dell'agente patogeno che transita nel tratto gastrointestinale e infetta le cellule epiteliali che rivestono il colon. Con l'aumento sbalorditivo della resistenza agli antibiotici e l'attuale mancanza di vaccini approvati, i protocolli di ricerca standardizzati sono fondamentali per studiare questo formidabile agente patogeno. In questo articolo, vengono presentate metodologie per esaminare la patogenesi molecolare della Shigella utilizzando analisi in vitro dell'aderenza batterica, dell'invasione e della replicazione intracellulare nelle cellule epiteliali del colon. Prima delle analisi dell'infezione, il fenotipo di virulenza delle colonie di Shigella è stato verificato mediante l'assorbimento del colorante rosso Congo su piastre di agar. Durante la coltura batterica possono essere presi in considerazione anche terreni di laboratorio integrati per simulare le condizioni in vivo . Le cellule batteriche vengono quindi utilizzate in un protocollo standardizzato per infettare le cellule epiteliali del colon nelle piastre di coltura tissutale a una molteplicità stabilita di infezione con adattamenti per analizzare ogni fase dell'infezione. Per i saggi di aderenza, le cellule di Shigella vengono incubate con livelli di terreno ridotti per promuovere il contatto batterico con le cellule epiteliali. Sia per i saggi di invasione che per quelli di replicazione intracellulare, la gentamicina viene applicata per vari intervalli di tempo per eliminare i batteri extracellulari e consentire la valutazione dell'invasione e/o la quantificazione dei tassi di replicazione intracellulare. Tutti i protocolli di infezione enumerano i batteri aderenti, invasi e/o intracellulari diluendo in serie i lisati di cellule epiteliali infette e placcando le unità formanti colonie batteriche rispetto ai titoli infettivi sulle piastre di agar rosso del Congo. Insieme, questi protocolli consentono una caratterizzazione e un confronto indipendenti per ogni fase dell'infezione da Shigella delle cellule epiteliali per studiare con successo questo patogeno.
Introduzione
Le malattie diarroiche causate da patogeni batterici enterici rappresentano un onere significativo per la salute globale. Nel 2016, le malattie diarroiche sono state responsabili di 1,3 milioni di decessi in tutto il mondo e sono state la quarta causa di morte nei bambini di età inferiore ai cinque anni(1,2). Il patogeno batterico enterico Gram-negativo Shigella è l'agente eziologico della shigellosi, una delle principali cause di decessi per diarrea in tutto il mondo3. La shigellosi causa ogni anno una significativa morbilità e mortalità nei bambini dei paesi a basso e medio reddito 4,5, mentre le infezioni nei paesi ad alto reddito sono collegate a focolai di asili nido, di origine alimentare e idrica 6,7,8,9. Lo sviluppo inefficace di vaccini10 e l'aumento dei tassi di resistenza antimicrobica (AMR)11,12 hanno complicato la gestione delle epidemie di Shigella su larga scala. I recenti dati dei Centers for Disease Control and Prevention mostrano che quasi il 46% delle infezioni da Shigella negli Stati Uniti ha mostrato resistenza ai farmaci nel 202013,14, mentre l'Organizzazione Mondiale della Sanità ha dichiarato la Shigella come patogeno prioritario della resistenza antimicrobica per la quale sono urgentemente necessarie nuove terapie15.
Le infezioni da Shigella si trasmettono facilmente per via oro-fecale dopo l'ingestione di cibo o acqua contaminati o attraverso il contatto umano diretto. La Shigella si è evoluta per essere un agente patogeno efficiente e adattato all'uomo, con una dose infettiva di 10-100 batteri sufficiente a causare la malattia16. Durante il transito dell'intestino tenue, la Shigella è esposta a segnali ambientali, come la temperatura elevata e la bile17. Il rilevamento di questi segnali induce cambiamenti trascrizionali per esprimere fattori di virulenza che aumentano la capacità dei batteri di infettare il colon umano 17,18,19. La Shigella non invade l'epitelio del colon dalla superficie apicale, ma piuttosto transita attraverso lo strato epiteliale dopo l'assorbimento in cellule microfold specializzate presentanti l'antigene (cellule M) all'interno dell'epitelio associato al follicolo 20,21,22. Dopo la transcitosi, le cellule Shigella vengono fagocitate dai macrofagi residenti. La Shigella sfugge rapidamente al fagosoma e innesca la morte delle cellule macrofagiche, con conseguente rilascio di citochine pro-infiammatorie 5,23,24. La Shigella invade quindi le cellule epiteliali del colon dal lato basolaterale, lisa il vacuolo macropinocitico e stabilisce una nicchia replicativa nel citoplasma 5,25. Le citochine pro-infiammatorie, in particolare l'interleuchina-8 (IL-8), reclutano leucociti neutrofili polimorfonucleati (PMN) nel sito di infezione, che indebolisce le giunzioni strette epiteliali e consente l'infiltrazione batterica del rivestimento epiteliale per esacerbare l'infezione basolaterale5. I PMN distruggono il rivestimento epiteliale infetto per contenere l'infezione, che si traduce nei sintomi caratteristici della dissenteria bacillare (sanguinolenta)5. Sebbene i meccanismi di invasione e replicazione intracellulare siano stati accuratamente caratterizzati, una nuova ricerca sta dimostrando nuovi importanti concetti nell'infezione da Shigella, tra cui la regolazione della virulenza durante il transito gastrointestinale (GI)17, l'aderenza19, il miglioramento dell'accesso basolaterale attraverso la permeabilità della barriera26 e il trasporto asintomatico nei bambini malnutriti27.
La capacità di Shigella spp. di causare malattie diarroiche è limitata agli esseri umani e ai primati non umani (NHP)28. Sono stati sviluppati modelli di infezione intestinale della Shigella per il pesce zebra29, i topi30, i porcellini d'India31, i conigli 21,32,33 e i maiali34,35. Tuttavia, nessuno di questi sistemi modello è in grado di replicare accuratamente le caratteristiche della malattia osservate durante l'infezione umana36. Sebbene siano stati stabiliti modelli NHP di shigellosi per studiare la patogenesi della Shigella, questi sistemi modello sono costosi da implementare e richiedono dosi infettive artificialmente elevate, fino a nove ordini di grandezza superiori alla dose infettiva degli esseri umani 37,38,39,40,41,42. Pertanto, il notevole adattamento della Shigella per l'infezione di ospiti umani richiede l'uso di colture cellulari di derivazione umana per ricreare modelli fisiologicamente rilevanti per un'accurata interrogazione della patogenesi della Shigella.
Qui, vengono descritte procedure dettagliate per misurare i tassi di aderenza, invasione e replicazione di Shigella all'interno delle cellule epiteliali del colon HT-29. Utilizzando questi protocolli standardizzati, i meccanismi molecolari con cui i geni di virulenza batterica e i segnali ambientali influenzano ogni fase dell'infezione da Shigella possono essere interrogati per comprendere meglio la relazione dinamica di interazione ospite-patogeno.
Protocollo
1. Preparazione di reagenti e materiali
NOTA: Tutti i volumi sono coerenti con un test che utilizza due piastre a 6 pozzetti.
- TSB mezzo: Aggiungere 0,5 L di acqua deionizzata (DI) a 15 g di brodo di soia triptico (TSB, vedi tabella dei materiali) e autoclave. Conservare a temperatura ambiente.
- Terreno di sali biliari (TSB + BS): Per preparare TSB contenente lo 0,4% (p/v) di sali biliari, risospendere 0,06 g di sali biliari (BS, vedere la tabella dei materiali) in 15 mL di TSB sterilizzato in autoclave. Sterilizzare con filtro utilizzando un filtro PES da 0,22 μm.
NOTA: I sali biliari sono costituiti da una miscela 1:1 di colato di sodio e desossicolato di sodio. Preparare i terreni freschi immediatamente prima dell'uso. - DMEM + 10% (v/v) FBS: Aggiungere 5 mL di siero fetale bovino (FBS) a 45 mL di Dulbecco's Modified Eagle Medium (DMEM). Conservare a 4 °C.
- DMEM + gentamicina: a una provetta da 50 mL, aggiungere 50 mL di DMEM e 50 μL di gentamicina da 50 mg/mL (vedere la tabella dei materiali).
NOTA: Preparare un'aliquota fresca e scaldare a bagnomaria a 37 °C prima di ogni esperimento. - PBS + 1% (v/v) Triton X-100: Aggiungere 150 μL di Triton X-100 a 15 mL di soluzione salina tamponata con fosfato (PBS).
NOTA: Preparare un'aliquota fresca e scaldare a bagnomaria a 37 °C prima di ogni esperimento. - Piastre indicatrici rosse TSB + Congo: Aggiungere 15 g di TSB, 7,5 g di agar selezionato e 0,125 g di colorante rosso Congo (vedi Tabella dei materiali) in un flacone da 1 L. Aggiungere 0,5 L di acqua deionizzata e autoclave. Versare 10-20 ml di terreno in singole piastre di Petri sterili (100 mm x 15 mm) e lasciare solidificare.
ATTENZIONE: Il rosso Congo è cancerogeno e una tossina riproduttiva. Assicurarsi che la manipolazione del rosso Congo avvenga utilizzando i dispositivi di protezione individuale appropriati. Consultare la scheda di sicurezza del prodotto per ulteriori informazioni.
NOTA: Circa 20 lastre sono realizzate con supporti rossi Congo da 0,5 L. I piatti possono essere preparati con 2-3 giorni di anticipo e lasciati capovolti a temperatura ambiente fino al momento dell'uso. Per la conservazione a lungo termine, posizionare le piastre invertite in manicotti di plastica a 4 °C per un massimo di 3 mesi. - DMEM + 10% (v/v) FBS e 5% (v/v) dimetilsolfossido (DMSO): aggiungere 42,5 mL di DMEM, 5 mL di FBS e 2,5 mL di DMSO a una provetta da 50 mL. Conservare a 4 °C.
2. Preparazione dei batteri
NOTA: Tutti i protocolli di coltivazione e conservazione della Shigella in laboratorio sono adattati da Payne, S. M.43.
ATTENZIONE: Le Shigella spp. sono patogeni del gruppo di rischio 244. Eseguire tutto il lavoro di laboratorio in un ambiente BSL-2, con ulteriori misure di sicurezza adottate per limitare le esposizioni accidentali dovute alla bassa dose infettiva di Shigella spp.
- Crescita della Shigella da stock congelati
- Trasferire una piccola quantità di coltura congelata dal flaconcino criogenico a una piastra di agar rosso TSB + Congo utilizzando un applicatore sterile.
- Sterilizzare a fiamma un ciclo di inoculazione e lasciarlo raffreddare. Strisciare l'inoculo avanti e indietro su un quadrante della piastra. Fiammeggiare l'anello, lasciarlo raffreddare, quindi strisciare dal primo quadrante al secondo quadrante della piastra. Ripetere l'operazione per striare l'inoculo nel terzo e quarto quadrante della piastra.
NOTA: In alternativa, striare l'inoculo utilizzando un applicatore sterile fresco tra ciascun quadrante. - Capovolgere la piastra e incubare a 37 °C per una notte.
NOTA: L'incubazione a temperature ≥37 °C è necessaria per l'espressione dei fattori di virulenza della Shigella necessari per l'osservazione del fenotipo45 del Congo rosso-positivo (CR+). Le colonie avirulente avranno un aspetto bianco e non saranno invasive. - Sigillare la piastra con pellicola di paraffina e conservare in frigorifero a 4 °C.
NOTA: Le colonie batteriche rimarranno vitali sulle piastre di agar per 1-2 settimane.
- Crescita notturna della Shigella in coltura liquida
- Aliquotare 3 mL di terreno TSB in provette sterili da 14 mL.
- Prelevare una singola colonia rossa (CR+) ben isolata utilizzando un applicatore sterile e risospendere in terreno liquido.
- Incubare le colture per una notte (16-18 h) a 37 °C con agitazione a 250 rotazioni al minuto (rpm).
3. Preparazione di cellule eucariotiche HT-29
NOTA: Tutti i volumi sono coerenti con un test che utilizza due piastre a 6 pozzetti. Le linee cellulari HT-29 sono state acquisite dall'American Type Culture Collection (ATCC). I protocolli di manutenzione HT-29 sono adattati dalle raccomandazioni ATCC46. Tutti i fluidi devono essere preriscaldati a bagnomaria a 37 °C prima dell'uso. Tutti i protocolli di manutenzione HT-29 devono essere eseguiti in una cabina di biosicurezza. Astenersi dal produrre bolle durante la miscelazione/lavorazione con le cellule HT-29 nei terreni per evitare cambiamenti drammatici del pH.
- Scongelamento di cellule HT-29 da brodo congelato
- Scongelare il flaconcino di cellule HT-29 a bagnomaria a 37 °C.
NOTA: Assicurarsi che il tappo rimanga completamente al di sopra dell'acqua per evitare contaminazioni. Lo scongelamento dovrebbe richiedere meno di 2 minuti. - Rimuovere il flaconcino dall'acqua immediatamente dopo che la coltura è stata completamente scongelata e decontaminare con etanolo al 70%. Assicurarsi che tutte le fasi da questo punto vengano eseguite utilizzando tecniche asettiche.
- Aggiungere tutto il contenuto del flaconcino a una provetta da centrifuga da 15 mL contenente 9 mL di DMEM + 10% FBS. Centrifugare a 125 x g per 5 min a temperatura ambiente.
- Travasare il surnatante in un contenitore di scarto e risospendere il pellet in 10 mL di DMEM caldo + 10% FBS. Trasferire le cellule risospese in un matraccio per coltura tissutaleda 75 cm 2 (T75) contenente 10 mL di DMEM caldo + 10% FBS (volume totale di 20 mL).
- Incubare le cellule a 37 °C con il 5% di CO2 fino a raggiungere il 90% di confluenza (circa 6-7 giorni).
NOTA: La confluenza è stimata attraverso l'approssimazione visiva.
- Scongelare il flaconcino di cellule HT-29 a bagnomaria a 37 °C.
- Semina di cellule HT-29
- Preriscaldare 20 mL di PBS e 50 mL di DMEM + 10% FBS a bagnomaria a 37 °C e preriscaldare 3 mL di tripsina-EDTA allo 0,25% (p/v) a temperatura ambiente.
- Una volta che le cellule HT-29 (dal punto 3.1) raggiungono il 90% di confluenza, travasare il terreno di coltura cellulare HT-29 dal matraccio T75 in un contenitore per rifiuti. Versare ~10 ml di PBS caldo nel pallone e agitare delicatamente per lavare. Travasare il PBS in un contenitore per rifiuti. Lavare nuovamente con PBS caldo e decantare.
- Aggiungere 2-3 mL di tripsina-EDTA allo 0,25% (p/v) e agitare delicatamente su tutta la superficie. Incubare a 37 °C con il 5% di CO2 per 4 min.
- Rimuovere il pallone dall'incubatrice e agitare delicatamente la tripsina-EDTA, assicurandosi visivamente che tutte le cellule si stacchino dalla superficie.
- Aggiungere immediatamente 6 mL di DMEM caldo + 10% FBS per disattivare la tripsina. Pipettare su e giù per mescolare accuratamente.
- Trasferire tutto il contenuto in una provetta da centrifuga da 15 mL e centrifugarla a 500 x g per 5 minuti a temperatura ambiente.
- Decantare delicatamente il surnatante in un contenitore per rifiuti e risospendere il pellet in 6 mL di DMEM caldo + 10% FBS.
- Immediatamente dopo la risospensione, trasferire 10 μL di cellule HT-29 sospese dal centro della coltura a una provetta PCR da 0,2 mL. Aggiungere 10 μL di colorante blu Trypan nella provetta PCR e mescolare.
- Aggiungere 10 μL di miscela di cellule HT-29/blu di tripano a un vetrino monouso della camera di conteggio delle cellule Countess (vedere la tabella dei materiali). Enumerare il numero di cellule vive e calcolare la vitalità cellulare.
NOTA: Quando si documenta il numero di cellule nel campione, leggere il numero sotto il conteggio delle cellule "vive", non il numero totale delle cellule. In alternativa, l'enumerazione delle cellule può essere eseguita manualmente utilizzando un emocitometro. - Le cellule HT-29 in sospensione del seme in un matraccio T75 fresco o in una piastra a 6 pozzetti.
- Per il pallone T75:
- Pipettare delicatamente per mescolare, quindi trasferire 2,5 x 106 cellule in un matraccio T75 fresco secondo l'equazione seguente:
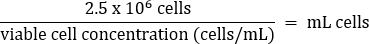
- Aggiungere terreno caldo DMEM + 10% FBS a un volume finale di 20 mL (concentrazione finale di 1,25 x 105 cellule/mL).
- Disperdere le cellule in modo uniforme sul pallone dondolando delicatamente avanti e indietro.
- Incubare a 37 °C con il 5% di CO2 fino a quando le cellule raggiungono l'80% di confluenza.
NOTA: Per una crescita ottimale, sostituire il terreno DMEM + 10% FBS nel pallone T75 ogni ~3 giorni. Travasare il terreno in un contenitore per rifiuti e aggiungere 10 ml di PBS caldo nel pallone. Agitare delicatamente il PBS e travasarlo nel contenitore dei rifiuti. Quindi aggiungere 20 mL di DMEM fresco e caldo + 10% FBS nel matraccio e rimettere nell'incubatore a 37 °C, 5% CO2 .
- Pipettare delicatamente per mescolare, quindi trasferire 2,5 x 106 cellule in un matraccio T75 fresco secondo l'equazione seguente:
- Per piastra a 6 pozzetti:
- Pipettare delicatamente per mescolare, quindi trasferire 5,85 x 106 cellule in una nuova provetta conica da 50 mL secondo l'equazione seguente:
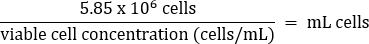
- Aggiungere terreno DMEM caldo + 10% FBS a un volume finale di 26 mL (concentrazione finale di 2,25 x 105 cellule/mL).
- Pipettare delicatamente per mescolare, quindi erogare 2 mL (4,5 x 105 cellule) in singoli pozzetti di piastre a 6 pozzetti.
- Disperdere le cellule in modo uniforme attraverso il pozzetto oscillando delicatamente su/giù e sinistra/destra 2-3 volte.
- Incubare a 37 °C con il 5% di CO2 fino a quando le cellule raggiungono l'80%-95% di confluenza (circa 3-4 giorni).
NOTA: per i saggi di invasione e replicazione intracellulare si raccomanda una confluenza dell'85%, mentre per i saggi di aderenza si raccomanda una confluenza del 90%-95%. Le cellule dovrebbero raggiungere ~85% di confluenza dopo 48 ore di incubazione con una concentrazione finale di circa 1 x 106 cellule/pozzetto. Possono essere necessari aggiustamenti del numero di cellule seminate e della durata dell'incubazione.
- Pipettare delicatamente per mescolare, quindi trasferire 5,85 x 106 cellule in una nuova provetta conica da 50 mL secondo l'equazione seguente:
- Per il pallone T75:
- Preparazione di brodi HT-29 congelati
- Aliquotare 1 mL di DMEM + 10% FBS + 5% di terreno DMSO in singole fiale criogeniche.
- Aggiungere 1 x 106 cellule HT-29 del passaggio 3.2.7 a ciascun flaconcino. Calcola il volume delle celle secondo la formula seguente:
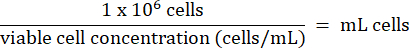
- Conservare le celle HT-29 a lungo termine a temperature inferiori a -130 °C in un congelatore a vapori di azoto liquido.
4. Saggio di aderenza
NOTA: Tutti i volumi sono coerenti con un test che utilizza due piastre a 6 pozzetti.
- Sottocoltura durante la notte delle colture Shigella tramite diluizione 1:50 in terreni freschi.
- Vortex, quindi aggiungere 100 μL di ciascuna coltura notturna a 5 mL di TSB fresco o TSB + BS in una provetta di coltura di dimensioni adeguate.
NOTA: Limitare il volume di coltura al <20% del volume del pallone di coltura o della provetta per garantire una corretta aerazione. - Incubare a 37 °C con agitazione a 250 giri/min fino a quando le cellule raggiungono una densità ottica (OD600) di 0,7 (fase intermedia della crescita della Shigella ); circa 2-2,5 h.
NOTA: Durante la sottocoltura, somministrare 50 mL di DMEM e un volume sufficiente di PBS per tutte le fasi di lavaggio e porre a bagnomaria a 37 °C. Lasciare che i supporti raggiungano i 37 °C prima dell'uso.
- Vortex, quindi aggiungere 100 μL di ciascuna coltura notturna a 5 mL di TSB fresco o TSB + BS in una provetta di coltura di dimensioni adeguate.
- Trasferire 2 x 108 unità formanti colonie (CFU) Shigella sottocoltivata in singole provette per microcentrifuga da 2 mL.
NOTA: 2 x 108 CFU corrispondono a circa 1 mL di cellule batteriche ad un OD600 di 0,7. Utilizzare le letture OD600 per approssimare CFU/mL in base alla calibrazione di ogni singolo spettrofotometro. - Lavare ogni campione di Shigella 2 volte con PBS.
- Celle a pellet per centrifugazione a 17.000 x g per 2 minuti a temperatura ambiente. Aspirare il surnatante, quindi aggiungere 1 mL di PBS caldo e risospendere bene il pellet, pipettando delicatamente il campione su e giù fino a quando la miscela non è completamente omogenea (8-10x).
- Ripetere il passaggio 4.3.1 1 volta in più.
- Celle di pellet mediante centrifugazione a 17.000 x g per 2 minuti a temperatura ambiente, aspirare il surnatante e risospendere i pellet in 2 mL di DMEM caldo.
NOTA: La concentrazione finale di batteri risospesi sarà di 1 x 108 CFU/mL.
- Vortex, quindi aggiungere 1 mL (1 x 108 CFU) di Shigella risospesa a ciascun pozzetto dei monostrati epiteliali del colon HT-29 preparati in piastre a 6 pozzetti (dal passaggio 3.2.10.2).
NOTA: Le infezioni vengono normalmente eseguite con una molteplicità di infezioni (MOI; rapporto tra cellule batteriche ed epiteliali) di 100. Per testare diversi MOI, diluire la Shigella risospesa in DMEM caldo alla concentrazione desiderata, quindi aggiungere 1 mL di batteri diluiti ai monostrati HT-29. Ad esempio, per testare un MOI di 10, diluire i batteri 1:10 aggiungendo 150 μL di 1 x 108 CFU/mL di batteri a 1,35 mL di DMEM caldo, quindi applicare 1 mL (1 x 107 CFU) alle cellule HT-29. - Incubare le piastre a 6 pozzetti a 37 °C con il 5% di CO2 per 3 ore.
- Durante l'incubazione, determinare il titolo dell'infezione batterica.
- Preparare diluizioni seriali 10 volte di cellule Shigella risospese (dal punto 4.3.3) in PBS.
- Fissare 100 μL delle diluizioni 1 x 10-5 e 1 x 10-6 su piastre rosse TSB + Congo e incubare per una notte a 37 °C.
NOTA: La placcatura di 100 μL dalle diluizioni 1 x 10-5 e 1 x 10-6 corrisponde a un fattore di diluizione finale rispettivamente di 1 x 10-6 e 1 x 10-7.
- Dopo l'incubazione, lavare i monostrati 4-5 volte con PBS.
- Aspirare il terreno da ciascun pozzetto.
NOTA: Quando si aspirano i fluidi da piastre a 6 pozzetti, guidare la punta dell'aspiratore lungo il lato inferiore dei pozzetti, cercando di evitare il contatto con le celle HT-29. - Aggiungere 1 ml di PBS caldo in ogni pozzetto e lavare delicatamente.
NOTA: Per lavare delicatamente i monostrati a 6 pozzetti con PBS, spostare la piastra su e giù e da un lato all'altro sul piano di lavoro. Il lavaggio delle piastre con un movimento circolare e/o la rimozione della piastra dalla superficie del piano di lavoro può causare la rimozione meccanica delle cellule dalla plastica. - Ripetere i passaggi 4.7.1 e 4.7.2 altre 4 volte.
- Aspirare il terreno da ciascun pozzetto.
- Rimuovere il PBS mediante aspirazione e lisare le cellule HT-29 aggiungendo 1 mL di PBS + 1% di Triton X-100 a ciascun pozzetto.
- Incubare le piastre a 6 pozzetti a 37 °C per 5 min.
- Utilizzare un raschietto per cellule o una punta di pipetta piegata per raschiare le cellule lisate dal fondo del pozzetto e trasferire l'intero 1 mL in una nuova provetta per microcentrifuga da 1,7 mL.
- Determinare il numero di batteri associati alle cellule.
- Vorticare ogni tubo (dal punto 4.10) per almeno 30 secondi per spostare ulteriormente la Shigella dalle cellule eucariotiche lisate.
- Preparare diluizioni seriali di 10 volte di lisati in PBS.
- Piastre da 100 μL delle diluizioni 1 x 10-2, 1 x 10-3 e 1 x 10-4 su piastre rosse TSB + Congo e incubare per una notte a 37 °C.
NOTA: La placcatura di 100 μL dalle diluizioni 1 x 10-2, 1 x 10-3 e 1 x 10-4 corrisponde a un fattore di diluizione finale rispettivamente di 1 x 10-3, 1 x 10-4 e 1 x 10-5.
5. Saggio di invasione
NOTA: Tutti i volumi sono coerenti con un test che utilizza due piastre a 6 pozzetti.
- Sottocoltura durante la notte delle colture Shigella tramite diluizione 1:50 in terreni freschi.
- Vortex, quindi aggiungere 100 μL di ciascuna coltura notturna a 5 mL di TSB fresco o TSB + BS in una provetta di coltura di dimensioni adeguate.
NOTA: Limitare il volume di coltura al <20% del volume del pallone di coltura o della provetta per garantire una corretta aerazione. - Incubare a 37 °C agitando a 250 rpm fino a quando le cellule raggiungono un OD600 di 0,7 (fase intermedia della crescita della Shigella ); circa 2-2,5 h.
NOTA: Durante la sottocoltura, aliquotare 50 mL di DMEM + 50 mg/mL di gentamicina e un volume sufficiente di PBS per tutte le fasi di lavaggio e porre a bagnomaria a 37 °C. Lasciare che i supporti raggiungano i 37 °C prima dell'uso.
- Vortex, quindi aggiungere 100 μL di ciascuna coltura notturna a 5 mL di TSB fresco o TSB + BS in una provetta di coltura di dimensioni adeguate.
- Trasferire 2 x 10 Shigella sottocoltivata da8 CFU in provette individuali da microcentrifuga da 2 mL.
NOTA: 2 x 108 CFU corrispondono a circa 1 mL di cellule batteriche ad un OD600 di 0,7. Utilizzare le letture OD600 per approssimare CFU/mL in base alla calibrazione di ogni singolo spettrofotometro. - Lavare i campioni di Shigella 1 volta con PBS.
- Celle a pellet per centrifugazione a 17.000 x g per 2 minuti a temperatura ambiente. Aspirare il surnatante, quindi aggiungere 1 mL di PBS caldo e risospendere bene il pellet, pipettando delicatamente il campione su e giù fino a quando la miscela non è completamente omogenea (8-10x).
- Ripetere il passaggio 5.3.1. 1x tempo supplementare.
- Celle di pellet mediante centrifugazione a 17.000 x g per 2 minuti a temperatura ambiente, aspirare il surnatante e risospendere i pellet in 2 mL di DMEM caldo.
NOTA: La concentrazione finale di batteri risospesi sarà di 1 x 108 CFU/mL.
- Vortex, quindi aggiungere 1 mL (1 x 108 CFU) di Shigella risospesa più 1 mL di DMEM a ciascun pozzetto dei monostrati epiteliali del colon HT-29 preparati in piastre a 6 pozzetti (dal passaggio 3.2.10.2).
NOTA: Le infezioni vengono normalmente eseguite con una molteplicità di infezioni (MOI; rapporto tra cellule batteriche ed epiteliali) di 100. Per testare diversi MOI, diluire la Shigella in sospensione in DMEM alla concentrazione desiderata, quindi aggiungere 1 mL di batteri diluiti ai monostrati HT-29. Ad esempio, per testare un MOI di 10, diluire i batteri 1:10 aggiungendo 150 μL di 1 x 108 CFU/mL di batteri a 1,35 mL di DMEM, quindi aggiungere 1 mL (1 x 107 CFU) alle cellule HT-29. - Per favorire il contatto batterico con le cellule HT-29, centrifugare le piastre a 6 pozzetti a 2.000 x g per 10 minuti a temperatura ambiente o 37 °C se è possibile regolare l'impostazione della temperatura.
NOTA: La centrifugazione favorisce il contatto batterico con le cellule HT-29, che bypassa la necessità di fattori di aderenza e consente ai batteri di invadere rapidamente le cellule. - Incubare piastre a 6 pozzetti a 37 °C con il 5% di CO2 per 45 min.
- Durante l'incubazione, determinare il titolo dell'infezione batterica.
- Preparare diluizioni seriali di 10 volte di cellule Shigella risospese (dal punto 5.3.3) in PBS.
- Fissare 100 μL delle diluizioni 1 x 10-5 e 1 x 10-6 su piastre rosse TSB + Congo e incubare per una notte a 37 °C.
NOTA: La placcatura di 100 μL dalle diluizioni 1 x 10-5 e 1 x 10-6 corrisponde a un fattore di diluizione finale rispettivamente di 1 x 10-6 e 1 x 10-7.
- Lavare accuratamente le cellule HT-29 infette 3 volte con 1 ml di PBS.
- Aspirare il terreno da ciascun pozzetto.
NOTA: Quando si aspirano i fluidi da piastre a 6 pozzetti, guidare la punta dell'aspiratore lungo il lato inferiore dei pozzetti, cercando di evitare il contatto con le celle HT-29. - Aggiungere 1 ml di PBS caldo in ogni pozzetto e lavare delicatamente.
NOTA: Per lavare delicatamente i monostrati a 6 pozzetti con PBS, spostare la piastra su e giù e da un lato all'altro sul piano di lavoro. Il lavaggio delle piastre con un movimento circolare e/o la rimozione della piastra dalla superficie del piano di lavoro può causare la rimozione meccanica delle cellule dalla plastica. - Ripetere i passaggi 5.8.1 e 5.8.2 altre 2 volte.
- Aspirare il terreno da ciascun pozzetto.
- Rimuovere il PBS mediante aspirazione, quindi aggiungere 2 mL di DMEM caldo integrato con 50 μg/mL di gentamicina in ciascun pozzetto e incubare per 30 minuti a 37 °C con il 5% di CO2 .
- Lavare accuratamente le cellule HT-29 infette 3 volte con 1 ml di PBS.
- Ripetere il passaggio di lavaggio 5.8.
- Rimuovere il PBS mediante aspirazione, quindi aggiungere 2 mL di DMEM caldo integrato con 50 μg/mL di gentamicina in ciascun pozzetto e incubare per 60 minuti a 37 °C con il 5% di CO2 .
- Lavare accuratamente le cellule HT-29 infette 3 volte con 1 ml di PBS.
- Ripetere il passaggio di lavaggio 5.8.
- Rimuovere il PBS mediante aspirazione e lisare le cellule HT-29 aggiungendo 1 mL di PBS + 1% di Triton X-100 a ciascun pozzetto.
- Incubare le piastre a 6 pozzetti a 37 °C per 5 min.
- Utilizzare un raschietto per cellule o una punta di pipetta piegata per raschiare le cellule lisate dal fondo del pozzetto e trasferire l'intero 1 mL in una nuova provetta per microcentrifuga da 1,7 mL.
- Determinare il numero di batteri intracellulari.
- Vorticare ogni tubo (dal punto 5.15) per almeno 30 s per spostare ulteriormente la Shigella dalle cellule eucariotiche lisate.
- Preparare diluizioni seriali di 10 volte di lisati in PBS.
- Piastre da 100 μL delle diluizioni 1 x 10-2 e 1 x 10-3 su piastre rosse TSB + Congo e incubare per una notte a 37 °C.
NOTA: La placcatura di 100 μL dalle diluizioni 1 x 10-2 e 1 x 10-3 corrisponde a un fattore di diluizione finale rispettivamente di 1 x 10-3 e 1 x 10-4.
6. Saggio di replicazione intracellulare
NOTA: Tutti i volumi sono coerenti con un test che utilizza due piastre a 6 pozzetti.
- Sottocoltura durante la notte delle colture Shigella tramite diluizione 1:50 in terreni freschi.
- Vortex, quindi aggiungere 100 μL di ciascuna coltura notturna a 5 mL di TSB fresco o TSB + BS in una provetta di coltura di dimensioni adeguate.
NOTA: Limitare il volume di coltura al <20% del volume del pallone di coltura o della provetta per garantire una corretta aerazione. - Incubare a 37 °C agitando a 250 rpm fino a quando le cellule raggiungono un OD600 di 0,7 (fase intermedia della crescita della Shigella ); circa 2-2,5 h.
NOTA: Durante la sottocoltura, aliquotare 50 mL di DMEM + 50 mg/mL di gentamicina e un volume sufficiente di PBS per tutte le fasi di lavaggio e porre a bagnomaria a 37 °C. Lasciare che i supporti raggiungano i 37 °C prima dell'uso.
- Vortex, quindi aggiungere 100 μL di ciascuna coltura notturna a 5 mL di TSB fresco o TSB + BS in una provetta di coltura di dimensioni adeguate.
- Trasferire 2 x 10 Shigella sottocoltivata da8 CFU in provette individuali da microcentrifuga da 2 mL.
NOTA: 2 x 108 CFU corrispondono a circa 1 mL di cellule batteriche ad un OD600 di 0,7. Utilizzare le letture OD600 per approssimare CFU/mL in base alla calibrazione di ogni singolo spettrofotometro. - Lavare i campioni di Shigella 1 volta con PBS.
- Celle a pellet per centrifugazione a 17.000 x g per 2 minuti a temperatura ambiente. Aspirare il surnatante, quindi aggiungere 1 mL di PBS caldo e risospendere bene il pellet, pipettando delicatamente il campione su e giù fino a quando la miscela non è completamente omogenea (8-10x).
- Ripetere il passaggio 6.3.1. 1x tempo supplementare.
- Celle di pellet mediante centrifugazione a 17.000 x g per 2 minuti a temperatura ambiente, aspirare il surnatante e risospendere i pellet in 2 mL di DMEM caldo.
NOTA: La concentrazione finale di batteri risospesi sarà di 1 x 108 CFU/mL.
- Vortex, quindi aggiungere 1 mL (1 x 108 CFU) di Shigella risospesa più 1 mL di DMEM a ciascun pozzetto di monostrati epiteliali del colon HT-29 preparati in piastre a 6 pozzetti (dal passaggio 3.2.10.2).
NOTA: Le infezioni vengono normalmente eseguite con una molteplicità di infezioni (MOI; rapporto tra cellule batteriche ed epiteliali) di 100. Per testare diversi MOI, diluire la Shigella in sospensione in DMEM alla concentrazione desiderata, quindi aggiungere 1 mL di batteri diluiti ai monostrati HT-29. Ad esempio, per testare un MOI di 10, diluire i batteri 1:10 aggiungendo 150 μL di 1 x 10 batterida 8 CFU/mL a 1,35 mL di DMEM, quindi applicare 1 mL (1 x 107 CFU) alle cellule HT-29. - Per favorire il contatto batterico con le cellule HT-29, centrifugare le piastre a 6 pozzetti a 2.000 x g per 10 minuti a temperatura ambiente o 37 °C se è possibile regolare l'impostazione della temperatura.
NOTA: La centrifugazione favorisce il contatto batterico con le cellule HT-29, che bypassa la necessità di fattori di aderenza e consente ai batteri di invadere rapidamente le cellule. - Incubare piastre a 6 pozzetti a 37 °C con il 5% di CO2 per 45 min.
- Durante l'incubazione, determinare il titolo dell'infezione batterica.
- Preparare diluizioni seriali di 10 volte di cellule Shigella risospese (dal passaggio 6.3.3) in PBS.
- Fissare 100 μL delle diluizioni 1 x 10-5 e 1 x 10-6 su piastre rosse TSB + Congo e incubare per una notte a 37 °C.
NOTA: La placcatura di 100 μL dalle diluizioni 1 x 10-5 e 1 x 10-6 corrisponde a un fattore di diluizione finale rispettivamente di 1 x 10-6 e 1 x 10-7.
- Lavare accuratamente le cellule HT-29 infette 3 volte con 1 ml di PBS.
- Aspirare il terreno da ciascun pozzetto.
NOTA: Quando si aspirano i fluidi da piastre a 6 pozzetti, guidare la punta dell'aspiratore lungo il lato inferiore dei pozzetti, cercando di evitare il contatto con le celle HT-29. - Aggiungere 1 ml di PBS caldo in ogni pozzetto e lavare delicatamente.
NOTA: Per lavare delicatamente i monostrati a 6 pozzetti con PBS, spostare la piastra su e giù e da un lato all'altro sul piano di lavoro. Il lavaggio delle piastre con un movimento circolare e/o la rimozione della piastra dalla superficie del piano di lavoro può causare la rimozione meccanica delle cellule dalla plastica. - Ripetere i passaggi 6.8.1 e 6.8.2 altre 2 volte.
- Aspirare il terreno da ciascun pozzetto.
- Rimuovere il PBS mediante aspirazione, quindi aggiungere 2 mL di DMEM caldo integrato con 50 μg/mL di gentamicina in ciascun pozzetto e incubare per 30 minuti a 37 °C con il 5% di CO2 .
- Lavare accuratamente le cellule HT-29 infette 3 volte con 1 ml di PBS.
- Ripetere il passaggio di lavaggio 6.8.
- Rimuovere il PBS mediante aspirazione, quindi aggiungere 2 mL di DMEM caldo con 50 μg/mL di gentamicina a ciascun pozzetto delle piastre a 6 pozzetti e incubare a 37 °C con il 5% di CO2 per il periodo di tempo desiderato per consentire la replicazione intracellulare (fino a 24 ore).
- Lavare accuratamente le cellule 2 volte con 1 mL di PBS.
- Ripetere il passaggio di lavaggio 6.8.
- Rimuovere il PBS mediante aspirazione e lisare le cellule HT-29 aggiungendo 1 mL di PBS + 1% di Triton X-100 a ciascun pozzetto.
- Incubare le piastre a 6 pozzetti a 37 °C per 5 min.
- Utilizzare un raschietto per cellule o un puntale per pipetta piegato per raschiare le cellule lisate dal fondo del pozzetto e trasferire l'intero 1 mL in una nuova provetta per microcentrifuga da 1,7 mL.
- Determinare il numero di batteri intracellulari.
- Vorticare ogni tubo (dal punto 6.15) per almeno 30 secondi per spostare ulteriormente la Shigella dalle cellule eucariotiche lisate.
- Preparare diluizioni seriali di 10 volte di lisati in PBS.
- Piastre da 100 μL di 1 x 10-2, 1 x 10-3 e 1 x 10-4 diluizioni su piastre TSB + Congo Red e incubare per una notte a 37 °C.
NOTA: La placcatura di 100 μL dalle diluizioni 1 x 10-2, 1 x 10-3 e 1 x 10-4 corrisponde a un fattore di diluizione finale rispettivamente di 1 x 10-3, 1 x 10-4 e 1 x 10-5.
Risultati
Sono stati eseguiti saggi di aderenza, invasione e replicazione intracellulare confrontando S. flexneri 2457T wild type (WT) con S. flexneri ΔVF (ΔVF), un mutante che si ipotizza regoli negativamente la virulenza della Shigella. Poiché la Shigella utilizza i sali biliari come segnale per regolare la virulenza 17,18,47, sono stati eseguiti esperimenti dopo la subcoltura batterica in terreni TSB e TSB integrato con lo 0,4% (p/v) di sali
Discussione
Questo protocollo descrive una serie di tre saggi standardizzati per studiare l'aderenza, l'invasione e la replicazione intracellulare delle cellule epiteliali intestinali della Shigella. Sebbene questi metodi siano semplicemente versioni modificate dei classici saggi di gentamicina utilizzati per studiare l'invasione e la replicazione intracellulare di vari patogeni batterici all'interno delle cellule ospiti 49,50,51, è necessario applicare considerazioni speciali quando si <...
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Il supporto per gli autori include il Dipartimento di Pediatria del Massachusetts General Hospital, il premio 2022A009041 del Comitato esecutivo per il finanziamento del supporto provvisorio alla ricerca, il R21AI146405 di sovvenzione del National Institute of Allergy and Infectious Diseases e il National Institute of Diabetes and Digestive and Kidney Diseases grant Nutrition Obesity Research Center di Harvard (NORCH) 2P30DK040561-26. I finanziatori non hanno avuto alcun ruolo nella progettazione dello studio, nella raccolta e nell'analisi dei dati, nella decisione di pubblicare o nella preparazione del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 μm PES filter | Millipore-Sigma | SCGP00525 | Sterile, polyethersulfone filter for sterilizing up to 50 mL media |
| 14 mL culture tubes | Corning | 352059 | 17 mm x 100 mm polypropylene test tubes with cap |
| 50 mL conical tubes | Corning | 430829 | 50 mL clear polypropylene conical bottom centrifuge tubes with leak-proof cap |
| 6-well tissue culture plates | Corning | 3516 | Plates are treated for optimal cell attachment |
| Bile salts | Sigma-Aldrich | B8756 | 1:1 ratio of cholate to deoxycholate |
| Congo red dye | Sigma-Aldrich | C6277 | A benzidine-based anionic diazo dye, >85% purity |
| Countess cell counting chamber slide | Invitrogen | C10283 | To be used with the Countess Automated Cell Counter |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | A a highly polar organic reagent |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 10569-010 | DMEM is supplemented with high glucose, sodium pyruvate, GlutaMAX, and Phenol Red |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F4135 | Heat-inactivated, sterile |
| Gentamicin | Sigma-Aldrich | G3632 | Stock concentration is 50 mg/mL |
| HT-29 cell line | ATCC | HTB-38 | Adenocarcinoma cell line; colorectal in origin |
| Paraffin film | Bemis | PM999 | Laboratory sealing film |
| Petri dishes | Thermo Fisher Scientific | FB0875713 | 100 mm x 15 mm Petri dishes for solid media |
| Phosphate-buffered saline (PBS) | Thermo Fisher Scientific | 10010049 | 1x concentration; pH 7.4 |
| Select agar | Invitrogen | 30391023 | A mixture of polysaccharides extracted from red seaweed cell walls to make bacterial plating media |
| T75 flasks | Corning | 430641U | Tissue culture flasks |
| Triton X-100 | Sigma-Aldrich | T8787 | A common non-ionic surfactant and emulsifier |
| Trypan blue stain | Invitrogen | T10282 | A dye to detect dead tissue culture cells; only live cells can exclude the dye |
| Trypsin-EDTA | Gibco | 25200-056 | Reagent for cell dissociation for cell line maintenance and passaging |
| Tryptic Soy Broth (TSB) | Sigma-Aldrich | T8907 | Bacterial growth media |
Riferimenti
- Karambizi, N. U., McMahan, C. S., Blue, C. N., Temesvari, L. A. Global estimated Disability-Adjusted Life-Years (DALYs) of diarrheal diseases: A systematic analysis of data from 28 years of the global burden of disease study. PloS one. 16 (10), e0259077 (2021).
- WHO. WHO methods and data sources for country-level causes of death 2000-2016. World Health Organization. , (2018).
- Kotloff, K. L. Shigella infection in children and adults: a formidable foe. Lancet Glob Health. 5 (12), e1166-e1167 (2017).
- Kotloff, K. L., et al. Burden and aetiology of diarrhoeal disease in infants and young children in developing countries (the Global Enteric Multicenter Study, GEMS): A prospective, case-control study. Lancet. 382 (9888), 209-222 (2013).
- Schroeder, G. N., Hilbi, H. Molecular pathogenesis of Shigella spp.: Controlling host cell signaling, invasion, and death by type III secretion. Clin Microbiol Rev. 21 (1), 134-156 (2008).
- Arvelo, W., et al. Transmission risk factors and treatment of pediatric shigellosis during a large daycare center-associated outbreak of multidrug resistant shigella sonnei: Implications for the management of shigellosis outbreaks among children. Pediatr Infect Dis J. 28 (11), 976-980 (2009).
- Kozyreva, V. K., et al. Recent outbreaks of Shigellosis in California caused by two distinct populations of Shigella sonnei with either increased virulence or fluoroquinolone resistance. mSphere. 1 (6), 1-18 (2016).
- Bowen, A., et al. Importation and domestic transmission of Shigella sonnei resistant to ciprofloxacin - United States, May 2014-February 2015. MMWR Morb Mortal Wkly Rep. 64 (12), 318-320 (2015).
- Tansarli, G. S., et al. Genomic reconstruction and directed interventions in a multidrug-resistant Shigellosis outbreak in Seattle, WA, USA: a genomic surveillance study. Lancet. 3099 (22), 1-11 (2023).
- Barry, E. M., et al. Progress and pitfalls in Shigella vaccine research. Nat Rev Gastroenterol Hepatol. 10 (4), 245-255 (2013).
- Increase in Extensively Drug-Resistant Shigellosis in the United States. CDC Health Alert Network. Centers for Disease Control and Prevention Available from: https://emergency.cdc.gov/han/2023/han00486.asp?ACSTrackingID=USCDC_511-DM100260&ACSTrackingLabel=HAN%20486%20-%20General%20Public&deliveryName=USCDC_511-DM100260 (2023)
- Shiferaw, B., et al. Antimicrobial susceptibility patterns of Shigella isolates in Foodborne Diseases Active Surveillance Network (FoodNet) sites, 2000-2010. Clin Infect Dis. 54, S458-S463 (2012).
- Centers for Disease Control and Prevention. COVID-19: U.S. Impact on Antimicrobial Resistance, Special Report 2022. Atlanta, GA: U.S. Department of Health and Human Services. CDC. , (2022).
- Centers for Disease Control and Prevention. Antibiotic resistance threats in the United States, 2019. CDC. 10 (1), (2019).
- WHO. Prioritization of pathogens to guide discovery, research and development of new antibiotics for drug-resistant bacterial infections, including tuberculosis. WHO. , (2017).
- DuPont, H. L., Levine, M. M., Hornick, R. B., Formal, S. B. Inoculum size in shigellosis and implications for expected mode of transmission. J Infect Dis. 159 (6), 1126-1128 (1989).
- Nickerson, K. P., et al. Analysis of Shigella flexneri resistance, biofilm formation, and transcriptional profile in response to bile salts. Infect Immun. 85 (6), 1-18 (2017).
- Faherty, C. S., Redman, J. C., Rasko, D. A. Shigella flexneri effectors OspE1 and OspE2 mediate induced adherence to the colonic epithelium following bile salts exposure. Mol Microbiol. 85 (1), 107-121 (2012).
- Chanin, R. B., et al. Shigella flexneri adherence factor expression in in vivo-like conditions. mSphere. 4 (6), e00751 (2019).
- Baranov, V., Hammarström, S. Carcinoembryonic antigen (CEA) and CEA-related cell adhesion molecule 1 (CEACAM1), apically expressed on human colonic M cells, are potential receptors for microbial adhesion. Histochem Cell Biol. 121 (2), 83-89 (2004).
- Wassef, J. S., Keren, D. F., Mailloux, J. L. Role of M cells in initial antigen uptake and in ulcer formation in the rabbit intestinal loop model of shigellosis. Infect Immun. 57 (3), 858-863 (1989).
- Sansonetti, P. J., Arondel, J., Cantey, J. R., Prévost, M. C., Huerre, M. Infection of rabbit Peyer's patches by Shigella flexneri: Effect of adhesive or invasive bacterial phenotypes on follicle-associated epithelium. Infect Immun. 64 (7), 2752-2764 (1996).
- Sansonetti, P. J., et al. Caspase-1 activation of IL-1beta and IL-18 are essential for Shigella flexneri-induced inflammation. Immunity. 12 (5), 581-590 (2000).
- Zychlinsky, A., Fitting, C., Cavaillon, J. M., Sansonetti, P. J. Interleukin 1 is released by murine macrophages during apoptosis induced by Shigella flexneri. J Clin Invest. 94 (3), 1328-1332 (1994).
- Sansonetti, P. J., Ryter, A., Clerc, P., Maurelli, A. T., Mounier, J. Multiplication of Shigella flexneri within HeLa cells: lysis of the phagocytic vacuole and plasmid-mediated contact hemolysis. Infect Immun. 51 (2), 461-469 (1986).
- Maldonado-Contreras, A., et al. Shigella depends on SepA to destabilize the intestinal epithelial integrity via cofilin activation. Gut Microbes. 8 (6), 544-560 (2017).
- Collard, J. -. M., et al. High prevalence of small intestine bacteria overgrowth and asymptomatic carriage of enteric pathogens in stunted children in Antananarivo, Madagascar. PLoS Negl Trop Dis. 16 (5), e0009849 (2022).
- Mattock, E., Blocker, A. J. How do the virulence factors of shigella work together to cause disease. Front Cell Infect Microbiol. 7, 1-24 (2017).
- Mostowy, S., et al. The zebrafish as a new model for the in vivo study of Shigella flexneri interaction with phagocytes and bacterial autophagy. PLoS Pathog. 9 (9), e1003588 (2013).
- Martinez-Becerra, F. J., et al. Parenteral immunization with IpaB/IpaD protects mice against lethal pulmonary infection by Shigella. Vaccine. 31 (24), 2667-2672 (2013).
- Shim, D. -. H., et al. New animal model of shigellosis in the Guinea pig: its usefulness for protective efficacy studies. J Immunol. 178 (4), 2476-2482 (2007).
- Marteyn, B., et al. Modulation of Shigella virulence in response to available oxygen in vivo. Nature. 465 (7296), 355-358 (2010).
- West, N. P., et al. Optimization of virulence functions through glucosylation of Shigella LPS. Science. 307 (5713), 1313-1317 (2005).
- Maurelli, A. T., et al. Shigella infection as observed in the experimentally inoculated domestic pig, Sus scrofa domestica. Microbial Pathog. 25 (4), 189-196 (1998).
- Jeong, K. -. I., Zhang, Q., Nunnari, J., Tzipori, S. A piglet model of acute gastroenteritis induced by Shigella dysenteriae Type 1. J Infect Dis. 201 (6), 903-911 (2010).
- Kim, Y. -. J., Yeo, S. -. G., Park, J. -. H., Ko, H. -. J. Shigella vaccine development: prospective animal models and current status. Curr Pharm Biotechnol. 14 (10), 903-912 (2013).
- Kent, T. H., Formal, S. B., LaBrec, E. H., Sprinz, H., Maenza, R. M. Gastric shigellosis in rhesus monkeys. Am J Pathol. 51 (2), 259-267 (1967).
- Shipley, S. T., et al. A challenge model for Shigella dysenteriae 1 in cynomolgus monkeys (Macaca fascicularis). Comp Med. 60 (1), 54-61 (2010).
- Higgins, R., Sauvageau, R., Bonin, P. Shigella flexneri Type 2 Infection in captive nonhuman primates. Can Vet J. 26 (12), 402-403 (1985).
- Oaks, E. V., Hale, T. L., Formal, S. B. Serum immune response to Shigella protein antigens in rhesus monkeys and humans infected with Shigella spp. Infect Immun. 53 (1), 57-63 (1986).
- Formal, S. B., et al. Protection of monkeys against experimental shigellosis with a living attenuated oral polyvalent dysentery vaccine. J Bacteriol. 92 (1), 17-22 (1966).
- Levine, M. M., Kotloff, K. L., Barry, E. M., Pasetti, M. F., Sztein, M. B. Clinical trials of Shigella vaccines: two steps forward and one step back on a long, hard road. Nat Rev Microbiol. 5 (7), 540-553 (2007).
- Payne, S. M. Laboratory cultivation and storage of Shigella. Curr Protoc Microbiol. 55 (1), 93 (2019).
- NIH Guidelines. NIH guidelines for research involving recombinant or synthetic nucleic acid molecules. NIH Guidelines. 2, 142 (2019).
- Maurelli, A. T., Blackmon, B., Curtiss, R. Loss of pigmentation in Shigella flexneri 2a is correlated with loss of virulence and virulence-associated plasmid. Infect Immun. 43 (1), 397-401 (1984).
- HT-29 cell line product sheet. ATCC Available from: https://www.atcc.org/products/htb-38 (2023)
- Sistrunk, J. R., Nickerson, K. P., Chanin, R. B., Rasko, D. A., Faherty, C. S. Survival of the fittest: How bacterial pathogens utilize bile to enhance infection. Clin Microbiol Rev. 29 (4), 819-836 (2016).
- Stensrud, K. F., et al. Deoxycholate interacts with IpaD of Shigella flexneri in inducing the recruitment of IpaB to the type III secretion apparatus needle tip. J Biol Chem. 283 (27), 18646-18654 (2008).
- Mandell, G. L. Interaction of intraleukocytic bacteria and antibiotics. J Clin Invest. 52 (7), 1673-1679 (1973).
- Elsinghorst, E. A. Measurement of invasion by gentamicin resistance. Methods Enzymo. 236 (1979), 405-420 (1994).
- Elsinghorst, E. A., Weitz, J. A. Epithelial cell invasion and adherence directed by the enterotoxigenic Escherichia coli tib locus is associated with a 104-kilodalton outer membrane protein. Infect Immun. 62 (8), 3463-3471 (1994).
- Dorman, C. J., McKenna, S., Beloin, C. Regulation of virulence gene expression in Shigella flexneri, a facultative intracellular pathogen. Int J Med Microbiol. 291 (2), 89-96 (2001).
- Porter, M. E., Dorman, C. J. Positive regulation of Shigella flexneri virulence genes by integration host factor. J Bacteriol. 179 (21), 6537-6550 (1997).
- Maurelli, A. T., Blackmon, B., Curtiss, R. Temperature-dependent expression of virulence genes in Shigella species. Infect Immun. 43 (1), 195-201 (1984).
- Schuch, R., Maurelli, A. T. Virulence plasmid instability in Shigella flexneri 2a is induced by virulence gene expression. Infect Immun. 65 (9), 3686-3692 (1997).
- Formal, S. B., Hale, T. L., Sansonetti, P. J. Invasive enteric pathogens. Rev Infect Dis. 5, S702-S707 (1983).
- Pál, T., Hale, T. L. Plasmid-associated adherence of Shigella flexneri in a HeLa cell model. Infect Immun. 57 (8), 2580-2582 (1989).
- Noben, M., et al. Human intestinal epithelium in a dish: Current models for research into gastrointestinal pathophysiology. United European Gastroenterol J. 5 (8), 1073-1081 (2017).
- Liévin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: lessons from cultured, fully differentiated human colon cancer cell lines. Microbiol Mol Biol Rev R. 77 (3), 380-439 (2013).
- Mitchell, D. M., Ball, J. M. Characterization of a spontaneously polarizing HT-29 cell line, HT-29/cl.f8. In Vitro Cell Dev Biol - Anim. 40 (10), 297-302 (2004).
- Gagnon, M., Zihler Berner, A., Chervet, N., Chassard, C., Lacroix, C. Comparison of the Caco-2, HT-29 and the mucus-secreting HT29-MTX intestinal cell models to investigate Salmonella adhesion and invasion. J Microbiol Methods. 94 (3), 274-279 (2013).
- Koestler, B. J., et al. Human intestinal enteroids as a model system of Shigella pathogenesis. Infect Immun. 87 (4), 00733 (2019).
- Ranganathan, S., et al. Evaluating Shigella flexneri pathogenesis in the human enteroid model. Infect Immun. 87 (4), (2019).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiol Spectr. 9 (1), 1-17 (2021).
- Perlman, M., Senger, S., Verma, S., Carey, J., Faherty, C. S. A foundational approach to culture and analyze malnourished organoids. Gut Microbes. 15 (2), 2248713 (2023).
- Pope, L. M., Reed, K. E., Payne, S. M. Increased protein secretion and adherence to HeLa cells by Shigella spp. following growth in the presence of bile salts. Infect Immun. 63 (9), 3642-3648 (1995).
- Faherty, C. S., et al. The synthesis of OspD3 (ShET2) in Shigella flexneri is independent of OspC1. Gut Microbes. 7 (6), 486-502 (2016).
- Ridlon, J. M., Kang, D. -. J., Hylemon, P. B. Bile salt biotransformations by human intestinal bacteria. J Lipid Res. 47 (2), 241-259 (2006).
- Köseoğlu, V. K., Hall, C. P., Rodríguez-López, E. M., Agaisse, H. The Autotransporter IcsA promotes Shigella flexneri biofilm formation in the presence of bile salts. Infect Immun. 87 (7), 1-14 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon