Method Article
Ingegneria delle proteine mediante display superficiale del lievito
In questo articolo
Riepilogo
Questo protocollo descrive i passaggi essenziali per condurre campagne di selezione della superficie del lievito per arricchire le varianti proteiche che si legano a un antigene di interesse.
Abstract
L'ingegneria proteica consente il miglioramento delle funzioni esistenti di una data proteina o la generazione di nuove funzioni. Uno degli strumenti più utilizzati e versatili nel campo dell'ingegneria proteica è il display della superficie del lievito, in cui un pool di proteine randomizzate viene espresso sulla superficie del lievito. Il legame tra fenotipo (ad esempio, legame della proteina mostrata con il lievito all'antigene di interesse) e genotipo (il plasmide che codifica per la variante proteica) consente la selezione di questa libreria per le proprietà desiderate e il successivo sequenziamento di varianti arricchite. Combinando la selezione delle biglie magnetiche con la selezione citofluorimetrica, è possibile selezionare e arricchire varianti proteiche con un maggiore legame a un antigene bersaglio. In particolare, oltre alla maturazione dell'affinità, il legame a un bersaglio può essere raggiunto anche senza alcuna affinità di legame iniziale. Qui, forniamo un protocollo passo-passo che copre tutte le parti essenziali di una campagna di selezione della visualizzazione della superficie del lievito e fornisce esempi di risultati tipici della visualizzazione della superficie del lievito. Dimostriamo che la visualizzazione della superficie del lievito è un metodo robusto e ampiamente applicabile che può essere stabilito in qualsiasi laboratorio di biologia molecolare con accesso alla citometria a flusso.
Introduzione
La visualizzazione della superficie del lievito è una delle tecnologie chiave nel campo dell'ingegneria proteica. Consente la selezione di varianti proteiche con le proprietà desiderate, come una migliore affinità o stabilità. Introdotta per la prima volta nel 19971, è una delle tecnologie di visualizzazione più comunemente utilizzate oltre al phage display 2,3, al ribosomidisplay 4 e al display delle cellule di mammifero 5,6,7. La proteina di interesse (POI) viene visualizzata sulla superficie delle cellule di lievito fondendola per ancorare le proteine. È disponibile una gamma di diverse proteine di ancoraggio e, più comunemente, il POI è fuso con il C-terminale della proteina di accoppiamento dell'agglutinina di lievito Aga2p 1,8. Inoltre, il POI è tipicamente affiancato da due tag, come un tag per emoagglutinina (HA-tag) e un tag c-myc, che consente il rilevamento del livello di visualizzazione utilizzando anticorpi marcati in fluorescenza e citometria a flusso (Figura 1A). Le tipiche campagne di selezione dei lieviti prevedono una combinazione di selezioni di biglie magnetiche e selezione citofluorimetrica. Le selezioni delle microsfere consentono la gestione di un numero elevato di cellule e l'arricchimento di varianti proteiche che si legano all'antigene bersaglio anche con basse affinità poiché le interazioni multivalenti con le perle caricate con l'antigene portano a effetti di avidità e, quindi, prevengono la perdita di varianti a bassa affinità (Figura 1B). L'analisi citofluorimetrica e la selezione offrono il vantaggio di visualizzare il legame delle varianti di POI visualizzate all'antigene marcato. Di conseguenza, le popolazioni leganti possono essere ordinate e coltivate, portando all'arricchimento di varianti proteiche con le caratteristiche desiderate durante diversi cicli di selezione. Inoltre, è possibile eseguire ulteriori cicli di mutagenesi casuale per aumentare ulteriormente la diversità e, quindi, la probabilità di trovare ulteriori mutazioni che contribuiscono all'affinità e/o alla stabilità della proteina.
L'esposizione della superficie del lievito presenta alcuni vantaggi, come (a) il macchinario di espressione eucariotico, che consente il ripiegamento ossidativo delle proteine e le modificazioni post-traduzionali eucariotiche (come la N-glicosilazione), (b) la normalizzazione dell'espressione dovuta al rilevamento dei due tag peptidici che fiancheggiano la proteina, (c) l'ispezione visiva dell'andamento della selezione mediante citometria a flusso (ad esempio, percentuale di cellule leganti e intensità di legame) e (d) la possibilità di analizzare singoli mutanti proteici su lievito (ad esempio, analizzando la termostabilità e l'affinità), presentando un'alternativa che consente di risparmiare tempo alla laboriosa espressione e purificazione delle proteine9. Infatti, sia le affinità (valori KD) che le stabilità (valori T50) delle proteine mostrate sulla superficie del lievito hanno mostrato buone correlazioni con i dati ottenuti utilizzando metodi biofisici e proteine solubili 9,10,11,12. Il display superficiale del lievito è stato impiegato per l'ingegnerizzazione di una varietà di proteine, ad esempio frammenti di anticorpi 13,14,15,16, il 10° dominio di fibronectina di tipo III 17,18, rcSso7d19,20 o noctine21. Allo stesso modo, sono state intraprese ricerche approfondite per ottimizzare i disegni delle librerie di lievito modificando le posizioni randomizzate e l'utilizzo del codone degli amminoacidi 17,22,23. La visualizzazione della superficie del lievito si è dimostrata efficace per l'ingegneria della stabilità 14,15,24,25, dell'affinità 18,26,27, dell'attività enzimatica 28,29,30,31 e dell'espressione proteica32. Inoltre, applicazioni più sofisticate come il legame condizionale in presenza o assenza di una piccola molecola sono state realizzate utilizzando il display di superficie del lievito20.
In questo protocollo, descriviamo tutti i passaggi essenziali per una campagna di selezione con display della superficie del lievito con l'esempio della libreria G4 (basata sul 10° dominio della fibronectina di tipo III, Fn3) selezionata contro l'antigene umano retinol-binding protein 4 (hRBP4) in presenza della piccola molecola A112020. Questa selezione è stata condotta per produrre un'interazione proteina-proteina che dipende da una piccola molecola che può essere utilizzata come interruttore molecolare. Da notare che, mentre sono possibili approcci alternativi con la visualizzazione della superficie del lievito, le tipiche selezioni di lievito di solito mirano a legarsi a un antigene bersaglio senza alcuna precedente affinità di legame. Copriamo tutte le fasi di una campagna di selezione del lievito, che prevede la coltivazione di una libreria di lieviti, la selezione delle sfere, la selezione citofluorimetrica e la maturazione per affinità mediante PCR soggetta a errori (epPCR). Pertanto, questo protocollo integra i precedenti protocolli di visualizzazione della superficie del lievito33,34 e può essere utilizzato come base per le selezioni di visualizzazione della superficie del lievito (Figura 1) con qualsiasi libreria di lievito e antigene target di scelta.
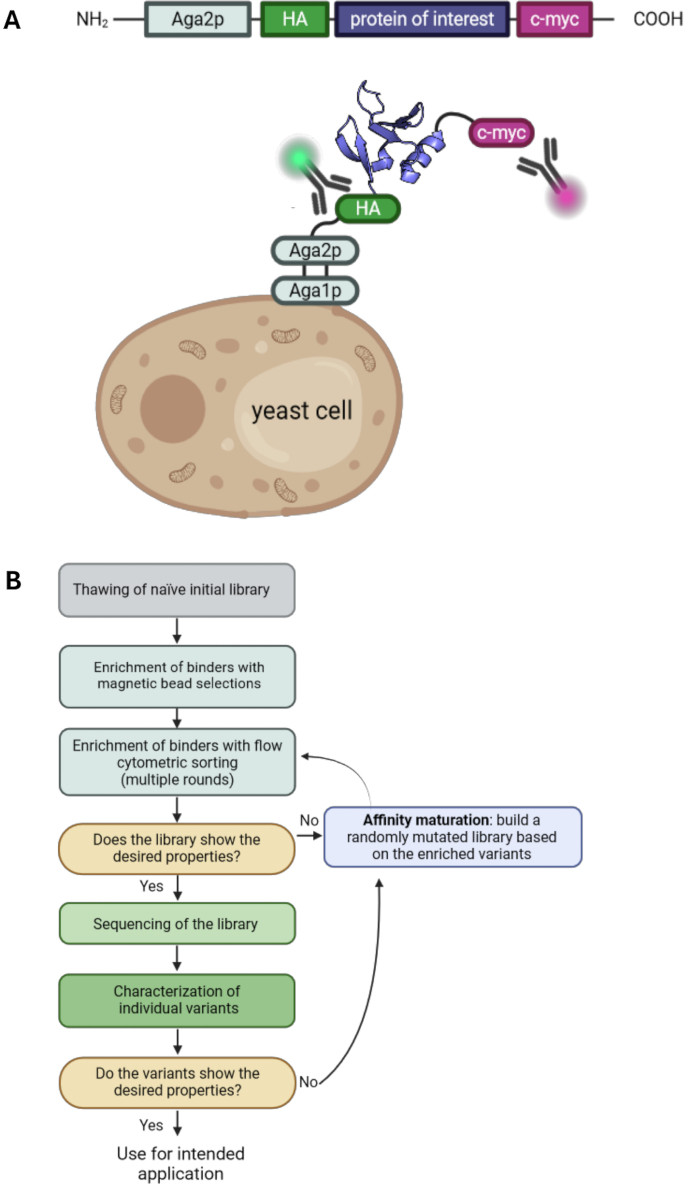
Figura 1: Principio della visualizzazione della superficie del lievito e un flusso di lavoro tipico per le selezioni della visualizzazione della superficie del lievito. (A) Il POI viene clonato in un vettore di visualizzazione della superficie del lievito e tipicamente affiancato da un HA N-terminale e da un c-myc-tag C-terminale. Il costrutto è fuso con la proteina di accoppiamento del lievito Aga2p per essere visualizzato sulla superficie. La proteina raffigurata è il legante ingegnerizzato "RS3" da PDB ID: 6QBA20. (B) Diagramma di flusso che illustra un flusso di lavoro tipico per le campagne di selezione della visualizzazione della superficie del lievito, che combinano l'arricchimento delle varianti proteiche con le proprietà desiderate mediante selezioni di perle e selezione citometrica a flusso, nonché epPCR per la maturazione per affinità. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Scongelamento e coltivazione di librerie di lieviti
- Scongelare una quantità sufficiente di aliquote della libreria di lieviti congelati a RT e diluire immediatamente in SD-CAA (vedi Tabella 1) fino a un OD600 di circa 1 (che corrisponde a 1 x 107 cellule/mL).
NOTA: Assicurati di coprire la diversità della libreria di almeno 10 volte. Pertanto, se la dimensione della libreria rappresenta 2,5 x 108 cloni (questa è la diversità della libreria G4 utilizzata in questo esempio), inoculare un minimo di 2,5 x 109 cellule in 250 mL di SD-CAA per raggiungere un OD600 di 1. Questo protocollo può essere avviato anche da colture liquide (cellule di lievito appena trasformate con una libreria). - Valutare il numero di cellule vitali.
- Prelevare un'aliquota di 100 μl da questa sospensione di lievito (con diametro esterno600 di 1) ed eseguire una serie di diluizioni 1:10 con 900 μl di terreno SD-CAA in provette per microcentrifuga.
- Preparare diluizioni fino a 10-5 e piastre 100 μL di diluizioni 10-3, 10-4 e 10-5 su piastre SD-CAA (Tabella 1). Incubare le piastre a 30 °C per 2-3 giorni. Assicurarsi che il numero di celle vitali dopo lo scongelamento superi la diversità della libreria di almeno 10 volte.
- Nel frattempo, continuare con la sospensione cellulare rimanente e coltivare la coltura di lievito contenente la libreria G4 in fiaschi di vetro per una notte a 30 °C agitando (180 giri/min). Dopo l'incubazione notturna, continuare con la sezione 2 (Induzione dell'espressione proteica sulla superficie del lievito).
- Dopo 2-3 giorni di incubazione, contare le colonie sulle piastre SD-CAA mediante ispezione visiva. Quando si calcola il numero finale di cellule vitali dopo lo scongelamento, considerare il fattore di diluizione e il rapporto tra la piastra (cioè 100 μL) e il volume di coltura originale (cioè 250 mL).
Cellule vitali/mL = numero di colonie su piastra × fattore di diluizione
Cellule vitali (totale) = (cellule vitali/mL) × volume di coltura
| Fluido/buffer | Componente | Concentrazione [g/L] | Commenti/Descrizione | |||
| SD-CAA | D-glucosio | 20 | Sciogliere tutti i componenti del terreno in 1000 mL di ddH2O e filtrare sterile con filtri sterili monouso da 0,22 μm. | |||
| Lievito a base di azoto | 6.7 | |||||
| Casminoacidi | 5 | |||||
| Acido citrico monoidrato | 7.4 | |||||
| Citrato trisodico diidrato | 10.83 | |||||
| SG-CAA | D-galattosio | 20 | Sciogliere tutti i componenti del terreno in 1000 mL di ddH2O e filtrare sterile con filtri sterili monouso da 0,22 μm. | |||
| D-glucosio | 2 | |||||
| Lievito a base di azoto | 6.7 | |||||
| Casamino acidi | 5 | |||||
| fosfato acido di sodio eptaidrato | 10.2 | |||||
| Sodio diidrogeno fosfato monoidrato | 8.56 | |||||
| Piastre SD-CAA | Sorbitolo | 182 | Sciogliere sorbitolo, fosfato acido disodico eptaidrato, sodio fosfato monobasico e agar-agar in 900 mL ddH2O e autoclave. Sciogliere e filtrare in modo sterile i componenti rimanenti in 100 mL di ddH2O e aggiungere quando il terreno autoclavato è tiepido. | |||
| fosfato acido di sodio eptaidrato | 10.2 | |||||
| Sodio diidrogeno fosfato monoidrato | 7.44 | |||||
| Agar-agar | 15 | |||||
| D-glucosio | 20 | |||||
| Lievito a base di azoto | 6.7 | |||||
| Casamino acidi | 5 | |||||
| YPD | Peptone | 20 | Preparare un brodo di D-glucosio 10x (200 g/L) e un filtrato sterile con filtri sterili monouso da 0,22 μm. Sciogliere il peptone e l'estratto di lievito in 900 mL ddH2O e in autoclave. Quando è tiepido, aggiungere 100 ml di D-glucosio. | |||
| Estratto di lievito | 10 | |||||
| D-glucosio | 20 | |||||
| Piastre YPD | Peptone | 20 | Preparare un brodo di D-glucosio 10x (200 g/L) e un filtrato sterile con filtri sterili monouso da 0,22 μm. Sciogliere il peptone, l'estratto di lievito e l'agar-agar in 900 mL ddH2O e in autoclave. Quando è tiepido, aggiungere 100 ml di D-glucosio. | |||
| Estratto di lievito | 10 | |||||
| D-glucosio | 20 | |||||
| Agar-agar | 15 | |||||
| PBSA | BSA | 1 | Sciogliere BSA in PBS e filtrato sterile con filtri sterili monouso da 0,22 μm. | |||
Tabella 1: Composizione dei supporti e dei buffer.
2. Induzione dell'espressione proteica sulla superficie del lievito
- Dopo l'incubazione notturna, le colture di lievito raggiungono in genere un OD600 tra 2-20. Misurare l'OD600 con un fotometro per determinare la densità della coltura di lievito.
NOTA: Le misurazioni OD sono lineari solo nell'intervallo 0,1-1. Pertanto, eseguire diluizioni (in genere 1:20 e 1:50) in SD-CAA per determinare l'OD600.- Diluire le cellule in SD-CAA a un OD600 di 1 e assicurarsi che la diversità sia coperta 10 volte (cioè, 2,5 x 109 celle vengono utilizzate per la diluizione della libreria G4). Continuare l'incubazione a 30 °C agitando per consentire alle cellule di raggiungere una fase di crescita esponenziale.
- Preparare le scorte di glicerolo (che coprono la diversità 50x) delle librerie di lievito (in SD-CAA contenenti il 15% di glicerolo) dalla coltura notturna. A tale scopo, centrifugare il numero richiesto di cellule di lievito (2000 x g, 3 min, 4 °C), scartare il surnatante e risospendere il pellet cellulare in 1 mL di terreno SD-CAA integrato con glicerolo al 15%. Congelare immediatamente le scorte di glicerolo a -80 °C.
NOTA: La vitalità delle cellule di lievito aumenta se le colture di lievito vengono coltivate in una fase stazionaria prima del congelamento. Nei cicli di selezione successivi, quando la diversità delle librerie è inferiore, in genere è sufficiente utilizzare 700 μL di coltura di lievito e mescolarla con 300 μL di SD-CAA integrato con il 50% di glicerolo. Qui, le colture di lievito mostrano tipicamente valori di OD600 di 15 nella fase stazionaria, ma il valore effettivo può dipendere dal fotometro in uso. Pertanto, la fase stazionaria può essere determinata mediante misurazioni ripetute di OD600 . Se il valore OD600 non aumenta durante un periodo di incubazione di 2 ore, si raggiunge una fase stazionaria.
- Centrifugare un volume adeguato di cellule per coprire la diversità 10 volte a 2000 x g per 5 minuti, 4-6 ore dopo la diluizione della coltura di lievito (i valori tipici di OD600 sono compresi tra 4-6) e scartare il surnatante.
- Risospendere il pellet della cella in SG-CAA (vedere la Tabella 1) per ottenere un OD600 di 1 e trasferirlo in un pallone di vetro nuovo.
NOTA: La risospensione in un mezzo contenente galattosio induce l'espressione della proteina sulla superficie da parte del promotore inducibile dal galattosio. - Incubare le colture di lievito per una notte a 20 °C agitando a 180 giri/min per indurre l'espressione superficiale del POI. Dopo l'induzione notturna, le colture di lievito raggiungeranno tipicamente valori di OD600 tra 2-5; se la temperatura di induzione viene aumentata a 37 °C, aspettarsi valori OD600 di 4-10.
NOTA: Riducendo la temperatura a 20°C si rallenta la crescita delle celle e in genere si ottengono livelli di visualizzazione migliori. Tuttavia, l'induzione a 37 °C potrebbe essere utile per aumentare la pressione di selezione verso mutanti stabilizzati35.
3. Primo giro di selezione delle librerie di lieviti (selezione positiva)
NOTA: Una procedura standard di selezione del cordone prevede 6 passaggi (Tabella 2).
| Giorno | Passo | |
| 0 | Cultura notturna | |
| 1 | Induzione dell'espressione proteica sulla superficie delle cellule di lievito | |
| 2 | Prima selezione del cordone con 1 selezione positiva | |
| 3 | Rimozione delle perle, passaggio, induzione dell'espressione proteica sulla superficie delle cellule di lievito e congelamento della libreria | |
| 4 | Selezione del secondo cordone con 3 selezioni negative e 1 positiva | |
| 5 | Rimozione delle perline e congelamento della libreria | |
Tabella 2: Sequenza temporale tipica per la conduzione delle selezioni di perline di una libreria di lieviti.
- Preparazione delle perline
- Preparare le perle (vedi Tabella dei materiali) per la prima selezione delle perline: Per ogni provetta, risospendere 10 μL di perle magnetiche leganti di biotina (4 × 105 perline/μL) in 990 μL di PBSA (Tabella 1) per il lavaggio, posizionare la provetta su una griglia magnetica per 2 minuti con il coperchio aperto. Rimuovere con cautela il surnatante. Ripetere la fase di lavaggio con 1 ml di PBSA.
NOTA: Se sono necessarie più provette di perle con lo stesso antigene biotinilato, possono essere preparate in una provetta in questa fase per ottenere una soluzione uniforme antigene-perlina. In questo caso, la quantità di antigene deve essere aumentata di conseguenza nella fase successiva. - Risospendere le microsfere in un volume totale di 1 mL di PBSA con 6,7-33 pmoli di antigene biotinilato (qui: 6,86 μL di hRBP4 con una concentrazione di 0,125 mg/mL) in una provetta da microcentrifuga da 1,5 mL.
NOTA: Mentre 6,7 pmoli di antigene sono sufficienti, se l'antigene non è limitante, se l'antigene non è limitante, se si dovrebbero usare 33 pmoli. Se la selezione viene eseguita per un antigene legato a un farmaco o ligando a piccola molecola, come in questo caso, questo farmaco/ligando deve essere presente durante la selezione e deve essere aggiunto in questa fase (qui: 5 μM A1120). - Incubare a 4 °C per 2 ore agitando delicatamente o ruotando.
NOTA: Le celle possono essere preparate durante questo passaggio. - Dopo l'incubazione e prima dell'aggiunta alle cellule, posizionare la provetta su una griglia magnetica per 2 minuti con il coperchio aperto. Rimuovere il surnatante e lavare le perle cariche di antigene con 1 mL di PBSA come descritto sopra. Ripetere la fase di lavaggio.
NOTA: Se viene selezionato un antigene legato a un farmaco o a un ligando, il tampone di lavaggio deve contenere questo farmaco o ligando in eccesso (qui: 5 μM A1120). - Risospendere le perle caricate con l'antigene in 50 μL di PBSA (in presenza del farmaco o del ligando, se applicabile).
NOTA: Se le stesse perle caricate con antigene sono state preparate per più provette, regolare il volume di conseguenza (per la libreria G4, il volume finale è di 100 μl di perle di resa per due provette).
- Preparare le perle (vedi Tabella dei materiali) per la prima selezione delle perline: Per ogni provetta, risospendere 10 μL di perle magnetiche leganti di biotina (4 × 105 perline/μL) in 990 μL di PBSA (Tabella 1) per il lavaggio, posizionare la provetta su una griglia magnetica per 2 minuti con il coperchio aperto. Rimuovere con cautela il surnatante. Ripetere la fase di lavaggio con 1 ml di PBSA.
- Preparazione delle cellule di lievito
- Misurare OD600 della coltura di lievito indotta come descritto sopra.
- Pellet celle sufficienti a coprire 10 volte la diversità della libreria (2000 x g, 10 min, 4 °C). Per la libreria G4, utilizzare 2,5 x 109 celle per la centrifugazione. Rimuovere il surnatante e lavare le cellule con 10 ml di PBSA.
- Centrifugare nuovamente (2000 x g, 10 min, 4 °C) e risospendere le cellule a una concentrazione di 1,25 x 109 cellule/mL nel volume appropriato di PBSA (qui: 2 mL) e trasferire in una provetta da microcentrifuga.
NOTA: Il volume di PBSA dipende dal numero di celle. - Centrifugare (2000 x g, 3 min, 4 °C) e risospendere in 950 μL di PBSA.
- Incubazione di cellule di lievito con perle caricate con antigene
- Aggiungere 50 μl di microsfere caricate con antigene a 950 μl di sospensione cellulare e incubare per 2 ore a 4 °C agitando o ruotando.
- Dopo l'incubazione, posizionare la soluzione cellula-antigene-perlina su una griglia magnetica con il coperchio aperto. Pipettare il liquido dal coperchio alla provetta per sottoporre l'intera coltura di lievito alla selezione magnetica. Attendere 2 minuti prima di scartare con cura le celle non legate. Lavare immediatamente con 1 mL di PBSA.
NOTA: Se le selezioni vengono condotte in presenza di un farmaco/ligando - in questo esempio A1120 - questo deve essere presente in PBSA. - Dopo la fase di lavaggio con PBSA, risospendere rapidamente le cellule in 1 mL di SD-CAA per evitare che si secchino.
- Aggiungere 200 mL di SD-CAA ed eseguire le diluizioni seriali come descritto sopra. Piastra da 100 μl su piastre SD-CAA e incubazione per 2-3 giorni a 30 °C per determinare la nuova diversità massima. Qui, la diversità della libreria G4 dopo la prima selezione di perline è di 1,25 x106 celle.
NOTA: Poiché la libreria originale è stata sovracampionata di 10 volte in questo ciclo di selezione, è ragionevole supporre che la maggior parte dei cloni sarà presente più volte nel pool arricchito dopo la selezione. Di conseguenza, la diversità reale sarà inferiore e questo numero dovrebbe essere considerato come un valore massimo. Di solito, la diversità si riduce di almeno 100 volte in questo primo turno di selezione. Incubare la coltura rimanente (200 mL) a 30 °C con agitazione (180 giri/min) per almeno 16 ore. Il protocollo può essere messo in pausa dopo l'incubazione notturna posizionando le cellule a 4 °C.
4. Rimozione delle perle e coltivazione prima del successivo giro di selezione delle perle
- Misurare OD600 come descritto sopra. Pellet celle sufficienti a coprire la diversità di almeno 10x (2000 x g, 3 min, RT). Scartare il surnatante e risospendere le cellule in 1 mL di SD-CAA.
- Trasferire in una provetta da microcentrifuga e posizionare su una griglia magnetica per 2 minuti con il coperchio aperto. Trasferire le cellule non legate in una nuova provetta da microcentrifuga e ripetere il processo di separazione.
- Diluire le cellule non legate in 100 mL di SD-CAA e incubare a 30 °C agitando a 180 giri/min fino a raggiungere un OD600 di 3-4.
NOTA: Se l'OD600 è inferiore a 4 prima della rimozione delle perle, le cellule possono essere indotte subito dopo senza incubazione. - Quando si raggiunge un OD600 di 3-4, centrifugare un numero sufficiente di cellule per coprire la diversità almeno 10x (2000 x g, 3 min, RT) e risospendere in SG-CAA a un OD600 di 1 per l'induzione dell'espressione proteica e incubare a 20 °C agitando a 180 giri/min per una notte.
- Quando la restante coltura di lievito SD-CAA ha raggiunto la fase di crescita stazionaria (OD600 superiore a 15), pellettare cellule sufficienti a coprire la diversità 50x (2000 x g, 3 min, RT). Congelare le cellule mediante centrifugazione (2000 x g, 3 min), seguita da risospensione del pellet cellulare in SD-CAA contenente il 15% di glicerolo e conservazione a -80 °C.
5. Secondo giro di selezione delle perle con 3 selezioni negative e 1 positiva
- Preparare le cellule e le sfere dell'antigene come descritto sopra. Preparare una soluzione di perle nude (senza aggiungere l'antigene) per le selezioni negative.
NOTA: Poiché vengono eseguite 3 selezioni negative, le perline possono essere preparate insieme per tutti e tre i giri. - Dopo il lavaggio, risospendere le perle dell'antigene in 50 μL di PBSA e risospendere le perle nude in 150 μL di PBSA (se sono state preparate per tutte e tre le selezioni negative).
- Per la prima selezione negativa, aggiungere 50 μl di perle nude lavate a 950 μl di cellule lavate in PBSA e incubare per 1,5 ore a 4 °C.
- Dopo l'incubazione, posizionare le provette contenenti le sospensioni di perle nude su una rastrelliera magnetica con il coperchio aperto. Pipettare il liquido nel coperchio nella provetta e attendere 2 minuti. Trasferire le cellule non legate in una nuova provetta da microcentrifuga e aggiungere 50 μl di perle nude lavate.
- Ripetere la procedura per altri due cicli di selezione negativa. Nel frattempo, preparare le perle caricate con antigene per la selezione positiva.
- Dopo 3 cicli di selezione negativa, aggiungere 50 μl di soluzione di perle caricate con antigene alle cellule e incubare per 2 ore a 4 °C.
- Posizionare le cellule contenenti le perle caricate con l'antigene su una rastrelliera magnetica con il coperchio aperto e pipettare il liquido presente nel coperchio nella provetta. Attendere 2 minuti prima di scartare le celle non legate.
- Eseguire tutti i passaggi rimanenti come descritto per la prima selezione dell'antigene-sfera. L'unica differenza rispetto alla prima selezione positiva (passaggio 3.3.2) è il numero di fasi di lavaggio: invece di lavare una volta con PBSA, lavare due volte la sospensione di perline con PBSA per aumentare il rigore della selezione.
NOTA: Di solito, la diversità viene nuovamente ridotta di 10x-100x. La massima diversità della libreria G4 dopo questa seconda selezione di perline era di 3,45 x 105 celle. Come notato in precedenza, si prevede che la diversità effettiva sia inferiore a causa del sovracampionamento della libreria.
6. Selezione delle librerie tramite smistamento citofluorimetrico
- Dopo l'induzione notturna dell'espressione superficiale in SG-CAA, misurare OD600 come descritto sopra. Pellet celle sufficienti a coprire 10 volte la diversità (2000 x g, 5 min, RT). Scartare il surnatante.
- Risospendere il pellet in PBSA e trasferirlo in provette per microcentrifuga. Utilizzare 3 x 107 celle per la colorazione per ogni provetta. Preparare tutti i tubi necessari, a seconda della diversità. Preparare una provetta di controllo per la colorazione senza l'antigene.
NOTA: Ad esempio, se è necessario ordinare 30 x 106 celle, pellettare 300 x 106 celle per ottenere un pellet di celle visibile e prevenire la perdita di celle nei passaggi successivi. Risospendere in 5 mL di PBSA e trasferire da 3 a 4 aliquote da 500 μL ciascuna in provette per microcentrifuga. Preparare circa 3 volte più cellule del numero che dovrebbe essere ordinato perché una frazione di cellule andrà persa durante il processo di colorazione. Se la selezione viene eseguita in presenza di un farmaco/ligando, preparare un ulteriore controllo con il solo antigene ma senza il farmaco/ligando. - Centrifugare nuovamente (2000 x g, 5 min, RT), risospendere il pellet in 200 μL di PBSA contenente l'antigene (hRBP4 con 5 μM A1120) e incubare per 1 ora a 4 °C.
NOTA: Tutti i passaggi da questo punto in poi devono essere eseguiti su ghiaccio e con reagenti freddi. Per prevenire la deplezione degli anticorpi o dell'antigene, tutti i reagenti devono essere aggiunti in eccesso stechiometrico rispetto al numero totale di proteine visualizzate con lievito nella provetta. In caso contrario, potrebbe esserci il rischio di limitare il legame delle varianti mostrate dal lievito a causa della limitata disponibilità di antigeni e/o anticorpi. Di solito, per il calcolo vengono utilizzate 5 x 104 molecole per cellula di lievito e si consiglia di aggiungere un eccesso stechiometrico di almeno 10 volte di antigene e anticorpi. Se l'antigene è molto limitato, l'eccesso può essere ridotto a 5 volte. Una spiegazione più dettagliata, inclusi esempi per i calcoli per evitare la deplezione del ligando, è fornita in un precedente protocollo9. - Dopo l'incubazione, centrifugare le cellule (2000 x g, 5 min, 4 °C) e lavare con 1 mL di PBSA (se durante le selezioni è presente un farmaco/ligando - come A1120 in questo caso - questo deve essere presente nel tampone di lavaggio).
- Risospendere le cellule in 100 μl di PBSA freddo contenente gli anticorpi per la colorazione e la rilevazione dell'antigene legato e incubare per 30 minuti a 4 °C.
- Utilizzare l'anticorpo anti-c-myc (diluizione finale 1:100) per rilevare i POI visualizzati a figura intera. Utilizzare anticorpi anti-HA (diluizione finale 1:50) per rilevare la quantità totale di proteine visualizzate. Utilizzare l'anticorpo Penta-His (diluizione finale 1:20) o la streptavidina (diluizione finale 1:200) per selezionare le cellule di lievito che si legano all'antigene solubile contenente rispettivamente un tag 6x HIS o biotina.
- Alternare i reagenti di rilevamento tra i cicli di smistamento per prevenire l'arricchimento di varianti proteiche che si legano al reagente secondario.
NOTA: È possibile utilizzare diverse combinazioni di fluorofori, ma Alexa Fluor 488 e Alexa Fluor 647 sono buone opzioni in quanto non richiedono una compensazione nella citometria a flusso. Per prevenire la deplezione degli anticorpi, dovrebbe essere aggiunto in eccesso stechiometrico rispetto al numero totale di proteine visualizzate nel lievito nella provetta. Di solito, i calcoli si basano su 5 x 104 molecole per cellula di lievito. Una spiegazione più dettagliata è fornita nei precedenti protocolli9.
- Dopo l'incubazione, centrifugare le cellule (2000 x g, 5 min, 4 °C) e aggiungere 1 mL di PBSA (contenente il farmaco/ligando, per la libreria G4 selezionata da hRBP4 5 μM di A1120) al pellet e centrifugare nuovamente. Rimuovere la maggior parte del surnatante e conservare solo 20-30 μl per evitare che il pellet si secchi.
- Risospendere il pellet in PBSA freddo solo subito prima della cernita e selezionare le cellule di lievito che mostrano molecole Fn3 con le proprietà desiderate (cioè, legandosi a hRBP4 in presenza di A1120).
NOTA: La strategia di gating è illustrata nella Figura 2A. - Separare le cellule direttamente nel terreno SD-CAA, aggiungere altro terreno SD-CAA dopo la cernita e incubare a 30 °C agitando a 180 giri/min.
NOTA: Nel caso in cui il volume aumenti significativamente dopo la cernita, è necessario eseguire una fase di centrifugazione (2000 x g, 5 min, RT) per rimuovere il liquido della guaina in eccesso e le cellule vengono risospese in un terreno SD-CAA fresco. Annotare il numero di cellule ordinate nella nuova provetta, poiché questa diventa la nuova diversità massima per il successivo ciclo di smistamento. Come notato sopra, l'effettiva diversità della biblioteca sarà inferiore a causa del sovracampionamento della biblioteca. - Ripetere gli ordinamenti citometrici a flusso per diversi cicli per vedere un arricchimento di varianti proteiche leganti nella libreria di lieviti (Figura 2B).
NOTA: Può essere utile confrontare le librerie fianco a fianco in un esperimento di citofluorimetria analitica. Nei cicli di selezione successivi, l'abbassamento della concentrazione dell'antigene può essere utile per discriminare tra leganti di affinità più bassi e più alti e per selezionare in modo specifico le varianti proteiche che mostrano un segnale di legame più elevato con il bersaglio a basse concentrazioni (Figura 2C). Nei turni di selezione successivi, è anche possibile effettuare la selezione per una maggiore termostabilità. A tale scopo, è stata eseguita la mutagenesi casuale della libreria mediante epPCR, come descritto nella Sezione 7. Preparare le cellule in modo simile ad altri cicli di selezione citofluorimetrica mediante induzione, determinazione di OD600 e lavaggio delle cellule. Mettere le cellule di lievito in PBSA su ghiaccio per 10 minuti, seguito da un'incubazione di 10 minuti in un termociclatore per lo shock termico a temperatura elevata. Successivamente, rimetterli sul ghiaccio per 10 minuti e procedere con il protocollo di colorazione. Le varianti stabilizzate resistono alla denaturazione termica e, quindi, mostrano un legame mantenuto dopo l'incubazione termica. Diverse temperature possono essere testate per lo shock termico, a seconda della termostabilità iniziale della proteina wildtype. In genere, sono adatte temperature comprese tra 40 e 80 °C. Tuttavia, al di sopra dei 48 °C, la vitalità delle cellule di lievito diminuirà drasticamente. Pertanto, è necessario estrarre i plasmidi dopo aver ordinato la libreria di lieviti con shock termico, come discusso nel passaggio 7.1. In questo caso, le cellule EBY100 devono essere trasformate di fresco con varianti arricchite prima di procedere al successivo ciclo di smistamento36. Un protocollo più dettagliato per aumentare la termostabilità può essere trovato altrove37. Se la denaturazione della proteina visualizzata è reversibile, questo metodo non funzionerà. In alternativa, indurre l'espressione proteica a 37 °C invece di 20 °C il giorno prima dello smistamento e selezionare le varianti proteiche che mostrano ancora un'efficiente visualizzazione a figura intera.
7. Maturazione di affinità con epPCR per introdurre mutazioni casuali
NOTA: La maturazione per affinità mediante epPCR può essere eseguita prima del primo ciclo di selezione citofluorimetrica o tra i cicli di selezione citofluorimetrica. Per la selezione della libreria G4 con hRBP4 in presenza di A1120, la maturazione per affinità è stata condotta prima del primo ciclo di selezione citofluorimetrica. Ciò dipende anche dalla dimensione della libreria dopo la selezione della perlina e dal segnale di legame che può essere rilevato con la citometria a flusso. In particolare, nei casi in cui le affinità dopo la selezione delle microsfere non sono sufficienti per ottenere un segnale negli esperimenti di citofluorimetria (perché l'antigene si dissocia rapidamente durante le fasi di lavaggio), l'epPCR può generare varianti migliorate che possono essere successivamente rilevate e selezionate tramite citometria a flusso.
- Eseguire un'estrazione del DNA del lievito miniprep dalla libreria di lievito arricchita utilizzando il kit commerciale plasmide miniprep II (vedi Tabella dei materiali) con le seguenti modifiche:
- Misurare la densità cellulare della coltura notturna, diluire a un OD600 di 0,2 al mattino e incubare a 30 °C agitando.
- Quando OD600 raggiunge 0,6, raccogliere 3 mL di coltura centrifugando le cellule a 600 x g per 2 minuti.
- Aggiungere 200 μl di soluzione 1 al pellet. Aggiungere 6 μl di zimoliasi. Risospendere il pellet mediante flicking o leggero vortice e incubare a 37 °C per 3 ore.
- Aggiungere 200 μl di soluzione 2 e mescolare delicatamente. Aggiungere 400 μl di soluzione 3 e mescolare delicatamente.
- Centrifugare alla massima velocità per 3 min.
- Trasferire il surnatante nella colonna di spin-I, centrifugare alla massima velocità per 30 s ed eliminare il flusso.
- Lavare con 550 μL di tampone di lavaggio contenente etanolo e centrifugare alla massima velocità per 2 minuti.
- Trasferire la colonna in una provetta da microcentrifuga pulita e centrifugare alla massima velocità per 2 minuti per rimuovere il tampone di lavaggio residuo.
- Trasferire la colonna in una nuova provetta da microcentrifuga, aggiungere 10 μL di acqua priva di nucleasi e centrifugare per 1 minuto alla massima velocità per eluire il DNA.
- Eseguire una PCR iniziale per amplificare il modello per l'epPCR.
- Amplificare il DNA mediante PCR utilizzando primer di ricottura all'esterno dell'inserto sul vettore di visualizzazione della superficie del lievito.
NOTA: Poiché molto DNA genomico di lievito viene eluito insieme al plasmide di interesse durante la miniprep del lievito, questo passaggio è raccomandato per ottenere un frammento di DNA pulito da utilizzare come modello per l'epPCR. - Per pCTCON2, utilizzare primer fwd 5'- GGCTCTGGTGGAGGCGGTAGCGGAGGCGGAG
GGTCGGCTAGC e rev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTTCGGATCC con i componenti di reazione descritti nella Tabella 3 e le condizioni cicliche illustrate nella Tabella 4. - Purificare la PCR con un kit per PCR e pulizia del DNA. Eluire in 20 μL di H2O privo di nucleasi.
- Amplificare il DNA mediante PCR utilizzando primer di ricottura all'esterno dell'inserto sul vettore di visualizzazione della superficie del lievito.
- Eseguire l'epPCR per introdurre mutazioni casuali.
- Eseguire una epPCR con analoghi nucleotidici con gli ingredienti indicati (Tabella 5) e le condizioni (Tabella 6).
- Eseguire l'intero prodotto epPCR su un gel preparativo di agarosio all'1% a 120 V per 45 minuti. Includere un pennarello e lasciare una corsia tra il prodotto epPCR e la scala. Asportare la banda desiderata corrispondente alla dimensione prevista dell'inserto, purificarla utilizzando un kit di estrazione del gel e misurare la concentrazione di DNA.
- Amplificare il DNA randomizzato mediante PCR.
- Amplificare 50 ng di DNA con una polimerasi Q5. Impostare 2 x 100 μL di reazione PCR per ogni elettroporazione (Tabella 7) con le condizioni indicate (Tabella 8). Utilizzare gli stessi primer utilizzati per la prima PCR.
NOTA: Sono necessarie grandi quantità di DNA per l'elettroporazione nel lievito. - Analizzare 5 μL del prodotto PCR su un gel di agarosio all'1%, eseguire a 120 V per 45 minuti.
- Amplificare 50 ng di DNA con una polimerasi Q5. Impostare 2 x 100 μL di reazione PCR per ogni elettroporazione (Tabella 7) con le condizioni indicate (Tabella 8). Utilizzare gli stessi primer utilizzati per la prima PCR.
- Purificare il DNA amplificato con precipitazione di etanolo.
- Raggruppare 2 x 100 μL insieme. Aggiungere 20 μl (10% del volume) di acetato di sodio 3 M filtrato sterile, pH 5,2.
NOTA: Questo tampone può essere preparato in anticipo e conservato per alcuni mesi presso RT. - Aggiungere 440 μl di etanolo al 100% (o almeno 2 volte il volume) e incubare a RT per 2 minuti.
- Centrifugare a 20.000 x g per 5 minuti a RT. Rimuovere il surnatante, aggiungere 500 μl di etanolo al 70% e mescolare brevemente.
- Centrifugare a 20.000 x g per 5 minuti a RT. Rimuovere il surnatante, aggiungere 500 μl di etanolo al 100% e mescolare brevemente.
- Centrifugare a 20.000 x g per 5 minuti a RT. Rimuovere il surnatante e lasciare asciugare il pellet di DNA sotto una cappa a flusso laminare fino a quando tutto l'etanolo non è evaporato.
NOTA: Utilizzare un blocco termico a 48 °C per accelerare questo processo. Il pellet di DNA potrebbe non essere sempre visibile. - Sciogliere il pellet di DNA essiccato in 10 μL di H2O privo di nucleasi.
NOTA: Il DNA deve essere disciolto nel minor volume possibile. Non misurare la concentrazione di DNA.
- Raggruppare 2 x 100 μL insieme. Aggiungere 20 μl (10% del volume) di acetato di sodio 3 M filtrato sterile, pH 5,2.
| Volume [μL] | Concentrazione finale | |
| 5x potenziatore Q5 | 10 | 1x |
| 5x buffer Q5 | 10 | 1x |
| Innesco fwd 10 μM | 2.5 | 0,5 μM |
| Primer rev 10 μM | 2.5 | 0,5 μM |
| dNTP 10 mM | 1 | 200 μM |
| Q5 polimerasi | 0.5 | 20 U/mL |
| DNA da lievito miniprep | 10 | |
| H2O senza nucleasi | 13.5 |
Tabella 3: Condizioni per la PCR di1a fase per l'amplificazione dei geni POI dal miniprep di lievito isolato.
| Passo | Temperatura | Ore |
| Denaturazione iniziale | 98 °C | 30 secondi |
| 25 cicli | 98 °C | 10 secondi |
| 72 °C | 30 secondi | |
| 72 °C | 30 secondi | |
| Estensione finale | 72 °C | 2 minuti |
| Tenere | 4 °C |
Tabella 4: Condizioni cicliche per la PCR di1a fase per l'amplificazione dei geni POI dal miniprep di lievito isolato.
| Volume [μL] | Concentrazione finale | |
| H2O senza nucleasi | fino a 50 | |
| 10x tampone Thermopol | 5 | 1x |
| Primer_fwd (10 μM) | 2.5 | 0,5 μM |
| Primer_rev (10 μM) | 2.5 | 0,5 μM |
| dNTP (10 mM) | 1 | 200 μM |
| 8-oxo-dGTP (100 μM) | 1 | 2 μM |
| dPTP (100 μM) | 1 | 2 μM |
| Prodotto PCR dalla 1a PCR | XX | 50 ng |
| Taq DNA polimerasi | 0.5 | 0,05 U/μL |
Tabella 5: Condizioni per l'epPCR che viene eseguita dopo l'amplificazione del DNA del POI con la PCR di1a fase.
| Passo | Temperatura | Ore |
| Denaturazione iniziale | 94 °C | 30 secondi |
| 15 cicli | 94 °C | 45 secondi |
| 60 °C | 30 secondi | |
| 72 °C | 1 minuto | |
| Estensione finale | 72 °C | 10 minuti |
| Tenere | 4 °C |
Tabella 6: Condizioni cicliche per l'epPCR.
| Volume [μL] | Concentrazione finale | |
| 5x potenziatore Q5 | 20 | 1x |
| 5x buffer Q5 | 20 | 1x |
| Innesco fwd 10 μM | 5 | 0,5 μM |
| Primer rev 10 μM | 5 | 0,5 μM |
| dNTP 10 mM | 1 | 200 μM |
| Q5 polimerasi | 1 | 20 U/mL |
| 50 ng di DNA | XX | |
| ddH20 | fino a 100 |
Tabella 7: Condizioni per la PCR dellaseconda fase per l'amplificazione del prodotto epPCR prima dell'elettroporazione delle celle EBY100.
| Passo | Temperatura | Ore |
| Denaturazione iniziale | 98 °C | 30 secondi |
| 25 cicli | 98 °C | 10 secondi |
| 72 °C | 30 secondi | |
| 72 °C | 30 secondi | |
| Estensione finale | 72 °C | 2 minuti |
| Tenere | 4 °C |
Tabella 8: Condizioni cicliche per la PCR dellaseconda fase per l'amplificazione del prodotto epPCR.
8. Linearizzazione del vettore di visualizzazione del lievito per l'elettroporazione
- Linearizzare il vettore di visualizzazione della superficie del lievito pCTCON2 (plasmide Addgene #41843).
- Eseguire un digest su larga scala e utilizzare questo vettore linearizzato digerito per tutte le ulteriori elettroporzioni.
- Digerire 2 x 200 μg di DNA plasmidico pCTCON2 con Sal I per 24 ore a 37 °C per una linearizzazione iniziale (Tabella 9).
- Digerire con NheI e BamHI per 24 ore a 37 °C per asportare l'inserto (Tabella 10).
- Infine, aggiungere 5 μL di ciascun enzima alla provetta (Sal I, NheI, BamHI) e incubare nuovamente per 24 ore a 37 °C.
- Eseguire il vettore digerito su un gel di agarosio preparativo. Asportare le bande e purificare con un kit di estrazione in gel. Eluire il vettore in 20 μL, misurare la concentrazione di DNA e regolarla a circa 2 μg/μL.
NOTA: La digestione del plasmide per un tempo prolungato garantisce una scissione sufficiente dell'inserto e si tradurrà in una minore dispersione dal vettore dopo l'elettroporazione.
| DNA | 200 μg |
| 10x CutSmartBuffer | 50 μl |
| Sal I-HF (NEB) | 30 μl (60 U) |
| H2O | Fino a 500 μl |
Tabella 9: Condizioni per la prima fase del digest su larga scala del vettore di visualizzazione della superficie del lievito pCTCON2.
| pCTCON2 (Sal I digerito) | 500 μl |
| 10x CutSmartBuffer | 37,5 μl |
| NheI-HF (NEB) | 15 μl (30 U) |
| BamHI-HF (NEB) | 15 μl (30 U) |
| H2O | fino a 875 μl |
Tabella 10: Condizioni per la seconda fase della digestione su larga scala del vettore di visualizzazione della superficie del lievito pCTCON2.
9. Elettroporazione di EBY100 con DNA randomizzato e vettore linearizzato
- Preparare le cellule di lievito EBY100 (ATCC, vedi Tabella dei materiali) estraendole da uno stock di glicerolo 3 giorni prima dell'elettroporazione. Poiché le cellule EBY100 non trasformate non contengono il vettore di visualizzazione della superficie del lievito, utilizzare piastre YPD a pieno terreno (Tabella 1). Incubare a 30 °C.
- Un giorno prima dell'elettroporazione, inoculare una singola colonia di EBY100 in 30 mL di terreno YPD appena preparato (Tabella 1). Incubare per una notte a 30 °C agitando a 180 giri/min.
NOTA: Il terreno YPD non deve essere più vecchio di 1 mese. - La mattina successiva, diluire la coltura notturna EBY100 a un OD600 di 0,2 in 100 mL di terreno YPD e incubare a 30 °C agitando a 180 giri/min.
NOTA: Un volume di coltura di 50 mL è sufficiente per 2 elettroporzioni. In genere, 100 ml vengono preparati per avere cellule sufficienti per i controlli (solo cellule e cellule più vettore). - Quando si raggiunge un OD600 di 1,3-1,5, pellettare le cellule in provette da 50 mL a 2000 x g per 3 minuti e rimuovere il surnatante.
- Risospendere il pellet in 25 mL di acetato di litio filtrato sterile da 100 mM (vedi Tabella dei materiali). Aggiungere DTT filtrato sterile appena preparato (1 M) (vedi Tabella dei materiali) a una concentrazione finale di 10 mM.
NOTA: Il volume dell'acetato di litio dovrebbe essere la metà del volume originale di YPD. Se necessario, ridurre o aumentare le prestazioni. L'acetato di litio (100 mM) può essere preparato in anticipo e conservato per alcuni mesi presso RT, ma il brodo DTT da 1 M deve essere preparato subito prima dell'uso e tenuto in ghiaccio. - Incubare le cellule a 30 °C agitando per 10 minuti.
- Centrifugare a 2000 x g per 3 minuti, scartare il surnatante e mettere le cellule sul ghiaccio.
NOTA: Tutti i passaggi seguenti devono essere eseguiti su ghiaccio e con reagenti, cuvette e provette per microcentrifuga raffreddati. - Risospendere il pellet in 25 mL di H2O. Centrifugare a 2000 x g per 3 min.
NOTA: Il volume H2O utilizzato deve essere la metà del volume YPD originale. - Risospendere in 350 μL di H2O sterile.
NOTA: Il volume totale dopo la risospensione deve essere di 500 μL. Se necessario, aggiungere altro H2O. - Preparare la libreria di DNA in parallelo: posizionare le provette per microcentrifuga contenenti gli inserti purificati con etanolo sul ghiaccio e aggiungere 4 μg di vettore pCTCON2 linearizzato.
- Aggiungere 250 μL di cellule al DNA e mescolare delicatamente tagliando. Trasferire in una cuvetta per elettroporazione da 2 mm pre-raffreddata e tenere in ghiaccio fino all'elettroporazione.
NOTA: Aggiungere i controlli (solo cellule senza DNA e cellule con vettore linearizzato solo senza inserto) per determinare il successo dell'elettroporazione. - Elettroporazione con cuvette per elettroporazione da 2 mm (vedi Tabella dei materiali) con le seguenti condizioni: protocollo a onda quadra, impulso singolo, 500 V, durata dell'impulso 15 ms.
NOTA: I cali tipici sono del 3%-9%. È possibile utilizzare altri elettroporatori, ma le condizioni indicate sono ottimizzate per lo strumento utilizzato in questo studio. - Salvare le cellule con 1 mL di YPD preriscaldato, trasferirle in una provetta da microcentrifuga preriscaldata e incubare senza agitare per 1 ora a 30 °C.
NOTA: Posizionare il blocco riscaldante accanto all'elettroporatore per assicurarsi che i tubi di recupero rimangano alla temperatura corretta. - Piastra 100 μl di diluizioni di celle 10-3, 10-4, 10-5 e 10-6 su piastre SD-CAA (vedi Tabella 1) e consentire la crescita di trasformanti a 30 °C.
NOTA: La diversità teorica della libreria si ottiene calcolando il numero di trasformanti al volume di coltura originale, qui 1 mL. Tipicamente, questo protocollo di trasformazione produce circa 107-10 8 cellule trasformate. - Pellettare le cellule rimanenti a 2000 x g per 3 minuti, risospendere in 200 mL SD-CAA e incubare a 30 °C agitando a 180 giri/min per una notte. Le cellule trasformate contenenti la libreria randomizzata con epPCR possono essere passate in SD-CAA e indotte per l'espressione superficiale in SG-CAA il giorno successivo o conservate a 4 °C fino a nuovo utilizzo. Congelare le scorte di glicerolo della libreria dopo epPCR dopo aver determinato la diversità. Poiché il giorno successivo una frazione di cellule sarà ancora costituita da cellule non trasformate che sono state trasferite nella procedura di trasformazione senza ottenere un plasmide, si raccomanda di passare almeno 20 volte la diversità della libreria nella prima fase di passaggio per compensare questo effetto.
10. Sequenziamento di librerie di lieviti dopo diversi cicli di selezione
- Estrarre il DNA del lievito eseguendo una miniprep di lievito come descritto sopra.
- Elettroporare cellule elettrocompetenti commerciali di E. coli 10-beta (vedi Tabella dei materiali) con il DNA isolato.
- Posizionare le cuvette per elettroporazione (1 mm, vedere la tabella dei materiali) e le provette per microcentrifuga sul ghiaccio. Preriscaldare le provette per microcentrifuga con 950 μL di terreno di crescita a 37 °C per recuperare le cellule dopo l'elettroporazione.
- Scongelare le cellule con ghiaccio e mescolare muovendo delicatamente il tubo. Trasferire 50 μl di cellule in una provetta da microcentrifuga refrigerata. Aggiungere 1 μL di DNA del lievito miniprep.
- Trasferire con cura la miscela cellula-DNA in una cuvetta di elettroporazione refrigerata.
NOTA: Evitare la formazione di bolle e assicurarsi che le cellule si depositino sul fondo della cuvetta. - Elettroporazione con un dispositivo di elettroporazione alle seguenti condizioni: 2,0 kV; 200 Ω; 25 μF.
NOTA: Le costanti di tempo tipiche sono 4,8-5,1 ms. È possibile utilizzare altri elettroporatori, ma le condizioni indicate sono ottimizzate per lo strumento utilizzato in questo studio. - Aggiungere immediatamente 950 μl di terreno di coltura preriscaldato a 37 °C (vedere la Tabella dei materiali) alle cuvette, mescolare delicatamente e trasferire nuovamente nelle provette calde per microcentrifuga.
NOTA: Posizionare il blocco riscaldante accanto all'elettroporatore per assicurarsi che i tubi di recupero rimangano alla temperatura corretta. - Incubare per 40 minuti a 37 °C e 300 giri/min. Piastra 10 μl, 100 μl e il resto delle cellule (dopo la centrifugazione) su una piastra LB selettiva preriscaldata (vedi Tabella 1). Incubare le piastre per una notte a 37 °C.
NOTA: Per il vettore pCTCON2, l'ampicillina viene utilizzata per la selezione degli antibiotici. - Preparare una copia-piastra con LB agar e l'antibiotico richiesto (qui viene utilizzata l'ampicillina): pipettare 200 μL di LB agar con 100 μg/mL di ampicillina in ogni pozzetto di una piastra da 96 pozzetti e lasciarla solidificare.
- Preparare una piastra di sequenziamento a 96 pozzetti con 50 μL di LB con 100 μg/mL di ampicillina per pozzetto.
- Prelevare una singola colonia dalle piastre e inoculare prima la piastra di copia e lo stesso pozzetto della piastra di sequenziamento.
NOTA: La piastra di copia assicura che i plasmidi desiderati possano essere facilmente ottenuti inoculando una coltura liquida ed eseguendo una miniprep. - Incubare la piastra di copia per una notte a 37 °C e quindi conservare a 4 °C.
- Incubare la piastra di sequenziamento per 3-4 ore a 37 °C agitando (180 giri/min) coperta con un foglio di plastica o alluminio.
- Sigillare la piastra di sequenziamento e inviarla per il sequenziamento insieme a 500 μL di primer di sequenziamento pCTCON2 da 10 μM (5'-CGTTTGTCAGTAATTGCGGTTCTC).
- Per l'analisi delle sequenze, tradurre le sequenze di DNA ottenute in sequenze di amminoacidi utilizzando un programma adatto a scelta, ad esempio EMBOSS Transeq38. Successivamente, utilizzare l'allineamento di sequenze multiple (MUSCLE)39 o uno strumento di allineamento simile per allineare le sequenze varianti tra loro e alla proteina wildtype parentale.
NOTA: Eseguendo il sequenziamento di librerie di diversi cicli di selezione (ad esempio, dopo un precedente ciclo di selezione, nonché dopo ulteriori cicli di selezione con concentrazioni di antigene più basse per arricchire le varianti ad alta affinità), è possibile identificare diverse varianti. Ciò dipende fortemente dal rigore dei cancelli applicati durante i giri di smistamento e, quindi, dalla diversità delle biblioteche.
Risultati
La libreria G4 è stata selezionata contro l'antigene hRBP4 legato al farmaco a piccola molecola A1120. La colorazione delle librerie per la selezione citofluorimetrica è stata eseguita come descritto nel Metodo 6 e la strategia di gating applicata è mostrata nella Figura 2A. Un gate iniziale includeva tutte le cellule in base alla morfologia cellulare e il secondo gate (istogramma di FSC-Width) mostrava una rigorosa strategia di gating che è stata applicata per selezionare singole cellule e rimuovere gli aggregati cellulari. Il terzo e ultimo gate ha mostrato la visualizzazione delle varianti proteiche (asse x) rispetto al legame dell'antigene (asse y). Sono state ordinate le cellule di lievito che mostravano sia segnali di visualizzazione che di legame. È importante sottolineare che il cancello di smistamento è stato impostato in modo rigoroso per arricchire i domini di legame con un segnale di legame elevato e quindi un'elevata affinità. Questa rigorosa selezione ha prodotto un arricchimento di cellule di lievito che si legano specificamente all'antigene bersaglio durante la campagna di selezione (Figura 2B). Nei successivi cicli di selezione citofluorimetrica, la concentrazione di antigene è diminuita di 10 volte (da 100 nM a 10 nM). Pertanto, il segnale di legame complessivo è stato ridotto e solo i leganti con un'elevata affinità erano ancora rilevabili e ordinati (Figura 2C).
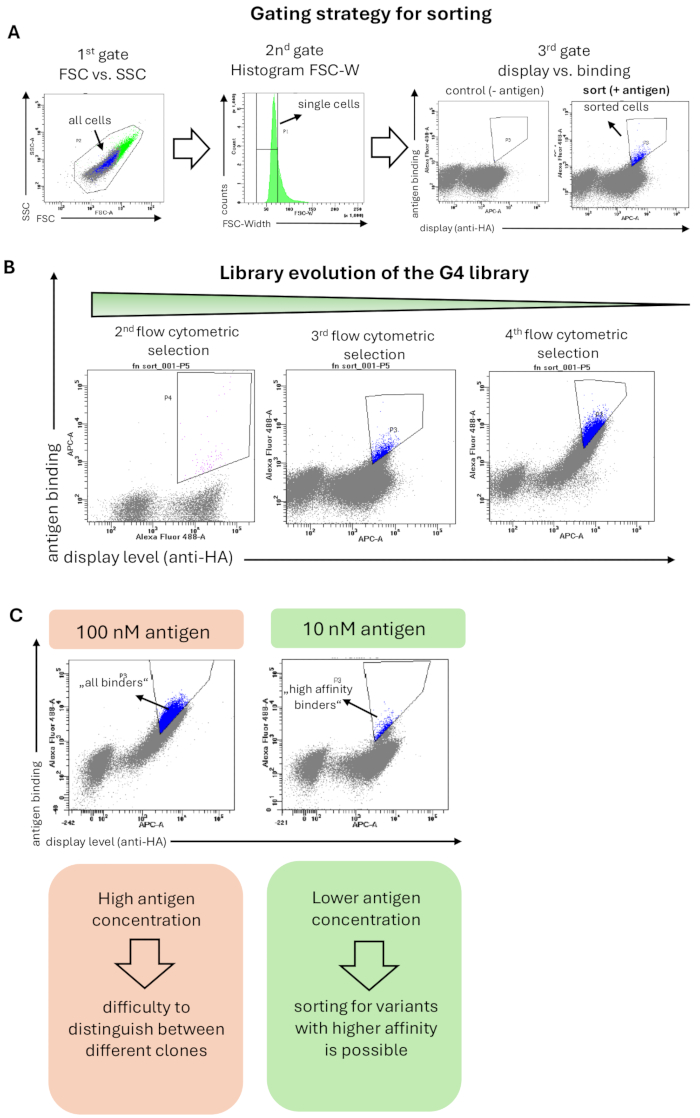
Figura 2: Risultati rappresentativi di una selezione della libreria G4 basata su Fn3 per il legame all'antigene (hRBP4 in presenza di A1120). (A) La strategia generale di gating per il sorting delle librerie di lievito. La prima porta (FSC vs. SSC) consiste nel selezionare tutte le cellule di lievito ed escludere gli eventi di dispersione; il secondo cancello (istogramma di FSC-W) mira a rimuovere gli aggregati cellulari e selezionare solo singole cellule di lievito. Il terzo gate traccia il livello di visualizzazione superficiale (rilevamento del tag HA o c-myc) rispetto al legame con l'antigene (qui hRBP4 in presenza di 5 μM A1120, rilevato dall'anticorpo anti-Hi). La libreria è stata inoltre colorata con solo anticorpi secondari (senza antigene), dove non è previsto alcun legame con l'antigene. Le celle ordinate sono evidenziate in blu. (B) Evoluzione della libreria G4 attraverso 3 cicli di smistamento citofluorimetrico. L'arricchimento della popolazione di legame può essere osservato ad ogni ciclo di selezione. (C) L'uso di concentrazioni di antigene più basse consente la selezione di varianti proteiche con una maggiore affinità verso l'antigene bersaglio. Dopo la riduzione della concentrazione dell'antigene (qui hRBP4) di 10 volte, compaiono diverse diagonali, che indicano la presenza di cloni con affinità maggiore (cellule selezionate, blu) o inferiore. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
La visualizzazione della superficie del lievito si è evoluta come uno dei metodi chiave utilizzati nell'ingegneria delle proteine. Sebbene sia comunemente impiegato per l'ingegneria dell'affinità 1,18,40,41, dell'espressione/stabilità 24,27,42,43 e dell'attività 28,44, ulteriori usi come la mappatura degli epitopi45,46 o la caratterizzazione dei singoli mutanti sulla superficie delle cellule di lievito9 sono possibili. In questo protocollo, forniamo i passaggi di base per l'avvio di una campagna di selezione della visualizzazione della superficie del lievito, inclusa la selezione con microsfere magnetiche e la selezione citofluorimetrica, nonché la diversificazione della libreria di lieviti mediante epPCR per la maturazione per affinità.
Un requisito essenziale per le selezioni convenzionali di visualizzazione della superficie del lievito è la disponibilità di proteine solubili di qualità sufficiente. Iniziare con una proteina bersaglio ben ripiegata con elevata purezza e uno stato di oligomerizzazione definito (cioè, la proteina monomerica dovrebbe essere presente solo come monomero) fornisce il più alto tasso di successo nella selezione di una variante proteica che si lega all'antigene bersaglio con elevata affinità. Un'alternativa per le proteine bersaglio difficili da esprimere sono le selezioni basate sulle cellule, che presentano una strategia ragionevole per aggirare questa limitazione47. Tuttavia, la visualizzazione della superficie del lievito offre molti vantaggi, come la possibilità di caratterizzare le varianti proteiche risultanti direttamente sulla superficie del lievito senza la necessità di eseguire il clonaggio laborioso e dispendioso in termini di tempo, l'espressione in un formato solubile e la purificazione delle proteine. Sia l'affinità che la stabilità delle varianti possono essere analizzate direttamente sulla superficie del lievito9.
In questo protocollo, mostriamo come la libreria G4 di varianti proteiche, più specificamente del 10° dominio di tipo III della fibronectina umana, sia stata selezionata per il legame con l'antigene hRBP4 in presenza della piccola molecola A1120. La combinazione di selezioni di biglie e smistamento citofluorimetrico ha prodotto un arricchimento delle varianti, che ha mostrato un aumento del legame con l'antigene bersaglio durante i cicli di selezione (Figura 2B). Abbiamo dimostrato che l'utilizzo di concentrazioni più basse di antigene consente la selezione di varianti proteiche ad alta affinità (Figura 2C). Tipicamente, le affinità che possono essere raggiunte con le selezioni di visualizzazione del lievito sono nell'intervallo nanomolare o addirittura picomolare18. Le affinità finali dipendono dall'antigene bersaglio, dal numero di cicli di selezione e dalla maturazione dell'affinità, dallo scaffold di legame utilizzato e dalla strategia di gating applicata. La caratterizzazione delle singole varianti proteiche non è trattata in questo protocollo, ma è spiegata in dettaglio nel nostro precedente lavoro9. Sebbene il display del lievito sia stato originariamente impiegato per l'ingegnerizzazione di frammenti di anticorpi come scFvs 1,40, il metodo è stato ampiamente utilizzato anche per proteine non basate su anticorpi10.
Per riassumere, il display della superficie del lievito è un potente strumento di ingegneria proteica che consente la generazione di varianti proteiche con proprietà nuove o migliorate, come il legame con quasi tutte le proteine bersaglio e/o una maggiore stabilità.
Divulgazioni
M.W.T. riceve finanziamenti da Miltenyi Biotec. Tutti gli autori sono inventori di domande di brevetto per tecnologie e proteine ingegnerizzate che sono state sviluppate utilizzando il display della superficie del lievito.
Riconoscimenti
Questo lavoro è stato sostenuto dal Fondo Scientifico Austriaco (Progetto FWF W1224 - Programma di Dottorato sulla Tecnologia Biomolecolare delle Proteine - BioToP e Progetto FWF ESP 465-B), dal Ministero Federale per gli Affari Digitali ed Economici dell'Austria, dalla Fondazione Nazionale per la Ricerca, la Tecnologia e lo Sviluppo dell'Austria alla Christian Doppler Research Association (Christian Doppler Laboratory for Next Generation CAR T Cells), e da donazioni private al St. Anna Children's Cancer Research Institute (Vienna, Austria). E.S. ha ricevuto una borsa di studio DOC dell'Accademia Austriaca delle Scienze presso l'Istituto di ricerca sul cancro dei bambini di Sant'Anna. Le figure sono state create con BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
Riferimenti
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon