È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazione dell'attività enzimatica degli enzimi deubiquitilanti associati a disturbi dello sviluppo neurologico tramite un saggio in vitro di scissione della catena dell'ubiquitina
In questo articolo
Riepilogo
Questo protocollo spiega come misurare l'effetto della variazione genetica associata ai disturbi dello sviluppo neurologico sull'attività dell'enzima deubiquitilante combinando la purificazione di proteine ricombinanti con saggi di scissione della catena dell'ubiquitina.
Abstract
I disturbi del neurosviluppo (NDD) sono associati a menomazioni della funzione del sistema nervoso, ma spesso rimangono poco compresi a livello molecolare. I disturbi discreti causati da singoli geni forniscono modelli per studiare i meccanismi che guidano lo sviluppo neurologico atipico. Varianti di geni che codificano per proteine della famiglia degli enzimi deubiquitilanti (DUB) sono associate a diverse NDD, ma è necessario determinare i meccanismi patogenetici dei disturbi guidati da queste varianti geniche. L'impatto delle varianti geniche sull'attività del DUB può essere determinato sperimentalmente utilizzando un saggio di scissione dell'ubiquitina in vitro indipendente dal substrato. Questo test non richiede la conoscenza dei substrati a valle per misurare direttamente l'attività catalitica. Qui, il protocollo per determinare l'impatto delle varianti geniche sull'attività enzimatica è modellato utilizzando la proteasi specifica per l'ubiquitina DUB 27, legata all'X (USP27X), che è mutata nella disabilità intellettiva legata all'X 105 (XLID105). Questa pipeline sperimentale può essere utilizzata per chiarire i meccanismi alla base dei disturbi dello sviluppo neurologico guidati da varianti nei geni DUB.
Introduzione
I disturbi del neurosviluppo (NDD) derivano da diverse eziologie con determinanti ambientali o genetici che guidano lo sviluppo atipicodel sistema nervoso 1. I test genetici di sequenziamento di nuova generazione hanno collegato un numero crescente di varianti nei geni correlati al sistema dell'ubiquitina con le NDD genetiche2. Il sistema dell'ubiquitina catalizza la legatura del piccolo modificatore proteico ubiquitina principalmente ai residui di lisina nei substrati proteici per guidare i cambiamenti nel comportamento cellulare, tra cui la localizzazione, la stabilità, le interazioni proteina-proteina o l'attività3. L'ubiquitinazione è mediata dagli enzimi E1 attivante, E2 coniugato e E3 ligasi4 ed è reversibile dall'attività degli enzimi deubiquitilanti (DUB) che catalizzano la scissione e la rimozione dell'ubiquitina dai substrati proteici5. L'ubiquitina può essere legata al substrato come monomero (monoubiquitinazione) o catene polimeriche (poliubiquitinazione) che si formano su uno qualsiasi dei sette residui di lisina (K6, K11, K27, K29, K33, K48, K63) o il residuo M1 dell'ubiquitina. Queste diverse topologie di catena dell'ubiquitina e le loro combinazioni creano un codice cellulare che è la chiave per la trasduzione del segnale6.
DUB come USP27X, USP7, USP9X, USP48, STAMBP, OTUD4, OTUD6B e OTUD5 sono stati associati a NDD 2,7,8,9,10,11. Per la maggior parte delle NDD, i meccanismi molecolari che guidano la patogenesi rimangono sperimentalmente indefiniti. Alcuni dei DUB che guidano i disturbi descritti di recente sono scarsamente conosciuti e mancano di letture cellulari note che possono essere utilizzate per valutare l'impatto della variazione genetica sulla funzione delle proteine. In vitro, i saggi di scissione della catena dell'ubiquitina superano questa limitazione in quanto l'attività DUB indipendente dal substrato può misurare l'impatto delle varianti geniche sull'attività enzimatica12.
I saggi in vitro per la scissione dell'ubiquitina sono stati utilizzati dagli anni '80. Questi saggi con substrati radiomarcati hanno permesso la scoperta dei primi DUB, tra cui l'isopeptidasi, identificata per la sua capacità di deubiquitilare l'istone H2A13, e l'ubiquitina carbossil-terminale idrolasi (UCH), identificata per la sua capacità di idrolizzare l'ubiquitina da una varietà di coniugati chimici 14,15,16. Inoltre, sono state utilizzate proteine o peptidi poliubiquitilati radiomarcati a lunghezza intera per identificare l'isopeptidasi T e diverse UCH e proteasi specifiche per l'ubiquitina (UBP) da eritrociti e muscolo scheletrico, rispettivamente 17,18,19,20. Le catene di ubiquitina di lunghezza e tipo di legame definite (tetra-ubiquitina legata a K48) sono state utilizzate per la prima volta per misurare l'attività DUB dell'isopeptidasi T21. Da allora, questo test è diventato il gold standard per misurare l'attività del DUB nelle analisi mutazionali22,23. Il perfezionamento di questo test consente attualmente la visualizzazione della scissione dell'ubiquitina tramite elettroforesi e colorazioni di gel convenzionali come il blu Coomassie, l'arancione SYPRO, la colorazione di rubino e argento o la rilevazione fluorescente o basata sull'immunoblotting12,24. Gli aspetti molecolari dell'attività del DUB, come la lunghezza minima della catena e la specificità del linkage 25,26,27,28, possono essere chiariti utilizzando catene di ubiquitina di diverse lunghezze (ad esempio, di-, tri-, tetra-ubiquitina) e linkage (K6, K11, K27, K29, K33, K48, K63, lineare) in saggi funzionali. Le varianti associate a NDD possono determinare difetti di attività DUB specifici del tipo di collegamento della catena dell'ubiquitina11.
Un saggio di scissione della di-ubiquitina che utilizza proteine DUB ricombinanti purificate può misurare direttamente l'impatto delle varianti NDD sull'attività DUB. USP27X, che è mutato nel disturbo da disabilità intellettiva legato all'X NDD 105 (XLID105)7,28, modella il processo qui presentato. Questo approccio consente di determinare come l'attività del DUB sia interrotta da varianti geniche in NDD associati a DUB esistenti e sconosciuti.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Il seguente protocollo può essere adattato per le proteine ricombinanti utilizzando vari tag di affinità espressi in diversi ceppi di cellule competenti. A seconda della proteina espressa, le condizioni di coltura e di espressione notturna possono richiedere l'ottimizzazione dell'OD600 all'induzione dell'espressione, al tempo di espressione, alla temperatura di espressione e alla concentrazione di IPTG. Una panoramica del protocollo è illustrata nella Figura 1. I dettagli dei reagenti e delle attrezzature utilizzate in questo studio sono elencati nella tabella dei materiali.
1. Trasformazione di cellule competenti di E. coli di Rosetta 2 con il plasmide ricombinante di espressione GST-USP27X
NOTA: Per mantenere la sterilità della coltura batterica, eseguire i passaggi in cui i contenitori dei terreni sono aperti sotto la fiamma di un bruciatore Bunsen. Per consentire un trasferimento ottimale dell'ossigeno, eseguire l'agitazione della coltura batterica in un agitatore da banco a temperatura controllata con un'orbita da 19 mm a 50 mm e una velocità di 200 giri/min29.
- Scongelare 20 μl di cellule di Rosetta 2 coli chimicamente competenti su ghiaccio immediatamente prima dell'uso. Aggiungere 1-10 ng del plasmide di espressione pGEX6P1-USP27X (codificante per USP27X marcato con glutatione-S-transferasi N-terminale (GST)30 e contenente resistenza all'ampicillina) e incubare 5 minuti su ghiaccio.
- Shock termico per 30 s a 42 °C in bagno asciutto. Incubare con ghiaccio per 2 min.
- Aggiungere 80 μl di terreno SOC a temperatura ambiente (RT). Incubare per 60 minuti a 37 °C con rotazione di 200 giri/min in un agitatore da banco con orbita da 19 mm a temperatura controllata.
- Piastra 50 μL di coltura su una piastra di agar LB integrata con 25 μg/L di cloramfenicolo e 50 μg/L di ampicillina. Incubare per 20 ore a 37 °C con il coperchio rivolto verso il basso in un incubatore a temperatura controllata.
2. Espressione batterica notturna di proteine ricombinanti da plasmidi di espressione
NOTE: Per mantenere la sterilità della coltura batterica, eseguire i passaggi in cui i contenitori dei terreni sono aperti sotto la fiamma di un bruciatore Bunsen. Per consentire un trasferimento ottimale dell'ossigeno, eseguire l'agitazione della coltura batterica in un agitatore da banco a temperatura controllata con un'orbita da 19 mm a 50 mm e una velocità di 200 giri/min29. Misurare la coltura OD600 utilizzando uno spettrofotometro.
- Preparare 1 L di Terrific Broth (TB) aggiungendo 47,6 g di TB Powder a 1 L di acqua ultrapura con glicerolo allo 0,4% in un pallone di coltura da 2 L. Autoclavare a medio per 30 minuti e raffreddare a RT.
- Prelevare una singola colonia di batteri trasformati e aggiungerla a 10 ml di terreno sterile LB integrato con 25 μg/L di cloramfenicolo e 50 μg/L di ampicillina. Incubare per 20 ore a 37 °C con rotazione di 200 giri/min in un agitatore da banco da 19 mm con orbita a temperatura controllata.
- Aggiungere 10 ml di coltura notturna a 1 litro di terreno TB integrato con 25 μg/L di cloramfenicolo e 50 μg/L di ampicillina (rapporto 1:100 tra coltura di partenza e coltura di espressione). Incubare a 37 °C con rotazione di 200 giri/min in un agitatore da banco con orbita di 19 mm a temperatura controllata fino a quando la coltura OD600 è compresa tra 0,5 e 0,6.
- Aggiungere 50 μM di IPTG alla coltura per indurre l'espressione, raffreddare a 16 °C e incubare per 20 ore a 16 °C con una rotazione di 200 giri/min.
- Cellule in pellet provenienti da coltura di espressione mediante centrifugazione per 20 minuti a ≥3.000 x g e 4 °C. Conservare il pellet a -80 °C per almeno 1 ora.
NOTA: A questo punto, l'esperimento può essere messo in pausa e riavviato in un secondo momento (preferibilmente la stessa settimana). Il pellet può essere conservato a lungo termine a -80 °C.
3. Purificazione delle proteine mediante colonna di affinità gravità-flusso
NOTA: La resina, il legame, il lavaggio, l'eluizione e i tamponi di conservazione appropriati per ogni purificazione dipenderanno dalla proteina ricombinante da purificare. Raccogli campioni dal pellet cellulare, dal surnatante, dal flusso, dalle frazioni di lavaggio e dalle frazioni di eluizione nel tampone SDS-PAGE. Eseguire la colorazione SDS-PAGE e Coomassie per i campioni per valutare il successo della purificazione. Eseguire la purificazione a 4 °C e maneggiare le frazioni con ghiaccio. La scissione delle etichette proteiche può essere eseguita all'interno o all'esterno della colonna utilizzando la proteasi appropriata per colpire il sito di scissione specifico della proteasi pertinente.
- Fissare la colonna di flusso a gravità vuota nel supporto della storta e riempirla con resina di glutatione agarosio. Utilizzare 2-3 mL di resina per purificare un pellet cellulare raccolto da 1 L di coltura di espressione.
- Lavare la colonna con un volume di letto di resina al 20% di etanolo. Lavare la colonna 3 volte con un volume di letto di resina di tampone MS500 (20 mM di Tris pH 7,5, 500 mM di NaCl, 0,5 mM di TCEP). Arrestare la colonna in modo che la resina rimanga ricoperta di tampone per evitare che si secchi durante la preparazione del pellet della cella.
- Scongelare il pellet della cella a 4 °C. Aggiungere 30 mL di tampone MS500 (20 mM di Tris pH 7,5, 500 mM di NaCl, 0,5 mM di TCEP, 60 mg di lisozima e una compressa di inibitore della proteasi) al pellet scongelato. Limare per 30 minuti a 4 °C con una leggera rotazione da un capo all'altro.
- Sonicare le cellule lisate in una provetta da centrifuga da 50 mL o in un becher di metallo su ghiaccio fino a quando il lisato scorre liberamente quando viene erogato da un puntale di pipetta. Centrifugare per 30 minuti a 4 °C e ≥20.000 x g per eliminare il surnatante.
NOTA: Determinare empiricamente le impostazioni del sonicatore. Impostare il sonicatore in modo tale che 120 s di tempo totale siano sufficienti per ridurre il lisato viscoso a un liquido libero e trasparente. - Decantare il surnatante in un becher e caricare il lisato chiarito sulla colonna. Far scorrere il lisato attraverso la colonna mediante il flusso per gravità mentre si raccoglie il flusso. Caricare la colonna con il flusso raccolto ed eseguirla attraverso la colonna.
- Lavare la colonna con almeno due volumi di letto di resina di tampone di lavaggio MS500 (20 mM di Tris pH 7,5, 500 mM di NaCl, 0,5 mM di TCEP) 5 volte. Raccogliere il flusso di lavaggio in frazioni di 5 mL. Aggiungere 1 μl di ciascuna frazione a 100 μl di reagente Bradford per verificare visivamente la presenza di proteine. Lavare fino a quando le proteine non legate non sono più presenti nell'ultimo lavaggio, aggiungendo ulteriori fasi di lavaggio se necessario.
- Recuperare la proteina eseguendo il tampone di eluizione MS500 (tampone MS500 integrato con 10 mM di glutatione e 10 mM di NaOH) attraverso la colonna, raccogliendo 5 mL di frazioni di eluizione. Verificare la presenza di proteine aggiungendo 1 μL di eluato a 100 μL di reagente Bradford. Interrompere la raccolta delle frazioni quando il reagente di Bradford non indica più la presenza di proteine.
- Eseguire lo scambio del tampone mediante precipitazione e centrifugazione. Far precipitare le proteine aggiungendo 2 volumi di 4 M di solfato di ammonio all'eluato, invertendo delicatamente fino a quando non diventa torbido, quindi centrifugando per 30 minuti a 4 °C e ≥20.000 x g e di nuovo per altri 5 minuti. Rimuovere il surnatante dopo ogni centrifugazione senza disturbare il pellet proteico.
- Risciogliere e conservare le proteine in MS500 integrato con glicerolo al 25%. Conservare i pellet proteici o le proteine in un tampone di stoccaggio a -80 °C.
4. Saggio in vitro di scissione della catena dell'ubiquitina
NOTA: Selezionare la lunghezza della catena dell'ubiquitina e i tipi di legame in base alla specificità del DUB descritta nei rapporti precedenti31 o determinata empiricamente. Se necessario, questo protocollo potrebbe essere utilizzato per testare l'attività del DUB wild-type di interesse su un pannello di catene di ubiquitina disponibili in commercio di lunghezza e tipo di legame definiti. Una quantità di catena di di-ubiquitina di 375-750 ng e una concentrazione di DUB di 1-2 μM possono essere utilizzati come punti di partenza per il test27.
- Preparare 10 tamponi di attivazione DUB (500 mM di Tris-HCl pH 7,5, 500 mM di NaCl e 100 mM di TCEP).
- Per ogni punto temporale per ogni DUB, preparare 10 μL totali di 2 μM di GST-USP27X purificato in 1x tampone di attivazione DUB (miscela DUB). Prepara i master mix e dividili in punti temporali.
- Incubare la miscela DUB per 10 minuti a RT.
- Aggiungere 7 μL di tampone di caricamento SDS-PAGE al tempo 0 prima di aggiungere le catene di ubiquitina per evitare l'avvio della reazione di deubiquitinazione.
- Per ogni punto temporale per ogni DUB, aggiungere 375 ng di catene di di-ubiquitina K-63 diluite in 10 μL di tampone di attivazione DUB 1x. Il volume totale è di 20 μl per reazione.
- Incubare le provette a 30 °C, fermando ogni punto temporale con 7 μL di tampone di caricamento SDS-PAGE.
- Eseguire SDS-PAGE con un gel gradiente al 4%-12% e immunoblot 7,32 per ubiquitina e USP27X per analizzare la variazione della presenza di mono-ubiquitina in punti temporali selezionati.
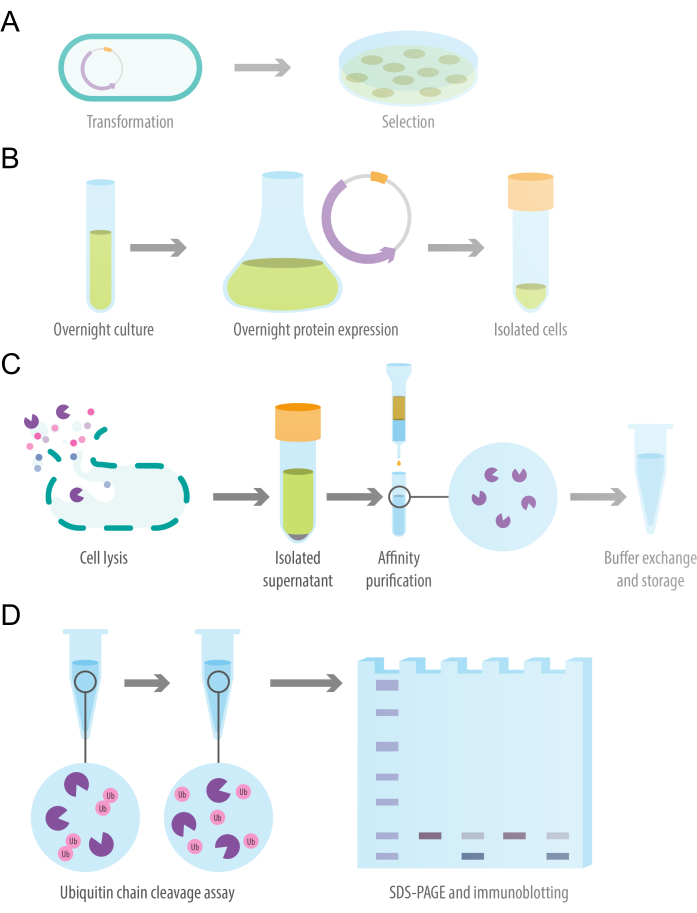
Figura 1: Schema del disegno dello studio. (A) Trasformazione di cellule di E. coli competenti con plasmide di espressione proteica ricombinante. (B) Espressione batterica notturna della proteina deubiquitilasi ricombinante. (C) Purificazione proteica della deubiquitilasi ricombinante utilizzando una colonna di affinità a flusso gravitazionale. (D) Saggio in vitro di scissione della catena dell'ubiquitina per valutare l'attività deubiquitilante. Clicca qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per determinare l'impatto delle varianti associate a XLID105 sull'attività catalitica di USP27X, le proteine USP27X wild-type marcate con GST e le varianti associate a XLID105 F313V, Y381H e S404N USP27X sono state purificate dai batteri. Queste varianti si trovano all'interno del dominio catalitico USP di USP27X (Figura 2A). Poiché in precedenza era stato riportato che USP27X scindeva le catene di ubiquitina K6331, le proteine USP2...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo articolo presenta un protocollo per l'espressione e la purificazione di DUB USP27X ricombinanti e un saggio in vitro di scissione della catena dell'ubiquitina per confrontare l'attività deubiquitilante di USP27X wild-type e proteine varianti associate a NDD. Questo test ha determinato che le varianti associate a XLID105 interrompono l'attività catalitica di USP27X7. Questa intuizione meccanicistica ci ha aiutato a definire XLID105 come un disturb...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questo lavoro è stato finanziato dai fondi di avvio di Sanford Research a FB e dalla sovvenzione NIH R01CA233700 a MJS. L'artwork è stato realizzato da Felipe G. Serrano (www.illustrative-science.com). Ringraziamo il Dr. Greg Findlay (Università di Dundee) per il plasmide GST-USP27X.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Amersham Protran 0.45 NC 200 mm × 200 mm 25/PK | Cytiva | 10600041 | |
| Ammonium sulfate | Fisher Scientific | AC205872500 | |
| Ampicillin | Fisher Scientific | BP1760 25 | |
| Anti- Ubiquitin (Mouse monoclonal) | Biolegend | Cat# 646302, RRID:AB_1659269 | (WB: 1:1000) |
| Anti-GST (Sheep polyclonal) | MRC-PPU Reagents and Services | Cat# S902A Third bleed | (WB: 1:1000) https://mrcppureagents.dundee.ac.uk/ |
| Baffled Culture Flasks 2 L | Fisher Scientific | 10-042-5N | |
| Bradford Reagent | Millipore Sigma | B6916-500ML | |
| Chloramphenicol | Gold Biotechnology | C-105-25 | |
| Complete, Protease Inhibitor tablets | Millipore Sigma | 5056489001 | |
| Econo-Column 1.5 × 5 cm | Bio-Rad | 7371507 | |
| Eppendorf ThermoMixer F1.5 | Eppendorf | 5384000020 | |
| Excel | Microsoft | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Glycerol | Genesee Scientific | 18-205 | |
| Illustrator | Adobe | https://www.adobe.com/products/illustrator.html | |
| Image Studio | LI-COR Biosciences | https://www.licor.com/bio/image-studio/ | |
| Inkscape | Inkscape | https://inkscape.org/ | |
| Invitrogen 4-12% NuPAGE 1mm 12 well gel | Thermo Fisher Scientific | NP0322BOX | |
| IPTG (Isopropyl-b-D-Thiogalactopyranoside) | Genesee Scientific | 20-109 | |
| IRDye 800CW Donkey anti-Goat IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32214 | (WB: 1:10000) |
| IRDye 800CW Donkey anti-Mouse IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32212 | (WB: 1:10000) |
| Isotemp Digital Dry Bath | Fisher Scientific | 88860022 | |
| K63 Di-Ubiquitin | South Bay Bio LLC | SBB-UP0072 | |
| LB Agar | Genesee Scientific | 11-119 | |
| LB Broth | Genesee Scientific | 11-118 | |
| Lysozyme | Gold Biotechnology | L-040-100 | |
| MaxQ 4000 Benchtop Orbital Shaker | Thermo Fisher Scientific | SHKE4000-7 | |
| MES-SDS Running Buffer | Boston Bioproducts Inc | BP-177 | |
| Mini Tube Rotator | Fisher Scientific | 88-861-051 | |
| NuPage LDS Sample buffer 4x | Thermo Fisher Scientific | NP0007 | |
| Odyssey Fc Imager | LI-COR Biosciences | 43214 | |
| PageRuler Plus Ladder | Thermo Fisher Scientific | 26620 | |
| pGEX6P1 human USP27X | MRC-PPU Reagents and Services | DU21193 | https://mrcppureagents.dundee.ac.uk/ |
| pGEX6P1 human USP27X F313V | Addgene | 225715 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X S404N | Addgene | 225717 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X Y381H | Addgene | 225716 | Koch et at 2024 (PMID: 38182161) |
| Pierce Glutathione Agarose | Thermo Fisher Scientific | 16100 | |
| PMSF (Phenylmethylsulfonyl fluoride) | Gold Biotechnology | P-470-10 | |
| Polysorbate 20 (Tween 20) | Fisher Scientific | AC233360010 | |
| Rosetta 2 Competent Cells | Millipore Sigma | 71402-M | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6060 | |
| SmartSpec 3000 | Bio-Rad | 170-2501 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Sodium chloride | Genesee Scientific | 18-216 | |
| Sonifier 250 | Branson | 100-132-135 | |
| Sorvall RC 6 Plus Centrifuge | Thermo Fisher Scientific | 46910 | |
| TCEP (Tris-(carboxyethyl) phosphine hydrochloride) | Gold Biotechnology | TCEP10 | |
| Terrific Broth Powder | Genesee Scientific | 18-225 | |
| Tris Base | Genesee Scientific | 18-146 | |
| XCell SureLock Mini-Cell and XCell II Blot Module | Thermo Fisher Scientific | EI0002 |
Riferimenti
- Morris-Rosendahl, D. J., Crocq, M. -A. Neurodevelopmental disorders: the history and future of a diagnostic concept. Dialogues Clin Neurosci. 22 (1), 65-72 (2020).
- Ebstein, F., Küry, S., Papendorf, J. J., Krüger, E. Neurodevelopmental disorders (NDD) caused by genomic alterations of the ubiquitin-proteasome system (UPS): the possible contribution of immune dysregulation to disease pathogenesis. Front Mol Neurosci. 14, 733012(2021).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annu Rev Biochem. 67 (10), 425-479 (1998).
- Pickart, C. M. Mechanisms underlying ubiquitination. Annu Rev Biochem. 70 (1), 503-533 (2001).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Yau, R., Rape, M. The increasing complexity of the ubiquitin code. Nat Cell Biol. 18 (6), 579-586 (2016).
- Koch, I., et al. USP27X variants underlying X-linked intellectual disability disrupt protein function via distinct mechanisms. Life Sci Alliance. 7 (3), e202302258(2024).
- Hao, Y. -H., et al. USP7 acts as a molecular rheostat to promote WASH-dependent endosomal protein recycling and is mutated in a human neurodevelopmental disorder. Mol Cell. 59 (6), 956-969 (2015).
- Fountain, M. D., et al. Pathogenic variants in USP7 cause a neurodevelopmental disorder with speech delays, altered behavior, and neurologic anomalies. Genet Med. 21 (8), 1797-1807 (2019).
- Santiago-Sim, T., et al. Biallelic variants in OTUD6B cause an intellectual disability syndrome associated with seizures and dysmorphic features. Am J Hum Genet. 100 (4), 676-688 (2017).
- Beck, D. B., et al. Linkage-specific deubiquitylation by OTUD5 defines an embryonic pathway intolerant to genomic variation. Sci Adv. 7 (4), eabe2116(2021).
- Cho, J., Park, J., Kim, E. E., Song, E. J. Assay systems for profiling deubiquitinating activity. Int J Mol Sci. 21 (16), 1-16 (2020).
- Matsui, S., Sandberg, A. A., Negoro, S., Seon, B. K., Goldstein, G. Isopeptidase: A novel eukaryotic enzyme that cleaves isopeptide bonds. Proc Natl Acad Sci USA. 79 (5), 1535-1539 (1982).
- Rose, I. A., Warms, J. V. B. An enzyme with ubiquitin carboxy-terminal esterase activity from reticulocytes. Biochemistry. 22 (18), 4234-4237 (1983).
- Wilkinson, K. D., Cox, M. J., Mayer, A. N., Frey, T. Synthesis and characterization of ubiquitin ethyl ester, a new substrate for ubiquitin carboxyl-terminal hydrolase. Biochemistry. 25 (21), 6644-6649 (1986).
- Pickart, C. M., Rose, I. A. Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides. J Biol Chem. 260 (13), 7903-7910 (1985).
- Hadari, T., Warms, J. V. B., Rose, I. A., Hershko, A. A ubiquitin C-terminal isopeptidase that acts on polyubiquitin chains: Role in protein degradation. J Biol Chem. 267 (2), 719-727 (1992).
- Woo, S. K., et al. Multiple ubiquitin C-terminal hydrolases from chick skeletal muscle. J Biol Chem. 270 (32), 18766-18773 (1995).
- Woo, S. K., et al. Purification and characterization of a new ubiquitin C-terminal hydrolase (UCH-1) with isopeptidase activity from chick skeletal muscle. J Biochem. 121 (4), 684-689 (1997).
- Baek, S. H., et al. Molecular cloning of a novel ubiquitin-specific protease, UBP41, with isopeptidase activity in chick skeletal muscle. J Biol Chem. 272 (41), 25560-25565 (1997).
- Wilkinson, K. D., et al. Metabolism of the polyubiquitin degradation signal: Structure, mechanism, and role of isopeptidase T. Biochemistry. 34 (44), 14535-14546 (1995).
- Hu, M., et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell. 111 (7), 1041-1054 (2002).
- Hu, M., et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14. EMBO J. 24 (21), 3747-3756 (2005).
- Gorka, M., Magnussen, H. M., Kulathu, Y. Chemical biology tools to study deubiquitinases and UBL proteases. Semin Cell Dev Biol. 132, 86-96 (2022).
- Faesen, A. C., et al. The differential modulation of USP activity by internal regulatory domains, interactors, and eight ubiquitin chain types. Chem Biol. 18 (12), 1550-1561 (2011).
- Mevissen, T. E. T., et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell. 154 (1), 169-184 (2013).
- Kwasna, D., et al. Discovery and characterization of ZUFSP/ZUP1, a distinct deubiquitinase class important for genome stability. Mol Cell. 70 (1), 150-164.e6 (2018).
- Hu, H., et al. X-exome sequencing of 405 unresolved families identifies seven novel intellectual disability genes. Mol Psychiatry. 21 (1), 133-148 (2016).
- Hartmann, I. Shaker Orbit - Revolving in Space Around the Samples? Eppendorf Lab Academy. Eppendorf Lab Academy. , at https://www.eppendorf.com/us-en/lab-academy/lab-solutions/others/shaker-orbit-revolving-in-space-around-the-samples (2020).
- Smith, D. B., Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene. 67 (1), 31-40 (1988).
- Ritorto, M. S., et al. Screening of DUB activity and specificity by MALDI-TOF mass spectrometry. Nat Commun. 5, 4763(2014).
- Bustos, F., Bustos, F. Immunoblotting using precast gels. , protocols.io (2024).
- Wingfield, P. T. Overview of the purification of recombinant proteins. Curr Protoc Protein Sci. 80, 6.1.1-6.1.35 (2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon