Method Article
ハイC:ゲノムの三次元構造を研究する方法。
* これらの著者は同等に貢献しました
Erratum Notice
要約
のHi - Cのメソッドは、クロマチンの相互作用(1)の公平な、ゲノムワイドな同定が可能になります。のHi - Cのカップル近接ライゲーションと超並列シーケンシング。結果のデータは、複数のスケールでのゲノムアーキテクチャを研究するために使用することができます:最初の結果は、染色体テリトリー、オープンおよびクローズクロマチンの分離、およびメガベーススケールでのクロマチン構造などの機能を同定した。
要約
染色体の三次元折りに近い空間的近接2月6日に、ゲノムを区分けすると、そのようなプロモーターおよびエンハンサーのような遠くの機能要素を、もたらすことができます。染色体の組織とゲノムの活動との関係を解読することは転写と複製のように、ゲノムのプロセスを理解する上で助けとなる。しかし、ほとんどは染色体が折り畳ま方法については知られている。顕微鏡は、遺伝子座を同時にまたは高解像度での大きな数字を区別することができません。日付に、染色体のコンフォメーションキャプチャ(3C)とその後の適応を用いた染色体の相互作用の検出は、ゲノムワイドな研究は不可能7-10を作り、標的遺伝子座のセットの選択が必要。
我々は、Hi - C、公平な、ゲノムワイドなファッションで長距離相互作用を同定することができる3Cの拡張を開発した。ハイCでは、細胞は、共有結合したDNA -タンパク質の架橋によって互いに結合される相互作用する遺伝子座を引き起こして、ホルムアルデヒドで固定されています。 DNAは、その後、制限酵素で断片化している場合、これらの遺伝子座は、リンクされたままになります。ビオチン残基が5'突出末端は次が記入されているとして組み込まれて、平滑末端ライゲーションは、架橋DNA断片間ライゲーションのイベントに有利な希釈条件下で行われる。核内で相互に近接してもともとのフラグメントのペアに対応ライゲーション産物のゲノムワイドなライブラリで、この結果、。各ライゲーション産物は、接合部の部位にビオチンが付いています。ライブラリが剪断され、接合部はストレプトアビジンビーズでプルダウンしています。精製された接合部は、その後、相互作用フラグメントのカタログで、その結果、ハイスループットシーケンサーを用いて分析することができます。
結果として得られる接触行列の直接分析は、染色体テリトリーの存在と小さな遺伝子が豊富な染色体の選択的結合として、ゲノム構成の多数の機能を明らかにする。アクセス可能な、オープンを含む小さい密集した区画、および活性クロマチンと閉鎖を含む、より緻密なコンパートメント、アクセス不能、および不活性クロマチン領域:相関分析は、ヒトゲノムは、2つのコンパートメントに分離されていることを示している、連絡先の行列に適用することができます。最後に、理論的な導出と計算機シミュレーションとの結合の接触行列のアンサンブルの分析は、、メガベーススケールでのHi - Cは、フラクタル球の立体構造と一貫性の機能を明らかにすることを明らかにした。
プロトコル
このメソッドは、で報告された研究で使用されてリーバーマン-エイデンら 、Science 326、289〜293(2009) 。
I.架橋、消化、DNA末端のマーキング、および平滑末端ライゲーション
- のHi - Cは、すべての3Cベースのメソッドに共通するスレッドであり、細胞、の架橋から始まります。開始するには、2 × 10 7および2.5 × 10 7、哺乳動物細胞、接着または懸濁液のいずれか、および架橋の細胞の間に成長する。 (細胞の架橋の詳細については、を参照してください:11
- ホモジナイザーを用いて550μLlysisbuffer(500μlの10mMトリス- HCl pH8.0の10 mMのNaCl、0.2%IGEPAL CA - 630および50μlのプロテアーゼ阻害剤)で細胞を溶解する。 5,000 rpmでクロマチンをスピンし、500μlの1X NEBuffer 2で2回ペレットを洗浄。
- 、1X NEBuffer 2でクロマチンを再懸濁し、5番号のチューブに分注し、362μlの最終容量に1X NEBuffer 2を追加します。 38μlを1%SDSを追加、10分間65℃で注意深く混合し、インキュベートする。直ちにインキュベーション後に戻って氷中にチューブを置きます。
- 44μlのトリトンX - 100と慎重にミックスを追加することにより、SDSを癒やす。回転しながら37℃で一晩をHindIIIとインキュベートの400台を追加してクロマチンを消化。
- 次のステップは、Hi - Cに固有のものであり、DNAをマークするビオチンと架橋フラグメントの平滑末端ライゲーションを行うことで終わるが含まれます。このステップでは、ライゲーションの接合は後で精製できるようになります。チューブ1は、ビオチン化のステップを受けてはならないし、代わりに別々に保つ必要がありますし、その消化を確保するための3Cコントロールとして機能し、ライゲーションの条件が最適であった。
- 制限酵素断片のオーバーハングを記入し、DNAが残りの4管でビオチンで終わるマーク、1.5μlの10mMのdATPを、1.5μlの10mMのdGTPを、1.5μLの10mM dTTPの、37.5μL0.4mMのビオチン- 14 - dCTPを追加する、とするチューブ2-5を10μl5U/μlクレノウ。注意深く混合し、37℃で45分間インキュベート℃に
- 氷上でチューブをセット。酵素を不活性化するために、チューブ1-5に10%SDSを86μlを加える。 ° Cを正確に30分間65℃チューブをインキュベートし、その後すぐに氷の上に置きます。
- ライゲーションは、架橋断片間ライゲーションのイベントを優先するために非常に希薄な条件で行われる。 [745μlの10パーセント7.61ミリリットルのライゲーションミックスを追加し、氷の上で作業をトリトンX - 100、745μlの10 ×ライゲーション緩衝液(500mMのトリス- HCl、pH7.5、100 mMのMgCl 2、100mMのDTT)、80μlの10 mg / mlのBSA 、80μlの100mMのATPおよび5.96ミリリットルの水] 5つのそれぞれに15 mlチューブの番号。対応する15 mlのチューブにそれぞれ消化クロマチンの混合物を転送する。
- 定期的な3Cライゲーションの場合は、チューブ1に10μlの1U/μlT4 DNAリガーゼを追加。平滑末端のHi - Cのライゲーションの場合は、チューブ2-5を50μlの1U/μlT4 DNAリガーゼを追加。反転でチューブを混合し、16で4時間全5チューブをインキュベート℃に
- 架橋が逆転し、蛋白質は、チューブ当たり50μlの10 mg / mlのプロテイナーゼKを追加し、65℃で一晩チューブをインキュベートすることにより分解されている翌日チューブあたり10 mg / mlのプロテイナーゼKさらに50μLを加え、さらに2時間、65℃でインキュベーションを続ける。
- 室温に反応混合物を冷却し、5 50 mlコニカルチューブに移す。フェノール抽出を行うことにより、これらのチューブでDNAを精製。 10ミリリットルフェノールpH8.0の2分間ボルテックスを追加。 3500 rpmで10分間チューブをスピンし、慎重に新しい50 mlチューブに可能な限り水相のを転送する。
- フェノールのpH 8.0を用いて抽出を繰り返す:クロロホルム(1:1)とエタノールを用いてDNAを沈殿させる。 (DNA精製の 詳細については、を参照してください:11
- エタノールの遠心分離してDNAを沈殿した後、450μlの1 × TE(10mMトリス- HCl pH8.0の、1mMのEDTA)で各DNAペレットを溶解する。 1.7ミリリットルの遠心チューブにDNAの混合物を転送する。
- クロロホルム抽出:精製の別のラウンドは2フェノールを行うことによって実行されます。クロロホルム(1:1)と1分間ボルテックス:500μlのフェノールのpH 8.0を追加します。 14,000 rpmで5分間、チューブを遠心し、新しいチューブに水相を移す。 2回目の抽出、-80、100%エタノールとインキュベート30分の2倍のボリュームをNaOAcの0.1 ×量を追加することでDNAを沈殿℃にした後
- 沈殿したDNAをスピンダウン後、70%エタノールで各DNAペレットを洗浄し、25μlの1 × TEの各DNAペレットを再懸濁します。 1μlの1 mg / mLのリボヌクレアーゼあたりチューブを追加し、37℃で30分間チューブをインキュベートすることにより、存在する可能性のあるRNA℃を低下させるプールチューブ2-5のハイCの内容は、まだ3Cコントロールなどのチューブ1を分離しておく。
- 今はHi - Cマーキングとライゲーション効率を検討する良い機会です。これらのコントロールは、Hi - Cのライブラリが成功するために起こっているかどうかの優れた指標です。
- 図書館の質と量を確認するには、0.8%アガロースゲル上3CとHi - Cのライブラリの両方から1:10希釈を2μlと6μlのアリコートを実行します。 (図2Aを参照してください)
- のHi - CマーキングとHi - Cのライゲーション効率がPCRダイジェストアッセイによって検証されます。成功したフィルインとHindIII部位の結紮は、(AAGCTT)を制限酵素NheI部位(GCTAGC)用サイトを作成します。つの近い制限断片から形成された特定のライゲーション産物は、PCR(として3C 11のテンプレートとして、各ライブラリの0.2μLを用いて増幅される。PCR産物はその後をHindIII、NheIをまたは両方で消化されています。2%ゲル上でサンプルを実行した後、3CとHi - Cのライゲーションのイベントの相対数は、カットとカットされていないバンドの強度を(図2Bを参照)定量することにより推定することができる。
- いくつかの断片が連結されていないでしょう:後でそれらをプルダウン避けるために、T4 DNAポリメラーゼのエキソヌクレアーゼ活性を使用して、これらのunligated末端からビオチンを削除します。
- 非連結DNAの末端にビオチン- 14 - dCTPをT4 DNAポリメラーゼのエキソヌクレアーゼ活性で除去されます。 1μlの10 mg / mlのBSA、10μlの10倍NEBuffer 2、1μlの10mMのdATPを、1μlの10mMのdGTPおよび5台を100μlの総体積で、T4 DNAポリメラーゼでのHi - Cライブラリの5μgを混合し、インキュベート12で混合℃で2時間。可能であれば、複数の5μgの反応が実行されます。
- 反応は、2μlの0.5 M EDTA pHが8.0を加えることによって停止されます。
- DNA、フェノールpH8.0のを浄化するには:クロロホルム(1:1)抽出、エタノール沈澱に続いて行われます。
- 上清を捨て、DNAペレットを100μlの水の全体積中に再懸濁させ、プールされます。
II。せん断とサイズの選択
- ハイスループットシーケンシングのためのビオチン化DNAに適したように、DNAはCovaris S2機器(デューティサイクル5、震度5、サイクル/バースト200、時間4サイクルの場合は60秒)で300〜500塩基対の大きさに剪断されている必要があります。
- せん断DNA末端を修復するには、14μlの10 ×ライゲーション緩衝液、14μL2.5mMのdNTPミックス、5μlのT4 DNAポリメラーゼ、5μlのT4ポリヌクレオチドキナーゼ、1μlのKlenow DNAポリメラーゼと1μlの水を加える。室温で30分間インキュベートする。
- インキュベーション後、メーカーの推奨にしたがってDNAを精製するためにキアゲンのアプライのカラムを使用してください。 15μlの1Xトリス低EDTA(:10mMのトリスpH8.0、0.1mMのEDTA TLE)で2回DNAを溶出する。その後、10μlの1mMのdATPを、2μlの水と3μlクレノウ(エキソ - )を5μlの10倍NEBuffer2を追加することによって、エンド修理DNAの3'末端にdATPを取り付けます。 37℃で30分間反応をインキュベート℃に
- Klenow断片を不活性化するために、氷上で反応° C、その後涼しい65℃20分間反応をインキュベートする。 speedvacを使用し、20μlのに反応容量を減らす。
- 次に、1 × TAE、1.5%アガロースゲルでDNAをロードし、80 90Vで3.5時間実行されます。 SYBRグリーンでゲルを染色した後、DarkReader上でDNAを可視化する。 300および500塩基対との間で物品のDNA断片は、ゲルの重量に応じて2から4列を使用して、キアゲンのゲル抽出キットでそれらを浄化する。 50μlの1X TLEでDNAを溶出する。
- QIAクイックカラムから溶出液を組み合わせると1x TLEと300μlのために、最終容量を持ち出す。最後に、量子ビット蛍光光度計を使用してクワント- ITアッセイによるDNA濃度を決定し、DNAの合計量を計算する。
III。ビオチンは、プルダウンやペアエンドシーケンシング
- プロトコルのこのセクションでは、ライゲーションの接合は、ペアエンドシーケンシングによりクロマチンの断片を相互作用の効率的な同定を可能にする、DNAのプールから精製されています。 DNAのLoBindチューブ内のすべての後続の手順を実行します。
- プルダウン液150は、400μlのトゥイーンバッファー(5 mMトリス- HCl pH 8.0に、0.5mMのEDTA、1MのNaCl、0.05%ツイーンTB)で2回磁性ストレプトアビジンビーズを再懸濁し、洗浄することにより、ビオチンのためにビーズを準備します。
これらのおよび将来の洗浄は、次の5つのステップで構成されています:- ビーズにバッファーを追加します。
- 新しいチューブに混合物を転送する
- 室温で3分間、サンプルを回転させる
- 磁性粒子の集配信装置を使用してビーズを取り戻す
- 上清を取り除く
- 300μlの2倍なしトゥイーンバッファー(2 × NTB:10mMトリス- HCl pH 8.0に、1mMのEDTA、2 M NaCl)中にビーズを再懸濁し、300μlのハイCのDNAと結合。のHi - C DNA標識されたビオチンは回転で15分間室温で混合物をインキュベートすることにより、ストレプトアビジンビーズに結合することができます。
- 磁性粒子のconcentrとストレプトアビジンビーズを結合したDNAを回収するator、そして上清を取り除く。 100μlの1 ×ライゲーション緩衝液に続く400μlの1X NTB(5mMのトリス- HCl pH 8.0に、0.5mMのEDTA、1 M NaCl)を、中のビーズを洗浄する。 50μlの1 ×ライゲーションバッファーでビーズを再懸濁し、新しいチューブに混合物を移す。
- イルミナペアエンドシーケンス用DNAを調製するために、ビオチン、前の手順2.6に算出した、プルダウン、および持っているのHi - CのDNA量を推定するために20でそれを分割するための入力として使用するDNAの合計量を取るプルダウンとライゲーション用に利用できるようになりました。ライゲーションに使用できるのHi - CのDNAのμgのあたりイルミナペアエンドアダプタの6ピコモルを追加。 DNAにアダプターを連結するために1200単位のT4 DNAリガーゼを使用してください。室温で2時間インキュベートする。
- のHi - CのDNA結合ビーズを回収し、400μlの1 × TBで二回ビーズを洗浄することにより、非結紮ペアエンドアダプタを取り外します。
- 200μlの後、50μlの1X NEBufferで次に2,3 ...、200μlの1XのNTBでビーズを洗浄する。最後の洗浄の後、50μlの1X NEBuffer 2でビーズを再懸濁し、新しいチューブに移す。
- シークエンシングのために十分なPCR産物を生成するために必要なサイクル数を決定するために、6、9、12または15サイクルで4つのテストのPCR反応をセットアップします。 (PCR増幅の詳細については、を参照してください:12、5%ポリアクリルアミドゲルおよびSYBR Green染色でPCR反応を実行するスプリアスバンドと400の間にスメアの存在の有無を確実にすることによって、最適なサイクル数を決定する - 。アダプターへのライゲーション後のせん断製品の長さは600塩基対、。
- PCRサイクルの最適な数を持つ大規模なPCRでのHi - C -ライブラリの結合ストレプトアビジンビーズの残りの部分を増幅する。別々のウェルからのプールのPCR産物を、ビーズを回収する。ゲル上で実行し、製造元の推奨にしたがって1.8倍のボリュームAmpureビーズでPCR産物の残りの部分を浄化するために別の大規模なPCR産物の1%をキープ。
- 50μlの1X TLEバッファーでDNAを溶出し、5%ポリアクリルアミドゲル上の元のPCR産物の1%のアリコートにAmpureビーズ精製PCR産物の1%を比較し、PCRプライマーの成功の除去を確保。
- 我々はまた、ハイCライブラリのクローニング1μlをお勧めし、サンガーシーケンシングを使用して、約100クローンの製品を決定する。これは、ハイCはPCR混合物中の読み取り配列可能の相対的な数を把握できるようになります。典型的な結果を得るためには、図3Bを参照してください。
- イルミナペアエンドシーケンスとシーケンスのHi - Cのライブラリを。独立して相互作用するクロマチンの断片を識別するために、MAQ(http://maq.sourceforge.net/)を使用してそれぞれの端に合わせます。
IV。代表的なのHi - Cの結果
- のHi - Cプロトコルは、品質管理基準を技術的にも実行されると考えることができるときは、次の結果が期待されています。
- 品質管理の手順は、両方3CとHi - Cのライブラリが10 KBを超えるなど、かなりタイトなバンドを実行することを明らかにする必要があります。 DNAのスメアが悪いライゲーション効率を示します。 3Cテンプレート(図2a参照)に比べて一般的に、ライゲーション効率は、Hi - Cのライブラリでは若干低いです。
- のHi - Cマーキングとライゲーション効率が3Cプライマーを用いて生成されたPCR産物の消化によって推定することができる。 3C接合は、NheI部位によってHindIIIで切断し、されていません。逆は、Hi - Cの接合についても同様です。このPCRダイジェストアッセイは、Hi - Cのアンプリコンの70%が(図2Bを参照)ライゲーションジャンクションのマーキング効率的に確認し、HindIIIでのNheIで切断し、されていないことを。示しています。
- シーケンスリードの分析は、比較して、ランダムに生成するためにHindIII制限部位にかなり近い位置合わせ、青と赤の線で示されている、染色体内および染色体間の両方の相互作用から読み取ることが表示されます(図3Aを参照)緑色に示すように、読み込みます。
- 成功した実験では、配列可能読み取りのペアの55%は染色体間相互作用を表しています。 (図3Bを参照してください)15パーセントは、離れてフラグメントの間に染色体内の相互作用未満20 kbを表し、30%以上離れて20キロバイトである染色体内読みペアです。このディストリビューションは、品質管理の形態として、前のハイスループットシーケンシングに、サンプリングされることがあります。約100クローンのクローニングとサンガーシーケンシングは、通常は十分です。
- クロマチンの相互作用は、x軸とy軸はゲノム順に遺伝子座を表し、各ピクセルは、それらの間の観察された相互作用の数を表すヒートマップ、として視覚化することができます。一般的に、非常にリニアなゲノムで互いに近い位置にあるDNA断片は、傾向が互いに頻繁にやり取りする必要があります。これは、著名な対角線として染色体内ヒートマップ(図4Aを参照)で見られます。
- 以下の結果は、ゲノムの組織のさまざまなレベルを明らかにするためにデータを分析のいくつかの方法を示します。接触probabilitのプロットY対ゲノムの距離が(図5aを参照)の接触の確率が最終的にプラトーに達し、ゲノム距離の関数として減少を示しています。すべての距離では、実線で示すように、染色体内の相互作用は、、破線で表される、染色体間相互作用の相対的な豊かされています。これは、直接染色体テリトリーの存在を意味する。
- 染色体のすべてのペア間で染色体間の接触の観察/期待値を計算すると、特定の染色体のペア間の選択的結合を明らかにする。小さな遺伝子が豊富な染色体が優先的に鮮やかな赤い色(図5bを参照)で示される、互いに相互作用する。
- 個々の染色体を調べることもできます。生のヒートマップは、観察/期待されるヒートマップで、その結果、遺伝子座のペア間のゲノムの距離を考慮に期待されるヒートマップを使って調整することができます。次に、相関行列は、観測/期待されるヒートマップの行や列を相関させることによって製造することができる。相関分析を使用して、それは2つの区画にヒトゲノムの偏析ことが示されている。これは、相関ヒートマップで格子縞のパターン(図4A - Dを参照)によって示されている。 (ハイ- Cデータの分析の詳細については、を参照してください:1。
- のHi - Cのデータを使用して、新しい洞察がメガベースのスケールでのクロマチンフォールディングに得られました。ポリマー凝縮の古典的なモデルは、均衡の小球にそのクロマチンパックを示唆している。距離の関数としての接触確率をプロットすると傾き(図6Aを参照)が約-1のゲノム距離とべき乗則としてその接触確率の尺度を示しています。これは平衡球の動作と一貫性ではなく、フラクタル球(図6B参照)と呼ばれる別の構造への期待と一致していません。
- ここでは、2つの球状構造が示されています。着色は青からシアン、緑、黄色、オレンジ、そして赤(図6C上部を参照してください)まで、1つのエンドポイントからの距離に相当する。平衡球とは異なり、フラクタル球はもつれを欠いている。フラクタル球では、輪郭に沿って近くに遺伝子座は、単色のブロックが存在する(図6Cの真ん中を参照)につながる、3Dに近くなる傾向があります。そのようなブロックは、平衡小球(図6C下部を参照)に記載されていません。
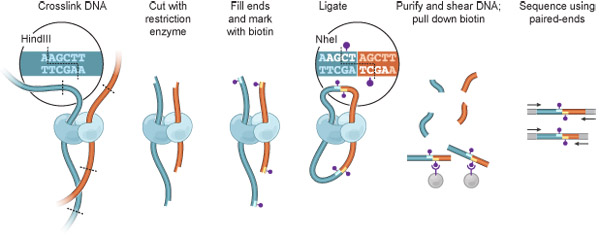
図1はHi - Cの概要。細胞は、空間的に隣接するクロマチンのセグメント(:;タンパク質、このような相互作用を媒介することができる、ライトブルーとシアンに示されている赤濃い青、DNA断片)との間に共有リンクでは、その結果、ホルムアルデヒドでクロスリンクされています。クロマチンは、制限酵素で消化され(ここで、HindIIIで、制限部位:破線は、挿入図参照)。結果として得られる粘着末端がビオチン化されそのうちの一つヌクレオチド、(紫のドット)が入力されています。ライゲーションは、分子内ライゲーションのイベントを好む非常に希薄な条件の下で実行され、HindIII部位は失われ、NheI部位は、(挿入図)が作成されます。 DNAを精製し、剪断、およびビオチン接合は、ストレプトアビジンビーズを用いて単離されています。相互作用フラグメントはペアエンドシーケンシングによって識別されます。

図2のHi - Cのライブラリの品質管理。 ()3CコントロールとHi - Cのライブラリの増加量を0.8%アガロースゲル上で分離された。両方のライブラリは、10 KBを超えるというタイトなバンドとして実行されます。ハイCライブラリの標準的なライゲーション効率が3Cテンプレートで観察されるものよりわずかに低く、およびHi - Cのレーンの塗抹標本で示されます。 (B)PCRダイジェストコントロール。つの近いの断片によって形成されたライゲーション接合が標準3C PCR条件を用いて増幅される。のHi - Cのライゲーション産物は、ライゲーション部位の消化によって、従来の3Cで製造されたものと区別することができる。のHi - Cの接合は、NheIのではなく、HindIIIで切断し、逆に3C接合についても同様です。のHi - Cアンプリコンの70%は、ライゲーションの接合部のマーキング効率的に確認し、NheIをでカットされた。二つの複製は、信頼性の高い定量性を確保するために行われた。

図3。のHi - Cは、品質コントロールをお読みください。 ()染色体内(青)と染色体間の両方に対応するフラグメントから読み込みます(赤)ランダムに生成されたリード(緑)と比較して、相互作用はHindIII制限部位にかなり近い位置に合わせます。読み取り染色体内および染色体間の両方は、高原が〜500bpの距離に達するまでの曲線は、HindIII部位からの距離が増加するにつれて急速に減少読み取ります。これは、配列決定のために使用される最大フラグメントサイズに対応しています。 (B)一般的に、配列可能読み取りのペアの55%は染色体間相互作用を表しています。 15パーセントは、フラグメント未満20キロバイトの間に染色体内の相互作用を表す離れて、30%以上の20キロバイトが離れている染色体内読みペアです。このディストリビューションは、品質管理の形態として、前のハイスループットシーケンシングに、サンプリングされることがあります。約100クローンのクローニングとサンガーシーケンシングは、通常は十分です。

図4相関分析は、核が2つのコンパートメントに分離されていることを示しています。 ()14番染色体上の染色体内の相互作用に対応するヒートマップ。各ピクセルは、1メガビットの軌跡と別の1 MBの遺伝子座の間のすべての相互作用を表し、強度は、読み取りの合計数(範囲:0から200ヒット)に対応しています。目盛りは10 MBごとに表示されます。ヒートマップは、強烈な対角と大きなブロックの星座の形で部分構造を示す。 (染色体14は、末端動原体であり、短い腕は表示されません。)指定されたゲノムの距離での遺伝子座のペアの平均接触確率を計算するためのHi - Cのデータセットを使用して、期待値の行列が生成されます(B)、対応がどうなるかにない長期的な構造がなければ観察。これら二つの行列の商は、観測された/予想される行列です(C)枯渇は、青と赤の[範囲:5(赤)〜0.2(青)]の濃縮に示されている場所。ブロックパターンは、より明白となります。 14番染色体に沿って遺伝子座の各ペアの染色体内相互作用プロファイルの間に:相関行列(D)は、相関関係は[-1(青)〜1(赤)の範囲]を示しています。印象的な格子縞のパターンは、染色体内の2つのコンパートメントの存在を示している。

図5。染色体テリトリーの存在と組織。 (A)に接触する可能性は最終的には〜90MB(青)でプラトーに達し、1番染色体上のゲノム距離の関数として減少する。染色体間の接触のレベルは、(黒ダッシュ)染色体の別のペアごとに異なります。1番染色体上の遺伝子座は10番染色体上の遺伝子座(緑のダッシュ)と21番染色体上の遺伝子座(赤ダッシュ)と相互作用する可能性が最も低いと相互作用する可能性が高いです。染色体間相互作用は、染色体内の相互作用の相対的な枯渇している。 (B)染色体のすべてのペア間で染色体間の接触の数観測/期待。赤は、濃縮を示し、青は空乏[範囲:2(赤)〜0.5(青)]を示しています。小さな、遺伝子が豊富な染色体は互いにより多くの相互作用する傾向がある。

図6。クロマチンの局所パッキングは、フラクタル球の動作と一貫性があります。 (A)ゲノム(青)の平均値、ゲノム距離の関数として確率にお問い合わせください。著名なべき乗スケーリングは500キロバイトと-1.08の傾き(シアンに示すようにフィット)と(斜線領域)7MBの間に見られる。 (B)平衡(赤)とフラクタル(青)小球の距離の関数としての接触確率のシミュレーション結果。フラクタル球のためのスロープは、私たちの新たな理論的な予測1を確認し、非常に近い-1(シアン)である。平衡小球のためのスロープは、事前の理論的な予想と一致している、-3 / 2です。平衡小球のためのスロープをHi - Cのデータでは見られないのに対し、フラクタル球のためのスロープは、密接に、ハイCの結果で観察される斜面に似ています。 (C) トップ:4000モノマー長く、ポリマー鎖が折りたたま。発色が青からシアン、緑、黄色、オレンジ、そして赤に至るまで、1つのエンドポイントからの距離に相当する中東:私たちのアンサンブルから引き出されたフラクタル球の典型的な例。フラクタル球はもつれを欠いている。 。平衡小球:輪郭に沿って近くにある遺伝子座は、表面上と断面下部で明らかになる大規模な単色ブロックの存在につながる、3Dに近くなる傾向があります。構造は非常に絡み合いです。等高線に沿ってすぐ近くにある遺伝子座(同じような色)は、3Dで近くである必要はない。
ディスカッション
私たちは、公平な、ゲノムワイドな方法で、クロマチンの相互作用をマッピングすることで、ゲノムの3次元構造を研究する方法を提示する。離れて前の仕事からこの技術を設定するどのような最も重要な実験的なステップは、 - 平滑末端ライゲーションの前に架橋された断片の末端制限でビオチン化ヌクレオチドの取り込みです。この手順を実行すると、正常にすべてのライゲーション接合の深いシーケンシングを可能にし、ハイCにその範囲とパワーを与えます。
読み取りの数は最終的に相互作用マップの解像度を決定します。ここでは、ヒトゲノムのための1 MBの相互作用マップが〜3000万配列可能読み取りを使用して表示されます。 nの係数が"万能"の解像度を増加させるために、読み取りの数は、nは 2倍に増やす必要があります。
のHi - Cの技術は容易にそのようなハイブリッドライブラリの生成(ゲノムの特定の部分を対象とする)とライゲーション後のクロマチン免疫沈降した後、キャプチャのような他の手法、(特定のタンパク質に関連付けられている地域のクロマチン環境を調べるために)と組み合わせることができる。
開示事項
謝辞
APエイデン、XRバオ、M.ブレナー、D.ガラ、W.絡まる、A. Jafferさん、A.メルニコフ、A.ミーレ、G. Giannoukos、C. Nusbaum、AJM Walhout、我々は議論やコードのためにA. Kosmrljに感謝、L.ウッド、とK.ゼリドビッチ議論のための、可視化とヘルプとL.ガフニーとB.ウォン。
ファニーメイとジョンヘルツ財団大学院奨学金、国防科学と工学大学院生フェローシップ、NSF大学院生フェローシップ、国立宇宙生物医学研究所、およびnoを付与することによってサポートされています。米国立ヒトゲノム研究所(NHGRI)(EL)からT32 HG002295、i2b2(生物学およびベッドサイドの統合のための情報)、ブリガムアンドウィメンズ病院(LAM)における生物医学コンピューティングのためのNIH -サポートされているセンター、助成金がない。 NHGRIからHG003143、とケック財団著名な若手学者賞(JD)。生とマッピングされたのHi - Cの配列データは、GEOのデータベース(で寄託されているwww.ncbi.nlm.nih.gov/geo/ )、無加盟。 GSE18199。追加の視覚エフェクトがご利用いただけますhttp://hic.umassmed.edu 。
資料
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
参考文献
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved