このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ブラッドフォードプロテインアッセイの線形化
要約
迅速かつ便利なブラッドフォードアッセイによるタンパク質定量の精度と感度は本質的な非線形性によって危険にさらされます。我々は、大幅に精度を向上させる簡単な線形化法を示して10倍程度アッセイの感度が向上し、大幅な洗剤による干渉を低減します。
要約
ブラッドフォードクマシーブリリアントブルーアッセイにおけるタンパク質のマイクログラム量の決定は、590nmでの吸光度を測定することによって達成される。この最も一般的なアッセイは、生化学的測定値の正規化の目的のために、細胞ライセート、細胞の分画、または組換えタンパク質サンプルの迅速でシンプルなタンパク質の定量が可能になります。しかし、本質的な非線形性は、この方法の感度と精度が低下します。それは標準的なアッセイ条件下で、590 nmと450 nmの吸光度測定の比はタンパク質濃度と厳密に線形であることが示されている。このシンプルな手順では、精度を向上させ、ダウンウシ血清アルブミンの50ngの10倍、許可定量化に関するアッセイの感度を向上させます。さらに、一般的に細胞ライセートを作成するために使用される界面活性剤によって導入された干渉が大幅に新しいプロトコルによって低減されます。線形方程式は、質量作用に基づいて開発され、ベールの法則は完璧に実験データをフィット。
プロトコル
Bradfordタンパク質のキャリブレーションのグラフの線形化:
一般的にブラッドフォードアッセイ1として知られているクマシーブリリアントブルータンパク質アッセイは、広くために迅速かつ便利なプロトコルだけでなく、その相対的な感度で使用されています。残念なことに、タンパク質濃度の広い範囲(図1)上の曲率の大きい程度がある。そのため、比較的高いタンパク質濃度の唯一の狭い範囲、20〜10 mg / mlのBSAは、次により良い線形回帰(図1、緑)に適合キャリブレーショングラフ、のために使用されます。しかし、非線形性が大きな誤差を避けるためにキャリブレーションのグラフの限られた範囲内に未知のサンプルのタンパク質濃度を必要とし、それも限られた範囲内で精度が低下します。非線形性は、タンパク質のマイクログラムの量が利用できない、特に深刻な問題を提示し、それは多くの場合、未知のサンプルの複数の希釈を必要とします。
オリジナルのブラッドフォードの論文で述べたように、"非線形性の源は、染料の2つの異なる色形のスペクトルに重なりがあるので。試薬自体にある"1。実際には、クマシーブリリアントブルー色素の3つの形式は、アッセイ2の通常の酸性pHでの酸塩基平衡状態にある。赤、青、緑のフォームは、470でそれぞれ590と650nm、(図2)吸光度の最大値を持っている。青は激しく594 nmの3、4(図2)で光を吸収する複合体を形成、タンパク質を結合するフォームです。ブラッドフォードはまた、1(図3)"より多くの色素がタンパク質に結合されている試薬のバックグラウンド値が継続的に減少している"と指摘した。したがって、我々はタンパク質の量を増やすとして590nmでの背景の削減を計算しようとしたタンパク質 - 色素複合体が吸収しない450nmの、吸光度の変化を測定することによって、追加されます。我々は、部分的にその減少の背景を発見ではなく、完全に、非線形性のためのアカウント(図4)。
我々は、その後、タンパク質-色素結合が平衡5にあるように、複合体形成だけではなく、自由なタンパク質の濃度だけでなく、無料の染料のそれに依存するため、遊離色素濃度の減少は、線形応答の別の歪みを生成するという仮説を立てた(図5)。考慮フリー染料の変数濃度に関連した両方の問題を踏まえ、我々はタンパク質濃度と吸光度測定、450 nmの(図6)以上590nmの比の間に直線関係を記述する数学的方程式を開発した。理論的および実験的研究の詳細な説明は、アナリティカルバイオケミストリー(Analytical Biochemistry)6の私たちの1996年出版で見つけることができます。数学的な方程式は、実験的にテストされ、全体のタンパク質濃度の範囲(図7)で直線の検量線が得られることがわかった。また、式はY軸切片6の正しいpH依存性値の独立した決定によっても検証されました。
改良されたブラッドフォードプロテインアッセイのための詳細なプロトコル、マイクロプレート吸光度リーダーを使用して:
- 標準、ウシ血清アルブミン0.1 mg / mlのストック溶液を調製。他の標準を選択しますが、同じ規格が全ての実験で使用する必要があることに注意してくださいすることができます。
- 脱イオン水で未知サンプルを希釈する。 5-50μg/ mlのを目指しています。アッセイの直線範囲のための明白な限界がないのでまだ、高いか低いタンパク質濃度は、許容範囲です。しかし、測定は、吸光度リーダーの直線範囲内でなければなりません。
- ブラッドフォード試薬(Bio - Rad社製)2.5倍の脱イオン水で希釈してください。
- 3つのウェルにBSAの原液(0-5μgのBSAの検量線を作成する)の0および10〜50μlを添加します。 100μl/ウェルを達するために脱イオン水で補う。
- 別の井戸では、三連で未知のサンプルの100μlを加える。未知のサンプルのいくつかの濃度は、精度を高めるために使用されることがあります。
- すべてのウェルに希釈したBradford試薬100μLを加える。総ボリュームは十分に200μl/です。
- 少なくとも5分ではなく、発色のための60以上の分を待ちます。
- ブランクは、脱イオン水(とではないだけでなく、ゼロタンパク質の色素)を200μlにする必要があります 。
- 590nmで450nmの吸光度を測定します。
- 590nmで正味吸光度値を割って、450 nmでのキャリブレーショングラフを準備します。ゼロタンパク質(染料のみ)の値をデータ点(図8)として含まれるべきであることに注意してください。
- 検量線(図9)の線形方程式に基づいて、未知サンプルの濃度を計算する。
代表的な結果:
590nmの吸光度値の比は、450 nmの上に590nmの単一波長で吸光度とは異なり、線形wはi番目のタンパク質濃度(図10)。
未知試料のタンパク質濃度は、単に検量線(図10、方程式)の線形方程式を用いて計算することができます。
しかし、精度を高めるには、いくつかの未知のサンプルの希釈液を測定して得られる。この目的のために、二つのグラフを用意。最初はX軸(図11、右)上μgのタンパク質を持つ標準のためのキャリブレーショングラフです。二つ目のグラフはX軸(図11、左)の元の未希釈サンプルの液で、未知のサンプル用です。染料のみの値は両方のグラフ(図11)に含まれるべきです。未知試料のタンパク質濃度は、未知試料と標準(図11、方程式)の斜面を分割することによって導出されます。
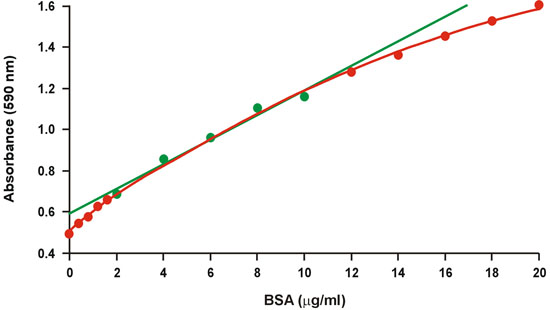
図1従来のブラッドフォードのキャリブレーショングラフ。直線性の範囲は、緑の記号で表されます。

図2。タンパク質-色素複合体の単独で色素のスペクトル。

図3。タンパク質-色素平衡。
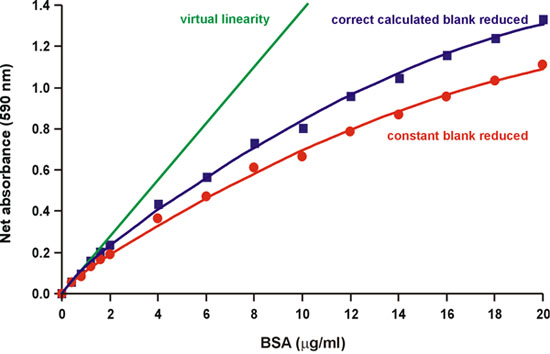
図4。正しい計算されたバックグラウンドの低減は、部分的に非線形性を減少させる。
図5。タンパク質-色素平衡。

図6。ブラッドフォードタンパク質アッセイの線形化のための数学的基礎。
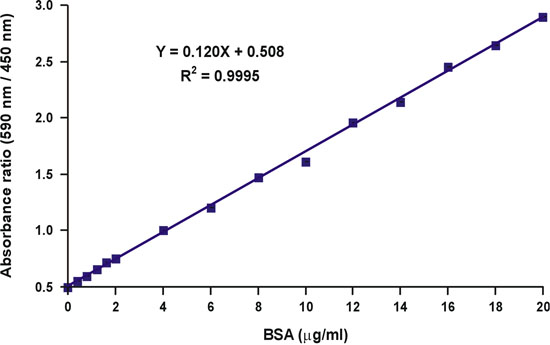
図7。ブラッドフォードのキャリブレーションのグラフの線形化。

図8。吸光度比の値の計算。

図9。線形ブラッドフォードのキャリブレーショングラフ。
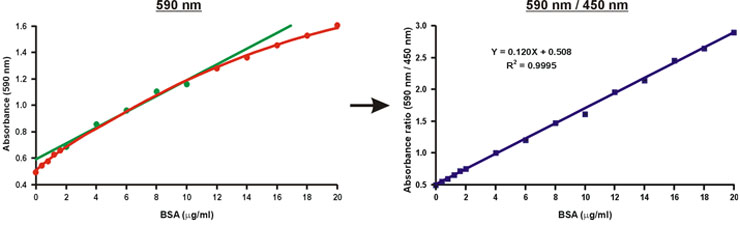
図10ブラッドフォードのキャリブレーションのグラフの線形化。
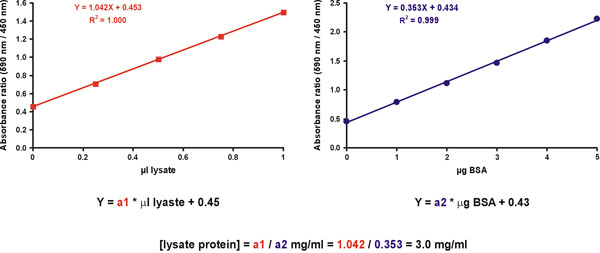
図11。未知のサンプル濃度の計算。
ディスカッション
ブラッドフォードタンパク質アッセイは、原因で、パフォーマンスと相対感度のその使いやすさに人気があります。プロトコルによって得られた距離、タンパク質全体の濃度以上の線形化は、未知のサンプルは、キャリブレーションのグラフの範囲内にする必要がないようにここでさらに、アッセイを簡素化発表。
重要なのは、改良されたプロトコルでは、さらにオリ?...
謝辞
本論文では、ここで説明する独自の研究をホストする故博士Zvi Selinger、の記憶に捧げられています。
資料
| Name | Company | Catalog Number | Comments |
| Bradford reagent | Bio-Rad | 500-0006 | |
| Bovine Serum Albumin | Amersco | 0332 | |
| Multiplate absorbance reader | BioTek | Synergy2 |
参考文献
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie blue G. Anal Biochem. 209, 258-266 (1993).
- Chial, H. J., Splittgerber, A. G. A comparison of the binding of Coomassie brilliant blue to proteins at low and neutral pH. Anal Biochem. 213, 362-369 (1993).
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Anal Biochem. 151, 369-374 (1985).
- Congdon, R. W., Muth, G. W., Splittgerber, A. G. The binding interaction of Coomassie blue with proteins. Anal Biochem. 213, 407-413 (1993).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: theoretical and experimental studies. Anal Biochem. 236, 302-308 (1996).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved