このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
in vivoおよびアダプター、クラスリンの相互作用のin vitro試験
要約
クラスリン依存性エンドサイトーシスは、貨物の選択とクラスリン被覆のアセンブリを調整するアダプタータンパク質に依存します。ここではモデルとして酵母エンドサイトーシスのアダプタータンパク質のSla1pを使用してアダプタクラスリンの物理的相互作用および生細胞イメージングのアプローチを研究するための手順を説明します。
要約
主要なエンドサイトーシス経路は、細胞表面からの輸送貨物は1-6エンドソームにいるクラスリン被覆小胞(CCVS)の形成を開始します。 CCVSは、そのクラスリンコート小胞の膜の多面体格子によって区別し、機械的な足場として機能している。クラスリンコートは個々のクラスリンtriskelionの複数形、三の重三軽鎖サブユニット7,8から構成されるクラスリンの可溶性形態から小胞形成の間に組み立てられます。三脚ともえ紋が直接細胞膜に結合する能力を持っていないため、クラスリン結合アダプタは、脂質および/ または膜タンパク質9で会合を介して膜に成形クラスリン格子をリンクするために重要です。アダプタには、パッケージの膜貫通タンパク質のような受容体などの貨物、、とお互いに、CCVの形成機構9の他のコンポーネントとやり取りすることができます。
20以上のクラスリンアダプターは、記載されているいくつかは、クラスリン依存性エンドサイトーシスに関与していると他の人は、トランスゴルジネットワークに局在または9をエンドソーム。 HIP1R(酵母Sla2p)を除いて、すべての既知のクラスリンアダプターは、クラスリン重鎖9のN末端-プロペラドメインに結合する。クラスリンアダプターの構造化されていない柔軟なリンカーで接続された折りたたまれたドメインで構成されるモジュラータンパク質である。これらのリンカー領域内で、短い結合モチーフはクラスリンのN -末端ドメインまたは小胞の形成機構9の他のコンポーネントとの相互作用を媒介する。二つの異なるクラスリン結合モチーフが定義されています:クラスリンボックスとW -ボックス9。コンセンサスクラスリンボックスシーケンスはもともとLとして定義されていた[L / I] [D / E / N] [L / F] [D / E] 10しかし、変種、その後11を発見されている。 W -ボックスは、シーケンスPWxxW(xは任意の残留物である)に準拠しています。
Sla1pは(アクチン結合タンパク質- 1で合成致死)もともとアクチン関連タンパク質として同定し、酵母細胞12のエンドサイトーシスのサイトで通常のアクチン細胞骨格の構造とダイナミクスのために必要ですれました。 Sla1pもNPFxDエンドサイトーシスのソートの信号を結合し、NPFxD信号13,14を担持する貨物のエンドサイトーシスに重要である。最近では、Sla1pはクラスリンボックス、LLDLQに似てモチーフを介してクラスリン結合することが実証された、バリアントクラスリンボックス(VCB)と呼ばれ、エンドサイトーシスのクラスリンアダプター15として機能する。さらに、Sla1pは生細胞の蛍光顕微鏡の研究16におけるエンドサイトーシスのコートに広く使用されているマーカーとなっています。ここでは、アダプタクラスリンの相互作用の研究のためのアプローチを記述するモデルとしてSla1pを使用してください。我々は、生細胞の蛍光顕微鏡に焦点を当てる、ダウンGST -プル、および共免疫沈降法。
プロトコル
1。 SLA1遺伝子にGFPのタグと選択マーカーの取り込み
SLA1遺伝子のオープンリーディングフレーム(Sla1p C -末端)の3'末端に直接GFPのタグを融合させると同時に、E.との遺伝子をマークするためにLongtine法17を適用するG418選択を可能にする大腸菌館r遺伝子。
- フォワード、5' - CA AGG CAA GCC AAC ATA TTC AAT GCT ACT GCA TCA AAT CCG TTT GGA TTC CGG ATC:鋳型としてプラスミドpFA6a - GFP(S65T)- kanMX6 18および以下のプライマーを用いたPCRによりDNA断片を生成するCCC GGG TTA ATT AA - 3'、およびリバース、5' - CA TAT AGC TTG TTT TAG TTA TTA TCC TAT AAA ATC TTA AAA TAC ATT AAT GAA TTC GAG CTC GTT TAA AC - 3'。下線の配列は、直前の(フォワードプライマー)とストップコドン(リバースプライマー)を以下のSLA1遺伝子の特定のセグメントに対応しています。 DNA精製に続いてアガロースゲル電気泳動によりPCR産物を分離する。
- 50ミリリットル文化S.を準備出芽酵母 SEY6210株(MAΤαURA3 - 52、LEU2 - 3、112 HIS3 -Δ200、TRP1 -Δ901、LYS2 - 801、SUC2 · -Δ9GAL - MEL)YPD 19は 、初期対数増殖期(OD 600 = 0.2〜0.6)で成長。 2000 × gで3分間、室温でスピン、滅菌水で2回洗浄。
- 酢酸リチウム手順20を用いて細胞に、ステップ1で得られたPCRフラグメントを変換します。 1ミリリットルYPD、30で4時間インキュベートしますシェーカーにCを追加し、1 mlの滅菌水で細胞を洗浄。
- 2〜3日間30℃でインキュベートしてG418耐性形質転換体、を選択するためにYPD - G418プレート上で細胞を広げる。 YPD - G418プレート上にコロニーとストリーク、それらを選択してください。
- コロニーPCRを用いて、GFP館モジュールが正しく相同組換えによりSLA1遺伝子配列に統合された形質転換体を識別する。 SLA1オープンリーディングフレームとGFP館モジュール内のアニールその逆方向プライマー内でアニールし、そのフォワードプライマーを使用してください。 PCR産物が予想される大きさを持っているし、シークエンシングにより確認するコロニーを特定する。
- 彼らはGFP蛍光を持って確認するために蛍光顕微鏡でコロニーを選別する。
2。 SLA1遺伝子の変異体クラスリンボックス(VCB)の変異
- 2段階のアプローチ15次のSLA1遺伝子(ヌクレオチド2407から2415)でAAALQの突然変異にLLDLQをご紹介。
- 最初に、PCRによってSLA1ヌクレオチド1207から1410と2427から2589を増幅し、それぞれ、pBluescriptKS中のNotI / BamHIおよびEcoRI / SalI部位にフラグメントをクローニングする。
- をBamHI / EcoRI部位URA3を含むPCRフラグメントにサブクローン。
- をNotI / SalIで開裂得られたプラスミドは、ゲル精製によって、そして第1部で生成されたSla1 - GFP株に酢酸リチウム形質転換により、それを導入、またはTVY614(MATA URA3 - 52 LEU2 - 3112 HIS3 -Δ200TRP1にURA3断片を分離-Δ901LYS2 - 801 SUC2 -Δ9PEP4::LEU2 PRB1::HISG PRC1::HIS3)21。
- ウラシルを欠く補足SDプレート上で細胞を広げる。裏+コロニーはVCBフラグメントを置換する適切に統合されたURA3を含むことをコロニーPCRとシークエンシングにより確認してください。
- 第二段階で、サブクローンSLA1ヌクレオチドpBluescriptKS中のNotI / SalI部位に1207年から2589年。クイックチェンジ- XLの部位特異的突然変異誘発キット(Stratagene)を使用して、AAALQにVCB残LLDLQを変異。シークエンシングにより確認してください。
- クリーブBsgI /阿給で得られた構築物、およびゲル精製により断片を含む変異体VCBを分離する。パート2.3で得られた株にpRS313(HIS3)22とBsgI /阿給フラグメントをCotransform。
- ヒスチジンを欠く補足SDプレート上で細胞を広げる。レプリカプレート彼+コロニーのため、変異体配列は、URA3を置換した細胞を同定するために、5 fluorotic酸、回生AAALQの突然変異にLLDLQを含むsla1遺伝子(sla1 AAA)を含む寒天培地上に。コロニーPCRとシークエンシングにより確認してください。
3。蛍光顕微鏡
- Sla1 - GFPとsla1 AAA - GFPは、OD 600 = 0.1から0.4に達するまで30℃で補ったSDメディアを4ml ·ダークでローテータのCのパート1と2で生成された酵母株を成長する。室温で1分間4000 × gで遠心機で文化の1mlをスピン。上清の950μlを捨てる。 ·残留液(50μl程度)で細胞を再懸濁します。
- ガラスのカバースリップと顕微鏡スライドとカバー上に細胞懸濁液の沈殿物3μlの。光からサンプルを保護します。
- マウントは回転、ディスク共焦点顕微鏡の100倍の油浸対物レンズにスライドさせます。
- GFPの蛍光を励起して検出するために適切なフィルタを使用して、明視野または488 nmのレーザーを使用してセルの正しい焦点面の位置を確認します。
- 少なくとも500ミリ秒の露光時間(または適切な値)を持つ細胞でSla1 - GFPとsla1 AAA - GFPのタイムラプス画像をキャプチャ150秒間に毎秒1画像のn個の間隔。期待される結果:punctaeラベル付けGFPは、細胞の膜を制限するの内面に表示されます数秒間持続する、と信号がすぐに(分解コートを示す)が消えるように、セルの中央に向かって移動します。
- スポットが、表示数秒間維持し、消滅する見されている画像の部分を選択します。タイムラプス画像の各フレームの画像のこのセクションをトリミング。
- 経過時間に対応する軸に沿ってフレームを合わせて150秒のタイムアウト経過の画像の各フレームの画像のこのセクションを表すカイモグラフを生成します。
- スポットがキモグラフに存在する秒数(コマ撮りのフレーム)に基づいて、各スポットの持続時間を計算する。各スポットは、それが消えているように、セルの内部に向かって"曲線"、コートのエンドサイトーシスと分解を反映する必要があることに注意。
- sla1 AAA - GFPと野生型Sla1 - GFPを比較。結果は統計的に有意であるかどうかを判断するために繰り返します。期待される結果:sla1 AAA - GFPのスポットが有意に長く(約80秒)被膜形成15の欠陥を示す野生型(〜30秒)よりも持続する。
4。酵母細胞質抽出液と総細胞抽出液の調製
- このようなTVY614 21などの適切な酵母菌株、とYPD 3リットルを接種し、30で成長OD 600 = 1-2に到達するまでシェーカーインキュベーター内° C。
- 3700 × gで20分間室温で酵母培養液を遠心してください。 、上清を捨て、滅菌水の3〜5 mlを追加し、ペレットが完全に再懸濁されるまで上下にピペッティングし。
- 250mlのプラスチックビーカーに液体窒素中〜150mlを注ぐ。凍結ペレットの凝集を避けるために、円を描くように液体窒素中に滴下して酵母を急速冷凍。必要に応じてより多くの液体窒素を追加し、ペレットを融解させてはいけない。
- その中に液体窒素を注ぐことによってステンレス鋼ブレンダー容器を冷やす。液体窒素がほぼ完全に蒸発させる。コールドブレンダー容器に凍結酵母ペレットを追加。冷たいゴム栓でブレンダー容器を閉じ、10秒間酵母のペレットを挽く。内容物を混合し、二回粉砕工程を繰り返すコンテナ3-4回転倒混和します。地面凍結酵母は粉のような外観を持つことになります。
- 液体窒素で漏斗及び50 mlコニカルチューブを冷やします。寒い漏斗を使用して、管にアース酵母を転送する。凍土の酵母は、-80 ° Cまたはすぐに使用でストアすることができます。
- GST -融合タンパク質のアフィニティーアッセイ(その5)、重量凍土3gを15mlコニカルチューブにTVY614酵母と室温の緩衝液3mlに再懸ための細胞質抽出液を得た(10mMのHEPES、pH 7.0の、プロテアーゼ阻害剤カクテル(Sigma)を含む150mMのNaCl、1mMのEDTA、1mMのDTT)。
- キャップと完全に融解し、氷上に置くまで、チューブを数回転倒。
- 4に抽出液を超遠心°30万x gで20分間を慎重にペレットが剥がれないようにピペットを用いてコニカルチューブに上清(細胞質抽出)を転送する。氷上で細胞質抽出液を保管してください。
- これは、ビーズをペッティング容易にするためにチップの先端をカットするのに便利です緩衝液Aで50%(v / v)のグルタチオン - セファローススラリー100μlを加え。 4℃で回転させ° Cで15分間、4℃で遠心分離によりビーズをスピンダウンで2 1,000 × gで分、および新しいチューブに上清(細胞質抽出物)を転送するためのC。予備入力の制御のための抽出物を50μl。
- 共免疫沈降実験(その6)のための全細胞抽出物を得るために、ひずみTVY614(WT SLA1)、TVY614バックグラウンドで2部で生成されたAAALQの突然変異にLLDLQを運ぶsla1 AAAひずみ、および削除を運ぶsla1菌株を使用してくださいGPY3130 23などのようなSLA1遺伝子。重量2コニカルチューブに対応する凍土の酵母のgと室温の緩衝液2mlで再懸濁します、プロテアーゼ阻害剤カクテル(Sigma社)、2%トリトンX - 100を含む。
- キャップと完全に融解し、定期的に反転して混合、10分間氷上でインキュベートするまで、チューブを数回転倒。
- 4でマイクロチューブとスピンに素材を転送° C 16000 XG(微量遠心機で最高速度)で15分間。慎重にペレットが剥がれないようにピペットを用いて氷上でコニカルチューブに上清(総細胞抽出液)を転送する。
- 緩衝液Aで50%(v / v)の蛋白質A -セファローススラリー100μlを追加する4時に回し℃で15分間、2 1,000 × gで分、および転送のためにスピンダウン4の遠心分離によりビーズ° CのCを上清(全抽出物)を新しいチューブへ。予備入力の制御のための抽出物を50μl。
5。 GST -融合タンパク質のアフィニティーアッセイ
- PCRフラグメントcontainiによって増幅NG Sla1pバリアントクラスリンボックス(VCB)(残基803から807)は、をpGEX - 5X - VCBを得るためにをpGEX - 5Xにそれを複製する。シークエンシングにより確認してください。
- テンプレートとクイックチェンジ- XLの部位特異的突然変異誘発キット(Stratagene社製)とをpGEX - 5X - VCBを使用して、をpGEX - 5X - vCBmut取得するためにAAALQにVCB残基をLLDLQ変異。シークエンシングにより確認してください。
- エクスプレスGSTとの融合蛋白質GST - VCBとE.におけるGST - vCBmut 大腸菌 (BL21 DE3)は、還元型グルタチオンで ビーズからグルタチオン-セファロース、溶出を用いて精製し、PBSに対して透析し、タンパク質濃度を決定する。
- ラベル3マイクロ遠心チューブ:GST、GST - VCB、およびGST - vCBmut、そして1mlのPBS、バッファ内の50%(v / v)のグルタチオン - セファローススラリー30μlのdialized GST、GSTの、および50μg/mlを追加する- VCB、またはGST - vCBmutの融合タンパク質。
- 結合を可能にするために30分間、室温で回転させます。
- パート4で説明したように調製 - - 各チューブに酵母細胞質抽出液の1 mlのバッファーAでビーズをPBSで2回と1回洗浄する。
- 、5000ペレットビーズにxgでチューブを4℃で1時間C、スピン10秒を回転させ上清を捨てる、とすぐにバッファAとバッファを使用してビーズを含む0.1%トリトンX - 100、および1時間を3回洗浄
- ビーズ1つのより多くの時間をスピンダウンし、できるだけ多くの液体を除去ゲルローディングチップを使用して。この時点でサンプルは後で続行するには-20℃で保存することができます。
- 96でインキュベート各チューブに2倍Laemmliサンプルバッファー15μlの、℃で5分間を、追加し、5,000 × gで10秒スピン
- クラスリン重鎖に対する抗体を用いたイムノブロットでサンプルを分析する。期待される結果:クラスリンが出ていて、GSTまたはGST - vCBmutにGST - VCBに結合する。また、GST融合タンパク質の同様の負荷を確認するためにSDS - PAGEでサンプルを分析する。
6。共免疫沈降アッセイ
- ラベル3マイクロ遠心チューブ:WT(野生型SLA1)、sla1 AAA、およびsla1Δ、そして1mlのPBSを加える、バッファ内の50%(v / v)のタンパク質-セファローススラリー液30μl、および2μgのウサギ抗Sla1p抗体。
- 30分間室温で回転させます。
- 含む緩衝液1%トリトンX - 100を含むPBSと1時間で二回ビーズを洗浄する。
- SLA1、sla1 AAA、およびsla1Δ:パート4で製造した対応する合計酵母抽出物の1 mlの。 4℃で1時間を回転させる
- スピン10秒は5,000で、ペレット、ビーズにXGの上清を捨てる、とすぐにバッファでビーズを3回洗浄を含む0.1%のTriton X - 100、および緩衝液Aで1時間
- ビーズ1つのより多くの時間をスピンダウンし、できるだけ多くの液体を除去ゲルローディングチップを使用して。この時点でサンプルは後で続行するには-20℃で保存することができます。
- 96でインキュベート各チューブに2倍Laemmliサンプルバッファー15μlの、℃で5分間を、追加し、5,000 × gで10秒スピン
- クラスリン重鎖に対する抗体を用いたイムノブロットでサンプルを分析する。期待される結果:クラスリンsla1 AAA、またはsla1Δサンプルのではなく、野生型Sla1pと共免疫沈降物。
7。代表的な結果
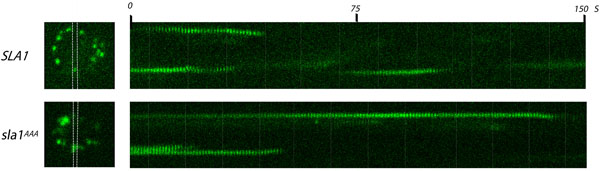
図1生細胞の蛍光顕微鏡によるエンドサイトーシスのサイトでSla1pクラスリンアダプターの分析。 Sla1 - GFPまたはAAALQの突然変異にLLDLQを運ぶsla1 AAA - GFPを発現する酵母細胞の映像と、対応するkymographs(右)から一つのフレーム(左)。野生型とSla1 - GFP変異体の両方は、内因性SLA1遺伝子座から発現させた。エンドサイトーシスのサイトでは、細胞の周辺部(左画像)で配布明るい点として観察されています。白い線の間の領域は、kymographsが生成された領域に相当する。動画は1フレーム/秒のフレームレートで撮影されました。野生型(SLA1)と比較してAAALQ変異体へLLDLQ(sla1 AAA)でSla1 - GFPの長い寿命に注意してください。
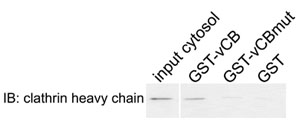
図2クラスリンとSla1p -バリアントクラスリンボックス間の物理的相互作用。 GSTはグルタチオンセファロースビーズに結合してから野生細胞質ゾル抽出物とインキュベートされたシーケンスLLDLQ(GST - VCB)、対応するAAALQ変異体(GST - vCBmut)、またはGST単独(GST)を含むフラグメントaa798 - 813をSla1pに融合酵母細胞のように入力します。関連するタンパク質がクラスリン重鎖のために(IB)イムノブロッティングにより溶出し、分析した。
ディスカッション
クラスリン被覆小胞(CCV)は、 トランスゴルジネットワークからエンドサイトーシスおよびトランスポートに参加し、真核細胞生物学の基礎となる保存経路をエンドソーム。このメソッドは用紙に記載されてのアプローチは、アダプタータンパク質とクラスリンとの間の特定の相互作用において、CCVの形成に関与する分子メカニズムを研究するために便利です。 GST -融合タンパク質の...
開示事項
謝辞
DFは、博士のフェローシップへのNSFのブリッジによってサポートされています。本研究で使用した顕微鏡は、コロラド州立大学から顕微鏡イメージングネットワークコアインフラ助成金によって部分的にサポートされています。著者の研究室でのクラスリンアダプターの作業は、CSUスタートアップ資金と米国心臓協会賞SDへ09SDG2280525でサポートされています
資料
| 材料 | 会社 | ||
|---|---|---|---|
| Name | Company | Catalog Number | Comments |
| クイックチェンジ- XL部位特異的突然変異誘発キット | ストラタジーン | ||
| プロテアーゼ阻害剤カクテル | シグマ |
参考文献
- Mellman, I., Warren, G. The road taken: past and future foundations of membrane traffic. Cell. 100, 99-112 (2000).
- Engqvist-Goldstein, A. E., Drubin, D. G. Actin assembly and endocytosis: from yeast to mammals. Annu Rev Cell Dev Biol. 19, 287-332 (2003).
- Conner, S. D., Schmid, S. L. Regulated portals of entry into the cell. Nature. 422, 37-44 (2003).
- Bonifacino, J. S., Traub, L. M. Signals for sorting of transmembrane proteins to endosomes and lysosomes. Annu Rev Biochem. 72, 395-447 (2003).
- Ungewickell, E. J., Hinrichsen, L. Endocytosis: clathrin-mediated membrane budding. Curr Opin Cell Biol. 19, 417-425 (2007).
- Doherty, G. J., McMahon, H. T. Mechanisms of endocytosis. Annu Rev Biochem. 78, 857-902 (2009).
- Kirchhausen, T. Clathrin. Annu Rev Biochem. 69, 699-727 (2000).
- Brodsky, F. M., Chen, C. Y., Knuehl, C., Towler, M. C., Wakeham, D. E. Biological basket weaving: formation and function of clathrin-coated vesicles. Annu Rev Cell Dev Biol. 17, 517-568 (2001).
- Owen, D. J., Collins, B. M., Evans, P. R. Adaptors for clathrin coats: structure and function. Annu Rev Cell Dev Biol. 20, 153-191 (2004).
- Dell'Angelica, E. C., Klumperman, J., Stoorvogel, W., Bonifacino, J. S. Association of the AP-3 adaptor complex with clathrin. Science. 280, 431-434 (1998).
- Dell'Angelica, E. C. Clathrin-binding proteins: got a motif? Join the network!. Trends Cell Biol. 11, 315-318 (2001).
- Holtzman, D. A., Yang, S., Drubin, D. G. Synthetic-lethal interactions identify two novel genes, SLA1 and SLA2, that control membrane cytoskeleton assembly in Saccharomyces cerevisiae. J Cell Biol. 122, 635-644 (1993).
- Howard, J. P., Hutton, J. L., Olson, J. M., Payne, G. S. Sla1p serves as the targeting signal recognition factor for NPFX(1,2)D-mediated endocytosis. J. Cell. Biol. 157, 315-326 (2002).
- Mahadev, R. K., Pietro, S. M. D. i., Olson, J. M., Piao, H. L., Payne, G. S., Overduin, M. Structure of Sla1p homology domain 1 and interaction with the NPFxD endocytic internalization motif. EMBO J. 26, 1963-1971 (2007).
- Pietro, S. M. D. i., Cascio, D., Feliciano, D., Bowie, J. U., Payne, G. S. Regulation of clathrin adaptor function in endocytosis: A novel role for the SAM domain. EMBO J. 29, 1033-1044 (2010).
- Kaksonen, M., Toret, C. P., Drubin, D. G. A modular design for the clathrin- and actin-mediated endocytosis machinery. Cell. 123, 305-320 (2005).
- Longtine, M. S., McKenzie, A., Demarini, D. J., Shah, N. G., Wach, A., Brachat, A., Philippsen, P., Pringle, J. R. Additional modules for versatile and economical PCR-based gene deletion and modification in Saccharomyces cerevisiae. Yeast. 14, 953-9561 (1998).
- Wach, A., Brachat, A., Alberti-Segui, C., Rebischung, C., Philippsen, P. Heterologous HIS3 marker and GFP reporter modules for PCR-targeting in Saccharomyces cerevisiae. Yeast. 13, 1065-1075 (1997).
- Robinson, J. S., Klionsky, D. J., Banta, L. M., Emr, S. D. Protein sorting in Saccharomyces cerevisiae: isolation of mutants defective in the delivery and processing of multiple vacuolar hydrolases. Mol Cell Biol. 8, 4936-4948 (1988).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J. Bacteriology. 153, 163-168 (1983).
- Vida, T. A., Emr, S. D. A new vital stain for visualizing vacuolar membrane dynamics and endocytosis in yeast. J Cell Biol. 128, 779-792 (1995).
- Sikorski, R. S., Hieter, P. A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae. Genetics. 122, 19-27 (1989).
- Piao, H. L., Machado, I. M., Payne, G. S. NPFXD-mediated endocytosis is required for polarity and function of a yeast cell wall stress sensor. Mol Biol Cell. 18, 57-65 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved