Method Article
前のDNA精製せずに植物や動物のサンプルの遺伝子型
要約
ここで紹介するダイレクトPCR法は、未精製の植物および動物組織の少量から直接PCR増幅を容易にします。
要約
ダイレクトPCR法は、未精製サンプルの少量から直接PCR増幅を容易にし、いくつかの植物や動物の組織( 図1)のためにここに例示されている。ダイレクトPCRは、彼らにこのような阻害剤の耐性が高いなどのユニークな特性を与える二本鎖DNA結合ドメインを含む特別に設計されたサーモサイエンティフィックPhusionとPHIRE DNAポリメラーゼ、に基づいています。
PCRベースの標的DNAの検出は、導入遺伝子の植物の遺伝子型解析と検証を含む、植物研究の多数のアプリケーションを持っています。植物組織からPCR法は、伝統的に高価なまたは有毒な試薬を必要とするかもしれない最初のDNA単離工程を伴う。プロセスは時間がかかり、クロスコンタミネーション1,2のリスクを増大させる。逆に、サーモサイエンティフィックPHIREプラントダイレクトPCRキットを用いて、標的DNAは簡単に前にDNAを抽出することなく検出することができる。モデルでは、ここに示さ派生切断増幅多型配列を解析した例(dCAPS)3,4は、 シロイヌナズナ植物の葉から直接実行されます。 dCAPSジェノタイピングアッセイは、SNP対立遺伝子特異的な制限エンドヌクレアーゼ消化によって3個の一塩基多型(SNP)を同定するために用いることができる。
いくつかの植物試料は、彼らがそのようなフェノール化合物としてPCRを妨害する成分を含んでいるため直接PCR法を用いている場合にはより挑戦的になる傾向があります。これらのケースでは、化合物を除去するために追加の手順が伝統的に2,5を必要とされる。ここでは、この問題は直接PCR増幅( 図1)に続いて迅速かつ簡単に希釈プロトコルを使用することによって克服される。標本はタンニンを含むフェノール化合物を大量に含んでいるため十五歳のオークの葉は厳しい植物のモデルとして使用されています。
マウスへの遺伝子導入は大まかdeveloの遺伝子の役割を研究するために使用されpment、生理学や人間の病気。これらの動物の使用は、通常のPCRで、導入遺伝子の存在をスクリーニングする必要があります。伝統的に、これはPCR解析用DNAは耳、尾やつま先組織6,7から精製されている間の時間がかかるのDNA単離工程を伴う。しかし、サーモサイエンティフィックPHIRE動物組織ダイレクトPCRキットトランスジェニックマウスとの事前のDNA精製せずに遺伝子型を決定することができます。唯一つのプライマーセットはサイズが大きく異なる2つのフラグメントの増幅のために使用されている挑戦的な例については、ここを実証されるように、このプロトコルトランスジェニックマウスのジェノタイピングでは、マウスの耳組織から直接得られる。
プロトコル
1。直接プロトコルを持つシロイヌナズナ植物個体のジェノタイピング
- シロイヌナズナ植物の葉の上に直接dCAPSジェノタイピングアッセイを開始するには、まず、 表1に示すようにPHIREプラントダイレクトPCRキットを用いて、20または50μlのPCR反応を定式化する。
- 次に、ハリスユニコアとハリス·カッティングマットを使用して、 シロイヌナズナ植物の葉から0.50 mmのパンチをカット。しっかりとパンチャーを持って、組織の中に最先端を押すと前後にパンチャーを回転させます。
- PCR反応混合物にパンチ、ディスクを取り出すにプランジャを押してください。サンプルはPCR溶液に落下し、管壁に付着しないことを確認します。
- 2パーセント次亜塩素酸ナトリウム溶液中に浸漬して、クロスコンタミネーションを防止するために、各サンプル間のパンチャーの最先端を清掃してください。プランジャーを押して、上下に数回、清潔なペーパータオルで刃先を拭いてください。カッティングマットもあるべきサンプル間でリンスした。
- 次に、 表2に記載のサイクリング条件を用いてPCR反応を実行するためのサーモサイエンティフィックピコピコ24ウェルサーマルサイクラーとサーモサイエンティフィックピコピコPCRプレートを採用しています。 PCR反応は、従来のPCRサーマルサイクラーで行うことができます。
- PCR後、植物材料をスピンダウンします。新しいマイクロチューブに上清5μlを移す。水4μlを、制限酵素、SSPIを1μl加え。穏やかに混合し、チューブの底に内容物を収集するために簡単にスピンダウンします。 37℃で1時間反応混合物をインキュベート℃に20分間65℃でインキュベートすることにより、制限酵素を不活性化する。
- 植物組織から直接DNAを増幅する場合、PCR産物を制限酵素消化酵素を妨げる可能性があり、植物やPCR由来成分が含まれています。したがって、それはどちらの希釈( 例えば 1:2または水で1:3)またはtの前にPCR産物を精製する必要があるかもしれません彼はそのようなサーモサイエンティフィックGeneJET PCR精製キットのような適当な市販のキットを用いて、例えば、その後の消化。
- 制限酵素消化後、アガロースゲル電気泳動で得られた断片を分析します。反応液に5倍のローディングバッファー2.5μlを添加し、得られた混合物の10μlをアガロースゲル電気泳動を行う。
2。 15歳のオークから特異的なDNA断片を増幅するには、希釈プロトコルを使用して葉
- 開始するには、オークの葉の2mmのパンチをカット。
- PHIRE工場ダイレクトPCRキットに付属の希釈緩衝液20μlにサンプルを置きます。パンチャーの最先端をきれいにし、前のようにマットをカット。
- 管の壁に簡単にそれを押すことによって、100μlのピペットの先端で葉のサンプルをつぶす。植物材料に応じて、希釈プロトコルは、時々サンプルを破砕せずにうまく動作します。
- tまで微量で簡単にサンプルをスピン彼の反応は、チューブの底にあります。
- 上清をPCRに直接使用することができ、またサンプルの種類に応じて、滅菌水で1:10または1:100に希釈することができる。 20μlのPCR反応の鋳型として清または希釈液の1μlに0.5μlを使用する。 表1に記載したように反応を準備します。
- 一度反応が用意されており、 表2に記載のサイクリング条件を用いてPCR反応を実行するためにサーモサイエンティフィックピコピコ24ウェルサーマルサイクラーとサーモサイエンティフィックピコピコPCRプレートを採用しています。 PCR反応は、従来のPCRサーマルサイクラーで行うことができます。
- PCR後、反応液に5倍のローディングバッファーを5μlを加え、得られた混合物の15μlをアガロースゲル電気泳動を行う。
3。希釈プロトコルを使用してトランスジェニックマウスのジェノタイピング
- mの2 mmのパンチを配置することによって、トランスジェニックマウスで希釈プロトコルを開始PHIRE動物組織ダイレクトPCRキットに付属DNARelease添加剤の0.5μlを含む希釈バッファー20μlにウーズの耳。
- 98℃2分のインキュベーションに続いて2分、℃の室温でサンプルをインキュベートする
- 反応はチューブの底になるまで遠心で簡単にサンプルをスピン。
- 表3に記載のように調製し、20μlのPCR反応の鋳型として清1μlを使用する。すぐに使用されるためにされていない任意の上清を新しいチューブに移し、-20℃で保存することができます
- 次に、 表4に記載のサイクリング条件を用いてPCR反応を実行するためのサーモサイエンティフィックピコピコ24ウェルサーマルサイクラーとサーモサイエンティフィックピコピコPCRプレートを採用しています。繰り返しになりますが、PCR反応は、従来のPCRサーマルサイクラーで行うことができます。
- PCR後、反応液に5倍のローディングバッファー5μlを追加し、アガロースゲル電気泳動ワットを行う得られた混合物のi番目の15μlの。
4。代表的な結果
dCAPS技術では目的のSNP内の制限部位はどちら標的DNAに1つまたはそれ以上のミスマッチを有するPCRプライマーを用いて導入するか、または破棄されます。 シロイヌナズナ植物個体の遺伝子型を直接葉のパンチからdCAPS技法を用いて行った。増幅産物を消化し 、各個体の遺伝子型を明らかに得られた断片をゲル電気泳動( 図2)により分析した。
この例では、 シロイヌナズナゲノム中に目的のSNP部位(Gまたは対立遺伝子)を含む160bpのPCR産物がPHIRE工場ダイレクトPCRキットを用いた植物の葉のパンチから増幅した。フォワードプライマーは、目的のSNP(対立)ではなく、が含まれていた標的DNA中SspIで固有の制限部位を、作成、3 '末端に1つのミスマッチを含んでいたSNPの他の対立遺伝子( 図2B)インチ
希釈プロトコルは、フェノール性化合物のその高い濃度のためにそのようなオークの葉のような挑戦的な植物材料、のために必要である。ここで説明する希釈プロトコルを使用して、葉緑体DNAの297塩基対の断片は、3ステップのプロトコルを用いて増幅した。様々な希釈とオークの葉の標本は、正と負の制御に加えて、 図3に示されています。
PHIRE動物組織ダイレクトPCRキットは、トランスジェニックマウス、1,500 bpおよび200 bpのための正しいサイズの製品の両方の豊かな収率で得られた、唯一つのプライマーセットが大きいサイズの差を持つ2つのフラグメントの増幅のために使用した挑戦的なプロトコルで採用された野生型マウス( 図4)。ヘテロ接合体マウスを用いた弱い上部のバンドは、同じプライマーペアの両方のテンプレート(対立遺伝子)の競争のためであるが、genotypる結果は明白です。
希釈プロトコルを使用して調製したサンプルの安定性もテストされています。結果は希釈サンプルは、少なくとも1年間は-20℃で保存することができることを示している。さらに、繰り返し凍結/融解サイクルが大幅に反応します ( 図5)には影響しなかった。また、ホットスタートTaqポリメラーゼおよび精製されたDNA( 図6)に比べPHIRE DNAポリメラーゼおよびダイレクトPCR法を用いたマウス耳組織サンプルからの堅牢な増幅が示されている。
| コンポーネント | 20μlの反応 | 50μlの反応 | 最終濃度。 |
| H 2 O | 20μlに追加 | 5に追加0μlの | |
| 2倍PHIRE植物のPCRバッファー | 10μlの | 25μlの | 1X |
| プライマー | μlのX | μlのX | 0.5μMで |
| プライマーB | μlのX | μlのX | 0.5μMで |
| PHIREホットスタートII DNAポリメラーゼ | 0.4μL | 1μlの | |
| サンプル/ダイレクトプロトコル | 0.50ミリメートルパンチ | ||
| サンプル/希釈プロトコル | 0.5から1μL |
表1。植物のPCRの反応条件は、このプロトコルで使用される。
| サイクルステップ | Tempです。 | 時間 | サイクル |
| 初期変性 | 98℃ | 5分 | 1 |
| 変性 *をアニーリング 延長 | 98℃ X℃で 72℃ | 5秒 5秒 20秒 | 40 |
| 最後の伸長 | 72℃ 4℃ | 1分 ホールド | 1 |
表2植物のPCR用のサイクリング条件は、このプロトコルで使用される。
* Tmは電卓でoscientific.com / pcrwebtools "ターゲット=" PHIREホットスタートII DNAポリメラーゼで使用するプライマーのTmを決定する際には、_blank "> www.thermoscientific.com / pcrwebtoolsが推奨されます。プライマーの推奨アニール温度が≤20 NTに等しいWebツールによって与えられる低いTmはプライマー> 20 NTの場合は、アニーリング温度を使用して3°Webツールによって与えられる低いTmよりC高い。
| コンポーネント | 20μlの反応 | 最終濃度。 |
| H 2 O | 20μlに追加 | |
| 2倍PHIRE動物組織PCRバッファー | 10μlの | 1X |
| プライマー | μlのX | 0.5μMで |
| プライマーB | μlのX | 0.5μMで |
| ペーハー怒りホットスタートII DNAポリメラーゼ | 0.4μL | |
| サンプル/希釈プロトコル | 1μlの |
表3動物の組織PCRの反応条件は、このプロトコルで使用される。
| サイクルステップ | Tempです。 | 時間 | サイクル |
| 初期変性 | 98℃ | 5分 | 1 |
| 変性 *をアニーリング 延長 | 98℃ X℃で 72℃ | 5秒 5秒 20秒≤1キロバイト 20秒/ KB> 1キロバイト | 40 |
| 最後の伸長 | 72℃ 4℃ | 1分 ホールド | 1 |
表4は、このプロトコルで使用される動物の組織PCRのサイクル条件。
で* Tmは電卓www.thermoscientific.com / pcrwebtoolsは PHIREホットスタートDNAポリメラーゼIIで使用するプライマーのTmを決定する際に推奨されます。プライマー≤20 ntのための推奨アニーリング温度はWebツールによって与えられる低いTmに等しい。プライマー> 20 NTの場合は、下のTmよりも℃高いWebツールによって与えアニール温度3を使用します。
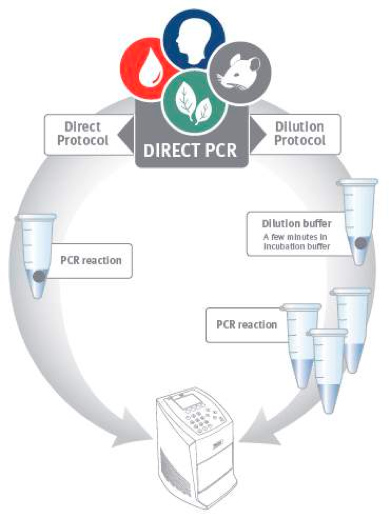
図1。ダイレクトPCRワークフロー。ダイレクトPCRは2代替のプロトコルを使用して実行できます。直接のプロトコルでは、試料の少量をPCに直接追加されますRの反応。希釈プロトコルは、試料物質からDNAを放出するためのPCRの前に簡単なプレインキュベーション工程を採用しています。
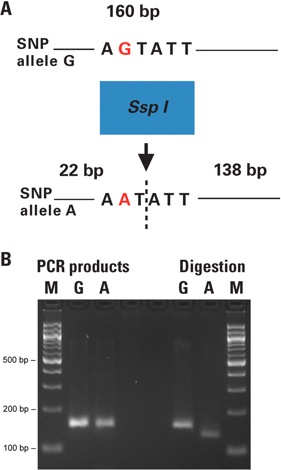
図2。直接リーフパンチからdCAPS技術とシロイヌナズナ植物個体の遺伝子型別。 A)は、図2Aは、実行dCAPSアッセイの原理を示す。目的のSNP内の制限部位はどちら導入または標的DNAへの1つまたはそれ以上のミスマッチを持つプライマーを用いたPCRによって除去される。 PCR産物を消化し 、電気泳動で分析したときに得られた断片は、それぞれの個体の遺伝子型を明らかにする。B)は 、植物の葉から0.50 mmのパンチは50μlのPCR反応に直接置かれている。 SNP部位(Gまたは対立遺伝子)を含む160bpのPCR産物がアレルにユニークなSSPIの部位を導入するプライマーを用いて増幅した。未精製のPCR産物はdigesだったSSPIの制限酵素でテッド。得られた断片を3%アガロースゲル上で分析した。レーンMはサイズマーカーで、レーンがあり、G各サンプルのSNP対立遺伝子に対応しています。得られた結果は、PCRや制限酵素処理(データは示していない)が続いてDNA抽出の従来の分析によって確認した。テンプレートDNAなしのネガティブコントロール反応を消化した(データは示していない)を実行する前に、テストの設定と実際のPCR反応と並行して別々のアガロースゲル上で実行に含まれていた。
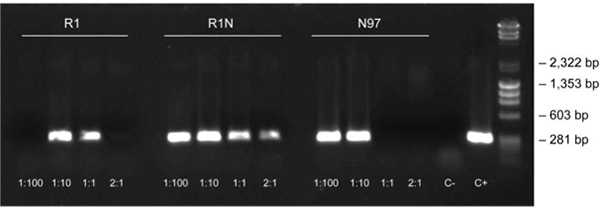
図3。希釈プロトコルはオークの葉のサンプルを使用した。希釈プロトコルが(1:100、1:10、1異なる希釈でオークの葉のサンプル(3ステップのプロトコル、62℃でアニール)の葉緑体DNAの297塩基対の断片を増幅するために使用されました:1と2:1)と、正と負の詐欺trols [C + +:C:いいえ、鋳型DNAとネガティブコントロール精製DNA( シロイヌナズナ )からポジティブコントロール]。サンプルの命名法は、試料捕集サイト(フィンランドの町)と番号のサンプルを表しています。
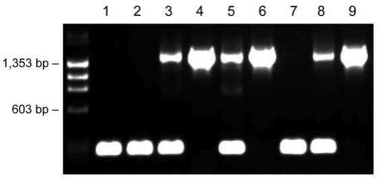
図4。希釈プロトコルを使用して1つのプライマー対を持つトランスジェニックマウスの遺伝子型別。9個々のマウス(レーン1-9)の耳の組織はDNARelease添加剤を含む希釈緩衝液20μlに入れた。室温および98℃でインキュベーションした後、上清1μlを20μlのPCR反応の鋳型として用いた。フラグメントサイズ:1500 BP(トランスジェニック)および200bp(野生型)。
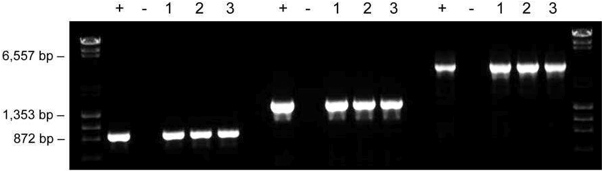
図5。サンプルは、希釈プロトコルに従って調製長期保存のために安定している。マウス耳組織のサンプルはincあっ DNARelease添加剤を含む希釈バッファー20μlにubated、繰り返し凍結/解凍(レーン1)にかけ、1年間(レーン2)、-20℃で保存し、またはPCR(レーン3)のために直ちに使用。増幅断片のサイズは900bpの、1500 bpと3200 bpであった。

図6。 PHIRE動物組織ダイレクトPCR KitはホットスタートTaq DNAポリメラーゼと結合し、市販のDNA精製システムよりも優れています。四リコン(0.5-1.5 KBは)PHIRE動物組織ダイレクトPCRキットの希釈プロトコルを使用して、マウスの耳組織から増幅した。比較のために、DNAは最初、市販のDNA抽出キットを用いて、同一のフラグメントは、製造業者の推奨に従ってホットスタートTaq DNAポリメラーゼを用いて増幅した精製した。のみPHIRE動物組織ダイレクトPCRキットで正常に増幅4つのすべてのアンプリコンがあった。
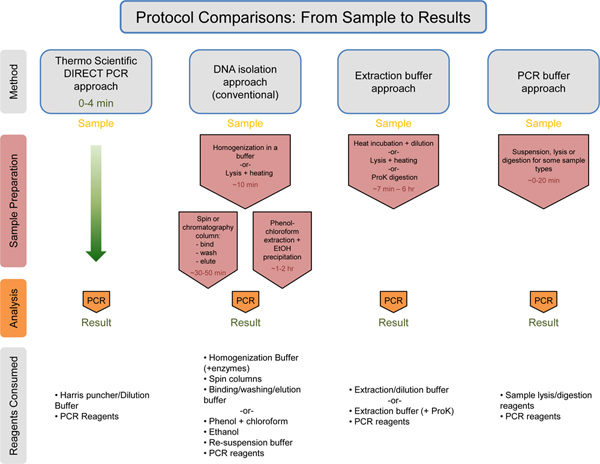
図7。回/消費試薬の推定を伴うワークフローのユニバーサル比較。サーモサイエンティフィックダイレクトPCR法は、DNA単離アプローチ、抽出バッファーのアプローチ、およびPCRバッファアプローチと比較されます。 PCRの前工程へのそれぞれのアプローチのための推定時間が赤文字で表示されています。サーモサイエンティフィックダイレクトPCR法は、PCRの前に試料調製のための唯一の0から4分かかります。各メソッドの下に、消費された試薬が記載されています。サーモサイエンティフィックダイレクトPCR法は他の方法と比較して試薬の最小量を使用しています。 拡大図を表示するには、ここをクリックしてください 。
ディスカッション
ここで示したダイレクトPCR法は、必要な時間を短縮し、未精製サンプルの少量から直接PCR増幅を可能にし、植物や動物のジェノタイピング( 図7)のワークフローを簡素化します。また、ここで示したダイレクトPCR法( 図3)との組み合わせで希釈プロトコルを使用することである。希釈プロトコルが困難なサンプル( 例えば高齢者の植物の葉は、干渉化合物を含有する植物種)または長い(またはGCリッチ)アンプリコンとをお勧めします。新しいダイレクトPCR実験を開始するときに、このプロトコルは、反応の最適化を可能にする、特に便利です。プロトコルは、このような組織材料および/または使用したプライマーの違いに起因する低い生成物の収率として時折困難に対処するためのツールとして機能することができます。また、同じサンプルから複数の反応を実行するとき、希釈プロトコルの使用はサンプル全体が一つに消費されないことを保証し反応。
PCRのための植物からDNAを精製するためには、規則は潜在的にPCR解析8に干渉する可能性があるさまざまなコンポーネントを使用して、DNA単離に続く細胞溶解を行うことでした。高フェノール含量、ポリビニルピロリドンを加えて特に難しい植物のため、伝統的に細胞溶解2,5続くDNAに結合する化合物を除去するために使用されていました。ここで明らかなように、これらの複雑な、時間のかかるステップは標的DNAを検出するためにPHIRE工場ダイレクトPCRキットを使用することによって回避することができます。細胞溶解とDNA分離ステップが直接シロイヌナズナ植物の葉( 図2)から、敏感dCAPS技術を使用して省略することができます。ここに示したように、希釈プロトコルは効果的にPCR法( 図3)に干渉することが問題の成分の存在を管理します。
伝統的に、動物タイピングするための前提条件であったisolati有機溶媒抽出、アルコール沈殿、遠心ステップ6,9,10続く皮膚や結合組織を消化するプロテイナーゼKとの長いインキュベーションによって特徴付け毒性、時間のかかるプロセスを通じて、動物組織からのゲノムDNAの上に。ここで示したように、このプロセスの簡素化は、トランスジェニックマウスは、従来のDNA精製( 図4)を使用せずに遺伝子型を特定することができるように、PHIRE動物組織ダイレクトPCRキットの使用によって達成される。唯一つのプライマーセットが大きいサイズの差を持つ2つの断片の増幅のために使用されたとして、この記事で紹介例は特に困難である。プロトコルの生得の難しさにもかかわらず、単純な希釈プロトコルの組み合わせは直接PCR法の使用は、2つのPCR産物( 図4)の高収率が得られた。
PCRベースの標的DNAの検出は、識別するために、遺伝子型解析を含め、研究に多くのアプリケーションを持っている開発、生理学と疾患における遺伝子の役割。特殊なDNAポリメラーゼの阻害剤の許容範囲により、プロトコルが前にDNAを精製することなく、最小限の時間で完了することができます。
開示事項
この記事への生産とフリーアクセスはサーモフィッシャーサイエンティフィック社が主催している
謝辞
シロイヌナズナサンプルと関係するPCRプライマーのために私達は教授試しプレーKangasjärviと彼のグループは、植物ストレス群、植物生物学、バイオサイエンス学科、ヘルシンキ大学に感謝します。伝統的な手法を用いた実験は、夫人あいりLamminmäkiによって実施された。
トランスジェニックマウスのサンプルが博士Jaana Vesterinen、生物医学/生化学研究所、ヘルシンキ大学から提供された。
ハリスユニコアとハリス·カッティングマットShundersonコミュニケーションズ株式会社の商標です。その他すべての商標は、サーモフィッシャーサイエンティフィック社およびその子会社の商標です。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名称 | 会社 | カタログ番号 | 注釈 |
| PHIREプラントダイレクトPCRキット | サーモフィッシャーサイエンティフィック | F-130 | 200反応液(50μlずつ) |
| PHIRE動物組織ダイレクトPCRキット | サーモフィッシャーサイエンティフィック | F-140 | 200 reations(50μLずつ) |
| ハリスユニコア0.35ミリメートル(ピンク) | サーモフィッシャーサイエンティフィック | F-180S / L | 数量:5月25日 |
| ハリスユニコア0.50ミリメートル(青) | サーモフィッシャーサイエンティフィック | F-185S / L | 数量:5月25日 |
| ハリスカッティングマット6.4×7.6センチメートル | サーモフィッシャーサイエンティフィック | F-190 | 数量:5 |
| SspIで | サーモフィッシャーサイエンティフィック | ER0771 | 100μL(100回分) |
| ピコピコ24ウェルサーマルサイクラー | サーモフィッシャーサイエンティフィック | TCP0024 | |
| 24ウェルPCRプレートピコピコ | サーモフィッシャーサイエンティフィック | SLP0241 | |
| GeneJET PCR 精製キット | サーモフィッシャーサイエンティフィック | K0701 K0702 | 50プレップ 250プレップ |
参考文献
- Doyle, J. J., Doyle, J. L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin. 19, 11-15 (1987).
- Kim, C. S., Lee, C. H., Shin, J. S., Chung, Y. S., Hyung, N. I. A simple and rapid method for isolation of high quality genomic DNA from fruit trees and conifers using PVP. Nucleic Acids Research. 25, 1085-1086 (1997).
- Neff, M. M., Neff, J. D., Chory, J., Pepper, A. E. dCAPS, a simple technique for the genetic analysis of single nucleotide polymorphisms: experimental applications in Arabidopsis thaliana genetics. The Plant Journal. 14, 387-392 (1998).
- Michaels, S. D., Amasino, R. M. A robust method for detecting single-nucleotide changes as polymorphic markers by PCR. The Plant Journal. 14, 381-385 (1998).
- John, M. E. An efficient method for isolation of RNA and DNA from plants containing polyphenolics. Nucleic Acids Research. 20, 2381-23 (1992).
- Wang, Z., Storm, D. R. Extraction of DNA from mouse tails. BioTechniques. 41, 410-412 (2006).
- Malumbres, M., Mangues, R., Ferrer, N., Lu, S., Pellicer, A. Isolation of High Molecular Weight DNA for Reliable Genotyping of Mice. BioTechniques. 22, 1114-1119 (1997).
- Yang, Y., Kim, J. Y., Soh, M. S., Kim, D. S. A simple and rapid gene amplification from Arabidopsis Leaves Using AnyDirect system. Journal of Biochemistry and Molecular Biology. 40, 444-447 (2007).
- Sambrook, J., Fritsch, E., Maniatis, T. Molecular Cloning: A Laboratory. , 2nd ed, CSH Laboratory Press. Cold Spring Harbor, NY. (1989).
- Meldgaard, M., Bollen, P. J. A., Finsen, B. Non-invasive method for sampling and extraction of mouse DNA for PCR. Laboratory Animals. 3, 413-441 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved