このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ヒト皮質下の高分解能構造磁気共鳴イメージング
要約
Here we present a protocol to determine the minimum number images that needed to be registered and averaged to resolve subcortical structures and test whether the individual layers of the LGN could be resolved in the absence of physiological noise.
要約
この研究の焦点は、人間の脳の生活に比べ死後脳の構造的MRIの解像度の限界をテストすることでした。 in vivoでの構造的MRIの解像度は、最終的に拍動、呼吸や頭の動きなど、生理学的ノイズによって制限されます。画像化ハードウェアは改善し続けているが、それはミリメートルスケールの構造を解決することは困難です。例えば外側膝状核(LGN)、通常6インターリーブ単眼層で構成されて視床における視覚リレーと制御核に、一次視覚感覚経路のシナプス。ニューロイメージング研究は確実に、厚さ1ミリメートル未満のサイズが小さいため、これらの層を区別することができていません。
死後脳の構造的MRIの分解能限界は、長い時間(〜24時間)にわたって平均化複数の画像を用いて試験しました。目的は、個々のLを解決することが可能であったかどうかを試験することでした生理的ノイズの不在下でのLGNのエアーズ。プロトン密度(PD)1加重パルスシーケンスを登録するのに必要な画像の最小数を決定するために、解像度および他のパラメータを変えて使用し、確実にLGNおよび他の皮質下の領域を区別するために平均しました。結果は、ヒトの脳の中で生きて取得した画像と比較した。 インビボでの被験体は、臨床用途に有用な皮質下構造を区別するために必要なPDスキャンの最小数の生理学的ノイズの付加的な効果を決定するためにスキャンしました。
概要
本研究の目的は、生理的なノイズのない状態での構造のMRIの解像度の限界をテストすることでした。プロトン密度(PD)強調画像を登録するために必要な画像の最小数を決定するために長い時間(2〜24時間のセッション)で死後脳において取得されたと皮質下構造を解決するために平均化しました。比較のために、PD加重画像もセッションの数を介して人間の生活に取得しました。特に、目的はそれは厚さ約1mm( 図1)、ヒトLGNのすべての6つの個々の層を、解決するために最良のシナリオでは可能であるかどうかを確認することでした。
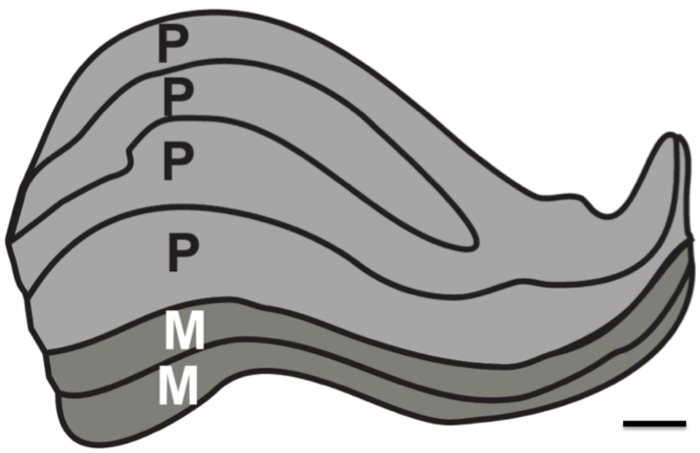
図1.人間の外側膝状核層。LGNの層構造の模式図。大細胞(M)層は、より大きな神経細胞で構成されています細胞の大きさと運動とコースアウトライン(層1-2、濃い灰色として描か)を解決するための責任がある小さいセル密度。小細胞性層(P)は(ライトグレーとして描か層4-6、)微形と色を解決する責任がある小さい神経細胞の大きさと、より大きな細胞密度で構成されています。スケールバー1ミリメートル。染色された人間のLGN 12に基づいて図。
行列のサイズが増加するとMRIの空間分解能が改善され、フィールド・オブビュー(FOV)とスライス厚が減少したとき。しかしながら、増加した解像度は、ボクセル体積に比例した信号対雑音比(SNR)に信号を減少させます。 SNRはまた、測定値の数の平方根に比例します。複数の画像が別々のイメージングセッションの数を介して取得することができるが、生きているヒトでは、最終的な解決は、呼吸、循環脈動や頭の動きとして、生理的なノイズによって制限されます。
高い-resolution(0.35ミリメートルの面内のボクセル)は、重み付けされたスキャンを取得したPD。 PDのスキャンは視床1の灰色と白のコントラストを強調し、T 1とT 2の影響を最小限に抑えた画像になります。その画像は、撮像ボリューム内のタンパク質や脂肪などの水と高分子の形でのプロトンの密度に依存しています。磁化2の高い縦成分には画像を明るく信号における組織結果の陽子数の増加。
それらは周囲の組織と皮質下構造のより高いコントラストを提供するので、PD加重スキャンを収集しました。そのようなT1-及びT2強調画像などの他のコントラストが、1,3ƒ決定されるように、より小さなコントラスト対雑音比にLGNのような皮質下構造を描写することが困難になります。
同様に、以前の研究は、ホルマリンのPD-強調画像は、死後脳がresulte固定発見しましたT1-と同様のグレーと白質画像強度3,4を持っていた T2強調画像と比較して、灰色と白質間のより高いコントラスト差でD。基礎となる生物物理学的な決定は、これらの違いを説明することができます。水素プロトンの(縦)T1およびT2(横)緩和時間は、組織内でどのように水の移動に依存しています。このような架橋タンパク質によるホルマリン作品として固定剤。固定剤が使用される場合、水の移動性との間の差は、異なる組織タイプの間で低減されます。脳組織内のプロトンの相対密度の差は、より良いコントラスト分化3,4を提供する 、固定して増加したのに対し、減少T1組織コントラストは、固定後に観察されています。
以前の研究では1.5 T 5,6,7を使用して 、PD加重スキャン中、および3のTスキャナ8,9でLGNを同定しました。これは正確の範囲を概説することができるようにこれらのスキャンを得ることが重要ですLGN。皮質下核の完全なカバレッジを維持するために、18 PD加重スライスは視床以内に得られました。各ボリュームは、2倍の解像度1024行列を再サンプリング(0.15 mmの面内ボクセルサイズ)、連結された、動きが補正され、皮質下構造の高解像度3D画像を生成するために平均しました。次のスライスの処方に必要なPD画像の最適な数は、生きているヒトにおいて15分未満にスキャン時間を短縮する、5ました。わずか1 PDイメージは3分未満にスキャン時間を短縮することが、明らかに死後脳で皮質下領域を区別するために必要とされた( 図2および3)。
全体ホルマリン固定死後脳標本は、82歳で心肺停止で死亡した女性からスキャンしました。医療記録の見直しは、彼女が持っていたことを明らかにした:子宮摘出で処理された慢性閉塞性肺疾患、狭心症、トリプルバイパス手術8年前に死に、子宮癌を7年前に死亡、高脂血症、緑内障、白内障手術へ。死後脳検体をC.Theの死後の脳は、画像品質の比較のために多くの時間をかけて同じイメージングプロトコルで、ならびに他のパラメータを用いてスキャンし、4℃で少なくとも3週間、10%中性緩衝ホルマリン中に浸漬固定しました。 。のみ最適化されたパラメータは、プロトコルのために説明します。
プロトコル
1.参加者と死後脳セットアップ
注:すべての画像は、32チャネルのヘッドコイル及び全てMRIスキャンで3 T MRIスキャナを用いて取得されたRT、約20℃で実施しました。すべての参加者は右利きであり、書面によるインフォームドコンセントを与えました。各参加者は、神経疾患の既往がないとの良好な健康状態にありました。実験プロトコルは、承認され、ヨーク大学人間の参加者審査委員会のガイドラインに従いました。
- 各参加者が記入し、MRI安全ガイドラインとニューロイメージングプロトコルを詳細に患者の同意書に署名して下さい。
- 各参加者のために、それぞれの耳に耳栓を配置し、頭部の動きを最小限に抑えるためにパッドと頭を固定します。
- 死後の脳イメージングのために、脳は神経画像の前に固定されており、MRI用ヘッドコイル内に収まる袋や容器内に収容されていることを確認します。 infに(優れた、そのz軸とヘッドコイルに死後脳を置き、erior)スキャナのボアと整列されます。脳幹(後部)は、スキャナベッドの足の方に向くようにします。
- 追加のサポートのための死後の脳の周りに真空クッションの手を置きます。
2.皮質下のローカライズと処方
注:視床は中脳および大脳皮質の間に位置する脳の中央付近に位置デュアルローブ構造体です。背側視床内にあり、人間のLGNは〜10mmの最大を拡張する小さな皮質下の構造体です。
- 新しい参加者を登録するには、MRIイメージングソフトウェアを開き、左上隅にある患者のタブをクリックします。その後、[登録]をクリックします。
- 適切な患者情報を入力した後、試験のタブをクリックします。
- ローカライザースキャンを取得するには、新しいプロトコルを作成するために、試験のエクスプローラ]タブをクリックします。画面上のセットアップウィンドウを観察し、ルーチンタブをクリックし、次のパラメータを入力します。取得時間28秒、取得行列160×160、1スライス、厚さ1.6mmの等方性ボクセルサイズ、FOV = 260ミリメートル、FoVの段階= 100%、スライス分解能= 69%、相とスライス部分相フーリエ= 6/8、TR = 3.15ミリ秒、TE = 1.37ミリ秒、角度= 8°フリップ。
- 視床内の皮質下核をカバーするだけでなく、( 図4)の構造を囲むローカライザーの上にPD画像を取得するために使用されるスライス選択ボックスをオーバーレイ。
3.高解像度の構造パラメータ
- 高解像度PD加重スキャンを取得するための新しいプロトコルを作成します。画面上のセットアップウィンドウで、ルーチンタブをクリックし、冠状方向で次のパラメータを入力します。取得時間179秒、取得行列512×512、0.3×0.3×1ミリメートル3ボクセルサイズ、TR = 3.25秒、TE = 32ミリ秒、フリップ角= 120°、インターリーブスライス取得は、FOVがで= 160ミリメートル、FOVは相= 100%、パラレルイメージング(GRAPPA)を読み取ります2の加速係数。
- 32ミリ秒で5第1のエコーのエコートレインの長さで、ターボスピンエコーシーケンスを使用して、このシーケンスの効果的なエコーです。 SNRを最大化するために、可能な最小、40ヘルツ/ピクセルへの帯域幅(BW)を減らします。 FOV = 160ミリメートルで、それぞれ厚さ1mmを、スキャン時間を短縮18スライスを選択します。このスラブが関心の皮質下の領域に十分なカバレッジを提供します。
注:皮質下構造の信頼性の識別については、上記のパラメータで5ランを取得します。総スキャン時間はわずか〜15分( 図5)です。脂肪抑制を採用していませんでした。
- 32ミリ秒で5第1のエコーのエコートレインの長さで、ターボスピンエコーシーケンスを使用して、このシーケンスの効果的なエコーです。 SNRを最大化するために、可能な最小、40ヘルツ/ピクセルへの帯域幅(BW)を減らします。 FOV = 160ミリメートルで、それぞれ厚さ1mmを、スキャン時間を短縮18スライスを選択します。このスラブが関心の皮質下の領域に十分なカバレッジを提供します。
- 死後の脳イメージングでは、皮質下構造の信頼性の高い識別が3.1( 図6)のように、同一の走査プロトコルに従ってのみ〜3分の合計時間でちょうど1回のスキャンで観察することができます。
4.画像解析
注:MRIデータを分析するために、自由に利用できるFMRIBのを使用ソフトウェアライブラリからダウンロードできます(FSL)パッケージ(https://www.fmrib.ox.ac.uk/fsl/)。
- ターミナルウィンドウを開き、NIfTIコンバータへのDICOMとNIfTI形式に各PDボリュームのスキャナからの生のDICOMファイルを変換します。数は、ダウンロードのために自由に利用可能である( 例えば 、https://www.nitrc.org/projects/mricron)。コマンドラインでは、各PD加重画像ランのディレクトリが続くdcm2nii入力します。
- ターミナルウィンドウで、元のPDスキャンのパラメータを得ます。 NIfTI形式のPDのスキャンに続いて、コマンドラインでfslinfo入力します。
- 2倍の解像度と元のPDスキャンからfslinfoからパラメータで指定された半分のボクセルサイズを持つ高解像度の空白画像ターゲットボリュームを作成します。次のようにこのコマンドのデータ入力の順序は次のとおりです。
fslcreatehd <データ型> <ヘッダ名>
注:例えば、3.1で説明したように次のパラメータを使用して、元のPDスキャンが収集されている場合( すなわち、512×512マトリックス、18スライス、0.3×0.3×1ミリメートル3ボクセルサイズ、TR = 3.25秒)、次のように入力コマンドウィンドウに:
fslcreatehd 1024 1024 36 1 0.15 0.15 0.5 3.25 0 0 0 4 blankhr.nii.gz - 単位行列を使用して変換を定義します。テキストファイルは次のようになります」identity.mat」として保存された任意のテキストエディタプログラムを入力:
0 0 0
1 0 0
0 1 0
0 0 1 - 1024マトリックスに512からの全解像度を倍に、それぞれオリジナルのPD加重ランをアップサンプリング、変換を適用するために浮気コマンドを使用し、0.15×0.15の解像度が得られ、各次元におけるボクセルサイズを半減0.5 mmの3×。各PDのボリューム用の端末ウィンドウで、実行ごとにオリジナルと出力の名前を変更し、次の浮気のコマンドを入力します。
浮気-interpシンク-in originalPD.nii.gz -ref blankhr.nii.gz -applyxfm -init identity.matアウトhighresPD.nii.gz
注:originalPD.nii.gzはソースボリュームで、所望の出力解像度blankhr.nii.gzで、highresPD.nii.gzは出力ボリュームの名前です。 - 新しいフォルダにすべての高解像度の画像を移動し、ターミナルウィンドウで、それに移動します。
- 各参加者のために、fslmergeを使用して単一の4DファイルにすべてのアップサンプリングのPD画像を連結します。ターミナルウィンドウタイプの場合:
fslmerge -t concat_highresPD * .nii.gz
注:これはconcat_highresPD.nii.gzと呼ばれる4Dファイルを作成します。 - モーションmcflirt 10を使用して連結されたファイルを修正します。このツールは、リニアのための自動化された堅牢な登録(アフィン)間およびインターモーダル脳画像を可能にします。選択より高い精度のためのさらなる最適化パスとしてSINC補間(内部)を利用し、4段階補正、。ターミナルウィンドウタイプの場合:
mcflirt -in concat_highresPD -out mcf_concat_highresPD.nii.gz -stages 4 -plots
注:これはmcf_concat_highresPD.nii.gzと呼ばれる4Dファイルを作成します。 - 最後に、fslmathsを使用して画像を意味し、3Dを作成します。ターミナルウィンドウタイプの場合:
fslmaths mcf_concat_highresPD.nii.gz -Tmean mean_highresPD.nii.gz
注:これは、高品質であるmean_highresPD.nii.gzと呼ばれる3Dファイルを作成します。 - fslviewコマンドを使用して、最終的な結果の3D高解像度の画像を視覚化。あなたのイメージがどこにあるのディレクトリには、ターミナルウィンドウで次のように入力します。
fslview mean_highresPD.nii.gz。」 - 問題のROIの強度プロファイルを検査します。 fslviewを使用して、ROIを作成します(これは、例えばLGNの領域にわたって描かれた縦線をすることができます)。 fslviewで高解像度のPD画像をロードします。 [ツール]タブをクリックし、その後のROIを描画するための画像を拡大する単一の画像タブをクリックします。その後、マスクを作成し、タブが続く[ファイル]タブをクリックします。興味のROI内の線を引きます。ファイル、[名前を付けて保存]をクリックしてROIを保存します。強度の比較、問題の他のROIのためのROI内の複数の領域のためのラインマスクを繰り返します。
- 画像の結果の強度を分析するためにAFNIの3dmaskdumpコマンドを使用します。画像がどこにあるかのディレクトリでは、あなたのROIマスクの(result_mask.txtとして与えられた)画像強度と位置を抽出するために、ターミナルウィンドウで次のコマンドを使用します。
3dmaskdump -o result_mask.txt -noijk -xyz -mask ROI_linemask.nii.gz PDaverage_image.nii.gz
結果
皮質下の視床内処方されると、PD強調画像は、スライス選択ボックス( 図4)内に収集されます。 SNRは、両方の死後およびインビボスキャンにおける平均の数を増やすことによって改善されました。画像品質を決定するために、異なるスキャンの平均値からのSNRは脳外のいくつかのエリアに標準偏差で平均脳領域の信号を分割して比較しました。 SNRがμ 組...
ディスカッション
この研究は、皮質下領域の高解像度PD強調画像を得るために収集し、分析技術で最適化されたプロトコルを記述しています。スキャンパラメータの数を試験し、SNRを向上させ、取得回数、高解像度の皮質下構造を決定することができることで重要なステップを減少させるために、マトリクスサイズ、ボクセルサイズ、帯域幅に関連する最も重要なものに改変しました。生きている人間の中に?...
開示事項
The authors have nothing to disclose.
謝辞
The authors acknowledge the following funding sources, the Natural Sciences and Engineering Research Council of Canada (NSERC), the Dorothy Pitts Research Fund (NG), and the Nicky and Thor Eaton Research Fund. The authors acknowledge Kevin DeSimone, and Aman Goyal and for their knowledge in MRI acquisition and analysis expertise.
資料
| Name | Company | Catalog Number | Comments |
| Magnetom Trio 3T MRI | Siemens (Erlangen, Germany). | ||
| Vacuum cushion hand | Siemens | Mat No: 4765454 | Manufactured by: Johannes-Stark-Stk. 8 D-92224 Amberg |
参考文献
- Devlin, J. T., et al. Reliable identification of the auditory thalamus using multi-modal structural analyses. NeuroImage. 30 (4), 1112-1120 (2006).
- Fellner, F., et al. True proton density and T2-weighted turbo spin-echo sequences for routine MRI of the brain. Neuroradiology. 36 (8), 591-597 (1994).
- Schumann, C. M., Buonocore, M. H., Amaral, D. G. Magnetic resonance imaging of the post-mortem autistic brain. J Autism Dev Disord. 31 (6), 561-568 (2001).
- Tovi, M., Ericsson, A. Measurements of T1 and T2 over time in formalin-fixed human whole-brain specimens. Acta Radiol. 33 (5), 400-404 (1992).
- Fujita, N., et al. Lateral geniculate nucleus: anatomic and functional identification by use of MR imaging. Am J Neuroradiol. 22 (9), 1719-1726 (2001).
- Bridge, H., Thomas, O., Jbabdi, S., Cowey, A. Changes in connectivity after visual cortical brain damage underlie altered visual function. Brain. 131 (6), 1433-1444 (2008).
- Gupta, N., et al. Atrophy of the lateral geniculate nucleus in human glaucoma detected by magnetic resonance imaging. Br J Opthalmol. 93 (1), 56-60 (2009).
- Dai, H., et al. Assessment of lateral geniculate nucleus atrophy with 3T MR imaging and correlation with clinical stage of glaucoma. Am J Neuroradiol. 32 (7), 1347-1353 (2011).
- McKetton, L., Kelly, K. R., Schneider, K. A. Abnormal lateral geniculate nucleus and optic chiasm in human albinism. J Comp Neurol. 522 (11), 2680-2687 (2014).
- Jenkinson, M., Bannister, P., Brady, M., Smith, S. Improved optimization for the robust and accurate linear registration and motion correction of brain images. NeuroImage. 17 (2), 825-841 (2002).
- Dietrich, O., Raya, J. G., Reeder, S. B., Reiser, M. F., Schoenberg, S. O. Measurement of signal-to-noise ratios in MR images: influence of multichannel coils, parallel imaging, and reconstruction filters. J Magn Reson Imaging. 26 (2), 375-385 (2007).
- Andrews, T. J., Halpern, S. D., Purves, D. Correlated size variations in human visual cortex, lateral geniculate nucleus, and optic tract. J Neurosci. 17 (8), 2859-2868 (1997).
- Pfefferbaum, A., Sullivan, E. V., Adalsteinsson, E., Garrick, T., Harper, C. Postmortem MR imaging of formalin-fixed human brain. NeuroImage. 21 (4), 1585-1595 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved