Method Article
の新生RNA FISHによる転写分析
要約
Trophoblast giant cells (TGCs) play a key role in the placenta to ensure a healthy pregnancy. We present a protocol for assessing the transcriptional status of genes in TGCs by nascent fluorescent in situ hybridization on cryostat sections of post-implantation embryos or short-term cultures of embryonic day 7 ectoplacental cones.
要約
胎盤は、1胚体外系統、栄養外胚葉に由来します。内部細胞塊を覆う極性栄養外胚葉が、後に二次TGCsに分化増殖し続けている間ペリ移植マウス胚盤胞では、壁画栄養外胚葉細胞は、初代栄養膜巨細胞(TGCs)に分化します。 TGCsは、胎盤の開発に重要な役割を果たし、妊娠の成功のために不可欠です。移植後の開発中に特定の遺伝子の転写調節の検討TGCs開発への洞察を与えることができます。極性栄養外胚葉由来妊娠(E7-7.5)の7-7.5日目の胚からの栄養膜円錐(EPC)の細胞は、二次TGCs 1に分化します。 TGCsの数は、この段階では非常に低いがTGCsはE7で胚のクリオスタット切片上で、 その場で研究することができます。二次TGCsを分析する代替手段E7胚から個々のEPCの短期培養を使用することです秒。我々は、新生転写産物を可視化するために、in situハイブリダイゼーション(RNA FISH)、蛍光を使用して、単一細胞レベルでのインビボおよびインビトロの両方において目的の遺伝子の転写状態を調査する手法を提案します。この技術は、遺伝子発現の直接的な読み出しを提供し、大endoreplicating細胞ひとつは、TGCの染色体の状態の評価を可能にします。実際、TGCsの分化の重要な特徴は、それらが細胞周期を終了し、endoreplication.Thisアプローチの複数ラウンドを受けることである常染色体および/または性染色体から発現される任意の遺伝子の発現を検出するために適用することができ、発達に重要な情報を提供することができますメカニズムだけでなく、胎盤の疾患。
概要
哺乳動物の栄養膜巨細胞(TGCs)、母体と胚組織間の障壁を形成します。彼らは、子宮に着床および受胎の侵入を媒介し、胎盤の形成のための開発に重要な役割を果たしています。彼らは、胚の成長と生存のために必要ないくつかの増殖因子(サイトカイン)およびプロラクチン/胎盤性ラクトゲンの家族やステロイドのホルモンを産生します。 TGCsはSとG相が交互に構成され、細胞周期、endocycle、で、大単核および倍数体細胞です。実際、TGCsは、任意の部門2せずにDNA合成の複数のラウンドを受けることができる細胞をendoreplicatingされています。他の胚及び胚外細胞型に比べTGCs インビボでの遺伝子の転写状態を調べるために、新生RNA FISHは、特定の移植後の段階でのクリオスタット切片3にインビボで行うことができます。 TGCsはtheiによる切片上で容易に認識されています大きなサイズとそれらの転写遺伝子活性を記録することができるが、初期の移植後の段階でそれらの数が少ないrは。 E7胚切片上TGCsの不足は、栄養外胚葉の開発中に遺伝子発現の調節を研究するために、差別TGCsを得るために、胚の栄養外胚葉組織の短期培養を行うために私たちを導きました。また、できるだけ生理として残り、樹立細胞株、すなわち栄養外胚葉細胞(TS)を食い止めるために、常に二次TGCsの発達メカニズムの生成を調査するのに適していない異常な遺伝子にTGCsの欠陥に関連する病理学的妊娠を研究するための貴重なツールを提供マウスでの規制。
栄養膜円錐(EPC)の栄養膜細胞は、二次TGCs 4の前駆体です。二次TGCsまで培養EPCの自発的分化は以前に5報告されています。しかし、一次TGCs、二TGCのdifferenの研究とは対照的にtiationはpresumablydue任意の母性や胚組織の自由EPC外植片を単離する難しさに、制限されたままです。私たちは、E7、ひとつは、TGCが非常に少ないですが、EPC前駆体から生成される可能性発達移植後の段階で個々の胚由来の二次TGCsのRNA FISHを行うためには、これらのメソッドを適応しました。核の一次転写産物を分析するためのRNA FISHは、二次TGCs上の単一細胞レベルのレベルで行われていませんでした。これは、転写の正確な分析を可能にし、移植後のステージ3でTGCsのエピジェネティックな不安定性を示すために使用されました。
哺乳類におけるエピジェネティクスの古典的な例は、X染色体不活性化(XCI)は聞いたラボ6で検討されています。このプロセスでは女性で2 X染色体の一方が不活性化されます。 Xist転写コートそれが雌の細胞で発現され、ほとんどの遺伝子のサイレンシングをトリガされたX染色体非コード。 RNAのFISHを使用して、X連鎖遺伝子の新生転写産物は、不活性X染色体(XI)上のXist RNAの蓄積はできる限り調査することができます。ここでは、移植後の胚の切片上およびEPCの短期培養にRNA FISHを実行するための手順について説明します。このプロトコルは、女性の胚性幹細胞および着床前胚7-11を分化にXCIを研究するために使用されたものから適合しました。我々は、in vitro TGCsと同様に、in vivoでの女性の胚でXCIの例を提供します。
プロトコル
動物の手順はキュリー研究所(CEEA-IC)のプロトコル(C 75-05-18)の承認制度的動物管理使用委員会に合わせて行われました。仕事はまた、遺伝子組換え生物(契約番号5549CA-I)の使用のための高等教育と研究のフランスの省からの承認の下で行われています。
凍結切片の調製
- シェイら 、12に記載されているように自然にF1 C57BL / 6のx DBA / 2Jマウスを排卵から胚を収集します。妊娠(E7)の7日目に、頚椎脱臼により8-12週齢のマウスを生け贄に捧げます。全体受胎すなわち脱落膜12を収集します。
- シェイらに記載されているように、E7の受胎を分離します。、12。 PBSを含む60ミリメートルペトリ皿にそれらを配置します。
- クリオスタット切片のためのE7マウス受胎をフリーズします。
- ガラスパスツールピップを使用して、高さが適合したアルミ箔ウェル1cmの( '自家製'を準備しますら)。この小さな容器の底部に組織凍結培地のドロップを堆積させます。正しい方向に受胎を堆積させる(すなわち、長手方向のセクションの受胎は、その水平方向に維持する必要があります)。
- 組織は凍結培地とよく含む受胎を埋めます。それが白くなるまで数秒間液体N 2中のブロックを浸す-それはゆっくりとフリーズすることを可能にするために液体N 2以上の蒸気中の鉗子でそれを一時停止します。続いて、(数ヶ月間保存することができます)-80℃で凍結バイアルとストアにブロックを転送します。
- クライオセクショニングする前に、30分間のクライオスタットに-20℃で受胎を含む凍結したブロックを配置します。 8ミクロンの厚さの凍結切片を実行します。デポジット効率的な添付ファイルを有効にするには、スライド上の4つのセクション。 18×18 mm 2のカバーグラスの下に収まるほど近いセクションを配置します。
- の品質をチェックセクション(無傷や傷なし)と実体顕微鏡で胚(縦断面)の向きとは、さらなる分析のために適切なセクションを選択します。可能な限り迅速にコプリンジャーにそれらを堆積し、(3.3.2を参照)RNA FISHを行います。
セカンダリTGCsの調製
- 受胎を分離し(1.1と1.2を参照してください)。
- E7栄養膜円錐(EPC)を解剖
- 実体顕微鏡と滅菌PBSを含むペトリ皿を使用してください。鉗子で脱落膜を解剖。ピアース離れて脱落膜の両側を引き裂くためのサンプルとオープン鉗子。
- 胚を殻から取り出します。適切な胚からEPCを分離するために、はさみのようなアクションで閉じられた微細な鉗子(デュモン第5号)のヒントを参考にしてください。絨毛膜と卵黄嚢から母体の組織から分離するだけでなく、完全にきれいサンプルを得るために特別な注意を払ってください。 PBSでEPCの外植片を洗浄します。
- 少しスプーンで、転送個体は4 - WELにEPCを解剖しました培地中の滅菌カバースリップを含むLプレート。
- EPCは短期文化からTGCsを求めよ。
- カバースリップを含む4ウェルプレートを準備します。火炎乾燥し、エタノール中に浸漬することによってカバーガラス丸い直径12mmを滅菌します。
- 各ウェルに0.5ミリリットルEPC培地を追加します(EPC培地:RPMI 1640、15%のFCS、0.1mMの2-メルカプトエタノール、および抗生物質を補充しました)。
- 預金シングル、培地と一緒にウェル中のカバースリップの中央に解剖-のEPC。カバーガラス上にEPCを適用するために微細な鉗子を使用してください。 37℃、5%CO 2で3-5日間Culturefor。個々の外植片を平坦化TGCs( 図2A)の単層として広がる伸長を形成しています。
3. RNA FISH
注:プロトコルは、Chaumeilらに胚性幹細胞(ESC)について記載したものに基づいて8と第一指と聞いた13されています。
- 高度なPrepa原液の比
- 3M酢酸ナトリウム緩衝液pH 5.2を準備します。
- 4倍生理食塩水クエン酸ナトリウム(SSC)で40%(w / v)のデキストラン硫酸ナトリウム、20X BSA、400mMのバナジルリボヌクレオシド複合体(VRC)を含む2×ハイブリダイゼーション緩衝液を準備します。
- PBS中のp-フェニレンジアミン、pHが9(w / v)の90%(v / v)のグリセロール、0.1%を含有する封入剤を準備します。
- 新たに調製されたソリューション
- PBSで3パーセント新たに調製したパラホルムアルデヒド(PFA)からなる固定液を準備します。 PBS中0.5%トリトンX-100を含有する透過液を調製すると、2mMのVRCを補充しました。 50%ホルムアミド(FA)と2×SSCを混合することにより、洗浄緩衝液を準備し、7.2から7.4にpHを調整します。
- 2×SSC中1μg/ mlのDAPI(4 '、6-ジアミジノ-2-フェニルインドール二塩酸塩)からなるDNAカウンター染色溶液を調製します。
- FISHのための準備中で固定し、透過処理
- カバースリップ上の細胞は、5メートルのために1×PBSで細胞を洗浄で。RTで固定液中で10分間(3%PFA)ソリューションを、カバースリップ上の細胞、またはスライド上の胚のセクションを修正しました。
- 1×PBSで細胞を3回洗浄します。氷氷のように冷たい透過性溶液中で5分間、細胞を透過性。 70%エタノールで細胞を3回洗浄します。 -20℃で70%エタノールで4スライド輸送箱に保管し4ウェルプレート中のカバースリップとスライド。
注:カバーガラスとスライドは-20℃で数ヶ月間保存することができます。板およびそれらを含むボックスは、エタノールの蒸発を避けるためにパラフィルムで密封されなければなりません。
- DNAプローブ標識
- 蛍光ヌクレオチドを使用して、ニックトランスレーションによってDNAプローブにラベルを付けます。製造業者の指示( 表1を参照)に従う8。
- (DNAの増幅のために、3.4.4を参照)50μlの反応ミックスのため、1-2プラスミドμgの、細菌人工染色体(BAC)または複数置換増幅(MDA)を追加し、17.5μlの水にDNAを2.5μlの0.2 mMのSR-、SG-、Cy5でdUTPを、10μlの10 mMの各dNTPミックス(のdGTP、dATPを、dCTPを)、5μlの10 mMのdTTPを、5μlの10×ニックトランスレーションバッファーと8μlのニックトランスレーション酵素。
- 暗所で15℃にて16時間インキュベートします。 -20℃で凍結することによって反応を不活性化します。 -20℃で数ヶ月のためのストアプローブ。
- MDA
注:BACのDNAについては、古典的な準備の後に得られたDNAの量が低いので、1はMDAを使用することにより、標識の前にDNA増幅のステップを使用することができます。市販のキットを使用し、製造業者の指示( 表1を参照)に従います。 - 9.5μlのサンプルバッファーで0.5μlのBAC DNAを混ぜます。 95℃で3分間加熱します。直ちに氷上にクエンチ。氷上で10分を残します。反応緩衝液/酵素混合物を準備します(9.5μlの+ 0.5μlの酵素ミックス)、氷上に保ちます。
- 10μlのDNAミックス+10μlの反応緩衝液/酵素ミックスを混ぜます。 30℃で少なくとも20時間インキュベートします。 65&#で試料10分間加熱して酵素を不活性化します176; C。 -20℃で保存する前に4℃に冷却したサンプル。
- 消化によってMDAを確認してください。 1μlのMDA、2μlの10×バッファー、2μlのHindIIIおよび15μlのH 2 Oを追加37℃で一晩インキュベートします。正確なDNA増幅を確認するために、0.8から1パーセントアガロースゲル上でゆっくり実行します。高分子量の明確なDNA断片をゲル上で観察されるべきです。
- プローブの準備
- カバースリップまたはスライドあたり0.1または1μgのプローブを使用してください。
注:競争は、 例えば、ほとんどのプローブを必要とする場合、彼らはそれ以外の場合は、クロスハイブリダイズ反復配列を含み、背景を増加させることができるように簡易ベッド-1 DNAの2-5μgのを追加します。- 沈殿のために、5μgのサケ精子DNA、1/10量5.2と3倍量のエタノールを3 M酢酸ナトリウムのpHを追加します。 16,000×gでスピンし、25分間、4°C。 70%エタノールでペレットを洗浄し、5分間再びスピンダウン。
- コンセントレータ/スピードバックで2分間乾燥したペレット。 100%の形で再懸濁ハイブリダイゼーションのために必要な半分の容量でアミド(カバースリップまたはスライド上の胚のセクションの7μlのための例えば、2.5μl)を。サーモミキサーで振とうしながら37℃で30分間置きます。 75℃で7分間の変性。
- 氷上でクエンチ、または競争は30〜60分間、37℃で直接置く必要な場合。 2Xハイブリダイゼーション溶液の等量のプローブ溶液を混合します。
- カバースリップまたはスライドあたり0.1または1μgのプローブを使用してください。
- ハイブリダイゼーションおよび洗浄
- 脱水; 5分ごとに、1×80%、1×95%と2倍の100%エタノールで逐次洗浄によるコプリンジャーのウェルでカバースリップ、スライド、。完全に乾燥したカバースリップとスライド。
- ハイブリダイゼーションのために、ハイブリダイゼーションミックスに直面している細胞を用いて、スライド上に5μlのプローブハイブリダイゼーション混合物を(3.5を参照)を適用し、カバースリップを下げます。スライド上のセクションについては、14μlのプローブハイブリダイゼーション混合物を適用する(3.5を参照)、18×18 mm 2のカバーガラスでカバーしています。
- に浸した加湿チャンバー(ティッシュペーパーに入れスライド50%FA / 2×SSC)、暗所で、37℃で一晩インキュベートします。
- ハイブリダイゼーション後の洗浄:
- それを緩めるために、スライド上のカバースリップ上に50%のFA / 2×SSCの1ミリリットルを追加します。スライド(細胞をこすりしないように注意しながら)から慎重にカバースリップを除去し、50%FA / 2×SSCを含む4ウェルプレートにそれをアップセル側に配置します。スライド上のセクションについては、同様の方法でカバースリップを除去し、50%FA / 2×SSCを含むコプリンジャーにスライドを配置します。
- それぞれ、カバースリップまたはスライドを42℃または44℃に予め加温した50%FA / 2×SSCで7分間3回の洗浄、それぞれを実行します。
- それぞれ、カバースリップまたはスライドを42℃または44℃で5分間ずつ予熱した2×SSCで3回の洗浄を行います。
- 室温で3分間に1μg/ mlのDAPIで2×SSCで洗浄することにより核を対比染色。 2×SSCで二回洗浄します。
- カバーガラスとスライドの取り付け
- 培養TGCsから小さなカバースリップのために、5&#を適用181; lは、スライド上のメディアをマウントします。ドロップの上にカバースリップ細胞側を下に置きます。
- セクションを有するスライドは、スライド上のメディアをマウント15μLを適用します。ドロップの上に22×22 mm 2のカバーガラスを配置します。
- 気泡を避けてください。過剰実装ソリューションを拭き取ってください。マニキュア少量のカバーガラスをシール。可能な場合は、画像を-20℃で数ヶ月に直ちにまたはストアアップのためにスライド。
4.顕微鏡および分析
- 蛍光顕微鏡(63X対物レンズ)を用いて0.3ミクロンで3D順次z軸画像を取得し、ImageJソフトウェア14を使用して、3D画像スタックの解析を行います。
結果
TGCs起因受胎での局在およびそれらの大きいサイズにDAPI染色時E7セクションに識別することができます。これは、縦断面の図1Aに示されています。 RNA FISHは、この胚体外系統にX染色体の不活性化を研究するために、このような胚の切片上で実施しました。
いくつかのスライドまたはカバースリップを同時に処理することができます。異なる遺伝子のRNAを標識するために、異なる染料を使用することによって、人は、同じ核内に異なる一次転写産物を検出することができます。インスタンスのXistは SG(緑シグナル)及びSR(赤色信号)に結合されたATRXに結合するための少なくとも2つのプローブは、一緒に混合することができます。女性のTGCの例は、2他の系統が表現されている図1B、適切な胚(E)及び内臓内胚葉(VE)に示されています。 TGCの核は、X CHROMをカバーするのXist RNAで示されていますアクティブになっている他のX染色体(Xaは)いくつかのピンポイントが表示されている間(西)不活性化されosome。 X連鎖遺伝子ATRXの一次転写産物は、内部複製のためにいくつかのコピーで( のXistによって装飾されていない)、アクティブX染色体から発現されます。これは、1アレルの発現例えば、1つのX染色体上のATRXの不活性化を示しています。
図2Bに示すように、ひとつは、TGCが、サイズが不均一であるので、唯一の最大のものが記録されています。二TGCsは、 図2Cに示すように、3つのプローブを使用することもできます。この場合、 のXist -SG(緑)は、二つのX連鎖遺伝子:G6PD -SR(赤)とHuwe1 -Cy(マゼンタ)は、同じ核内で同時に分析しました。ここに示されている(と以前に二次TGCsに示す3)としてG6PDは monoallelically Huwe1が biallelicallyそのPRを実証表現され発現されるがXCIを脱出するoperty。内部複製は、いくつかのピンポイントすなわち、新生転写物のコピーでも明らかです。
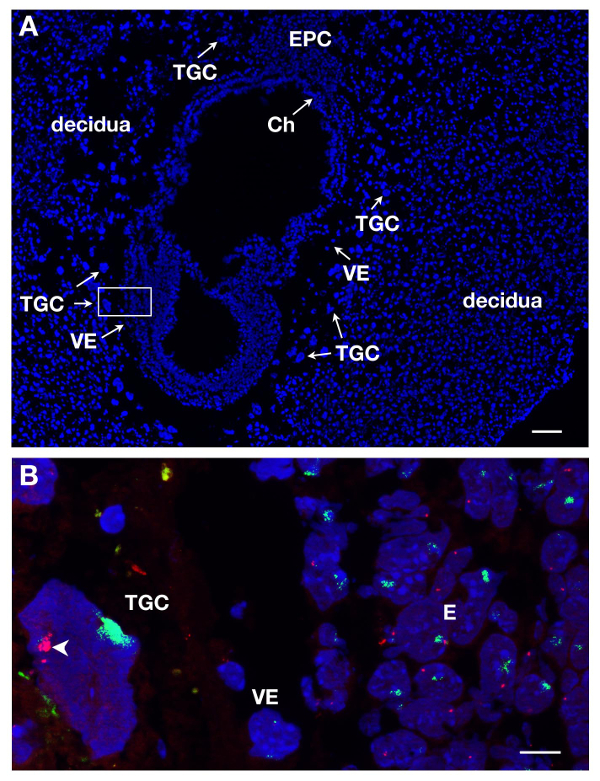
E7女性の胚セクションからTGCs図1.一次転写産物発現。(A)DAPIで染色したE7の受胎の縦断面図。胚および胚体外組織は母組織、脱落膜に囲まれています。異なる系統の局在は、5Xの目的とDAPI染色の際に行われます。 TGCsはサイズが大きいために同定されています。 Chの、絨毛膜、E、胚、EPC、栄養膜円錐、VE、臓側内胚葉、TGC、栄養膜巨細胞。スケールバー=100μmです。 (B)(63X対物レンズ)で箱入りイン領域の高倍率のRNA FISH。ATRX一次転写物の赤とのXist RNAに緑の中を3系統= E上で可視化され、TGCをVE。皮下=10μmのエールバー。 XistコーティングされたX染色体上のin vivoで ATRXが monoallelically表現されるTGC(第Xa、矢じり)およびサイレント(西)の例。第Xaに見られるいくつかのATRX信号は、内部複製によるものである。ATRX = BACクローンRP23-260I15。 この図の拡大版をご覧になるにはこちらをクリックしてください。
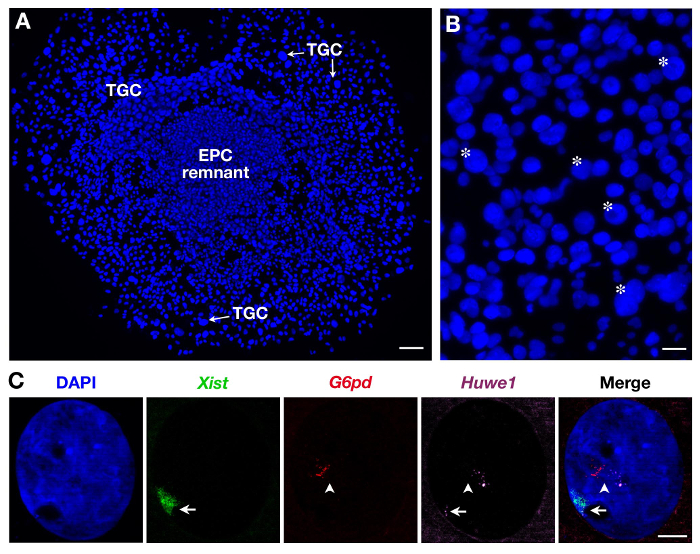
図2. EPC由来の二次TGCsにおける一次転写物発現。(A)成長している二次TGCs(×5の対物レンズ)の一般的な見解。スケールバー=100μmです。 (B)アスタリスクは、そのサイズ(10×対物レンズ)に従ってTGCsの例を示しています。 =60μmのスケールバー。 (C)3プローブ( のXist土間を用いた二次女性TGC RNA FISHの例西)()赤とマゼンタのいくつかのピンポイントを、G6PDとHuwe1一次転写物の内部複製を示す:緑色で、不活性化X染色体をカバーインチG6PDが monoallelically発現されるが、Huwe1式は、この核内の二対立遺伝子であるHuwe1 = BACクローンRP24-157H12; G6PD = BACクローンRP23-13D21。西=矢印;第Xa =矢じり(63X対物レンズ)。スケールバー=10μmである。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
新生RNA FISHは、異なる発生段階の胚組織において転写活性の単一細胞解析のための簡単かつ高感度な方法を表しています。このアプローチのパワーは、形態学的基準に従って、任意の特定の段階で異なる胚系統を識別するための能力です。しかし、これはまた、最小のバックグラウンド蛍光が存在することを必要とします。任意のこのような背景に挑戦異なる胚領域および細胞型の同定をレンダリングします。最小限のバックグラウンドを確保するために、このプロトコルで2つの重要なステップがあります。最初の凍結切片の品質であり、第二は、遺伝子発現のレベルおよびプローブの品質に依存RNA FISH、効率(信号対雑音比)です。後者については、プローブは、したがって、常にセクションで使用する前にカバーガラス上で培養した細胞(胚性幹細胞または体細胞)で試験されています。
このプロトコルでは、目を提供します電子技術は、サンプル調製(クライオスタット切片及び胚性外植片の初代培養物)の二種類からTGCsのRNAのFISH分析を実行するために必要。このような単一細胞分析を評価するために、異なる移植後の段階での遺伝子発現の動的変化を可能にします。
我々は(E7-7.5移植後の段階での)二次TGCsを生成する記述方法は、移植後の段階でTGCsで構成されている胚体外系統のX染色体不活性化パターンを研究するために、我々の研究室で使用されました。げっ歯類の胚体外開発はTGCsの分化に依存します。実際、TGCsは、胎盤、したがって、胚発生に必須です。 TGC分化における欠陥は、(参照15を参照のレビューのために)胚性致死を引き起こします。 RNA FISHを使用してTGCsの転写活性は、私たちは、マウスの開発3中にこのような細胞型の異常なX染色体不活性化の状態を実証することができました。
取得し、二次TGCsを研究するためにここに提示される方法は、正常および変異マウスの両方で、彼らはの一部である重要な胚体外組織の開発に分子経路を研究するために適用することができます。これらのアプローチは、他の哺乳動物におけるTGC発達を調査するために適合させることができます。我々の分析は、私たちは、インビボで TGCsにし、EPCの外植片由来のin vitro TGCsに様々な遺伝子の転写活性を評価することができ、野生型マウス胚の使用を含んでいました。この方法は、トランスジェニックマウスの分析および/または培養培地中の阻害剤分子を添加することによって拡張することができます。私たちは、このような成長を開発している間、4-5日ごとに個別に培養することができる初代マウス開発の早い段階で表示されTGCs、E3.0のblastocyts、として、TGCsの別の種類にRNA FISHのこのメソッドを正常に使用していますICMは、大きな一次TGCs 16,17に囲まれています。異なるため、新生転写産物遺伝子と同様のXistを可視化し、同一のRNA FISH法3を用いて定量することができました。
合わせた免疫蛍光法およびRNA FISHはまた、インビトロで培養TGCs 3と同様クリオスタット切片上で行うことができます。これは、一次転写物が分解を非常に受けやすいとRNaseを含まない化合物の絶対的な要件があるような方法は、非常に堅牢であることを示しています。 IF / RNA FISHは同時に与えられたセルでは、転写レベル、細胞局在およびタンパク質発現に関する追加情報を提供します。 、パラフィン包埋組織の切片を以前に我々の手で、組織形態18を維持しながら、ヒト腫瘍における新生RNAを検出するために使用されてきたが、クライオスタット切片は、新生RNAおよび免疫蛍光中の抗体による検出のために必要なエピトープの両方を保存するためのより適切です。
イム以下、RNA FISHに加えて、munofluorescence、染色体DNAのFISHはまた、RNA FISH 3のために使用されるものと同様の蛍光色素で標識されたプローブを用いた二次TGCsに行うことができます。蛍光プローブは、いずれかのプラスミド/フォスミドまたはBACの、ここで使用されるような蛍光dUTPsで標識し、Chaumeil ら 、8で説明することができます。あるいは、蛍光標識されたオリゴヌクレオチドは、19を使用することができます。 DNA FISHは、鎖特異性を必要としないので、オリゴヌクレオチドプローブは、標的領域内の2つの相補鎖のいずれかを標的とするように設計することができます。シグナル増幅を特定し、アンププローブの2つの連続ラウンドによって達成される分枝DNAプローブは、また、FISH信号20の信号対雑音比を向上させるために使用することができます。我々のDNAベースのプローブにおいて信号品質、特異性および使いやすさの間のより良いトレードオフを保証するが、代替的に、例えば、リボプローブのようなRNAプローブを使用することができます。
最後に、3D画像acquisitiこれらの大きなセルで必要な空間情報を得るために必須であり、そのようなDeltavision(GEH)などのデコンボリューションを有するそのようなApotome顕微鏡などの蛍光顕微鏡、または落射蛍光顕微鏡のさまざまな方法を使って行うことができる、または他の顕微鏡の適切な撮像組織切片、および大きいため(>20μM)TGCs。単一コピーDNA遺伝子座のために、新生RNA FISHまたはDNA FISHにより検出された点状信号は容易に共焦点顕微鏡により検出されないことに留意すべきです。
結論として、私たちはここで説明する方法は、発生コンテキストでTGSの詳細な分析のためだけでなく、他の疾患の状況の両方に有用であるはずです。げっ歯類におけるTGC発達および機能に関与する多くの遺伝子は、転写因子、プロテアーゼおよび細胞接着分子21として、げっ歯類とヒトの間で保存されています。マウスTGCsは、胎盤開発ANを調節する遺伝子を研究するための細胞モデルでありますdはしたがって、ヒト胎盤疾患への洞察を与えます。また、原因これらの細胞はendoreplicatingされ、いくつかの癌細胞はendocycleプログラムを係合するため、遺伝子増幅法に加えて、我々は、記述の方法はまた、癌細胞中での不安定性をゲノムにつながるメカニズムを探るために必要な単一細胞研究に有用であるべきであるという事実に。
開示事項
The authors have nothing to disclose.
謝辞
私たちは、原稿、動物飼育施設やユニットのイメージングプラットフォームを読み取るためのイラストのヘルプのためのソフィーGournet、ジュリーChaumeilに感謝します。この作品は、プログラム«INVESTISSEMENTSドールアベニール»下で支援を受けた参照ANR-10-LABX-0044とANR-10-IDEX-0001から02 PSL、EpiGeneSys FP7なしでフランス政府によって起動され、ANRにより実施しています。 EHへエクセレンスの257082ネットワーク、ERC上級調査官賞なし。 250367とEU FP7 SYBOSSはありません付与します。著者らは光で助けをキュリー研究所、フランス・バイオイメージング(ANR-10-INSB-04)のメンバー、の遺伝学と発生生物学部門(UMR3215 / U934)の細胞と組織イメージングプラットフォームを感謝したいEHへ242129顕微鏡。
資料
| Name | Company | Catalog Number | Comments |
| Stereomicroscope | Nikon | SMZ 1500 | |
| Stereomicroscope | Zeiss | Stemi SV6 | |
| Scissors Pascheff-Wolff | Moria | MC19 | |
| Dumont #5 forceps | Roth | PK78.1 | |
| 4-well tissue culture dishes | Nunc | 176740 | |
| 60 mm Petri dishes Falcon | Dutsher, France | 353004 | |
| 100 mm Petri dishes Falcon | Dutsher, France | 353003 | |
| Coverslips 18 mm x 18 mm | VWR | 631-1331 | |
| Coverslips 22 mm x 22 mm | VWR | 631-0125 | |
| 12 mm glass round coverslips | Harvard apparatus | 64-0712 | |
| Slides Superfrost plus | VWR | 631-9483 | |
| 4-slide Transport box Lockmailer | Dutsher, France | 40684 | |
| Cryotubes 1.8 ml Corning | Fisher Science | 10418571 | |
| Glass Coplin staining jars | Fischer Scientific | W1561L | |
| TissueTek O.C.T compound | VWR | 4583 | |
| RPMI 1640 medium | Invitrogen | 61870 | |
| Phosphate buffered saline (PBS) | Sigma-Aldrich | D1408 | 10x is used |
| Water | Sigma-Aldrich | W3500 | |
| Paraformaldehyde | Panreac Quimica, Spain | 141451 | 3% in PBS |
| Triton-X-100 | Euromedex | 2000-A | 0.5% final |
| Vanadyl ribonucleoside complex (VRC) | New England Biolabs, USA | S1402S | |
| Sodium dextran sulfate | Sigma-Aldrich | D8906 | |
| Bovine serum albumin (BSA) | New England Biolabs, USA | B9001S | |
| Formamide | Sigma-Aldrich | 47671-1L-F | aliquots kept at -20 °C |
| Illustra TempliPhi Kit Construct (Kit MDA) | Dutsher, France | 25-6400-80 | |
| Nick translation kit | Abbott, USA | 07J00-001 | |
| 20x SSC buffer concentrate | Sigma-Aldrich | S6639 | |
| Spectrum green dUTP | Abbott, USA | 02N32-050 | |
| Spectrum red dUTP | Abbott, USA | 02N34-050 | |
| Cy-5 dUTP | Dutsher, France | PA55022 | |
| Mouse Cot-1 DNA | Invitrogen | 18440016 | |
| DNA, MB grade | Invitrogen | Roche | DNA from fish sperm |
| 4′,6-diamidino-2-phenylindole dihydrochloride | Sigma-Aldrich | D9564 | DAPI |
| Glycerol | Sigma-Aldrich | G9012 | |
| p-phenylenediamine | Sigma-Aldrich | 695106 | |
| Centrifuge 5417R | Eppendorf, Germany | molecular biology grade | |
| Eppendorf concentrator plus | Eppendorf | ||
| Eppendorf Thermomixer comfort | Eppendorf | ||
| Liquiport Liquid pump | KNF Neuberger, Trenton, USA | ||
| Shake'N'Bake Hybridization oven | Boekel Scientific, USA | ||
| Cryostat | Leica | CM3050 |
参考文献
- Cross, J. C. Genetic insights into trophoblast differentiation and placental morphogenesis. Sem. Cell Dev. Biol. 11, 105-113 (2000).
- Zybina, E. V., Zybina, T. G. Polytene chromosomes in mammalian cells. Int. Rev. Cytol. 165, 53-119 (1996).
- Corbel, C., Diabangouaya, P., Gendrel, A. -. V., Chow, J. C., Heard, E. Unusual chromatin status and organization of the inactive X chromosome in murine trophoblast giant cells. Development. 140, 861-887 (2013).
- Rossant, J., Tamura-Lis, W. Effect of culture conditions on diploid to giant-cell transformation in postimplantation mouse trophoblast. J. Embryol. Exp. Morphol. 62, 217-227 (1981).
- El-Hashash, A. H., Kimber, S. J. Trophoblast differentiation in vitro: establishment and characterization of a serum-free culture model for murine secondary trophoblast giant cells. Reproduction. 128, 53-71 (2004).
- Chow, J. C., Heard, E. X inactivation and the complexities of silencing a sex chromosome. Curr Opin Cell Biol. 3, 359-366 (2009).
- Chaumeil, J., Okamoto, I., Heard, E. X-chromosome inactivation in mouse embryonic stem cells: analysis of histone modifications and transcriptional activity using immunofluorescence and FISH. Methods Enzymol. 376, 405-419 (2004).
- Chaumeil, J., Augui, S., Chow, J. C., Heard, E. Combined immunofluorescence, RNA fluorescent in situ hybridization, and DNA fluorescent in situ hybridization to study chromatin changes, transcriptional activity, nuclear organization, and X-chromosome inactivation. Methods Mol. Biol. 463, 297-308 (2008).
- Okamoto, I., Otto, A. P., Allis, C. D., Reinberg, D., Heard, E. Epigenetic dynamics of imprinted XCI during early mouse development. Science. 303, 644-664 (2004).
- Okamoto, I., Arnaud, D., Le Baccon, P., Otte, A. P., Disteche, C. M., Avner, P., Heard, E. Evidence for de novo imprinted X-chromosome inactivation independent of meiotic inactivation in mice. Nature. 438, 369-373 (2005).
- Patrat, C., Okamoto, I., Diabangouaya, P., Vialon, V., Le Baccon, P., Chow, J., Heard, E. Dynamic changes in paternal X-chromosome activity during imprinted X-chromosome inactivation in mice. Proc. Natl. Acad. Sci. 106, 5198-5203 (2009).
- Geijsen Shea, K., N, Dissection of 6.5 dpc mouse embryos. J. Vis. Exp. (2), e160 (2007).
- Pollex, T., Piolot, T., Heard, E. Live-cell imaging combined with immunofluorescence, RNA, or DNA FISH to study the nuclear dynamics and expression of the X-inactivation center. Methods Mol. Biol. 1042, 13-31 (2013).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- Hemberger, M. IFPA award in placentology lecture - characteristics and significance of trophoblast giant cells. Placenta. 29, 4-9 (2008).
- Carney, E. W., Prideaux, V., Lye, S. J., Rossant, J. Progressive expression of trophoblast-specific genes during formation of mouse trophoblast giant cells in vitro. Mol. Reprod. Dev. 34, 357-368 (1993).
- Shin, J., et al. Maternal Rnf12/RLIM is required for imprinted X-chromosome inactivation in mice. Nature. 467, 977-981 (2010).
- Capodieci, P., Donovan, M., Buchinsky, H., Jeffers, Y., Cordon-Cardo, C., Gerald, W., Edelson, J., Shenoy, S. M., Singer, R. H. Gene expression profiling in single cells within tissue. Nat Methods. 9, 663-665 (2005).
- Beliveau, B. J., Joyce, E. F., Apostolopoulos, N., Yilmaz, F., Fonseka, C. Y., McCole, R. B., Chang, Y., Li, J. B., Senaratne, T. N., Williams, B. R., et al. Versatile design and synthesis platform for visualizing genomes with Oligopaint FISH probes. Proc. Natl. Acad. Sci. 109, 21301-21306 (2012).
- Kenny, D., Shen, L., Kolberg, J. A. Detection of viral infection and gene expression in clinical tissue specimens using branched DNA (bDNA) in situ hybridization. J. Histochem. Cytochem. 50, 1219-1227 (2002).
- Cross, J. C., Baczyk, D., Hemberger, M., Hugues, M., Simmons, D. G., Yamamoto, H., Kingdom, J. C. Genes, development and evolution of the placenta. Placenta. 24, 123-130 (2003).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved