このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
免疫蛍光によるDNA修復タンパク質相互作用の可視化
要約
DNA損傷の後、ヒト細胞はゲノムの完全性を回復するために不可欠な修復経路を活性化する。ここでは、DNA修復タンパク質を検出し、その空間的および時間的なリクルートメントを分析し、DNA損傷部位におけるタンパク質とタンパク質相互作用を問い合わせた手段として、間接的な免疫蛍光の方法を説明する。
要約
哺乳類の細胞は、常に化学物質、放射線、および自然発生する代謝副産物にさらされ、特定のタイプのDNA侮辱を引き起こす。遺伝子毒性物質は、DNA骨格を損傷したり、破壊したり、個々の塩基の化学的性質を改変したりする可能性があります。DNAの侮辱に続いて、DNA損傷応答(DDR)経路が活性化され、修復に関与するタンパク質がリクルートされる。損傷の種類を感知し、適切な修復応答を活性化する要因の多くが関与しています。DDR因子を正しく活性化して採用しないと、ゲノム不安定性を引き起こし、癌を含む多くのヒト病理の根底にあります。DDRタンパク質の研究は、遺伝子毒性薬物応答および薬剤耐性の細胞メカニズムに関する洞察を提供することができる。
インビボでタンパク質を可視化する方法は、直接観察、対象タンパク質を蛍光タンパク質でタグ付けし、それをライブイメージングでフォローすること、または固定サンプル上の間接的な免疫蛍光を得る方法です。蛍光タグ付きタンパク質の可視化により、時間の経過とともに正確なモニタリングが可能になりますが、N-またはC末語で直接タグ付けすると、タンパク質の局在や機能が妨げられる可能性があります。それらの未修飾のタンパク質の観察は、内因性のバージョンが好ましい。DNA修復タンパク質をDNA侮辱にリクルートすると、その濃度は局所的に増加し、特定の抗体を用いた間接的な免疫蛍光によって可視化できるグループ(病巣)を形成します。
タンパク質病巣の検出は直接的な相互作用の決定的な証拠を提供するものではありませんが、細胞内のタンパク質の共局在化は、それらが損傷部位に再グループ化し、複雑な形成に必要なイベントのシーケンスを知らせることができることを示しています。タンパク質の野生型または変異型を発現する細胞における病巣空間的重複の注意深い分析は、DNA修復機能に重要な機能的ドメインに関する貴重な手がかりを提供することができる。最後に、タンパク質の共同局在化は、細胞内の共免疫沈降法によって検証できる可能性のある直接的な相互作用、または精製されたタンパク質を用いた直接プルダウンを示す。
概要
ヒト細胞は、様々な起源の様々なDNA損傷剤に常に曝露されています。外因性源は主に放射線、化学物質(化学療法剤および抗生物質を含む)、ウイルスへの暴露で構成され、主な内因性源にはDNA複製および酸化ストレスの誤りが含まれる。遺伝子毒性暴露の直接的な影響は、ストレスおよび暴露用量に応じて、修飾された塩基から潜在的に致死的なDNA二本鎖破断(DSB)まで及ぶ。最終的には、修復されていないか、誤って修復されたDNA損傷は、突然変異の蓄積、ゲノムの再配置、ゲノム不安定性を引き起こし、最終的に発癌につながる1。哺乳類細胞は、特定の種類のDNA損傷を認識する複雑な経路を進化させ、,細胞周期の進行と同期して、それらをタイムリーに修復しました。
電離放射線(IR)はDNA二重らせんを損傷し、DNA損傷の最も有害な形態の1つである二本鎖破断(DSB)を生み出します。MRN(MRE11、RAD50、NBS1)複合体はDNAのセンサーとして機能し、変異したプロテインキナーゼ運動失調症(ATM)4,5,5を活性化する。DNAによってATMの最初の活性化に続いて、ATMは、キーイベント、ヒストンバリアントH2AX6のリン酸化を開始し、休憩の部位でDDRイベントのカスケードをトリガします。残基S139上のH2AXリン酸化は、それをγH2AXに活性化し、DNA病変66、7、8、97,8,の周りのメガベースまでの領域に広がる。このイベントはDNAのアクセシビリティを高め、他のDNA修復タンパク質の募集と蓄積につながる7.γH2AXは、DSBを取り巻く豊富かつ特異的に誘導されるため、特定の抗体を用いて容易に可視化することができ、DNA修復分野におけるDSBの代理マーカーとして一般的に使用されています。破断がシグナル化されると、細胞はDNA修復経路を活性化し、DNA損傷を処理します。タンパク質MDC1(DNA損傷チェックポイントタンパク質1のメディター)はγH2AX1010に直接結合し、ATM11とまたNBS111,13,13と相互作用する。これは、DSB で MRN 複合体の濃度を増加し、正の ATM フィードバック ループを発生させることに貢献します。γH2AXは、ブレークが修復されると迅速に除去され、結果的に、DSBクリアランスのモニタリングが可能となる。顕微鏡検査が続き、時間の経過とともにγH2AXの減少は、残留破断およびDNA修復効率の間接的な測定を提供する。
真核細胞は、いくつかの経路によってDSBを修復することができ、2つの主なものは非相同のエンドコング(NHEJ)と相同組換え(HR)である(図1)。NHEJは本質的に拡張相同性を使用せずにDNA二本鎖末端をリリューションし、細胞周期14、15,15を通して動作する。HRはSおよびG2段階において優勢となり、それ以外は、修復14、16のための相同のテンプレートとして姉妹クロマチを必要とするため、16抑圧される。NHEJとHRの間の経路選択は、姉妹クロマチドの物理的近接性に依存するだけでなく、NHEJを阻害するDNA末端切除17の延長にも依存する。
相同性依存性DSB修復は、ブレークエンドからの5'鎖のヌクレオ分解によって開始され、3'一本鎖DNA(ssDNA)尾部を生成し、5'-3'切除と呼ばれるプロセスである。MRN複合体は、DNA末端切除を開始し、さらに切除はBLM/EXO1(ブルームシンドロームタンパク質/エキソヌクレアーゼ1)またはBLM/DNA2(DNA複製ATP依存性ヘリカーゼ/ヌクレアーゼ)18、19、20、21、22と組み合わせて処理される。18,19,20,21,22DNA末端切除は、MRN複合体23との直接的相互作用およびBRCA1(乳癌タイプ1感受性タンパク質)24,25,25の採用を通じてCtIP(CtBP相互作用タンパク質)によって増強される。23複製タンパク質A(RPA)は、露出したssDNAに速やかに結合し、次いでリコンビナーゼタンパク質RAD51によって変位して、相同の探索と鎖浸潤を触媒する核タンパク質フィラメントを形成する26、27、28。27,2826
切除の開始は、修復経路選択のための重要なステップです。切除が開始されると、DNA末端はKu70/Ku80ヘテロ二量体(NHEJ経路の成分)による結合のための貧弱な基質となり、細胞はHR17、29、3029,30にコミットされる。17Ku70/Ku80ヘテロダイマーはDSB末端に結合し、DNA-PKcsおよびp53結合タンパク質1(53BP1)29,30をリクルートする。29,3053BP1はG1における切除障壁として機能し、NHEJ31,32を促進しながらHRを遮断するが、S32相でBRCA1依存的に除去され、結果的に切除が33、34,34に起こることを可能にする。したがって、53BP1とBRCA1はDSB修理において反対の役割を果たし、53BP1はNHEJファシリテーターであり、BRCA1はHRを通じて修理するための休憩を可能にします。
研究室では、DSBの形成は、電離放射線(IR)によって誘導することができる。この例では、4 Gy、1 Gy、2 Gyの高用量を利用して、大量のDSBを作成し、豊富なタンパク質による病巣形成の分析に適しています。使用される放射線の種類と線量は、DNAおよび細胞内の異なる病変につながる可能性があることに注意することが重要です:IRはDSBを誘導する一方で、一本鎖の破断または塩基修飾を引き起こす可能性があります(照射線形エネルギー伝達(LET)およびDNA損傷の種類に関する参照については、35,36を参照してください)。35,イオン化放射線誘発病巣(IRIF)形成の運動学及びそれらのクリアランスを決定するために、活性化されたDDR8、9、37、38の損傷および8,9,37逆転の修復を38示す、病巣形成は、電離放射線後の異なる時点で監視することができる。全ての主要DNA損傷タンパク質の活性化とクリアランスのタイミングは39と知られており、多くは主要事象の代理マーカーとして調査されている。例えば、切れ切除の代理としてssDNAに対して高い親和性を有するpRPA、MRNタンパク質(MRE11、RAD50、NBS1)およびエキソヌクレアーゼも切除効率を評価するために使用することができる。RAD51、BRCA1、BRCA2(乳癌タイプ2感受性タンパク質)、およびPALB2(BRCA2のパートナーおよびローカライザー)をモニタリングしてHR効率を評価できるが、Kuタンパク質または53BP1の存在は、NHEJのマーカーとして使用されている(図1)。
DNA修復機械のタンパク質が互いに破断をリクルートし、超複合体で組み立てると、DNAタンパク質とタンパク質タンパク質相互作用は、時間の経過とともに個々の局在化に従い、タンパク質の共局在化を分析することによって推測され、細胞4040、41、4241,42の重複するシグナルによって視覚化される。細胞株では、ゲノム編集またはプラスミドベースの変異体の過剰発現による特定のDNA修復遺伝子における点突然変異または欠失の導入により、特定の残基の調査が可能であり、DNA損傷の認識におけるその可能な役割(例えば、γH2AXとの共局在化)または複雑なアセンブリ(別のタンパク質または複数のタンパク質との共局在化)、ならびにDNA修復への影響を可能にする。ここでは、時間の経過に伴うγH2AX病巣に従ってDSBの形成と分解能を調べるため、間接免疫蛍光を用いる。また、DSB修復における主要なプレーヤーによる病巣形成および共局在化分析の一例を提示する:p53結合タンパク質1(53BP1)32。32前述のように、53BP1は、DNA修復経路選択の中心と考えられている。53BP1蓄積およびγH2AXとの共局在化に続いて、細胞周期相、DNA損傷蓄積、およびDSBsの修復に使用される経路に関する貴重な情報を提供する。間接免疫局在化の目的は、細胞株中のDNA損傷修復の効率を評価することであり、この研究ではIRに続く、または細胞内の様々なストレスにさらされた後、DNA架橋から複製フォークの閉塞まで(DNA損傷剤のリストを表1に提供する)
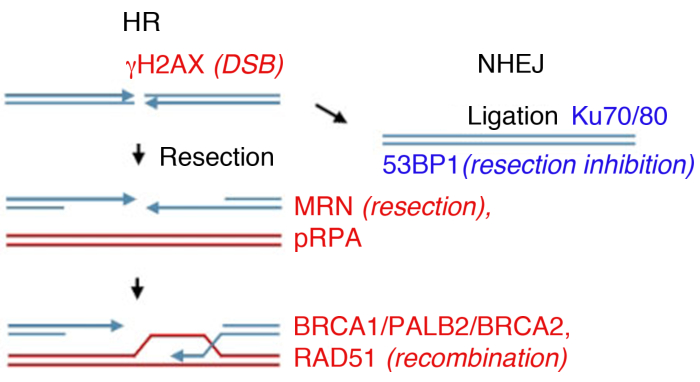
図1:DNA二本鎖切断(DSB)修復経路。
DSB修復には、相同組換え(HR、左)と非相同端結合(NHEJ、右)の2つの主要経路が含まれます。破断後、タンパク質は、ブレーク(γH2AX)をマークするために活性化され、末端切除(MRN)に参加し、切除されたssDNA(pRPA)をコーティングし、組み換えを促進する(BRCA1、PALB2、BRCA2、RAD51)、または切除を制限し、NHEJ(53BP1)を促進する。他のタンパク質は損傷修復に関与するが、リストされているタンパク質は日常的に間接的な免疫蛍光が続いている。 この図の大きなバージョンを表示するには、ここをクリックしてください。
| DNA損傷剤 | 作用機序 | 推奨用量 |
| γ線/X線 | 放射線 制御されていない細胞効果を持つ二本鎖破断の形成 | 1-4 Gy |
| 36アルイオン | 放射線 二本鎖破断の形成 | 270 keV/μm |
| α粒子 | 放射線 二本鎖破断の形成 | 116 keV/μm |
| ブレオマイシン | DNA合成阻害剤 | 0.4-2 μg/mL |
| カントーテシン | トポイソメラーゼIの阻害剤 | 10-200 nM |
| シスプラチン | アルキル化剤 (イントラストランドクロスリンクを誘導する) | 0.25-2 μM |
| ドキソルビシン | インターカラット トポイソメラーゼIIの阻害剤 | 10-200 nM |
| エトポサイド | トポイソメラーゼIIの阻害剤 | 10 μM |
| ヒドロキシウレア | DNA合成阻害剤 (リボヌクレオチド還元性による) | 10-200 μM |
| メチルメタンスルホン酸 | アルキル化剤 | 0.25-2 mM |
| マイトマイシン C | アルキル化剤 | 0.25-2 μM |
| 紫外線(UV)ライト | チミジン二量体の形成 (DNA鎖の歪みを発生させる) | 50-100 mJ/cm2 |
表1: 遺伝子毒性剤.DNA損傷剤の例は、それらの作用機序および示唆された作業集中に基づいて誘発される損傷である。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. HeLa細胞培養
- 16-18時間50°Cで1 M HClで丸いガラスカバーリップを事前に扱います。ddH2Oでリンスし、100%EtOHで保管してください。
- 細胞培養培地を調製する:DMEM培地に10%(v/v)FBSを加える。
- 37 °Cで18 mmの丸いガラスカバースリップを持つ無菌12ウェルプレートで4.0 x 10 4細胞/ウェルを成長させ、加湿インキュベーターで5%CO2を栽培します。280%の合流率(約24時間)に細胞を成長させます。
2. 放射線による細胞処理(IR)
- 二本鎖破断の形成を誘導するには、細胞を4Gyγ照射(対照:照射なし、t=0)に曝露する。ガンマセル-40では、これは毎分0.815 Gyで4.54分に相当します。
- 適切な時間長さ(ここで選択した時間ポイントt=1、2、4、16時間)の加湿インキュベーターで37°Cおよび5%CO2 の細胞をインキュベートする。
3. 核抽出と細胞固定
- ストックソリューションを準備する:0.2 Mパイプ(pH 6.8)、5 M NaCl、2 M2スクロース、1 M MgCl 2、0.1 M EGTA(pH 8.0)。
- 核抽出バッファー(NEB)を準備する:10 mM PIPES(pH 6.8)、100 mM NaCl、300 mMスクロース、3 mM MgCl2、1mM EGTA(pH 8.0)、0.5%(v/v)トリトンX-100をddH2に溶解するまで溶解します。2
- 4%(v/v)パラホルムアルデヒド(PFA)を調製する:16%PFA水溶液の10 mLを30 mL PBSに溶解する。完全に溶解するまで混ぜます。
- 適切な時点(t=0,1,2,4,16h)で、細胞を1mLのPBSで2回洗浄する。PBS を完全に削除します。
- 細胞核抽出のために各ウェルに200μLのNEBを加える(細胞質は分解され、核だけが残る)(図2)。室温で2分間インキュベートし、完全に取り除きます。
注:2分を超えないようにしてください。
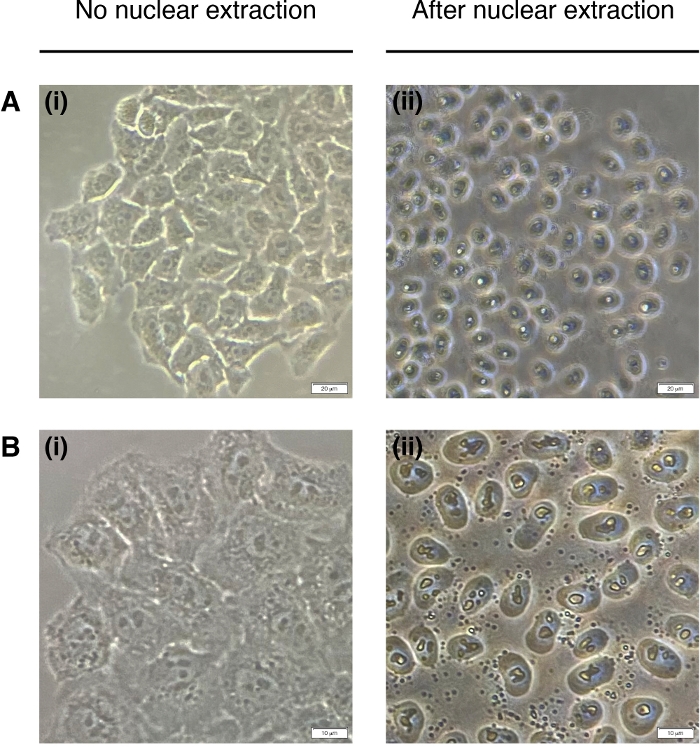
図2:核抽出
核抽出前(左)とポスト(右)の細胞の代表的な画像。細胞質は消化されるべきであるが、核構造は抽出後もそのまま残すべきである(右)。(A) 20倍の倍率。スケールバー= 20 μmと(B)40倍の倍率;スケールバー= 10 μm この図の大きなバージョンを表示するには、ここをクリックしてください。
- PBSの1 mLで細胞を洗浄します。PBS を完全に削除します。PBSを慎重に追加し、細胞はこのステップで非常に壊れやすいです。
- 細胞固定のために各ウェルに4%(v/v)PFAの200 μLを加える。4°Cで10分間インキュベートする。 PFA を完全に削除します。
- PBSを1 mL追加します。
メモ:細胞は4°CでPBSに保存することができます。
4. 免疫蛍光染色
- ブロッキング溶液を準備する:PBSで5%BSA(w/v)を溶解し、0.3%(v/v)Triton X-100を追加します。完全に溶解するまで混ぜます。
- 希釈バッファーを準備する:PBSで1%BSA(w/v)を溶解し、0.3%(v/v)トリトンX-100を追加します。完全に溶解するまで混ぜます。
- ブロックの場合は、各ウェルに200 μLのブロッキングソリューションを追加します。室温で2時間、または4°Cで16〜18時間インキュベートする。
注:ヤギ抗体を使用する場合は、ブロッキング溶液に5%のヤギ血清を加えます。 - 希釈緩衝液中の希薄な一次抗体(1:500;抗体リストについては 表2 参照)及びボルテックスが十分に混合されるまで。
注:ヤギ抗体を使用する場合は、希釈バッファーに1%ヤギ血清を加えます。 - 湿度/インキュベーションボックスに、パラフィルムを付着します。10 μLの一次抗体を追加します(1つのドロップで)。カバースリップの 1 つのエッジをドロップに合わせ、ゆっくりとパラフィルムの上に下げて、液体が全体に広がるようにします(可能であれば泡を避けてください)。室温で2時間インキュベートする。
- PBSで1分間カバースリップを3回洗います。
- 希釈緩衝液中の二次抗体(最終濃度:2 μg/mL)とボルテックスを十分に混合するまで希釈した。
- 一次抗体について説明したとおりに10μLの二次抗体を適用する。室温で2時間インキュベートする。
メモ:光から保護します。
| 抗体 | 会社 | 参照 | ソース |
| 53BP1 | セルシグナリング | 4937 | ウサギ |
| アンチマウスIgG H&L (アレクサ・フルーター 647) | アブカム | ab150103 | ロバ |
| アンチホスホヒストンH2A.X(Ser139)、クローンJBW301 | ミリポア | 05-636 | マウス |
| アンチラビットIgG H&L (アレクサフルーオール488) | アブカム | ab150081 | ヤギ |
表2: 使用する抗体.本研究で用いた抗体のリスト
- PBSで1分間カバースリップを3回洗います。
- H2 Oで2カバーリップを1分間洗います。
- DAPIを用いたカウンターステインDNA:300 nM DAPIの10μL(抗体について説明する)を適用し、室温で30分間インキュベートし、グリセロールベースの取り付け媒体でガラススライドに取り付けます。あるいは、DAPIを含む市販のアンチフェードマウントメディアを1滴(10μL)スライドに追加し、カバースリップを適用します。カバースリップを軽く押し、ペーパータオルで周囲の余分な液体を取り除きます。
- 透明なマニキュアでカバーリップをシールし、20分間乾燥させます。
- スライドは4°Cで保存します。
5. 画像の取得
- 60x対物レンズに浸漬油を一滴入れます。DAPI を使用して、アイピースを介して核を見つけます。
- XYZ画像取得の場合、取得ソフトウェアを開き、パラメータを選択: スキャナタイプ: ガルヴァーノ;スキャナモード:往復;画像サイズ:512×512;PMT モード: VBF;PMT平均:フレーム(4回);PMT シーケンシャル スキャン: 行。
- 染料と検出器を選択します。
チャネル (CH1),染料(DAPI),検出器 (SD1)
チャネル (CH2), 染料 (アレクサフルーター 488), 検出器 (HSD3)
チャンネル(CH3)、染料(アレクサフルーター647)、検出器(HSD4) - 「Z」で [オン ]を選択します。
- ライブ画像を調整します。ライブウィンドウのライブボタンを押します。
- フォーカスを調整し、レーザー強度(%)、感度(HV)、ゲイン、オフセットを「PMT」ツールウィンドウで調整します。
- レーザー強度(%)を調整:明るさと漂白のために。レーザー強度が高いほど信号は強くなりますが、検体は光漂白剤になります。
- 感度(HV):ノイズレベルを調整します。HVが高いほど信号は強くなりますが、高すぎると画像が騒がれになります。
注:常に電圧を一定に保ちます。 - オフセットを調整します: 背景レベル。
- Z スタックの 開始/終了 (15 スライス) を選択します。
- フォーカスを調整し、レーザー強度(%)、感度(HV)、ゲイン、オフセットを「PMT」ツールウィンドウで調整します。
- 取得を開始します。
- 画像を保存するフォルダを選択します。 LSMのスタート ボタンを押して、画像の取得を開始します。 [シリーズ完了 ]ボタンを押して、画像の取得を完了します。
6. データ分析
- 画像解析の場合は、解析ソフトウェアを開きます。
- [バッチ]ツール ウィンドウを押し、分析する画像を選択して、出力フォルダを選択します。
- [解析] ツール ウィンドウを押して[投影]を選択します(15 スライスからの最大強度投影が表示されます)。
- [ 入出力の設定] で、作成するバッチを選択します。
- 処理する画像を処理するには、[ 処理] を押します。
- イメージを .tiff ファイルとしてエクスポートします。
- 核病巣定量の場合は、CellProfiler を開きます。
- γH2AX および 53BP1 病巣定量パイプラインを開きます ( 補足情報を参照)。
- テーブルソフトウェアを使用したグラフデータ。
- 共同ローカリゼーション分析の場合は、CellProfiler を開きます。
- 共局在パイプラインを開きます ( 補足情報を参照)。スプレッドシート ファイルが作成され、希望の場所に保存されます。ただし、グラフ自体は自動的に保存されません。結果を記録するために、ウィンドウのスナップショットを作成することをお勧めします。
- テーブルソフトウェアを使用したグラフデータ。
Access restricted. Please log in or start a trial to view this content.
結果
2日目、または24時間ポストシード細胞をカバースリップに当てはめ、細胞は1つの分裂を受け、80%コンフルエントである。DNA修復タンパク質の特定のノックダウンまたは突然変異は、倍増時間を増加させるか、または細胞を遺伝子毒性治療に感作することができ、それに応じて播種密度と治療用量を調整する必要があります。最良の働き条件を決定するために、時間の経過とともにγH2AXのウェ...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
顕微鏡によるDNA損傷修復のタイミングと効率の分析は、正常細胞および癌などのヒト病理において、DNA修復機械がどのように機能するかを理解するために不可欠であることが証明されている。
リン酸化されたバージョン(γH2AX、pRPA、pRAD50など)で活性化タンパク質の検出を可能にする特異的抗体の開発(例えば、γH2AX、pRPA、pRAD50など10、23、39、43)23
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは開示するものは何もない。
謝辞
この作品は、サンアントニオ地域財団からの助成金によって支えられました。メイズがんセンターは、NCIがんセンターサポートコアグラントP30 CA054174によってサポートされています。スティーブン・ホロウェイが試薬を調達してくれたことに感謝し、宇宙と顕微鏡の能力を提供してくれたSung研究所に感謝します。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
参考文献
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600(2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757(2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428(2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740(2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, sup1 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819(2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved