Method Article
ゼブラフィッシュオプティクスカップ形態形成の4次元イメージング
要約
このプロトコルは、ゼブラフィッシュ早期眼の開発におけるトトラベリングおよび多次元イメージングのアプローチを記述する。レーザー走査共焦点顕微鏡によるラベリング、埋め込み、4次元(4D)イメージング、および光学カップ形態形成の解剖機構のためのデータセットの取得を最適化するための考慮事項について述べる。
要約
視覚システム機能は精密な組織および器官の構造の確立を必要とする。脊椎動物の目では、構造的欠陥は視覚障害の一般的な原因であるが、眼形態形成のメカニズムはまだ十分に理解されていない。胚性眼の基本的な組織は脊椎動物全体で保存され、したがってゼブラフィッシュ胚の生画像化は、正常および病理学的状態下でリアルタイムで目の発達を直接観察するための強力なアプローチとなっている。動き、形態、相互作用、分裂、死を含む動的細胞プロセスを胚で可視化することができる。ゼブラフィッシュにおける初期の眼の発達における細胞下構造とタイムラプス共焦点顕微鏡の均一標識法を開発した。このプロトコルは、1細胞ゼブラフィッシュ胚への注入のためのキャップ付きmRNAを生成し、光学小胞段階で胚を取り付け(受精後12時間、hpf)、レーザー走査共焦点顕微鏡で光学カップ形態素の多次元タイムラプスイメージングを行い、複数のデータセットが同じイメージングセッションで連続して取得されるようにする方法を概説する。このようなアプローチでは、セルの追跡、体積測定、3 次元 (3D) レンダリング、視覚化など、さまざまな目的に使用できるデータが得られます。私たちのアプローチは、野生型と遺伝子変異条件の両方で、光学カップの開発を促進する細胞および分子メカニズムを特定することを可能にします。これらの方法は、他のグループによって直接採用されるか、またはゼブラフィッシュの眼の発達の多くの追加の側面を視覚化するために適応することができる。
概要
脊椎動物の目の発達は、将来の脳神経エピテリウムからの眼小胞の出現(または膣)から始まる。その後、視小胞は一連の組織形状の変化を受け、伸び、膣化して光学カップを生成する。光学カップにおいて、神経網膜および網膜色素上皮は、いずれも神経エピテリウムに由来し、表面外皮に由来する新生レンズを包み込む。全体のプロセスは、神経エピテリウム、外胚葉、間葉系細胞集団の間で調整された、複雑な一連の細胞および組織の動きおよび分子シグナル伝達を必要とする。これらの初期事象は眼の基本的な構造を確立し、虹彩や角膜形成を含む眼の発達の後のステップは、初期の組織化に精緻化されている。早期の眼の発達と形態形成の混乱は、アノフタルミア、微小眼科、コロボーマを含むヒトの多数の視覚障害状態の根づく。視用カップ形態形成を支配する細胞および分子メカニズムのロックを解除することは、視覚システムの発達とこれらのプロセスが苦しくなったときに生じる病理学的状態をさらに理解するために重要です。
脊椎動物の目の発達と形態形成の理解は、古典的な組織学的研究から、マウス、ひよこ、カエル、および魚1、2、3、4、5を含む様々なモデル生物における胚学的および遺伝的アプローチに至るまでの膨大な量の研究から生まれた。この研究機関は、初期の眼の発達を調節する分子メカニズムを確立しましたが、歴史的には、眼の形態形成、すなわち3D構造の出現に対する理解が乏しく存在しています。これらの知見の大部分は、離散的な時点で切除された胚をイメージングすることから来た。これは2次元の組織形態のビューを提供するのに十分であるが、形態形成は動的な、3Dプロセスである。時間の経過とともに組織の形状が3次元でどのように変化し、単一細胞がどのように振る舞い、そしてそれらの挙動が3D組織形状の変化にどのように寄与するかを決定するためには、異なるアプローチが必要である。
この知識のギャップに対処する1つの解決策は、臓器が形になるにつれて細胞や組織をリアルタイムで動的に観察できるライブイメージングです。残念ながら、これは胚発生の制約のために多くのモデル生物で容易に実現可能ではありません。例えば、マウスやひよこの胚(子宮内または卵殻内で発達する)は容易にアクセスされず、多くの生きた胚は光学的に透明ではなく、重大な光散乱を引き起こし、画像を取得できる深さを制限する。ゼブラフィッシュは、外部発達および透明胚を有し、眼形態形成6、7、8、9、10、11、12、13、14、15、16、17、18、19の生画像化を行うユニークな機会を提供する。十分なトランスジェニックおよび変異線も利用可能であり、新しいトランスジェニックおよび変異体20、21、22、23、24を生成するためのツールも利用可能である。また、ゼブラフィッシュにおいて、12時間の時間枠(受精後12〜24時間、hpf)を超えて、全体のプロセスのイメージングが可能となる。
細胞下構造の遺伝的にコード化された重要な標識を可能にする蛍光タンパク質のファミリーの拡大と最適化、顕微鏡法の改善と革新によって、ライブイメージングの取り組みが加速されました。ここで説明するプロトコルは、スピニングディスク共焦点顕微鏡、選択的平面照明顕微鏡(SPIMおよびその変異体)、および他のより専門的な顕微鏡法を含むゼブラフィッシュ胚発生をイメージングするための他の現在のアプローチではなく、レーザースキャン共焦点顕微鏡を使用する。開発中のゼブラフィッシュの目では、回転ディスク共焦点顕微鏡は、組織の深いイメージングには十分ではないことがわかりました。SPIM は非常に高速なイメージング時間を誇り、より広く使用されていますが、ビジュアライゼーションと分析のために大規模なデータセットを処理することは依然として課題です。対照的に、レーザースキャン共焦点顕微鏡は、特に光学ハードウェアの組み立てに関する専門知識を欠いている個人のために、簡単にアクセスできます。レーザースキャン共焦点顕微鏡の普及が、多くのラボで役立つことを期待しています。
ここでは、光学カップ形態形成の4Dデータセットを、膜およびクロマチン用の胚の トト 標識に使用する方法と、画像取得用のレーザー走査型共焦点顕微鏡( 図1で示す)を用いる方法を説明する。ここで使用される蛍光タンパク質(EGFP-CAAXおよびH2A.F/Z-mCherry)は、単一細胞分解能を有する組織標識を提供するために選択された。このプロトコルで生成されたデータセットを、さまざまな画像解析や可視化機能に使用します。このプロトコルは、他の細胞内構造が望まれる場合に容易に適応することができる。細胞形状の可視化のために選ばれた細胞膜標識:我々は、EGFP-CAAXを用い、H-rasの最後の21個のアミノ酸を、プレニル化シグナル配列として、EGFP13のC末流に融合させる。他の血漿膜標的蛍光タンパク質(例えば、膜貫通融合またはミリストア化)も同様に働く可能性が高い。核をマークするために、我々はH2Aを選択しました。F/Z-mCherry, mCherryがヒストンタンパク質に融合している13.これにより、有糸分裂のスピンドルの向きを含む細胞分裂が容易に可視化されます。
ライブイメージングアプローチでは、信号対雑音比、軸解像度、サンプル保存を最大化しながら、イメージング速度を向上させるトレードオフを考慮する必要があります。私たちは、画像品質と1回の実行で画像化することができる胚の数を最大化する方法を最適化しました。多くの場合、目標は、ホモ接合性変異胚における光学カップ形態形成を画像化するものであり、これは、視カップ形態形成の発症時およびヘテロ接合の子孫(胚の25%が所望の遺伝子型である)の野生型と区別できない可能性がある。画像取得を最適化し、その後多重化することにより、ホモ接合変異型胚のデータセットをキャプチャする可能性が高くなります。
時間分解、またはボリュームデータ(Zスタック)の取得頻度は、タイムラプスイメージングの重要な側面です。このようなデータセットの目的に応じて、速度に関する要件が異なります。当初、このプロトコルは手動4Dセルトラッキングのために開発されました。均一に標識された組織内の個々の細胞の追跡は、1つの細胞が時間の経過とともに連続的に追跡されているという確信を与えるのに十分な時間分解能を必要とします。我々は、ゼブラフィッシュの視用カップ形態形成のZスタックは、少なくとも12時間にわたって3.1分ごとに取得しなければならないことを発見した。ここでは、2.5分ごとに4~5個の胚のZスタックを取得できるように、レーザー走査共焦点顕微鏡での取得を最適化しました。
Z ステップ サイズの確立は、3D レンダリングと視覚化の場合、Z ステップ サイズが XY ピクセル次元に等しい等方性データが理想的なプロトコル最適化の重要なステップでした。実際には、イメージング時間とフォトブリーチングの制約を与えられ、ライブサンプルでこのようなタイムラプスデータを取得することは非常に困難です。したがって、適切なZステップサイズを決定することは、実験のレンダリングと可視化のニーズにとって重要であり、具体的には、速度を維持し、光の漂白を防ぎながら軸情報を最大化するために必要なX:Y:Zボクセル比が必要です。このプロトコルでは、確立されたボクセル比は1:1:3.5(0.6 μm x 0.6 μm x 2.1 μm Zステップサイズで、40倍長い作動距離の水浸漬目標を使用した場合)でした。Z深度130~140μmを取得すると、適切な時間分解能と少ない光の切り落としを伴うボリュームデータが得られます。
上で議論したように、このプロトコルは、ゼブラフィッシュの光学カップ形態素の4Dイメージングに特異的であり、細胞膜およびクロマチンに対して標識されたトトの胚、およびレーザー走査共焦点顕微鏡を用いる。以下のプロトコルは、さまざまな実験やニーズに簡単に適応できます。まず、細胞内構造に関しては、生細胞マーカーが存在するあらゆる構造を画像化することができる。次に、ここでの焦点は光学カップ形態形成のみに焦点を当てているが、タイムラプスイメージングは、眼の発達の他の段階に適応することができ、例えば、神経新生25、26、27、28、29、30、31、32、33。イメージング後の開発では、胚固定化(自発的な筋肉活動が約24hpfから始まる)、色素沈着(24時間前後に出現し始める)、組織サイズ(眼の神経発生時に体積が著しく成長する)、イメージング速度(対象プロセスの速度に応じて調整する必要があります)を考慮する必要があります。これらの考慮事項はすべて、容易に管理できます。プロトコルは非常に柔軟です。ここでの特定のプロトコルの詳細に加えて、目の発達の他の側面をライブイメージングに興味のある人を支援する一般的な原則があります。
プロトコル
ゼブラフィッシュを用いた全ての方法は、クリステン・クワン博士の動物プロトコル「ゼブラフィッシュにおける視覚システム開発の細胞・分子メカニズム」のもとでカバーされ、ユタ大学の施設動物のケアと使用委員会(IACUC)によって承認されています。
1. キャップRNA合成
- インビトロ転写用のDNAテンプレートを生成します。
- 反応混合物の100 μL体積で10μgのDNAを消化してDNAテンプレートをリニアライズします。典型的な反応は、酵素3 μLと10μLのバッファーを用いて10μgのDNAを消化し、水で100μLに反応量を持たします。
注: ダイジェストの典型的なテンプレートは、pCS2 ベクター、たとえば、pCS2-EGFP-CAAX または pCS2FA H2A.F/Z-mCherry 膜およびクロマチン標識に関して、ここで説明します。この場合、プラスミドDNAは、それぞれ、酵素NotIを使用して消化される。ユーザーはスターの活動に注意する必要があります。この場合、高忠実度の酵素が推奨され、市販されている。 - 反応を一晩37°Cでインキュベートし、DNAが消化されて完了するようにします。
- メーカーのプロトコルに従って PCR 精製キットを使用して制限ダイジェストをクリーンアップします。30 μL ddH2O. で DNA を溶出し、線形化された DNA を -20 °C に保存し、必要に応じて使用します。
注:DNAの消化量と溶出量は約0.3 μg/μLです。これは、dnaの〜2 μgをテンプレートとして使用して、体外転写の〜5ラウンドで十分です。
- 反応混合物の100 μL体積で10μgのDNAを消化してDNAテンプレートをリニアライズします。典型的な反応は、酵素3 μLと10μLのバッファーを用いて10μgのDNAを消化し、水で100μLに反応量を持たします。
- インビトロ転写キットを使用して、インビトロでキャップされたRNAを転写します。pCS2 テンプレート (ここで説明する方法) については、SP6 キットを使用します。
- インビトロ転写反応を組み立てる:10x反応バッファーの2 μL、2xリボヌクレオチドミックスの10 μL、10x酵素ミックスの2 μLを用いて、DNAテンプレートの2μgまたは最大6 μLを消化します。
- 37°Cで2〜4時間以上インキュベートします(より長い時間は、より大きな収率につながります)。必要に応じて、酵素ミックスの1 μLは、インキュベーション期間の途中で補うことができます。
- 1 μLのRNaseフリーDNaseを加え、37°Cで15分間インキュベートして、DNAテンプレートを消化します。
- メーカーのプロトコルに従ってRNA精製キットを使用して、キャップされたRNAを精製します( 材料表を参照)。RNaseフリーH2Oの100 μLを持つエルテ。
注:このアプリケーションにはβメルカプトエタノールの添加は必要ありません。このプロトコルに記載されている方法は簡単ですが、代替試薬を使用してRNAを精製することもできます。 - RNAを沈殿します。
- 溶出したRNAに3 M RNaseフリーNaOAcの10 μLと2.5ボリューム(約275 μL)の氷冷RNaseフリー100%EtOHを加えます。
- -20°Cで15〜30分間インキュベートします。4°Cで高速で15分間スピンし、RNAをペレットします。
注:ペレットはチューブの壁に対して見える必要があります。このステップでチューブが遠心分離機でどのように整列されているかに注意して、ペレットがスピンの終わりにある場所を予測するのに役立ちます。 - EtOHを注射器で慎重に取り出し、加熱、過剰乾燥、またはペレットの除去を避けるように注意してください。ペレットをRNaseフリーH2Oの20 μLで再懸濁します。
- RNA をチェックして、合成が成功していることを確認します。1 μLを使用して分光光度計の濃度をアッセイし、1%アガロースゲルで0.5 μLを実行して、低分子量スメアではなく1つまたは2つの離散バンドをチェックします。インビトロ転写反応の収率は〜1 μg/μLである必要があります。
- アリコートでRNAを-20°Cまたは-80°Cで保存します。 RNAは、注射の直前まで希釈されません。
2. 1細胞ゼブラフィッシュ胚のマイクロインジェクション
注:RNAあたり200〜300 pgを注入して、光学カップ形態形成全体にわたってクロマチンと細胞膜のユビキタス発現を得る。5~10 μLのRNA希釈液を調製し、2.5~5 μL/針をロードします。針が壊れた場合に余分な計画を立てる。
- 注射のための事前準備.
- マイクロインジェクション用の1細胞胚の配向と向きを容易にする射出型皿を準備する。E3(標準的な胚培地)で2%アガロースを溶かし、ペトリ皿に注ぎます。熱いアガロースの上に射出型( 材料表を参照)を慎重に浮かべ、凝固するにつれてアガロースのトラフの刻印を生成します。アガロースが固まったら、射出成形を取り除く。
注:射出型の皿は数ヶ月間使用することができる。E3でカバーし、使用しない場合は4°Cで保存してください。 - マイクロインジェクション針を引っ張ります。ガラスの毛細血管を長いテーパーに引き上げ、針の引き手を使用してマイクロインジェクション針を作ります。使用する毛細血管の種類に固有の機械をプログラムします。1.0 x 0.78 mm のホウケイ酸毛細管では、熱 = 546 °C、プル = 130、速度 = 70、時間 = 90 (図 2F) を使用します。ペトリ皿に針を保管し、モデリング粘土でそれを確保します。
注:引っ張るレシピは、機械とフィラメントによって異なり、新しいフィラメントは常に較正する必要があります。引っ張りの各ラウンドは2本の針を作り出す(図2G);偶発的な休憩や将来の実験の場合に十分な針を生成するためにこれを数回行います. - キャップされたRNAを希釈して注射します。EGFP-CAAXおよびH2A-mCherry RNAAsの両方を、RNaseフリーH2Oと1μLのフェノールレッドを全容量5μL(フェノールレッドの最終濃度は0.1%)で200-300 ng/μL濃度に希釈します。混合物をフリックし、総体積を収集するために簡単にスピンダウン。希釈したRNAを注入する前に氷の上に保管してください。
- マイクロインジェクション用の1細胞胚の配向と向きを容易にする射出型皿を準備する。E3(標準的な胚培地)で2%アガロースを溶かし、ペトリ皿に注ぎます。熱いアガロースの上に射出型( 材料表を参照)を慎重に浮かべ、凝固するにつれてアガロースのトラフの刻印を生成します。アガロースが固まったら、射出成形を取り除く。
- 1細胞胚の注入
- 魚が繁殖し始めたら、卵が受精することを確実にするために〜15〜20分を許可します。この間、P10およびP10マイクロローダの先端を使用して、RNA希釈液の2.5〜5 μLで針をバックロードします。
- ピコインジェクターを使用して、眼球レチクル(マイクロメートルキャリブレーションスライドも十分)を使用して針を較正し、液滴の体積(球の体積=(4/3)*π*半径^3)を測定します。射出量が1nLになるように射出時間と圧力を調整する(図2I)。
- ローラー/転写ピペットを使用して、注入金型に卵を慎重にロードします(図2J)。有用であれば、鉗子を使用して、単一の細胞が注射の前または間に見えるように胚を転がす。マイクロマニピュレータを使用するのがユーザーの好みです。
- 卵黄ではなく細胞を標的とする1細胞段階で胚を注入する(図2M)。これは、発達中の胚の均一な標識を保証します。
- 胚を所望の段階まで上げる(標準のステージング34に従って12 hpfより前)。注入された胚は発達が遅れるので、わずかに高い温度(29.0-29.5°C)で上げて時間を補います。午後の間に、胚をチェックし、クラッチの健康を維持するために死んでいるものを取り除きます。
3. タイムラプスイメージング用の視小胞ステージゼブラフィッシュ胚の取り付け
- 取り付ける前に、E3で1.6%低溶融アガロースを調製してください。複数のイメージング実験を計画する場合は、低融解アガロースの約20mLを調製し、室温で保存します。胚取り付けの日に、これを溶かして、取り付ける前に42°Cの熱ブロックに入れることができるチューブに新鮮なアリコート(1〜5mL)を生成します。
- 実装前に蛍光顕微鏡を用いた蛍光の注入および全体的な明るさを得るためのスクリーニング胚。理想的なサンプルは、強力なEGFPとmCherry蛍光を有し、正しい発達段階にあるでしょう(図3B - B')。
- 蛍光顕微鏡を用いた胚のスクリーニング。EGFPとmCherry両方の蛍光を強く発現する胚を選んで実装します。
- 11 hpf である胚を選択します。胚34を適切にステージングするためにスマイトを数える。11 hpfでは3つのスマイトがあり、12 hpfによって6つのスマイトがあるはずです。
注:12 hpf(11 hpf)より前の胚を取り付けることによって、サンプルはタイムラプスが始まるときに12 hpfで適切にステージングされます。
- 取り付け前のデコリオネート胚。これを手動で細かい鉗子で行うか、プロナーゼ(2 mg/mL)を使用して化学的に行ってください。この若い胚では、寒天コーティングされた皿の中でデコールリオネーションが行われ、胚が空気水界面に接触しないことが不可欠である。
- ガラスローラーピペットを使用して、胚を吸い上げ、胚がガラスパスツールピペットの先端に座るようにできるだけ多くのE3を排出します。
- 胚を熱ブロックからアガロースのチューブに落とします。数秒間アガロースに沈ませ、次にアガロースを最初に吸い上げ、次に胚を吸い上げ、胚がピペットの先端に残っていることを確認します(図3C - C')。
- 解剖スコープの下でイメージング用のガラス底皿を置きます。胚とアガロースをガラス底皿のアガロース液滴に排出する。鉗子を使用して、胚が底面下(ガラス底の頭の上)になるように、非常に迅速に配向します。アガロース液滴を硬化させます(図3D - D')。この段階の胚は壊れやすいので、このステップは迅速かつ慎重に行う必要があります。損傷した胚は、タイムラプスイメージングプロセスを生き残るものではありません。
注:この実験では、すべての胚の向きを一貫して行うことが重要です。タイムラプスを行う場合、胚は、光学小胞が成長するための追加のスペースを維持しながら、割り当てられた視野内に収まる必要があります。最適な方位は、すべての胚の前部後方軸を「垂直」に、または時計面の12時と6時に沿って整列させることです(図3G)。 - 10~12個の胚の間にマウントし、これは画像化される2倍以上であり、共焦点で評価されると最良のサンプルを選択することができます(図3E - E')。サンプルは、年齢、蛍光標識の均一性、および取り付け方向の精度について評価されます。
- 胚を取り付けた後、ピペットよりアガロースが皿の底を完全に覆い、それによって単一の大きなアガロースディスクに分離したアガロース液滴のすべてを包み込む(図3F - F')。十分な量のアガロースは、個々の胚液滴またはディスク全体が皿の底から持ち上がり、視界から浮かび上がらないようにします。
- 硬化したら、E3でアガロースを重ね、タイムラプスイメージング実験の期間中、サンプルを水分補給します(建物の湿度や環境に依存する懸念)。
注:トリケーヌは、これらの胚が十分に若いので、これらの特定のタイムラプスイメージング実験には必要ありません。しかし、必要に応じて、そして胚の古い段階で作業する場合、トリケーヌはアガロース自体に添加され、メディアに重ねることができます。 - 紙の上に、タイムラプスの設定中に簡単に参照するための胚の地図を描きます。これは、後で各サンプルの遺伝子型と位置を関連付けるために重要です。
4. レーザー走査共焦点顕微鏡による多重位置共焦点タイムラプス
注:このタイムラプスイメージングプロトコルは、メーカーのソフトウェアを搭載したレーザー走査型共焦点顕微鏡で使用するように設計されました( 材料表を参照)。このシステムはZスタックの急速な獲得を可能にするピエゾZ段階装置が装備されている。このプロトコルで使用されるレーザーラインは、488 nmアルゴンイオンレーザーとDPSS 561 nmレーザーです。561 nmレーザーは、mCherry蛍光色素を撮像するのに適しています:それはmCherry励起(587 nm)のピークに十分近く、十分に動力を与えられています。
- 共焦点を設定します。
- コンフォーカルの電源を入れ、デスクトップコンピュータにログインします。
- piezo Z ステージインサート (図 4B)を取り付け、取得ソフトウェアを起動します。
- 取得モード内のソフトウェアで、ドロップダウンメニューを使用して488および561レーザーをオンにします(図4F)。レーザーはウォームアップに数分かかるので、このステップは時間を節約するために早めに行われます。
注: 取得パラメータは次のとおりです: Zスタック、時系列、および位置ボックスをチェックします。フレームサイズを512(X)×384(Y)、スキャン速度を9に、スキャンモードを双方向に、ズームを0.7に、ピンホールを60.2(1.63エアリーユニット〜=1.6 μmセクション)に設定し、Z間隔を2.1 μmに設定します。これにより、303.6 μm x 227.7 μm の画像サイズと、0.59 μm のピクセルサイズが得られます。488 および 561 レーザーは、チェックされ、同じトラックに割り当てられる必要があります。EGFP検出範囲は494-545 nmで、mCherry検出範囲は598-679である(図4F)。 - ガラス皿の底部を水の屈折率に合致する浸漬媒体で自由にコーティングし、気泡を避けるように注意する(図4C)。40x水の目的を選択し、目的に浸漬媒体の小さな滴を適用します(図4D)。ステージインサートでガラス底の皿を固定し、E3を適用して胚を一晩湿らせておき、モデリングクレイを使用してプラスチック製の蓋を皿の上に密封します(図4E)。料理と接触する目的を上げます。
- サンプルをスクリーニングし、タイムラプスの準備をします。
- [取得]タブから[場所]タブに切り替え、電球記号をクリックして、透過光をオンにします。透過光で、ジョイスティックで最初のサンプルを見つける:目で、まずサンプルを光のビームに移動し、次に接眼レンズを中心に使用して、光学小胞に焦点を当てます。
- [取得] タブに戻ります。Z-stack、時系列、および位置の各ボックスがオンになっていることを確認し、レーザーが使用できるようにウォームアップされていることを確認します。
- [フレーム サイズ]を 512 (X) x 384 (Y) に設定し、[スキャン速度] を9 に、[スキャン モード] を [双方向] に設定し、[ズーム]を 0.7 に設定します。
- チャネルの見出しの下で、488および 561 レーザーを Power 2.0 およびGain 550 に割り当てることから始めます(これは後で調整できます)。ピンホールを60.2(1.63エアリーユニット~=1.6μmセクション)に設定します。
- [連続]ボタンを使用してスキャンを開始します。細かいフォーカスノブを使用して、サンプルをZ軸に配置し、フレーム内のX-Y軸を配置します。スキャンを停止します。
- [ 位置 ] 見出しの下にある [追加 ] をクリックして、最初のサンプルに対する XYZ 情報を保存します (図 4F)。これは、サンプルがタイムラプスされる場合にのみ行います。強力な蛍光と最適な取り付けのためのサンプルを選択してください。
- [場所検索] タブに切り替え、次のサンプルに移動し、サンプルの選択について選択的に手順 4.2.5 ~ 4.2.6 を繰り返します。
- サンプル位置を確定し、z 範囲を割り当て、次に開始タイムラプスを割り当てます。すべてのサンプルを選択し(2.5分の合計時間間隔内で4-5サンプルを画像化することが可能)、位置を保存したら、各サンプルを通過し、表示フレーム内のXYZ位置を調整します。
- 最初の位置をハイライト表示し、[ 移動先] をクリックします。連続的にスキャンしながら、フレーム内の視小胞を並べています。眼小胞と脳に対して相対的な前および遠位領域に十分なスペースを残します。
- 次に、細かいフォーカスノブでZ方向を移動しながら 「最初に設定 」と「 最後に設定 」を選択して、最初と最後のZスライスを割り当てます。これは、光学カップの成長に対応するので、〜63の総スライス数を維持します。成長のための部屋を可能にするために、視小胞の腹側に余分な部屋を提供します。
- 最初と最後の Z スライスを設定したら 、C ボタンをクリックして Z スタックの中央に移動します。両方のレーザーのレーザーパワーとゲインを調整します。可能であれば、レーザーパワーを5以下に保ち、必要に応じてより高いゲインを使用します。スキャンを停止します。[更新] をクリックして、位置情報を 更新します。
- [位置 2]をクリックして次の位置に移動します。割り当てられた各ポジションについて、ステップ 4.3.1 から 4.3.3 を繰り返します。レーザーパワーとゲインは、すべてのサンプルが十分に照らされるように設定する必要があります。
- 位置情報とレーザー設定の両方が確定したら、時系列設定を割り当てます。[ 時系列 ] の見出しの下で、[ 時間間隔 ] を 2.5 分に、 サイクル を 300 に割り当てます (図 4F)。サイクル数は、光学カップの開発が完了したときにタイムラプスが24 hpf段階を超えて進むような計算されます。
- タイムラプスを開始するには、[ 開始] をクリックします。最初の完全なサイクルを監視する:タイマーを使用して、1つの完全なサイクル(すべての位置のイメージング)が2.5分未満であることを確認し、各位置が正しく見えることを確認します。顕微鏡は一晩独立して動作し、監視する必要はありません。
注意:コンフォーカルルームは温度制御されていますが、室温は20〜25°Cです。 機器の走行とレーザースキャンにより、ステージ上の温度は、形態学的ランドマークで視覚的にアッセイされるように、この予想速度で胚の発達が進むので、28.5°Cに近いように見えます。 - これらの設定では、タイムラプスは12.5時間続きます。タイムラプスが完了したら、ファイルを保存します。これは、大きい(〜40 GB)になり、取得ソフトウェアを使用して保存した後、個々の位置に分離することができます。
結果
蛍光RNAの注入は細胞内標識を可能にする
細胞の血漿膜とヒストンにラベルを付けるRNAは、光学カップ形態形成の根本となる個々の細胞形態および動きを捕捉するために注入される。図2は、1細胞段階でゼブラフィッシュ胚をマイクロ注入するプロセスの各工程を示す。簡単に言えば、ゼブラフィッシュが交配され(図2E)、胚が採取され、射出型にロードされる(図2J)。マイクロインジェクション針が戻り充填され(図2H)、胚が単一の細胞(図2K-M)に直接注入され、均一な標識を得るために必要である(図2M、図4I-I'、ムービー1)。均一な標識に加えて、堅牢な蛍光が観察され、発達は摂動されずに進行する(図3B-B')。
効果的なマウントは、12 - 24 hpfからのOCMのタイムラプスイメージングに不可欠です
長期間にわたって発生する光学カップ形態素を画像化するには、理想的な胚を選択し、埋め込み中に各サンプルを適切に配置することが重要です。図3は、11個のhpf胚の取り付けに成功した詳細な説明を示しています。注射された胚は、最初に十分な蛍光をスクリーニングする(図3B',B',F'個々の胚はアガロース(図3C、C')に浸漬され、アガロースの個々の滴にドーサリーに取り付けられる(図3D、D')。すべてのサンプルはアガロースの集合ディスクに取り付けられます(図3E-F')。胚が正しくステージングされ、十分に蛍光を発し、十分に取り付けられているとき(図3G)、サンプルは、時間経過の間、画像化フレーム内に留まり、臓器全体を画像化することを可能にする(図4G-G''、I-I''、ムービー1)。これらのステップのいずれかを達成しないと、タイムラプスイメージングに最適なサンプルではない胚が取り付けられます。回転度のわずかな変動は、タイムラプス中の胚の成長方向に劇的な影響を与えます。図3に示す、過度に回転した胚(図3I)は、より後回しになる胚と、最も前部組織のみを観察できる回転が少ない胚(図3J)を含む。取り付け時の適切な回転に加えて、フレームの向きに関して取り付けられたサンプルは、タイムラプスが成功する可能性が高い(図4G-G''、I-I''、ムービー1)。最適なサンプルは、時計面上の12時と6時に沿って前後軸を持つ皿の上に垂直に向けられているものである(図3G)。最適ではないサンプルは、水平または対角線のような、この軸上に向いていないサンプルです(図3H)。これらのサンプルは、タイムラプスが進むにつれてイメージングフレームから成長し、さらなる分析には使用できません(図4H-H''、J-J'')。
将来の分析に適したタイムラプスデータセットの取得
正確に注入され、上げられ、取り付けられた胚は、共焦点画像サンプルを成功させる。図4は、時間経過に最適なサンプルを示しています:蛍光も存在し、サンプルは12 hpfです。イメージング用のzスタックは、最初のスライスが光学小胞に対して単に背部になるように割り当てられ、最後のスライスは12 hpf光学小胞の腹側境界を越えていくつかのスライスを残します(図4G-G'')。腹側に対するこのバイアスは、腹側方向の組織成長のための余分なスペースを提供する。これらの要因は、満たされると、最適なタイムラプスをもたらす(図4I-I''、ムービー1)。最適でないサンプルはモザイクまたは薄暗い蛍光を有し、すでに12hpf(視球形態形成が進行中である場合)、またはZスタックまたはXYフレーム内に位置が悪い(図4H-H')を有する。これらの基準が満たされない場合、タイムラプスは組織の発達時に組織の3Dボリューム全体をキャプチャしなくなり、さらなる分析に使用できなくなります(図4J-J''')。
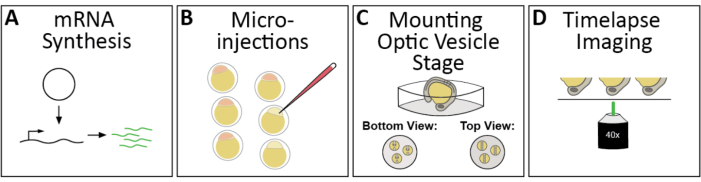
図1:ゼブラフィッシュ光学カップ形態形成の4Dタイムラプスイメージングのワークフロー (A)目的の細胞下構造を蛍光的に標識し、適切なキャップされたmRNAを合成する。(B) 1細胞段階でゼブラフィッシュ胚にmRNAをマイクロインジェクトする。(C)胚は、背もたたく、または反転した共焦点顕微鏡のためにヘッドダウンして、光学小胞段階で、受精後〜11時間または3スマイト段階に取り付けられる。(D)単一のタイムラプスイメージングセッション中に複数の胚を順次イメージングすることができる。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:1細胞ゼブラフィッシュ胚をマイクロ注入するための視覚的なガイド。(A)注射キットに含める項目(時計回り):使い捨てニトリル手袋でキット全体を保持できる中型ボックス。マイクロインジェクション針を含む料理;鉗子;鉱物油のアリコートとパラフィルムの正方形をカット。マイクロローディングのヒント;氷上の希釈されたRNA;アガロース射出成形型;マーカー;ローラー/転写ピペット;とP10ピペット。(B)マイクロインジェクションセットアップ: 外部の圧縮ガス源に接続されているピコインジェクターの隣にある顕微鏡を解剖する。ピコインジェクタにはフットペダルとマイクロマニピュレーターが取り付けられています。マイクロマニピュレーターは針のホールダーが装備され、顕微鏡の段階の上の位置に置かれる。(C)片側に90度の角度、もう一方の側に45度の角度を持つ溝の6列を含むアガロース射出型。(D) ピコインジェクタフロントパネル。圧力表示は7.8 PSIを読み取る;これは、Pインジェクトとラベル付けされたノブで調整することができます。下には、注入する圧力を手動でトリガーする押しボタンがあり、注入時間を変更したり、液体を針に取り除いたり、充填したり、保持したりします。注入時間は08(8 x 10ミリ秒に相当)に設定されます。出力ホースはPに接続され、注射針に取り付けられます。圧力計のソーススイッチは、射出圧力を設定しながらP注入に設定され、注射が行われていないときに針に適用される基底圧力を調整するためにPバランスに切り替えることができます。Pバランスレギュレータは Pインジェクトレギュレータの隣にあります。電源ボタンが点灯し、マシンの電源が入っている。(E)成虫ゼブラフィッシュは、受精卵から成魚を分離し、容易に収集できるメッシュ底を持つ入れ子タンクを含む小さな繁殖タンクで交配される。水位は浅い水の条件を模倣するために下がり、タンクの仕切りは、交配行動を開始するために除去されます。(F)マイクロピペット(針)の引き手の前部パネル:針を引くために使用される特定の条件を示すディスプレイが含まれています。圧力設定は P = 500 で、プログラムが編集された最後の日時、プログラム番号、HEAT = 546、PULL = 130、VEL = 70、TIME = 90 です。(G)ガラスの毛細管をマイクロピペット(針)の引き手に挿入し、所定の位置に固定する。各プログラムされたプルランは、2つの長いテーパー針(赤い矢印)をもたらします。(H) RNA希釈は針にバックロードされる。フェノールレッド染料は、溶液の容易な視覚化を可能にする。(I) 針の先端を折った後、RNAのボーラスは解剖範囲の眼球レチクルを用いて測定される。この立体顕微鏡では、合計30倍の倍率で6ハッシュマークは1nL体積に等しい。(J)受精卵はアガロース射出成形型に装填され、ローラーピペットを用いて金型のトラフに沿って分布する。(K)1細胞段階胚を含む射出型を顕微鏡下に配置し、胚を順次注入する。(L)注射針は、単一細胞を標的とする各胚に挿入される。(M) 細胞内に入ると、フットペダルは、調節された量の圧力を引き起こし、1 nLのRNAを胚の細胞に放出する(フェノールレッドによって視覚化される) 。スケールバー:I = 0.1ミリメートル。L, M = 0.5 mm.この図の大きなバージョンを表示するには、ここをクリックしてください。

図3:イメージング用の胚を取り付けるための視覚的なガイド。(A)取り付け(左から右へ):42°Cにプログラムされたヒートブロック、低溶融アガロースの加熱チューブ。ローラーピペット;鉗子のペア;デコリオネート胚を含むアガロースコーティングされた皿。金属ハロゲン化物蛍光灯に接続された解剖実視的な顕微鏡。(B,B', B'')EGFP-CAAX mRNAおよびmCherry-H2A mRNAを注入した11hpf胚は、それぞれ明視野下、GFP蛍光およびmCherry蛍光の下で示される。(C, C')先端に座っているアガロースと胚を含むガラスパスツールピペット。C'は拡大表示です。(D,D')ガラス底の皿の上の低溶融アガロースの液滴に取り付けられた1つの胚は、顕微鏡の段階と顕微鏡の接眼を通して両方から見た。(E,E')ガラス底の皿に低溶融アガロースの個々の液滴に取り付けられた12個の胚は、顕微鏡の段階と顕微鏡接眼眼を通して見た。(F,F',F',F'')すべての取り付けられた胚は、顕微鏡の段階と明るい分野で示された顕微鏡の接眼眼点の両方から見た皿の底を満たすためにアガロースの完全な層で重ねられます。F''GFP蛍光;そしてF''mCherry蛍光。(G)12 hpf の最適サンプルは、背もてに取り付けられ、同じ平面に両方の視小胞を持つ 12 と 6 時に沿って垂直方向に向けられる。(H)最適でないサンプル:背部および光学小胞は互いに平面に取り付けられているが、このサンプルは対角軸に向けられ、開発が進むにつれてフレームサイズから成長する。(I) 最適でないサンプル: このサンプルは、過度に回転し、後回しではなく、結果として、視小胞の前部がタイムラプスで捕捉されない。(J)最適でないサンプル: このサンプルは、回転が少なくて、後部の小胞の後部がタイムラプスに捕捉されない結果として、後方のマウントではありません。点線は各視小数を示します。スケールバー:B = 0.1ミリメートル。D' = 1.5 mm;E', F' = 2.5 mm;G = 0.1 mm.この図の大きなバージョンを表示するには、ここをクリックしてください。
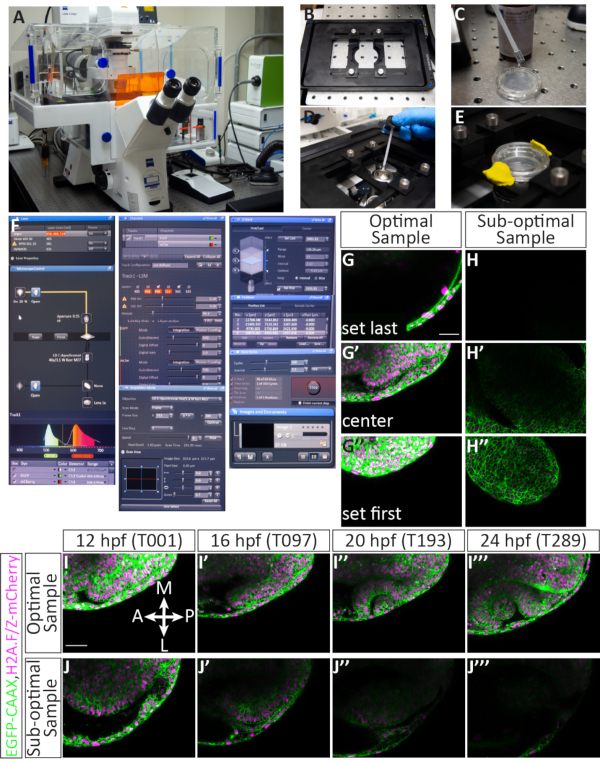
図 4: 複数のポジションのタイムラプスと潜在的な結果を設定します。 (ある) レーザー走査共焦点顕微鏡。(B)ピエゾZステージインサート。(C) アガロースに埋め込まれた胚を含むガラス底皿の底部は、水の屈折率に一致する浸漬培地にコーティングされる。(D) 浸漬媒体の液滴は、40x Wの目的(長時間作動距離)に置かれる。(E)皿は、ステージインサートで所定の位置に保持され、E3はアガロース層の上に追加され、蓋はモデリング粘土で固定されています。(F) 取得ソフトウェア設定: 希望のレーザーラインは、ドロップダウンメニューからオンになります。EGFP-CAAX RNAおよびH2A-mCherry RNAを注入した胚の場合、アルゴンおよびDPSS 561-10レーザーがオンになります。赤いハイライトは、アルゴンレーザーがウォーミングアップしていることを示しています。目的の上にサンプルを見つけるために、透過光は顕微鏡のコントロールパネルを通してオンにされる。EGFPとmCherryの両方がトラック1(同時イメージング用)に割り当てられ、各検出器の範囲が設定されています。ここで、EGFP範囲は494-545 nmに設定され、mCherryの範囲は598-679 nmに設定される。下に チャンネル ・レーザーパワーを設定できます。レーザーが温まった後、電力を5.0まで上げることができ、ゲイン(マスター)を調整することができます。ピンホールは60.2に設定されており、これは1.63エアリーユニットまたは1.6 μmのセクションに相当します。下に 取得 メニューの場合、フレームサイズは 512 x 384、スキャン速度は 9.0、平均は双方向、ズームは 0.7 に設定されます。下に Zスタック メニューでは、Z軸の最初と最後の位置が設定され、コンフォーカルがスキャンされます。間隔(ステップサイズ)は2.1 μmに設定されています。最初と最後の Z 位置を選択する間、63 スライスの標準数は一般的に維持されます。これは、目の全体的な成長に対応します。「ピエゾを使う」オプションが選択されています。下に 位置 メニューの各選択された位置は、割り当てられた番号とX、Y、Zの位置を含むリストされます。追加、更新、削除など、イメージに対する胚の数(別々の位置)を制御する追加のボタンがあります。下に 時系列 メニューの場合、サイクルは300(取得されるZスタックの数)に設定され、間隔(時間ステップ)は2.5分に設定されます。設定が確定すると、startを押してタイムラプス取得が開始されます。ソフトウェアは、Zスタックのスライス、サイクル、および現在スキャンされている位置を表示します。タイムラプスが完了すると、ファイルは、フロッピー ディスクのアイコンをクリックして保存されます。 画像とドキュメント パネル。(G-J) 細胞膜 (green)は、EGFP-CAAXおよび細胞核(クロマチン、 magenta) は H2A-mCherry RNA でラベル付けされています。(G,G',G'') 最適にマウントされたサンプルの最初と最後の Z スライスを設定する例。最初のZスライス(後側)は裏側の外皮を犠牲にして来、最後のZスライス(腹側)は腹側の成長に対応するために、視縁の下にあります。(H,H',H'') 最適でないサンプルの最初と最後の Z スライスを設定する例。サンプルはmCherry蛍光が弱く、斜めに取り付けられ、現像型光学カップがタイムラプスの間フレームにとどまる可能性が低い。(私は、私は、私は、私''') 最適サンプルからのタイムラプスの例Zスタックの中央からの単一のスライス画像は、4つのポイント(T値)で63スライスボリューム全体から撮影されます。Br, 脳;NR, 神経のレチナ;RPE、 網膜色素上皮;ル、レンズ。(J,J',J',J'') 最適でないサンプルからのタイムラプスの例。この場合、サンプルはZ面から移動し、前視カップの斜め部分のみが捕獲された。スケールバー:G、I = 50 μm。 この図の大きなバージョンを表示するには、ここをクリックしてください。
動画1:最適な野生型胚における光学カップ形態形成のタイムラプス。 EGFP-CAAX(原形質膜、 緑色)およびH2Aで標識された野生型胚の背部図。F/Z-mCherry (クロマチン、 マゼンタ).タイムラプスは~12~24 hpfで、4Dデータセット全体から1つの共焦点セクションが含まれています。時間間隔は z スタック間で 2.5 分で、毎秒 22.5 フレームで表示されます。この映画をダウンロードするには、ここをクリックしてください。
ディスカッション
ここでは、視カップ形態形成の toto 標識および4Dタイムラプスイメージングのためのプロトコルについて説明する。私たちは、異なる細胞内区画をマークするために蛍光タンパク質をコードするキャップRNAを生成するプロセスを進めます。ゼブラフィッシュ1細胞胚を注入する;多重化イメージングのためのアガロースに11 hpf胚を埋め込む。そして、光学カップ形態形成(12-24 hpf)の間に複数の胚の4Dデータセットを取得する。
これらの情報密度の高いデータセットでは、無数の質問に答えることができます。4Dデータは、さまざまな方法で視覚化し、定量的に分析することができます。このプロトコルの範囲外ですが、達成できるものの種類の例として、目標と標準アプリケーションの一部をここに挙げてください。もちろん、定量的な画像解析方法は絶えず開発されており、市販のツールとカスタムメイドのツールの両方を使用できます。そのような方法を使用していない場合は、取得したデータセットが選択した定量的分析アプローチに適していることを確認するために、何らかの最適化を行う準備をする必要があります。
4D データセットの視覚化と定量的な評価は、ファイルのサイズが原因で困難になる場合があります。取得ソフトウェアを使用してデータセットを個別の胚に分割し、ImageJ/Fiji を使用して、コンフォーカル ファイルを商用形式からより標準的な tif スタックに変換し、各時間ポイントを別のファイルとして保存することができます。これにより、ファイル サイズが小さくなり、ファイル形式が標準化されます。各タイムポイントからの個々の光学セクションは、ImageJ/Fiji を使用して2D(XY)タイムラプスとして組み立てることができ、データの迅速な2D可視化と評価が可能になります。映画1は正確にこれを示す例です:図4I–I''に示す最適サンプルのタイムラプスとして時間をかけて組み立てられた単一の光学セクション。そこから、3Dおよび4Dビジュアライゼーションのために、我々は一般的に、自由に利用できるが、特定のグラフィックスカードの要件を持っているFluoRender35、36を使用しています。FluoRender を使用すると、任意の軸で 3D レンダリングデータを回転させ、時間の経過とともにデータセットを 4D で視覚化し、任意の平面でカットアウェイを生成し、回転とビジュアライゼーションをムービーまたは一連の tif イメージとして保存できます。
定量分析の面では、答えるべき多くの質問があります。私たちは、光学カップ形態形成の根底にある細胞挙動を理解するという独自の目標を支援するソフトウェアを社内で開発しました。私たちのプログラム、LongTrackerは、セル13を追跡するための位置のプロキシとして核信号を使用しています。これらのデータを使用して、セルが移動するタイミング、場所、および方法を決定できます。細胞の移動速度と距離。細胞が分裂する頻度。当社独自のソフトウェアに加えて、4Dセルトラッキング用の複数の市販およびカスタムメイドのオプションがあります。我々はまた、神経残の37内の細胞のセグメンテーションを行い、細胞の形状と組織を定量化するプログラムLongAxisを開発しました。ただし、LongAxis に入力されるデータセットは、高解像度で取得された単一の Z スタックです。永続的な課題は、細胞を自信を持ってセグメント化し、その形態を外挿するのに十分な解像度のタイムラプスデータセットを生成することです。その他の1つは、細胞形状の直接可視化のためにKaedeなどの光変換可能な蛍光素を用いたモザイク(スパース)ラベリングであり、我々および他の人たちが発達眼8、11、13で行ったように。これにより、セルのセグメンテーションの問題が簡単になり、FluoRender での 3D レンダリングを通じてセルの形状とサイズを簡単に定量できます。
このプロトコルの各ステップは、私たちの目的のために特別に最適化されました。このプロトコルの特異性はいくつかの制限をもたらします:書かれたプロトコルは、ゼブラフィッシュの目の発達(例えば、眼の神経新生など)、他の眼構造、他の発達段階、または他の細胞内区画の他の側面をイメージングするために最適化されていません。埋め込みの向き、イメージングの速度、ラベルはすべて、生物学的な質問に答えるように設計されています。画像の神経新生を画像化するために、例えば、ここで説明する胚の後方方向は、目的の特定の特徴の可視化を可能にしないことがあり、かつ、イメージングの速度が適切でない場合がある。プロトコルの多くの側面は、自分の特定の興味や目標に応じて、さまざまなニーズに合わせて適応および変更することができます。まず、RNA注射を使用すると、ラベリングプロセスが非常に柔軟になります。蛍光タンパク質融合構築物は、対象となる細胞下の構造を標識するために使用することができ、RNA注入量は、標識を最適化するために変化させることができる。光変換可能な蛍光系Kaedeの研究に基づいて、RNA注射は12 hpf11,13で終わった翻訳のバーストをサポートしているように見えます。RNA注入による高レベルの蛍光タンパク質発現は、光漂白と戦うことができるが、そのようなアプローチは、目的の蛍光標識の持続的な発現をサポートしていない。後の段階で胚を画像化する場合など、持続的な発現が必要な場合、トランスジェニックラインはオプションであり、ゼブラフィッシュで新しいラインを構築することは簡単です24.
次に、プロトコルは、開発の後期段階に適応することができます。顔料は後の段階で画像化を妨げる可能性があるため、胚をフェニルチオ尿素(PTU)で処理して顔料形成を阻害したり、色素合成のための遺伝子変異体を使用することができる。胚のけいれんを防ぐために、トリケーヌはアガロースおよび胚培地オーバーレイ溶液に添加することができ、アガロース率は必要に応じて調節することができる。目が大きくなるにつれて、取り付け方向を変更する必要があるかもしれません。ここでは、胚をドーサリーに取り付けますが、後の段階では、目的の構造に応じて横または前に取り付ける方が理にかなっているかもしれません。空間的スケールと時間スケールの異なるプロセスが発生するため、画像取得の Z ステップと時間ステップを最適化することもできます。これらの機能は、実際には各ラボのニーズに合わせて経験的にしか決定できません。
最後に、このプロトコルは、目の発達の初期段階で比較的高い割合のアガロースに埋め込まれた胚を用いて、レーザー走査共焦点顕微鏡用に特別に開発されました。眼の発達中に異なる時間または異なる場所でイメージングしている場合、このプロトコルは目的のプロセスに適応する必要があります。現在、多くの異なるイメージング手法が可能であり、光学エンジニアによって開発されています。各アプローチは、イメージング用の胚を埋め込み、取り付けるさまざまな方法から、さまざまなファイルサイズやフォーマットにまで、独自の課題をもたらします。最大限の空間解像度と時間分解能がフォトブリーチ、光毒性、および膨大なファイルサイズとバランスを取る最適化プロセスをガイドするための導入での考慮事項を概説します。これらの一般的な原則は、上記の実用的な情報に加えて、他の人が目の発達における多くのオープンな質問を研究するためのタイムラプスイメージングアプローチを確立するのに役立つことを願っています。
開示事項
著者らは開示するものは何もない。
謝辞
私たちは、このプロトコルのタイムラプスアプローチと議論に取り組むために、クワンラボの過去と現在のメンバーに感謝しています。この作業は、NIH(R01 EY025378およびR01 EY025780からKMKまで)によってサポートされました。F31 EY030758 から SL;および T32 GM007464 を MAC にします。
資料
| Name | Company | Catalog Number | Comments |
| Delta TPG Dish 0.17mm clear | Bioptechs | 0420041500C | coverglass bottom for optical compatibility |
| Disposable Pasteur Pipettes | VWR | 14672-608 | overall length 14.6 cm (53/4") |
| Dumont #5, #55, or #5SF Forceps | Fine Science Tools | 11254-20 | For style #5: straight tip, 0.05 x 0.01 mm tip, inox, 11 cm long |
| Flaming/Brown Micropipette Puller | Sutter | P-97 | |
| Immersol W | Carl Zeiss Microscopy | 4449690000000 | immersion fluid for water-emission objectives, halogen-free, low fluorescence |
| Microinjection Mold | Adaptive Science Tools | TU-1 | Six ramps, one 90 degree and one 45 degree beveled side. |
| Microloader Tips | Eppendorf | 930001007 | Microloader, tip for filling glass microcapillaries, 0.5 – 20 µL |
| mMessage mMachine SP6 Transcription Kit | Invitrogen | AM1340 | contains SP6 Enzyme Mix, 10X Reaction Buffer, 2XNTP⁄CAP Solution, GTP Solution, pTRI-Xef, TURBO DNase, Ammonium Acetate Stop Solution, Lithium Chloride Precipitation Solution, and Gel Loading Buffer and are all stored at –20°C. Nuclease-free Water may be stored at any temperature. |
| Nikon SMZ645 Stereo Microscope | Nikon | SMZ645 | used for injecting embryos (see Fig. 2B) |
| NotI-HF enzyme | NEB | R3189S | comes with Gel Loading Dye, Purple (6X) |
| NuSieve GTG Agarose | Lonza | 50081 | fine resolution, low melt agarose |
| Objective LD "C-Apochromat" 40x/1.1 W Korr M27 | Carl Zeiss Microscopy | 421867-9970-000 | |
| Olympus SZX16 Stereo Microscope | Olympus | SZX2-ZB16 | used for screening, embedding embryos (see Fig. 3A) |
| Phenol red solution | Sigma-Aldrich | P0290 | 0.5% in DPBS, sterile filtered, endotoxin tested, cell culture tested |
| Pico-liter Microinjector | Harvard Apparatus | PLI-100 | Femtoliter to microliter injections; digital readouts for injection count, time, and pressure; contains 5 pressures including inject, balance, clear, fill, and hold |
| Pipette Pump Pipettor | SP Bel-Art | F37898 | fits a disposable pasteur pipette, contains thumbwheel on side for aspirating or dispensing and plunger for quick emptying |
| Premium Thin Wall Borosilicate Glass Capillaries | Warner Instruments | G100T-4 | 1.0 x 0.78 mm |
| QIAquick PCR Purification Kit | Qiagen | 28106 | contains QIAquick Spin Columns, Buffers, Collection Tubes (2 ml) |
| RNase-free H2O | Invitrogen | AM9906 | DNase-Free, Molecular Biology Grade, RNase-Free |
| RNeasy Mini Kit | Qiagen | 74104 | contains RNeasy Mini Spin Columns, Collection Tubes (1.5 ml and 2 ml), RNase-free Reagents and Buffers |
| Stage Attachment Z PIEZO WSB 500 | Carl Zeiss Microscopy | 432339-9000-000 | |
| Stage Insert Z PIEZO WSB Universal | Carl Zeiss Microscopy | 432339-9030-000 | |
| Zeiss LSM 880 with Airyscan | Carl Zeiss Microscopy | N/A | inverted Axio Observer microscope |
| Zen Black 2.3 SP1 software | Carl Zeiss Microscopy | N/A | Zeiss Efficient Navigation (ZEN) controls all light microscope systems from Zeiss |
参考文献
- Adler, R., Canto-Soler, M. V. Molecular mechanisms of optic vesicle development: complexities, ambiguities and controversies. Developmental Biology. 305 (1), 1-13 (2007).
- Chow, R. L., Lang, R. A. Early eye development in vertebrates. Annual Review of Cell and Developmental Biology. 17, 255-296 (2001).
- Fuhrmann, S. Eye morphogenesis and patterning of the optic vesicle. Current Topics in Developmental Biology. 93, 61-84 (2010).
- Martinez-Morales, J. R., Wittbrodt, J. Shaping the vertebrate eye. Current Opinion in Genetics and Development. 19 (5), 511-517 (2009).
- Yang, X. J. Roles of cell-extrinsic growth factors in vertebrate eye pattern formation and retinogenesis. Seminars in Cell & Developmental Biology. 15 (1), 91-103 (2004).
- Bogdanovic, O., et al. Numb/Numbl-Opo antagonism controls retinal epithelium morphogenesis by regulating integrin endocytosis. Developmental Cell. 23 (4), 782-795 (2012).
- Bryan, C. D., Casey, M. A., Pfeiffer, R. L., Jones, B. W., Kwan, K. M. Optic cup morphogenesis requires neural crest-mediated basement membrane assembly. Development. 147 (4), (2020).
- Bryan, C. D., Chien, C. B., Kwan, K. M. Loss of laminin alpha 1 results in multiple structural defects and divergent effects on adhesion during vertebrate optic cup morphogenesis. Developmental Biology. 416 (2), 324-337 (2016).
- Cavodeassi, F., Ivanovitch, K., Wilson, S. W. Eph/Ephrin signalling maintains eye field segregation from adjacent neural plate territories during forebrain morphogenesis. Development. 140 (20), 4193-4202 (2013).
- England, S. J., Blanchard, G. B., Mahadevan, L., Adams, R. J. A dynamic fate map of the forebrain shows how vertebrate eyes form and explains two causes of cyclopia. Development. 133 (23), 4613-4617 (2006).
- Gordon, H. B., et al. Hedgehog signaling regulates cell motility and optic fissure and stalk formation during vertebrate eye morphogenesis. Development. 145 (22), (2018).
- Ivanovitch, K., Cavodeassi, F., Wilson, S. W. Precocious acquisition of neuroepithelial character in the eye field underlies the onset of eye morphogenesis. Developmental Cell. 27 (3), 293-305 (2013).
- Kwan, K. M., et al. A complex choreography of cell movements shapes the vertebrate eye. Development. 139 (2), 359-372 (2012).
- Martinez-Morales, J. R., et al. Ojoplano-mediated basal constriction is essential for optic cup morphogenesis. Development. 136 (13), 2165-2175 (2009).
- Miesfeld, J. B., et al. Yap and Taz regulate retinal pigment epithelial cell fate. Development. 142 (17), 3021-3032 (2015).
- Nicolas-Perez, M., et al. Analysis of cellular behavior and cytoskeletal dynamics reveal a constriction mechanism driving optic cup morphogenesis. eLife. 5, (2016).
- Picker, A., et al. Dynamic coupling of pattern formation and morphogenesis in the developing vertebrate retina. PLoS Biology. 7 (10), e1000214 (2009).
- Rembold, M., Loosli, F., Adams, R. J., Wittbrodt, J. Individual cell migration serves as the driving force for optic vesicle evagination. Science. 313 (5790), 1130-1134 (2006).
- Sidhaye, J., Norden, C. Concerted action of neuroepithelial basal shrinkage and active epithelial migration ensures efficient optic cup morphogenesis. eLife. 6, (2017).
- Driever, W., et al. A genetic screen for mutations affecting embryogenesis in zebrafish. Development. 123, 37-46 (1996).
- Haffter, P., et al. The identification of genes with unique and essential functions in the development of the zebrafish, Danio rerio. Development. 123, 1-36 (1996).
- Nusslein-Volhard, C. The zebrafish issue of Development. Development. 139 (22), 4099-4103 (2012).
- Hoshijima, K., et al. Highly efficient CRISPR-Cas9-based methods for generating deletion mutations and F0 embryos that lack gene function in zebrafish. Developmental Cell. 51 (5), 645-657 (2019).
- Kwan, K. M., et al. The Tol2kit: a multisite gateway-based construction kit for Tol2 transposon transgenesis constructs. Developmental Dynamics. 236 (11), 3088-3099 (2007).
- Almeida, A. D., et al. Spectrum of Fates: a new approach to the study of the developing zebrafish retina. Development. 141 (9), 1971-1980 (2014).
- Baye, L. M., Link, B. A. Interkinetic nuclear migration and the selection of neurogenic cell divisions during vertebrate retinogenesis. The Journal of Neuroscience. 27 (38), 10143-10152 (2007).
- Clark, B. S., et al. Loss of Llgl1 in retinal neuroepithelia reveals links between apical domain size, Notch activity and neurogenesis. Development. 139 (9), 1599-1610 (2012).
- Das, T., Payer, B., Cayouette, M., Harris, W. A. In vivo time-lapse imaging of cell divisions during neurogenesis in the developing zebrafish retina. Neuron. 37 (4), 597-609 (2003).
- Kay, J. N., et al. Transient requirement for ganglion cells during assembly of retinal synaptic layers. Development. 131 (6), 1331-1342 (2004).
- Norden, C., Young, S., Link, B. A., Harris, W. A. Actomyosin is the main driver of interkinetic nuclear migration in the retina. Cell. 138 (6), 1195-1208 (2009).
- Suzuki, S. C., et al. Cone photoreceptor types in zebrafish are generated by symmetric terminal divisions of dedicated precursors. Proceedings of the National Academy of Sciences of the United States of America. 110 (37), 15109-15114 (2013).
- Wan, Y., et al. The ciliary marginal zone of the zebrafish retina: clonal and time-lapse analysis of a continuously growing tissue. Development. 143 (7), 1099-1107 (2016).
- Yoshimatsu, T., et al. Presynaptic partner selection during retinal circuit reassembly varies with timing of neuronal regeneration in vivo. Nature Communications. 7, 10590 (2016).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental Dynamics. 203 (3), 253-310 (1995).
- Wan, Y., Otsuna, H., Chien, C. B., Hansen, C. An interactive visualization tool for multi-channel confocal microscopy data in neurobiology research. IEEE Transactions on Visualization and Computer Graphics. 15 (6), 1489-1496 (2009).
- Wan, Y., Otsuna, H., Chien, C. B., Hansen, C. FluoRender: an application of 2D image space methods for 3D and 4D confocal microscopy data visualization in neurobiology research. IEEE Pacific Visualization Symposium. , 201-208 (2012).
- Carney, K. R., Bryan, C. D., Gordon, H. B., Kwan, K. M. LongAxis: a MATLAB-based program for 3D quantitative analysis of epithelial cell shape and orientation. Developmental Biology. 458 (1), 1-11 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved