Method Article
クライオEM実験のための情報管理システムgP2S
要約
gP2SはクライオEM実験の追跡のためのウェブアプリケーションです。主な機能と、アプリケーションのインストールと構成に必要な手順について説明します。設定が完了すると、アプリケーションは、陰性染色およびクライオEM実験に関連するメタデータを正確に記録することができます。
要約
極低温電子顕微鏡(cryoEM)は、タンパク質標的の結晶学が必ずしも達成可能ではなく、cryoEMが構造ベースのリガンド設計をサポートする代替手段を提供するため、多くの創薬プロジェクトの不可欠な部分となっています。多数の異なるプロジェクトを扱う場合、そして各プロジェクト内で、潜在的に多数のリガンド-タンパク質の共構造を扱う場合、正確な記録を急速に保つことは困難になります。サンプル調製、グリッド調製、顕微鏡検査段階など、各ターゲットに対して多くの実験パラメータが調整されます。したがって、正確な記録保持は、長期的な再現性を可能にし、特にクライオEMワークフローのステップが異なるオペレータによって実行される場合に効率的なチームワークを促進するために非常に重要です。この課題に対処するために、我々はgP2Sと呼ばれるcryoEMのためのウェブベースの情報管理システムを開発しました。
アプリケーションは、サンプルから最終的なアトミック モデルまでの各実験をプロジェクトのコンテキストで追跡し、アプリケーション内で管理される一覧、または外部で別のシステムに保持されます。消耗品、装置、プロトコル、ソフトウェアのユーザー定義制御語彙は、cryoEMワークフローの各ステップを構造化された方法で記述するのに役立ちます。gP2S は、広く構成可能であり、チームのニーズに応じて、スタンドアロン製品として存在するか、プロジェクト管理ツールと REST API を介して統合する、タンパク質や小分子リガンドの生産を追跡するアプリケーション、またはデータ収集と保存を自動化するアプリケーションを使用して、科学アプリケーションのより広範なエコシステムの一部である可能性があります。主要な実験メタデータやパラメータ値を含む各グリッドおよび顕微鏡セッションの詳細を登録することができ、各実験アーティファクト(サンプル、グリッド、顕微鏡コピーセッション、マップなど)の系統が記録されます。gP2Sは、チームの正確な記録保持を可能にするcryoEM実験的ワークフローオーガナイザーとして機能し、オープンソースライセンスの下で利用可能です。
概要
クライオエム施設での情報管理
2014年頃から、極低温電子顕微鏡(cryoEM)1施設の数は爆発的に増加しており、世界中に少なくとも300のハイエンドシステムが設置され、製薬会社を含む2つ以上のハイエンドシステムが、創薬におけるcryoEMの役割の増大を反映しています3。これらの施設の使命と、データ追跡と管理のためのそれらの要件は4.一部の人々は、例えば、全国のcryoEMセンターは、EMグリッドを受信し、データセットを収集し、おそらくいくつかの自動画像処理の後に、構造決定のためにユーザーにデータを返すことを担当しています。このような施設では、グリッドの証明、ユーザー提案または付与との関連付け、およびグリッドからデータセットへの系統を追跡することは非常に重要ですが、タンパク質サンプルの精製方法や最終的な構造決定プロセスなどの他の要因は、関連性が低いか、まったく関係ありません。地元の学術施設などの他の施設では、各エンドユーザーが独自のサンプルとグリッドを準備し、顕微鏡を行い、生データとその処理を管理し、結果を公開する責任があります。この役割はエンド ユーザーまたは主任調査員によって果たされるため、このような施設のメタデータ追跡には厳密な必要はありません。
当社のクライオエム機能では、サンプル、グリッド、データ収集および処理プロトコル、および結果(マップ、モデル)の取り扱いと最適化が、少数の開業医グループに多くのプロジェクトにわたって集中化されています。これは、実験的な(メタ)データ管理の課題を提示します。原子モデルからタンパク質やリガンドの正確な同一性に至るまで、構造の実験的系統は、グリッド調製パラメータおよびデータ収集プロトコルを介して正確に捕捉され、保存されなければならない。これらのメタデータは、多くの人間のオペレータが利用できるようにする必要があります。例えば、画像処理を行う人は、タンパク質を精製したり、cryoEMデータ自体を収集したりしていないにもかかわらず、タンパク質のどのコンストラクトが使用され、イメージングパラメータが何であるかを知る必要があるかもしれません。自動データ管理デーモンなどの情報システムは、ディレクトリ名を正しく体系的に割り当てるために、現在データを収集しているプロジェクトを特定する必要があります。
クライオエム機能をサポートするために、いくつかの情報管理システムが利用できます。EMEN2 5 は電子ラボ ノートブック、情報管理システム、およびビジネス プロセス管理ツールの一部の要素を組み合わせた EMEN25です。多くのシンクロトロンで使用されているISPyB6は、もともと結晶学用のX線ビームをサポートするために構築され、現在ではクライオEMデータ収集もサポートしています。Scipion7は、画像処理ワークフローを記録し、例えばパブリックリポジトリEMPIAR8、9を介して共有することができ、また、オンザフライクライオEMデータ処理を可能にするためにISPyBと統合されている画像処理パッケージの豊富で強力なラッパーです。
ここでは、精製タンパク質や小分子リガンドから最終原子モデルまでのワークフローをサポートするために構築された最新かつ軽量のクライオEM情報管理システムであるgP2S(ジェネンテックタンパク質から構造への)について説明します。
gP2S の概要
gP2Sは、クライオEMラボやマルチユーザー、マルチプロジェクト施設の正確な記録保持を容易にする、ユーザーフレンドリーなWebベースのクライオエム情報管理システムです。プロジェクト、機器、消耗品、プロトコル、サンプル、グリッド、顕微鏡セッション、画像処理セッション、マップ、およびアトミックモデルのエンティティ、それらの関係、および関連メタデータが追跡されます。また、ユーザーは、任意で、ファイルの添付ファイルを含むフリーテキストのコメントを追加することができ、gP2Sに登録された任意のエンティティの豊富な注釈を可能にします。フロントエンドはタッチスクリーンデバイスでの使用を容易にするように設計されており、12.9インチiPad Proで広範囲にテストされており、サンプルとグリッド(図1)を準備しながらラボベンチでgP2Sを使用できるようにし、顕微鏡、画像の処理、またはモデルの堆積時にコンピュータでgP2Sを使用することが可能です。フロントエンドの各ページは、可能な場合はパラメータを有効なデフォルト値に事前設定することで、手動でのデータ入力を減らすことを目的としています。
gP2S のバックエンドには、多くの REST API (REpresentational 状態転送アプリケーション プログラミング インターフェイス) エンドポイントが搭載されており、既存のワークフローやスクリプトに gP2S を統合することが可能です。データ モデルは、分岐を含む、分岐、複数の顕微鏡セッションからのデータを 1 つのデータ処理セッションにマージする、複数のマップを生成する 1 つのデータ処理セッションなど、負の汚れと cryoEM ワークフローを正確にキャプチャできるように設計されています。
システムアーキテクチャ
gP2S は、従来の 3 層アプリケーションです (図 2)。このモジュール式アーキテクチャでは、システムは 3 つの個別の層に分割され、それぞれが個別の業務を行う責任を負い、それぞれが他のシステムとは独立して交換可能または変更可能です。(1) プレゼンテーション層(またはフロントエンド)は、ウェブブラウザ(ChromeとSafariで広範囲にテスト)を介してユーザーアクセスを提供し、ワークフロー要素(データ検証を含む)を作成および変更し、実験データを個々のエンティティ、プロジェクトベースのリスト、および完全なワークフローレポートとして表示します。(2) サービス層 (またはバックエンド) は、ユーザー インターフェイスとストレージ システムの間の仲介レイヤとして機能します - コア ビジネス ロジックを保持し、フロントエンドで使用されるサービス API を公開し、ユーザー認証用のデータ ストレージおよび LDAP (ライトウェイト ディレクトリ アクセス プロトコル) システムと統合し、外部システムとの追加統合の基礎を提供します。(3) パーシスタンス層 (データアクセス) は、実験データ、ユーザーコメント、および添付ファイルの保存を担当します。
主要な技術とフレームワーク
gP2Sアプリケーションの開発、構築、メンテナンスを容易にするために、プロジェクトではいくつかの技術とフレームワークが使用されました。最も重要なものは、Vue.jsフロントエンド用2.4.210、 バックエンド用のTomcat 8サーバーが埋め込まれたSpringBoot 1.311 です。アプリケーションは、ストレージ用に MySQL 5.7 および MongoDB 4.0.6 データベースを使用し、認証に LDAP12 を使用します。既定では、これらのコンポーネント パーツはすべて 1 つのアプリケーションとして出荷およびデプロイされます。
合計で、アプリケーションは直接的または間接的に何百もの異なるライブラリを使用します。最も顕著なものは 、表1にリストされています。
データモデル
gP2S データモデルでは、3 つのタイプのエンティティを区別できます (図 3): 実験中に収集されたデータに関連するワークフロー エンティティ (サンプルセッションや顕微鏡検査セッションなど)。すべてのプロジェクトで共通のデータを記述する機器およびプロトコルエンティティ(例えば、顕微鏡またはガラス化プロトコル)。システムで支持的または技術的な役割を果たす他のエンティティ (コメントやデフォルト値など)。
ワークフロー データ ツリーのルートは、プロジェクト エンティティです。すべてのプロジェクトは、サンプルエンティティを作成するためのビルディングブロックであるタンパク質および/またはリガンドの数で構成されています。各サンプルは、複数のグリッドを作成するために使用することができ、その結果、顕微鏡セッション(顕微鏡観察セッションごとに1グリッド)で使用されます。後者は、1 つ以上のマップを生成できる処理セッションに割り当てられます。ツリーの最後のエンティティは、1 つまたは複数のマップを使用して作成されたアトミック モデルです。結果として、タンパク質からモデルまで、すべてのワークフロー関連エンティティは、常にその祖先を介して特定のプロジェクトにバインドされます。このような設計では、フロントエンド モジュールまたは API を使用した外部システムで処理しやすいデータ集計が作成されます。
ワークフロー データに加えて、実験で使用される機器や、グリッドの準備中に従ったプロトコルを記述するエンティティがあります。これらのエンティティを定義することは、グリッド、顕微鏡、および処理セッションなどの実験的なワークフローエンティティを作成するための前提条件です。
「Other」と総称される最後のタイプのデータエンティティは、技術的な目的(添付ファイルやデフォルト値など)に使用されます。このカテゴリには、ワークフローまたは機器/プロトコル エンティティにリンクできるコメント エンティティが含まれます。
ソフトウェアの可用性
gP2S のオープンソースバージョンは、apache ライセンス バージョン 2.026の https://github.com/arohou/gP2S から入手できます。gP2S を実行する Docker イメージは、https://hub.docker.com/r/arohou/gp2s から入手できます。gP2Sのクローズドソースブランチは、ロシュ&ジェネンテックで開発を続けています。
gP2S アプリケーションの実行
gP2S を実行する方法は、ドッカーコンテナーとして、またはスタンドアロン Java アプリケーションとして実行する方法の 2 つがあります。最適な選択は、ターゲットのデプロイメント環境によって異なります。たとえば、ユーザーの特定のニーズに合わせてコードをカスタマイズまたは拡張する機能が必要な場合は、アプリケーション全体を最初に再構築する必要があります。この場合、gP2S をスタンドアロンアプリケーションとして実行することをお勧めします。
ドッカーコンテナ
gP2S アプリケーションの操作を開始する最も簡単な方法は、それを Docker サービスとして実行することです。そのために、専用の Docker イメージが準備され、Docker Hub リポジトリ (「https://hub.docker.com/r/arohou/gp2s」) で公開されています。gP2S イメージの実行は、MySQL データベースと MongoDB データベース、および LDAP サーバーへのアクセスに依存します。非運用環境では、これらの依存関係をすべて、gP2S アプリケーションと共にマルチコンテナー Docker アプリケーションとして実行することをお勧めします。これをシームレスにするために、最終的な環境のすべての必要な構成を含む docker-compose ファイル (https://github.com/arohou/gP2S/blob/master/docker-compose.yml) が gP2S GitHub リポジトリ (https://github.com/arohou/gP2S) で準備および提供されています。次のドッカーイメージは依存関係です: mysql27、 mongodb28、 apacheds29.
既定の構成では、docker コンテナーを削除すると、エンティティと添付ファイルの両方を格納されているすべてのデータが削除されます。データを保持するには、ドッカーボリュームを使用するか、gP2S アプリケーションを専用のデータベースインスタンス(MySQL および MongoDB)に接続する必要があります。ApacheDS LDAP サーバーコンテナには、事前に構成された管理者ユーザ (パスワード: シークレット) が付属しています。これらの資格情報は、Docker サービスとして実行される場合、gP2S アプリケーションにログインするために使用する必要があります。実稼働環境では、同じ docker-compose ファイルを使用して、サービスとして gP2S (および必要に応じて他のコンテナー) を Docker Swarm コンテナー オーケストレーション プラットフォームにデプロイできます。
適切な構成に関するすべての詳細を含む、gP2S を Docker コンテナーとして実行する完全なプロセスは、gP2S GitHub リポジトリで説明され、次のトピックをカバーしています。
• すべての依存関係を持つドッカー化 gP2S アプリケーションを実行する。
gP2S アプリケーション、データベース、および LDAP へのアクセス。
• gP2S サービスを新しいバージョンで更新する。
• gP2S アプリケーションを削除する。
• データ永続性の設定。
ドッカー化された gP2S アプリケーションを専用データベースまたは LDAP サーバーに接続する。
• 構成の詳細
スタンドアロン Java アプリケーション
gP2S アプリケーションを実行する別のオプションは、自己完結型 Java パッケージを作成することです。Docker コンテナーを実行できない場合は、この方法を使用する必要があります。gP2S アプリケーションをビルドするには、Java 開発キットバージョン 8 以上をインストールする必要があります。ビルドプロセス全体は、GitHub リポジトリのコードベースで提供される Maven ツールによって管理されます。ビルド構成は、最初にフロントエンドパーツをビルドし、次にそれをバックエンドソースにコピーしてから、最終的なアプリケーションとしてビルドする準備ができています。このように、完全に機能する gP2S パッケージを準備するために、他のツールやライブラリをインストールする必要はありません。デフォルトでは、ビルドの結果は JAR パッケージ (ローカルに格納) と Docker イメージ (Maven pom.xml ファイルで構成されたリポジトリにプッシュされます) です。外部システム (データベースと LDAP サーバー) に接続するために必要な情報は、パッケージを構築する前に適切な構成ファイルで指定する必要があることを覚えておくことが重要です。
gP2S JAR パッケージが作成されると、システムをホストする Tomcat アプリケーション・サーバーを含め、アプリケーションの実行に必要なすべての依存関係と構成情報が含まれます。パッケージが複数の構成ファイルでビルドされた場合は、再構築せずに異なるモードで実行できます。
gP2S GitHub リポジトリには、gP2S をスタンドアロン アプリケーションとして構築および実行するプロセスの詳細な説明が含まれており、次のトピックについて説明します。
• Maven ツールを使用した gP2S の構築
• 組み込みデータベースを使用した構築と実行
• Docker コンテナとしてデプロイされた依存関係を使用して構築および実行する
• 専用データベースを使用した構築と実行
• 認証の構成
プロトコル
1. 作業用の gP2S のセットアップ
- gP2S にログオンします。ログインが成功すると、メイン画面が表示されます。
注: 右上にユーザー名が表示されます - ログアウトするには、これをクリックします。左側のナビゲーション バーは、プロジェクト セレクタ (上) 、cryoEM ワークフローを定義する実験エンティティタイプ (サンプル、グリッド、顕微鏡セッション、処理セッション、マップ、およびモデル) を一覧表示するナビゲーション項目のセット、アプリケーションの [設定] セクションへのリンクで構成されます。 - 実験を記録する前に、cryoEM 機能で使用されているプロジェクト、機器、消耗品、ソフトウェア、およびプロトコルに関する情報を設定します。設定は、新しいツールやプロジェクトを追加したり、既存のエントリを編集したりすることで、いつでも更新できます。ただし、gP2S のすべてのエンティティと同様に、一度作成した設定エンティティは削除できません。
2. 少なくとも 1 つのプロジェクトを構成する
- [プロジェクト>設定] に移動します。
- [ 新しいプロジェクトの作成] をクリックします。
- プロジェクト ラベルを入力します。
- [ 保存 ]をクリックします。
3. 少なくとも 1 台の表面処理機を構成します。
注:表面処理機は、EMグリッドの表面特性を変更するために使用されます - 最も一般的には、グロー放電器またはプラズマクリーナーです。
- [ 機器] セクションから、[ 表面処理機]を選択します。
- [ 新しいコンピュータの作成]をクリックします。
- 後でマシンを識別するために使用するラベルを入力します。
- 製造元、モデル、および場所を提供します。
- [ 保存 ]をクリックします。
4. 少なくとも 1 つのグリッドタイプを登録します。
注:グリッドタイプは、特定のバッチやグリッドの多くではなく、グリッド(例えば、「300メッシュ銅グリッド上の2-μmの穴の多いカーボンフィルム」)のモデルを識別するためのものです
- [ 消耗品 ] セクションで 、[ グリッドの種類]を選択します。
- [ 新しいグリッドタイプの作成] をクリックします。
- [グリッド タイプ]ラベル、[製造元]および[説明]を入力します。
- [ 保存 ]をクリックします。
5. 少なくとも1台のガラス化機を登録する
- [ 機器] セクションで、[ ガラス化マシン]を選択します。
- [ 新しいコンピュータの作成] をクリックします。
- 製造元、モデル、および場所を提供します。
- [ 保存 ]をクリックします。
6. 少なくとも1枚のブロッティング紙を登録
- [ 消耗品 ] セクションで [ブロッティング紙] を選択します。
- [ 新しいブロッティング用紙を作成]をクリックします。
- ブロッティング用紙ラベル、製造元、およびモデルを入力します。
- [ 保存 ]をクリックします。
7. 少なくとも 1 つの Cryo ストレージ デバイスを登録する
- [ 機器] セクションで、[ クライオ記憶装置]を選択します。
- [ 新しいストレージ デバイスの作成]をクリックします。
- デバイスの製造元、モデル、および場所を入力します。
- トグル スイッチを設定して、追加したストレージ デバイスがシリンダ、チューブ、ボックスを備えるかどうかを指定します。
注:それが行われる場合、gP2Sは、ユーザーが個々のグリッドの格納場所を記録したときに、後で関連するシリンダー、チューブおよび/またはボックス識別子を指定することができます。上記の機器と消耗品をセットアップすると、表面処理、負の汚れ、ガラス化の3種類のプロトコルを作成できます。
8. 少なくとも 1 つの表面処理プロトコルを登録する
- [ プロトコル] セクションで、[ 表面処理] を選択します。
- [ 新しいプロトコルの作成 ] をクリックします。
- プロトコルを識別するラベルを入力します。
- 表面処理機の 1 つを選択します。
- このプロトコルで使用する設定:放電の持続時間、電流と極性、圧力、大気中の添加剤を指定します。
- [ 保存 ]をクリックします。
9. 少なくとも 1 つの負の汚れプロトコルを作成します。
- [ プロトコル] セクションで 、[ 負の汚れ] を選択します。
- [ 新しいプロトコルの作成 ] をクリックします。
- プロトコル ラベルを入力します。
- その名前、pH、および重金属塩の濃度の値を与えることによって、汚れを記述します。
- ブロットする前に染色のインキュベーション時間を指定します。
- プロトコルの自由書式の説明を入力します。
- [ 保存 ]をクリックします。
10. 少なくとも 1 つのグリッド凍結プロトコルを登録する
- [ プロトコル] セクションで 、[ ガラス化] を選択します。
- [ 新しいプロトコルの作成 ] をクリックします。
- プロトコル ラベルを入力します。
- ドロップダウン リストから、該当する [ガラス化機械] を選択します。
- このプロトコルで使用するブロッティング用紙を選択します。
- 次に、残りの実験情報を提供する:相対湿度、温度、ブロット力、ブロット数、ブロット時間、待ち時間、ドレイン時間、サンプルアプリケーションの数。
- 自由書式の説明を入力します。
- [ 保存 ]をクリックします。
注: プロトコルを設定した後、クライオと負の染色の両方のグリッドを作成できます。gP2Sを使用して、顕微鏡セッションから始めてワークフローの次のステップを記録するには、顕微鏡、電子検出器、サンプルホルダーを構成する必要があります。
11. 少なくとも1つの顕微鏡を登録する
- [機器]セクションで、[顕微鏡]を選択します。
- [新しい顕微鏡を作成]をクリックします。
- 顕微鏡ラベルにタイプします。
- 製造元、モデル、および場所を提供します。
- 80、120、200、300 kVのプリセットリストから、この顕微鏡で構成され、使用できる加速電圧を選択します。
- コンデンサ("C2")と取り付けられている目的絞りのリストを指定します。注:各タイプに対して、最大4つの絞りスロットを設定することができ、そのうちの1つは、この顕微鏡のデフォルトの絞りとして指定されています。目的の絞りの場合、1つ以上のスロットが位相プレートによって取り込まれることを示し、その場合は直径パラメータが無効になります。
- この顕微鏡にオートローダーが装備されているか、サイドエントリーホルダーが必要かを示します。
- 顕微鏡にエネルギーフィルターが取り付けられているかどうかを示します。
- 抽出電圧、ガンレンズの設定、スポットサイズ、エネルギーフィルタのスリット幅(該当する場合)のデフォルト値を指定します。指定された値は、ユーザーが顕微鏡セッションを作成するときに使用されます。
12. 電子検出器を1つ以上登録する
- [機器]セクションで、[電子検出器]を選択します。
- [ 新しい電子検出器の作成] をクリックします。
- ラベル、製造元、およびモデルを入力します。
- この検出器が取り付けられている顕微鏡をドロップダウンリストから選択します。
- この顕微鏡と検出器の組み合わせのために較正された少なくとも1つの倍率を追加します。
- [倍率] で、[ 新規追加] を選択します。
- 公称倍率と較正倍率の両方を指定します。
- 必要なすべての倍率設定に対して、上記の手順を繰り返します。これらの倍率設定は、後で顕微鏡セッションを記録するユーザー用のドロップダウンセレクターで使用できるようになります。
- チェックボックスを使用して、検出器が電子カウント、線量分画、および超解像度に対応しているかどうかを指定します。
- 最後に、検出器の追加仕様を提供します:電子カウント数係数(入射電子によって登録されたカウントの平均数)、各ピクセルの線形次元(μm)、およびピクセルの行と列の数。
- [保存] をクリック します。
13. サイドエントリーサンプルホルダを必要とする顕微鏡が1つ以上ある場合は、gP2Sに使用可能なサンプルホルダーを登録してください。
- [ 機器] セクションで、[ サンプルホルダ]を選択します。
- [ 新しいホルダーの作成] をクリックします。
- ラベル、製造元、モデル、および場所を入力します。
- サンプルホルダの最大傾き(度数)を指定します。
- チェックボックスを使用して、極低温EMグリッドを保持できるかどうか、および二重軸傾斜が可能かどうかを指定します。
- ドロップダウン リストから、このホルダーを使用できるすべての顕微鏡を選択します。
注:これにより、ユーザーがサイドエントリー顕微鏡を使用して顕微鏡セッションを登録する際に、関連するホルダーのみがリストに表示されます。 - [ 保存 ]をクリックします。
14. 各マイクロコピーセッションに関連付けられたディレクトリ名の設定で、gP2Sが従うパターンを指定します。
注: 顕微鏡セッション中に記録された画像データの保存用のディレクトリ名を gP2S に自動的に生成させると便利です。これにより、ストレージ ディレクトリの体系的で情報が豊富な名前付けが保証されます。各顕微鏡セッションに関連付けられたディレクトリ名を設定する際に、gP2S が従うパターンを指定します。
- [管理] セクションで [ 設定] を選択します。
- ディレクトリ名パターン文字列を編集します。
注: この文字列には、プロジェクト ラベル、グリッド ID、グリッド ラベル、顕微鏡セッション ラベル、顕微鏡セッション ID、顕微鏡セッション開始日、顕微鏡セッション開始時刻、および顕微鏡ラベルの変数が含まれます。これらの変数以外のディレクトリ名パターンには、ほとんどの文字が含まれている場合があります。たとえば、既定のディレクトリ名パターンは ${グリッドラベル}_${顕微鏡スタート日付}_${プロジェクトラベル}_${顕微鏡ラベル}_グリッド_${グリッドID}_セッション_セッション_$\顕微鏡セッションID} です。これで、顕微鏡セッションまで、およびマイクロコピーセッションを含む実験エンティティの登録を可能にする十分な設定が構成されました。
15. ユーザーが利用できる画像処理ソフトウェアを登録します。
注: これにより、処理セッションと後のエンティティタイプ (マップおよびモデル) の登録が可能になります。
- [ 画像処理 ] を選択します。
- [ 新しい画像処理ソフトウェアの作成] をクリックします。
- ソフトウェアの名前を入力します。
- ユーザーが使用できるすべてのバージョンを一覧表示します。
- [ソフトウェア バージョン] で、[ 新規追加] を選択します。
- ソフトウェアバージョンを入力します。
注: これにより、ユーザーは画像処理セッションを登録するときに、結果に到達するために使用したソフトウェアのバージョンを正確に指定できます。これで gP2S の必要な設定が完了します。ユーザーは、次のセクションで説明するように、電子顕微鏡実験を記述するキーメタデータを正確にキャプチャできるはずです。
結果
全体的なデザインとナビゲーションパターン
gP2S アプリケーションはプロジェクト指向であり、エンティティはプロジェクトのコンテキスト内でのみ作成できます。関連するプロジェクトは、アプリケーションの左上隅近くにあるドロップダウンから最初に選択されます。便宜上、プロジェクトのリストはフィルタリング可能で、最近使用したプロジェクトを上部に表示してソートされます。プロジェクトを選択すると、このプロジェクトに関連付けられている各タイプのエンティティの数が、左側のナビゲーション バーのワークフロー セクションに表示されます。ユーザーは、ワークフロー エンティティの種類 (たとえば、顕微鏡セッション) をクリックして、選択したプロジェクト内のエンティティの一覧を表示できます (図 4)。このリストは、各エンティティ、ラベル、作成日時、作成したユーザーの名前、このエンティティに関するコメントが行われたかどうかを示す、最大 6 つの主要なメタデータ フィールド (たとえば、各顕微鏡セッション: グリッド、画像の数、開始時間と終了時間、および使用された顕微鏡と検出器) で構成されます。リストされたエンティティのいずれかを選択すると、すべての先祖エンティティの要約リストを含む、このアイテムで使用可能なすべての情報がリストされた詳細ページが開きます (たとえば、顕微鏡セッションの場合は、その親グリッドとサンプルが一覧表示されます)。これにより、たとえばアトミック モデルからサンプルの詳細へのシングル クリック ナビゲーションを有効にするなど、エンティティの "系統" を非常に迅速に移動できます (図 5) 。さらに、gP2S の任意のエンティティは、その詳細ページの右上にある 「コメント」を選択して、フリーテキストのコメントを入力し、オプションで 1 つ以上のファイルを添付することでコメントできます。
サンプル準備
ワークフローの最初のステップで、サンプルについて説明します。これを行うには、まず、少なくとも1つの成分(タンパク質またはリガンド)を定義します。
新しいタンパク質を追加するにはタンパク質ラベルのみが必要ですが、タンパク質をよりよく記述するのに役立ちます PUR ID を追加します (精製識別子の場合)。このフィールドは、任意のテキストを受け入れ、ロット番号やロット番号を含めたり、バーコードラベルの場所として使用したりできます。gP2S がタンパク質登録システムと統合するようにカスタマイズされている場合 (説明を参照)、PUR ID を自動的に検証し、この多くのタンパク質に関する詳細情報を取得および表示するために使用できます。リガンドの場合、ラベルと在庫濃度は必須情報です。その他のフィールドはすべてオプションで、コンセプト(バーコード、共通名、またはその他のリガンド識別子)とバッチ/ロット識別子が含まれます。繰り返しますが、gP2Sがリガンド登録システムと統合するように構成されている場合、コンセプトとロット識別子を使用して、リガンドを記述する外部に保存されたデータ(例えば、その化学構造、アッセイ結果)を取得して表示することができます。
サンプルは、タンパク質とリガンドの任意の組み合わせとその最終濃度によって定義されます。必要に応じて、インキュベーション時間や温度、バッファ、フリーテキストプロトコル記述など、サンプルの他の実験詳細を指定する。
グリッドの準備
サンプルの準備ができたら、[グリッド] に移動します。リスト内の各 Grid のラベルの下に、グリッドタイプ(cryo または stain)とそのグリッドが使用可能かどうかを示す 1 つまたは 2 つの色付きタグが表示されます。新しいグリッドを作成するには、[ 新しいグリッドの作成] を選択します。ラベルを入力し、使用するグリッドタイプと表面処理プロトコル(例えば、グロー放電)を選択します。次に、クライオまたはネガティブな汚れグリッドを準備するかどうかを指定し、前に選択したグリッド準備タイプに応じて、ドロップダウンリストから、ネガティブステインプロトコルまたはガラス化プロトコルが設定されている、事前設定済みの準備プロトコルのいずれかを選択します。次に、ドロップダウン リストから適切なサンプルを選択し、トグル スイッチを使用してサンプルが使用可能なままかどうかを示します (詳細は後述)。選択したサンプルを希釈または濃縮する場合は、「希釈/濃縮」トグルを使用してこれを指定し、関連する希釈係数または濃度係数を指定します。グリッドに適用するボリューム(μL)を指定し、必要に応じてインキュベーション時間を記録することもできます。最後に、グリッドの格納場所を定義します。負のグリッドの場合は、ストレージボックスのラベル/番号とグリッドの位置をボックス内に記録します。クライオ グリッドの場合は、まず一覧からストレージ デバイスを選択し、使用可能で適切なフィールド (設定で既に定義した Cryo ストレージ デバイスのプロパティに応じて、円柱、チューブ、ボックス)の情報を入力します。
上記のワークフローの一部であるサンプルとグリッドは、在庫管理システムの一部です。この機能は、コンポーネントがまだ使用可能かどうかを追跡します。
- タンパク質またはリガンドは、サンプルレベルから利用不可にすることができます。サンプルを作成する際に、Sample のコンポーネントに対して「最後のドロップ」を選択すると、それらのコンポーネントは、将来の使用に使用できないものとしてマークされます。
- 選択したサンプルは、2 つのトグル スイッチの 1 つを使用して使用できないものとしてマークできます。(サンプルの下)または「サンプルは、さらなる使用のために利用可能ですか?(グリッドの下)
- グリッドの可用性を管理するには、[グリッドがストレージに返される]([コピーセッション]) を使用します。デフォルトでは、この値は負の色のグリッドでは「はい」、cryoEM グリッドでは「いいえ」に設定されています。
データ収集
グリッドを登録したら、gP2Sで顕微鏡セッションを作成してデータ収集実験を登録します。顕微鏡セッションは、アプリケーションによって追跡される最も複雑な実験エンティティであり、基本情報、顕微鏡の設定、露出設定、顕微鏡制御の4つのセクションに編成されています。
最初のセクションには、顕微鏡セッションラベル、その開始と終了の日付と時刻、画像化されたグリッド、顕微鏡、検出器、サンプルホルダー(該当する場合)、収集された画像の数などの基本的な情報が含まれています。新しい顕微鏡セッションを作成する場合、システムは自動的に開始日時に入力します。終了日時はオプションです。これは、実験がまだ進行中の間にセッションがシステムに登録され、その終了時刻が正確に分かっていないためです。終了日時が不明な場合は、手動で入力するか、[今]ボタンを使用して現在の日付と時刻を入力します。別の方法は、gP2Sが任意の顕微鏡上で複数の未完成の顕微鏡セッションを許可していないという事実を利用することです。同じ顕微鏡で新しい顕微鏡セッションを開始すると、以前に開始したセッションはすべて自動的に終了済みとしてマークされます。
次の手順で、[グリッド] を選択します。ドロップダウン リストには、現在のプロジェクトで使用可能なすべてのグリッドが表示されます。グリッドを選択すると、その基本情報の一部(誰がいついつ作成されたか、どのサンプルがそれに適用されたか)が表示されます。選択されているグリッドのタイプに応じて、顕微鏡セッションはリストビューで「ステイン」または「クライオ」としてマークされます。
デフォルトでは、現在のプロジェクトで最後に使用された顕微鏡が事前に選択されています。特定の顕微鏡にオートローダーとして定義されたサンプル挿入機構がある場合、これはサンプルホルダーとして表示される情報です。ただし、選択した顕微鏡がサイド入力ホルダーの使用を必要とする場合は、この顕微鏡で動作するように設定されたサンプルホルダーのリストから使用するホルダーを選択します(選択されたグリッドがクライオグリッドの場合は、クライオ可能なホルダーのみがリストされます)。
顕微鏡セッションフォームの2番目のセクションには、抽出と加速度電圧、銃レンズ、C2絞りの直径、客観的な絞り、エネルギーフィルタのスリット幅などの顕微鏡設定に関する情報が含まれています。通常の使用時に、ユーザーが通常デフォルト値から逸脱する必要がないため、これらの設定は変更されることはほとんどありません。
顕微鏡セッションの3番目のセクションには、露出設定に関する情報が含まれています。このセクションでは、次のメタデータが記録されています:拡大(ピクセルサイズ)、スポットサイズ、照らされた領域の直径、露出時間、およびナノプローブ、カウントモード、用量分画および超解像度を使用したかどうか(カウントモード、用量分画および超解像度設定は、選択された検出器がこれらの特徴を有する場合にのみ有効になる)。線量分画を用いた場合、フレーム数および露光率も記録される。
利便性のために、実験的に重要なパラメータの数は、その場で計算され、形式内で表示されます:最終的な画像ピクセルサイズ(Å)、露光率(電子/Å2/s)、総露出(電子/Å2)、フレーム時間(複数可)およびフレームあたりの露出(電子/Å2)。。
顕微鏡セッションの4番目と最後のセクションを使用して、最小ターゲットと最大ターゲットのアンダーフォーカス、およびホールあたりの露出数を記録できます。
gP2Sの顕微鏡セッションは、スクリーニングやデータ収集の目的で、あらゆる種類の顕微鏡作業を登録するために使用できますが、ユーザーにデータ収集セッションの登録に集中するよう求めるのは十分で効率的であり、グリッドが品質管理のために簡単に検査されるだけで、必ずしも顕微鏡セッションとして登録する必要はありません。
画像処理
画像処理作業は、gP2Sに処理セッションエンティティとして記録されます。各処理セッションは、ドロップダウンリストから選択する必要がある1つ以上の顕微鏡セッションに関連しています。使用されたソフトウェアパッケージ(プログラムとバージョン)、顕微鏡写真の数、および選択されたパーティクルの数を示します。必要に応じて、処理のディレクトリーの名前を記録します。
マップデポジション
1つ以上の3次元の再構築が得られたら、マップをgP2Sに堆積させることができる。各マップは、処理セッションに関連付けられており、実際のマップ ファイル (通常は MRC 形式のファイルですが、gP2S では任意のファイル タイプを使用できます) とキー メタデータ: ピクセルのサイズ (Å)、サーフェス レンダリングに推奨される等等角レベル、適用されるシンメトリ、マップの作成に使用されるイメージの数、推定解像度で構成されます。: その最高と最悪の部分だけでなく、平均グローバル解像度で。マップは、フィルター処理、マスク、リサンプリング、または洗練されたバージョンのリレーションシップの種類を使用して、互いに関連付けられます。このような関連付けを登録する場合は、関係のタイプを選択します(例えば、「''または「フィルタ版」のフィルタ版です)。
モデルの堆積
アトミックモデルを取得すると、関連プロジェクトの gP2S のモデル セクションに入金できます。gP2S の最初のリリースのモデル機能はベアボーンです: 実際のモデル ファイル (通常は PDB または mmCIF ファイル) 以外、モデルが派生した解像度 (Å) とマップ (またはマップのリスト) のみが必要です。さらに、モデルが以前に預託されたモデルの洗練されたバージョンであることを示す可能性があります。モデル検証を含む追加の機能は開発中であり、将来的にはgP2Sのオープンソースバージョンに追加される可能性があります。
レポート
gP2Sへのアクセス権を持たない、またはファイルシステムにアーカイブされる可能性のある共同編集者に配布する要約文書を生成する必要がある場合があります。gP2S は、各エンティティ詳細ビュー ページの右上にあるプリンタ アイコンを使用して、この目的のためのレポート機能を提供します。これにより、エンティティとその先祖エンティティ (すべてのコメントを含む) を記述するすべてのメタデータを含む、印刷可能な PDF ファイルが生成されます。この機能は、最終的な原子モデルの系統を経るすべてのデータとメタデータが、顕微鏡セッションとGrid(s)を介して特定のタンパク質および低分子リガンドロットまでさかのぼるすべてのデータとメタデータが単一の文書で利用可能になるため、モデル堆積に続いて特に価値があります。

図 1.ガラス化ラボベンチでiPad上で動作するgP2S。 ユーザーインターフェイスは、実習での使用と正確なメタデータ入力を容易にするタッチスクリーンを使用して操作用に設計されています。 この図の大きなバージョンを表示するには、ここをクリックしてください。
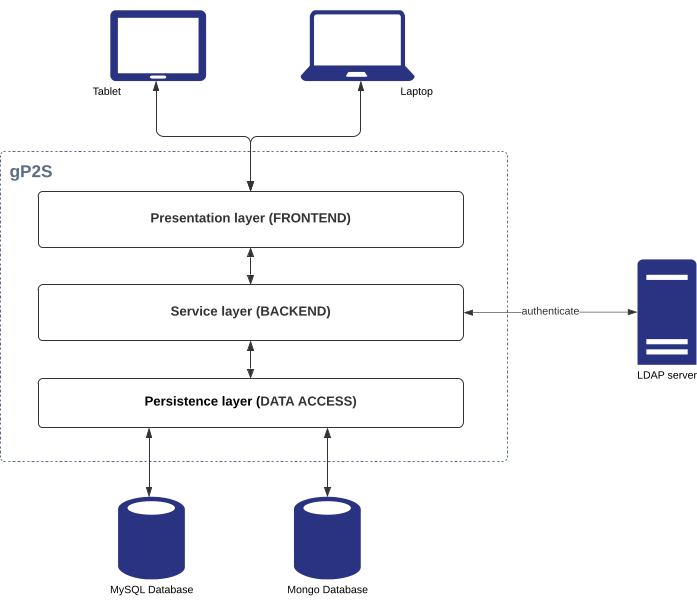
図2:gP2S システムアーキテクチャgP2S は従来の3層組織に従い、データストレージ用に 2 つのデータベースサーバ、ユーザ認証用の LDAP サーバを使用しています。 この図の大きなバージョンを表示するには、ここをクリックしてください。
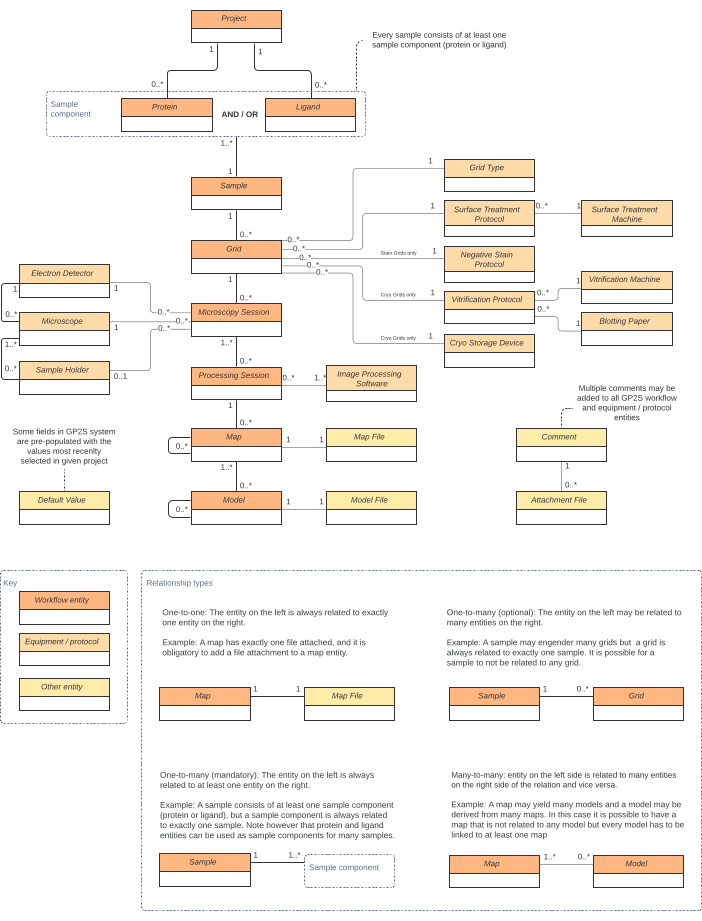
図3: gP2Sデータモデル エンティティは長方形 (ワークフロー エンティティの場合は濃いオレンジ、機器とプロトコルのオレンジ、その他のエンティティタイプの場合は黄色) として表示され、それらの関係は連続する線で示されます (1 対 1、1 対多、多対多)。 この図の大きなバージョンを表示するには、ここをクリックしてください。
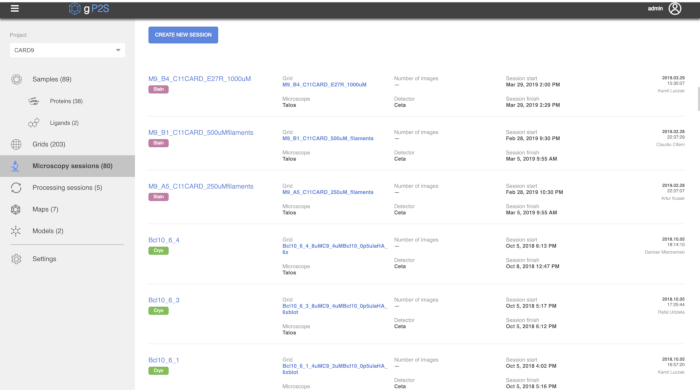
図 4.顕微鏡セッションリストビュー。 このビューでは、選択したプロジェクト (このスクリーンショットの"CARD9")の下に登録されているすべての顕微鏡セッションが一覧表示されます。緑色または紫色のタグは、室温(負の染色)と極低温顕微鏡セッションを区別し、各セッションを記述するいくつかの重要なメタデータがリストされています(例えば、登録したユーザーは右端にあります)。顕微鏡セッションの名前をクリックすると、そのセッションの詳細ビューが開きます(モデルの詳細図を 図5に示します)。 この図の大きなバージョンを表示するには、ここをクリックしてください。
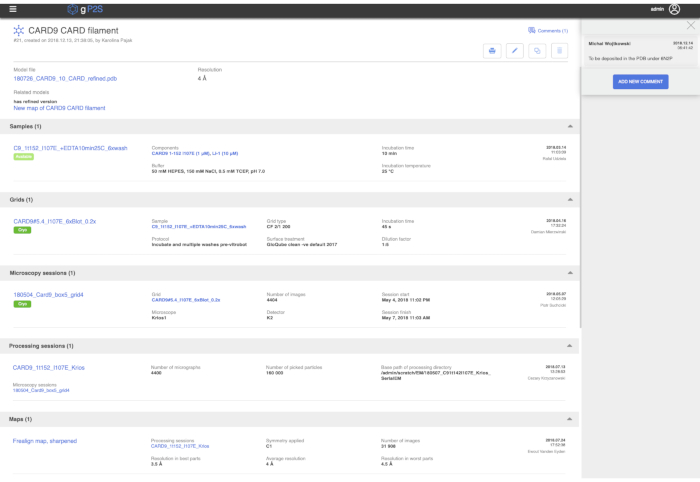
図 5.モデル詳細ビュー。 ページの上部には、選択したモデルで使用可能なメタデータが表示されます。右側のコメントウィンドウは、クロス(右上)または左の「コメント(1)」をクリックすると非表示にすることができます。以下の一連のアイコンは、PDF レポート(プリンターアイコン、メインテキストを参照)の生成、エントリの編集(鉛筆アイコン)、または複製(二重長方形のアイコン)を可能にします。ページの下部には、このモデルの降順となるすべてのエンティティの構造リストが含まれています( サンプルからマップまで)。 この図の大きなバージョンを表示するには、ここをクリックしてください。
| ライブラリまたはフレームワークの名前 | 種類 | バージョン |
| アパッチ | LDAP サーバー | 0.7.0 |
| 港湾労働者 | 開発ツール | 該当する |
| 要素 | 図書館 | 1.4.10 |
| 冬眠する | 図書館 | 5.0.12 |
| ジャワ | プログラミング言語 | 1.8+ |
| ジャワスクリプト | プログラミング言語 | エクマスクリプト 2017 |
| JUnit | 図書館 | 4.12 |
| カルマ | 図書館 | 1.4.1 |
| メイブン | 開発ツール | 3+ |
| モンゴデータベース | DB サーバー | 4.0.6 |
| データベース | DB サーバー | 5.7 |
| ノード.js | フレームワーク | 6.9.1 |
| SASS (ノードサス) | 図書館 | 4.5.3 |
| スプリングブーツ | フレームワーク | 1.3 |
| スワッガー UI | 図書館 | 2.6.1 |
| 雄猫 | アプリケーション サーバー | 8.5.15 |
| ヴュー.js | フレームワーク | 2.4.2 |
| ヴュー・クリエ | 開発ツール | 2.6.12 |
表 1.gP2S で使用されるライブラリとフレームワーク
ディスカッション
gP2S を適切かつ一貫して使用すると、構造化データ モデルと定義済みボキャブラリを使用して重要な実験メタデータの記録を強制することで、高品質のメタデータを適切に記録することができますが、この付加価値は、ラボで高いレベルのコンプライアンスが達成された場合にのみ完全に実現されます。上記のプロトコルは、これを達成する方法をカバーしていません。効果的な施行技術は、顕微鏡オペレータがgP2Sに登録されていないグリッド上のデータを収集することを拒否することであることがわかりました。これは非常に迅速にコンプライアンスを推進し、詳細かつ正確な実験の詳細と企業の記憶の大規模な体の次の数ヶ月間の出現のための地面を築きました。数ヶ月の使用の後、gP2Sに格納されたメタデータのコーパスの価値は、明示的な介入なしにコンプライアンスが高いままであったことをほとんどのユーザーに明らかにしました。
この集合メモリを十分に活用するには、gP2Sに格納されたメタデータが外部システムからアクセス可能であり、実験データ(顕微鏡写真)と結果(マップとモデル)に簡単に関連付けられる必要があります。上記のプロトコルは、gP2Sを他の情報学およびデータ処理システムと統合する方法を説明していません。最も簡単なのは、gP2SのバックエンドREST APIを介した潜在的な統合であり、gP2Sの変更は必要ありません。たとえば、データ収集検出器を制御する各コンピュータは、顕微鏡セッション管理RESTコントローラの下でgP2Sのエンドポイント「getItemBy顕微鏡」を定期的に問い合わせ、顕微鏡観察セッションが顕微鏡上で進行中かどうかを確認するスクリプトを実行します。その場合、スクリプトは gP2S から適切なデータストレージディレクトリ名を取得し(上記の「設定」ページで構成)、この名前を使用してローカルデータストレージデバイス上にディレクトリを作成します。これにより、データストレージディレクトリの体系的な命名が保証され、誤字入力による誤りのリスクが軽減されます。
gP2Sの公開版のソースでコメント化されていますが、gP2Sが外部システムのデータを消費するさらなる統合も可能です。私たちの研究室では、gP2Sの展開は(i)プロジェクト管理システムと統合され、gP2Sで構成された各プロジェクトを会社全体のポートフォリオプロジェクトにリンクさせ、ポートフォリオからのメタデータをgP2S内に表示することができます。(ii) タンパク質登録システムは、gP2Sに添加された各タンパク質が、局所的に保存された識別子を介して、タンパク質の証明を詳述する完全な記録にリンクされるように、関連する分子生物学、発現系および精製の詳細を含む。(iii)低分子化合物管理システムは、gP2Sがその化学構造などの各リガンドに関する重要な情報を表示することを可能にする。これらの統合を有効にするために必要なコードの変更については、gP2S リポジトリ (https://github.com/arohou/gP2S) から入手できる README-BUILD.md ドキュメントの「統合」セクションで説明します。
gP2S の現行バージョンには制限があり、その中からは、過度に単純化されたデータ モデルと構造 (Model) のデポジションのフロントエンドが最初に挙げられており、その中から 1 つ目は制限があります。これは、本格的な構造の堆積および検証機能が現在X線結晶学のサポートと共に開発中であるため、gP2Sのリリース版では意図的に「ベアボーン」状態に置かれました。もう 1 つの設計上の決定は、特権やアクセス許可システムを実装しないことでした: gP2S のすべてのユーザーは、その機能とデータに対して同等のアクセス権を持っています。これは、競合する利益と機密性の要件を持つユーザーグループにサービスを提供する施設にとっては不十分な選択になるかもしれませんが、私たちの施設にとっては懸念事項ではありませんでした。
当社の社内版gP2Sの開発は進行中であり、ここで説明したオープンソース版が他のcryoEMグループに役立ち、将来提案やコードの改善に貢献する可能性があることを期待しています。今後の価値の高い開発は、例えば、ラボ機器(ガラス化ロボット、電子顕微鏡)、ソフトウェア(例えば画像処理メタデータを収穫する)、外部の公共リポジトリ(構造堆積を容易にする)との統合に焦点を当てることができる。
ラボやクライオエムの施設でgP2Sを日常的に使用することで可能になる高品質のメタデータの体系的な収集は、数年間にわたって複数のプロジェクトを並行して起訴する能力に大きなプラスの影響を与える可能性があります。ますます共有され、集中化されたクライオEMグループや施設が確立されるにつれて、gP2Sなどの情報管理システムの必要性は今後も高まり続けると予想しています。
開示事項
すべての著者は、ロシュまたはその子会社ジェネンテックの請負業者または従業員です。
謝辞
著者らは、プロジェクトの設立以来、プロジェクトに取り組んできたgP2S開発チームの他のすべてのメンバーに感謝します: ラファウ・ウッジエラ, セザリー・クシジャノフスキ、プシェミスワフ・スタンコフスキ、ヤチェク・イェムスキ、ピオトル・スチッキ、カロリーナ・パヨンク、エウト・ヴァンデン・アイデン、ダミアン・ミエルツヴィングスキ、ミハウ・ヴォイトコフスキ、ピオトル・ピクサ、アンナ・スルダッカ、カミル・ウチャク、アルトゥール・クサック。また、レイモンド・ハとクラウディオ・シフェリがチームを組み立て、プロジェクトを形作ってくれたことに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| n/a | n/a | n/a | n/a |
参考文献
- Cheng, Y., Grigorieff, N., Penczek, P. A., Walz, T. A Primer to Single-Particle Cryo-Electron Microscopy. Cell. 161 (3), 438-449 (2015).
- . High-End Cryo-EMs Worldwide Available from: https://www.google.com/maps/d/u/0/viewer?mid=1eQ1r8BiDYfaK7D1S9EeFJEgkLggMyoaT (2021)
- Renaud, J. -. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Alewijnse, B., et al. Best practices for managing large CryoEM facilities. Journal of Structural Biology. 199 (3), 225-236 (2017).
- Rees, I., Langley, E., Chiu, W., Ludtke, S. J. EMEN2: An Object Oriented Database and Electronic Lab Notebook. Microscopy and Microanalysis. 19 (1), 1-10 (2013).
- Delagenière, S., et al. ISPyB: an information management system for synchrotron macromolecular crystallography. Bioinformatics. 27 (22), 3186-3192 (2011).
- dela Rosa-Trevín, J. M., et al. Scipion: A software framework toward integration, reproducibility and validation in 3D electron microscopy. Journal of Structural Biology. 195 (1), 93-99 (2016).
- . EMPIAR deposition manual Available from: https://www.ebi.ac.u/pdbe/emdb/empiar/depostion/manual/#manScipion (2021)
- Iudin, A., Korir, P. K., Salavert-Torres, J., Kleywegt, G. J., Patwardhan, A. EMPIAR: a public archive for raw electron microscopy image data. Nature Methods. 13 (5), 387-388 (2016).
- . Vue.js Available from: https://vuejs.org (2021)
- . Spring Boot Available from: https://spring.io/projects/spring-boot (2021)
- . Lightweight Directory Access Protocol Available from: https://ldap.com (2021)
- . Vue CLI Available from: https://cli.vuejs.org (2021)
- . Element, A Desktop UI Library Available from: https://element.eleme.io (2021)
- . Sass Available from: https://sass-lang.com/ (2021)
- . Node.js Available from: https://nodejs.org/ (2021)
- . Java Available from: https://www.java.com/ (2021)
- . Hibernate Available from: https://hibernate.org (2021)
- . Swagger UI Available from: https://swagger.io/tools/swagger-ui/ (2021)
- . JUnit Available from: https://junit.org/junit4/ (2020)
- . Apache Maven Project Available from: https://maven.apache.org/ (2020)
- . MySQL Available from: https://www.mysql.com/ (2020)
- . mongoDB Available from: https://www.mongodb.com/ (2020)
- . Apache license, version 2.0 Available from: https://www.apache.org/licenses/license-2.0 (2004)
- . mysql Docker Official Image Available from: https://hub.docker.com/_/mysql (2021)
- . mongo Docker Official Image Available from: https://hub.docker.com/_/mongo (2021)
- . openmicroscopy apacheds Available from: https://hub.docker.com/r/openmicroscopy/apacheds (2021)
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved