Method Article
深層学習に基づくクライオ電子線トモグラムのセグメンテーション
要約
これは、1つのトモグラムの一部をトレーニング入力として使用して、クライオ電子線トモグラムの多クラスセグメンテーションのためのマルチスライスU-Netをトレーニングする方法です。このネットワークを他のトモグラムに推測する方法と、サブトモグラムの平均化やフィラメントトレースなどのさらなる分析のためにセグメンテーションを抽出する方法について説明します。
要約
クライオ電子線トモグラフィー(クライオET)により、研究者は天然の水和状態の細胞を現在可能な最高の解像度でイメージングできます。ただし、この手法にはいくつかの制限があり、生成するデータの分析には時間がかかり、困難になります。1つの断層撮影に数時間から数日かかることがありますが、顕微鏡では1日に50個以上の断層撮影を簡単に生成できます。現在のクライオETのディープラーニングセグメンテーションプログラムは存在しますが、一度に1つの構造をセグメント化することに制限されています。ここでは、マルチスライスU-Net畳み込みニューラルネットワークをトレーニングして適用し、クライオトモグラム内で複数の構造を同時に自動的にセグメント化します。適切な前処理により、これらのネットワークは、トモグラムごとに個々のネットワークをトレーニングすることなく、多くのトモグラムに対して堅牢に推論できます。このワークフローは、ほとんどの場合、セグメンテーション時間を30分未満に短縮することにより、クライオ電子断層撮影の分析速度を劇的に向上させます。さらに、セグメンテーションを使用して、細胞コンテキスト内のフィラメントトレースの精度を向上させ、サブトモグラム平均化のための座標を迅速に抽出することができます。
概要
過去10年間のハードウェアとソフトウェアの開発は、クライオ電子顕微鏡(クライオEM)1,2の「分解能革命」をもたらしました。より優れた、より高速な検出器3、データ収集を自動化するソフトウェア4,5、および位相プレート6などの信号ブーストの進歩により、大量の高解像度クライオEMデータの収集は比較的簡単です。
クライオETは、天然の水和状態で細胞の微細構造に関する前例のない洞察を提供します7、8、9、10。主な制限はサンプルの厚さですが、トモグラフィー11のために厚い細胞および組織サンプルを薄くする集束イオンビーム(FIB)ミリングなどの方法の採用により、クライオETでイメージングできるものの視野は絶えず拡大しています。最新の顕微鏡は1日に50トモグラムをはるかに超えるトモグラムを生成することができ、この速度は迅速なデータ収集スキームの開発により増加すると予測されています12,13。クライオETによって生成された膨大な量のデータを分析することは、このイメージングモダリティのボトルネックのままです。
断層情報を定量的に分析するには、最初に注釈を付ける必要があります。従来、これには専門家による手作業によるセグメンテーションが必要であり、時間がかかります。クライオトモグラムに含まれる分子の複雑さによっては、専用の注意に数時間から数日かかる場合があります。人工ニューラルネットワークは、セグメンテーション作業の大部分をわずかな時間で実行するようにトレーニングできるため、この問題に対する魅力的なソリューションです。畳み込みニューラルネットワーク(CNN)は、コンピュータビジョンタスク14に特に適しており、最近、クライオ電子断層撮影15,16,17の分析に適応されています。
従来のCNNでは、何千もの注釈付きトレーニングサンプルが必要ですが、生物学的画像解析タスクでは不可能なことがよくあります。したがって、U-Netアーキテクチャは、ネットワークを正常にトレーニングするためにデータ拡張に依存し、大規模なトレーニングセットへの依存を最小限に抑えるため、このスペース18 で優れています。たとえば、U-Netアーキテクチャは、1つのトモグラムのほんの数スライス(4つまたは5つのスライス)でトレーニングでき、再トレーニングなしで他のトモグラムに対して堅牢に推論できます。このプロトコルは、Dragonfly 2022.119内で電子クライオトモグラムをセグメント化するためにU-Netニューラルネットワークアーキテクチャをトレーニングするためのステップバイステップガイドを提供します。
Dragonflyは、深層学習モデルによる3次元画像のセグメンテーションや解析に使用される商用開発のソフトウェアで、学術的に自由に利用できます(地理的な制限があります)。高度なグラフィカルインターフェイスを備えているため、専門家でなくても、セマンティックセグメンテーションと画像のノイズ除去の両方でディープラーニングの力を最大限に活用できます。このプロトコルは、人工ニューラルネットワークをトレーニングするためにDragonfly内でクライオ電子線トモグラムを前処理して注釈を付ける方法を示し、大規模なデータセットを迅速にセグメント化するために推測できます。さらに、フィラメントトレースやサブトモグラム平均化のための座標抽出などのさらなる分析のためにセグメント化されたデータを使用する方法について簡単に説明します。
プロトコル
注: Dragonfly 2022.1 には高性能ワークステーションが必要です。システムの推奨事項は、このプロトコルに使用されるワークステーションのハードウェアとともに 、材料表 に含まれています。このプロトコルで使用されるすべての断層撮影は、3.3〜13.2 ang/pixのピクセルサイズから4倍にビニングされます。代表的な結果に使用されたサンプルは、この機関の倫理基準に沿った動物飼育ガイドラインに従う会社( 資料表を参照)から入手しました。このプロトコルで使用されるトモグラムとトレーニング入力として生成されたマルチROIは、 補足ファイル1 (https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct にあります)にバンドルデータセットとして含まれているため、ユーザーは必要に応じて同じデータを追跡できます。Dragonflyは、ユーザーがトレーニング済みのネットワークを共有できるInfinite Toolboxと呼ばれるオープンアクセスデータベースもホストしています。
1. セットアップ
- 既定のワークスペースの変更:
- このプロトコルで使用されているワークスペースを反映するようにワークスペースを変更するには、メインパネルの左側にあるシーンのビュープロパティセクションまで下にスクロールし、[凡例を表示]の選択を解除します。[レイアウト] セクションまで下にスクロールし、[単一シーン] ビューと [4 つの等しいビュー] ビューを選択します。
- デフォルトの単位を更新するには、[ ファイル]、[環境設定。開いたウィンドウで、 デフォルトの単位 をミリメートルから ナノメートルに変更します。
- 便利なデフォルトのキーバインド:
- [Esc]を押すと、2D ビューに十字線が表示され、3D ビューで 3D ボリュームの回転が可能になります。[X]を押すと、2D ビューの十字線が非表示になり、3D ビューで 2D 移動と 3D ボリューム変換が可能になります。
- 十字線にカーソルを合わせると、クリックしてドラッグできる小さな矢印が表示され、他の 2D ビューの表示平面の角度を変更できます。
- Z キーを押して両方のビューでズーム状態に入り、ユーザーが任意の場所をクリックしてドラッグしてズームインおよび ズーム アウトできるようにします。
- 4 ビュー シーンのビューをダブルクリックして、その ビュー のみにフォーカスします。もう一度ダブルクリックすると、4 つのビューすべてに戻ります。
- [ プロパティ ] タブのすべてを ORS オブジェクトとしてエクスポートして、進行状況を定期的に保存し、簡単にインポートできるようにします。リスト内のすべてのオブジェクトを選択し、[ エクスポート] |ORS オブジェクトとして。ファイルに名前を付けて保存します。または、[ ファイル|セッションの保存。ソフトウェアで自動保存機能を使用するには、[ ファイル]|環境設定 |自動保存。
2.画像のインポート
- 画像をインポートするには、[ファイル] |画像ファイルをインポートします。[追加] をクリックし、イメージ ファイルに移動して、[開く] |次へ |終了。
注: ソフトウェアは .rec ファイルを認識しません。すべての断層撮影には.mrc接尾辞が必要です。提供されたデータを使用する場合は、代わりに [ファイル]|オブジェクトをインポートします。 トレーニング.ORSObject ファイル に移動して [開く] をクリックし、[ OK] をクリックします。
3. 前処理(図1.1)
- カスタム強度スケールを作成します(データセット間で画像強度を調整するために使用されます)。ユーティリティに移動 |ディメンション単位マネージャー。 左下の [+ ] をクリックして、新しい 寸法単位を作成します。
- 対象のすべての断層画像に含まれる高輝度(明るい)および低強度(暗い)の特徴を選択します。ユニットに名前と略語を付けます(たとえば、このスケールでは、基準ビーズを0.0標準強度に設定し、背景を100.0に設定します)。カスタム寸法単位を保存します。
注意: カスタム強度スケールは、異なる時間または異なる機器で収集されたにもかかわらず、すべてのデータが同じ強度スケールにあることを保証するために作成およびデータに適用される任意のスケールです。信号が収まる範囲を最もよく表す明るい特徴と暗い特徴を選択してください。データに基準がない場合は、セグメント化される最も暗い特徴量(たとえば、タンパク質の最も暗い領域)を選択するだけです。 - 画像をカスタム強度スケールに調整するには、画面の右側にある [プロパティ] 列でデータセットを右クリックし、[強度スケールの調整] を選択します。画面の左側にある [メイン] タブで、[プローブ] セクションまで下にスクロールします。適切な直径の円形プローブツールを使用して、トモグラムの背景領域のいくつかの場所をクリックし、平均数を[生の強度]列に記録します。基準マーカーに対して繰り返し、[キャリブレーション]をクリックします。必要に応じて、コントラストを調整して、[メイン]タブの[ウィンドウレベリング]セクションにある[領域]ツールを使用して構造を再び表示します。
- 画像フィルタリング:
注意: 画像フィルタリングは、ノイズを低減し、信号をブーストすることができます。このプロトコルは、このデータに最適に機能するため、ソフトウェアに組み込まれている3つのフィルターを使用しますが、使用可能なフィルターは多数あります。目的のデータの画像フィルタリングプロトコルに落ち着いたら、セグメンテーションの前にすべての断層撮影に まったく同じプロトコル を適用する必要があります。- 左側の メイン タブで、[ 画像処理パネル]まで下にスクロールします。[ 詳細設定 ]をクリックして、新しいウィンドウが開くのを待ちます。[ プロパティ ] パネルで、フィルター処理するデータセットを選択し、データセットの左側にある 目の アイコンをクリックして表示します。
- [操作]パネルから、ドロップダウンメニューを使用して、最初の 操作 の [ヒストグラムイコライゼーション ]( [コントラスト ]セクションの下)を選択します。[ 操作の追加] を選択します。 |ガウス分布 ( スムージング セクションの下)。 カーネル ディメンション を 3D に変更します。
- 3 番目の操作を追加します。次に、[ シャープ解除 ]( [シャープ] セクションの下)を選択します。出力はこれのままにしておきます。すべてのスライスに適用し、フィルタリングを実行してから、 画像処理 ウィンドウを閉じてメインインターフェイスに戻ります。
4. トレーニングデータの作成(図1.2)
- トレーニング領域を特定するには、まず [データ プロパティ] パネルの左側にある目のアイコンをクリックして、フィルター処理されていないデータセットを非表示にします。次に、新しくフィルター処理されたデータセットを表示します (データセット-ヒストEq-ガウス-アンシャープという名前が自動的に付けられます)。フィルタリングされたデータセットを使用して、関心のあるすべての特徴を含むトモグラムのサブ領域を特定します。
- 関心領域の周囲にボックスを作成するには、左側の メイン タブで、[ 形状 ]カテゴリまで下にスクロールし、[ ボックスの作成]を選択します。 4 つのビュー パネルで、異なる 2D 平面 を使用してボックスの端をガイド/ドラッグし、すべての寸法で関心領域のみを囲みます。 データ リストで [ボックス ] 領域を選択し、目のシンボルの横にある 灰色の四角形 をクリックして、見やすくするために境界線の色を変更します。
注: 2D U-Net の最小パッチ サイズは 32 x 32 ピクセルです。400 x 400 x 50 ピクセルは、開始するのに妥当なボックス サイズです。 - マルチROIを作成するには、左側で [セグメンテーション ]タブを選択します 。[新規 ] をクリックし 、[マルチ ROI として作成] をオンにします。クラスの数が、対象のフィーチャの数 + バックグラウンド クラスに対応していることを確認します。マルチROI トレーニングデータ に名前を付け、ジオメトリがデータセットに対応していることを確認してから、[ OK]をクリックします。
- トレーニングデータのセグメント化
- ボックス化された領域の境界内になるまでデータをスクロールします。右側のプロパティメニューでマルチROIを選択します。マルチROIの最初の空白のクラス名をダブルクリックして名前を付けます。
- 2Dブラシでペイントします。左側の セグメンテーション タブで、[ 2Dツール ]まで下にスクロールし、 円形のブラシを選択します。次に、ドロップダウンメニューから[ アダプティブガウス ]または [ローカルOTSU ]を選択します。ペイントするには、 左Ctrl キーを押しながらクリックします。消去するには、 左シフトを押し たままクリックします。
注:ブラシには、現在選択されているクラスの色が反映されます。 - マルチROIの各オブジェクトクラスについて、前の手順を繰り返します。ボックス化された領域内のすべての構造が完全にセグメント化されているか、ネットワークによってバックグラウンドと見なされることを確認します。
- すべての構造物にラベルを付けたら、マルチROIのバックグラウンドクラスを右クリックし、[ラベル付けされていないすべてのボクセルをクラスに追加]を選択します。
- マスクという名前の新しい単一クラスのROIを作成します。ジオメトリがフィルター処理されたデータセットに設定されていることを確認し、[適用] をクリックします。右側のプロパティタブで、ボックスを右クリックし、[ROIに追加]を選択します。マスクROIに追加します。
- マスクを使用してトレーニング データをトリミングするには、[プロパティ] タブで、Ctrl キーを押しながら [トレーニング データのマルチ ROI] と [マスク ROI] の両方を選択します。次に、[ブール演算] セクションのデータ プロパティ リストの下にある [交差] をクリックします。新しいデータセットに [トリミングされたトレーニング入力] という名前を付け、ジオメトリがフィルター処理されたデータセットに対応していることを確認してから、[OK] をクリックします。
5.反復トレーニングのためのセグメンテーションウィザードの使用(図1.3)
- トレーニング データをセグメンテーション ウィザードにインポートするには、まず [ プロパティ ] タブでフィルター処理されたデータセットを右クリックし、[ セグメンテーション ウィザード ] オプションを選択します。新しいウィンドウが開いたら、右側の 入力 タブを探します。 マルチROIからフレームをインポート をクリックし、 トリミングされたトレーニング入力を選択します。
- (オプション)視覚的なフィードバックフレームを作成して、トレーニングの進捗状況をリアルタイムで監視します。
- セグメント化されていないフレームをデータから選択し、[ + ] をクリックして新しいフレームとして追加します。フレームの右側にある 混合 ラベルをダブルクリックし、[ 監視] に変更します。
- 新しいニューラル ネットワーク モデルを生成するには、[モデル] タブの右側にある [+] ボタンをクリックして新しいモデルを生成します。リストから [U-Net] を選択し、[入力ディメンション] で [2.5D スライス] と [5 スライス] を選択し、[生成] をクリックします。
- ネットワークをトレーニングするには、SegWiz ウィンドウの右下にある [トレーニング] をクリックします。
注:トレーニングは、進行状況を失うことなく早期に停止できます。 - トレーニング済みネットワークを使用して新しいフレームをセグメント化するには、U-Net トレーニングが完了したら、新しいフレームを作成し、[ 予測(Predict )] (右下) をクリックします。次に、予測フレームの右上にある上矢印をクリックして、セグメンテーションを実際のフレームに転送します。
- 予測を修正するには、 Ctrl キーを押しながら 2 つのクラスをクリックして 、一方のセグメント化されたピクセルを他方に変更します。両方のクラスを選択し、 ブラシ でペイントして、いずれかのクラスに属するピクセルのみをペイントします。少なくとも 5 つの新しいフレームのセグメンテーションを修正します。
注:両方のクラスが選択されているときにブラシツールでペイントすると、通常のようにShiftキーを押しながらクリックで消去する代わりに、最初のクラスのピクセルが2番目のクラスのピクセルに変換されます。Ctrl キーを押しながらクリックすると、その逆が実行されます。 - 反復トレーニングの場合は、[ トレーニング(Training )] ボタンをもう一度クリックし、ネットワークがさらに 30 から 40 エポックまでトレーニングできるようにし、その時点でトレーニングを停止し、ステップ 4.5 と 4.6 を繰り返してトレーニングの別のラウンドを行います。
注: このようにして、1 つのデータセットを使用してモデルを反復的にトレーニングおよび改善できます。 - ネットワークを公開するには、パフォーマンスに満足したら、セグメンテーション ウィザードを終了します。公開(保存)するモデルを自動的に尋ねるダイアログボックスで、成功したネットワークを選択して名前を付け、公開して、セグメンテーションウィザードの外部でネットワークを使用できるようにします。
6.ネットワークを適用します(図1.4)
- 最初にトレーニング トモグラムに適用するには、[ プロパティ ] パネルでフィルター処理されたデータセットを選択します。左側の [セグメント化 ] パネルで、[ AI を使用したセグメント] セクションまで下にスクロールします。正しいデータセットが選択されていることを確認し、ドロップダウンメニューで公開したばかりのモデルを選択して、[ セグメント |すべてのスライス。または、[ プレビュー ] を選択して、セグメンテーションの 1 スライス プレビューを表示します。
- 推論データセットに適用するには、新しいトモグラムをインポートします。ステップ3(図1.1)に従って前処理します。 [セグメンテーション ] パネルで、[ AI を使用したセグメント ] セクションに移動します。新しくフィルタリングされたトモグラムが選択されたデータセットであることを確認し、以前にトレーニングしたモデルを選択して、[ Segment] |すべてのスライス。
7.セグメンテーションの操作とクリーンアップ
- 最初に、ノイズと目的のフィーチャをセグメント化したクラスの 1 つを選択して、ノイズをすばやくクリーンアップします。 右クリック |プロセス島 |ボクセル数で削除 |ボクセル サイズを選択します。小さく(~200)始めて、徐々にカウントを増やしてほとんどのノイズを取り除きます。
- セグメンテーション補正を行うには、Ctrl キーを押しながら 2 つのクラスをクリックして、それらのクラスに属するピクセルのみをペイントします。セグメンテーションツールでCtrlキーを押しながらクリック+ドラッグすると、2番目のクラスのピクセルが最初のクラスに変更され、Shiftキーを押しながら+ドラッグしてその逆になります。これを続けて、誤ってラベル付けされたピクセルをすばやく修正します。
- 接続されたコンポーネントを分離します。
- クラスを選択してください。マルチROIでクラスを右クリック |[接続されたコンポーネントを分離 ]: 同じクラスの別のコンポーネントに接続されていないコンポーネントごとに新しいクラスを作成します。 Multi-ROIの下にあるボタンを使用すると、クラスを簡単にマージできます。
- ROIをバイナリ/TIFFとしてエクスポートします。
- マルチROIでクラスを選択し、右クリックしてROIとしてクラスを抽出します。上のプロパティパネルで、新しいROIを選択し、右クリック|エクスポート |バイナリとしてのROI(すべての画像を1つのファイルにエクスポートするオプションが選択されていることを確認します)。
注:ユーザーは、IMODプログラムtif2mrc20を使用して、tiffからmrc形式に簡単に変換できます。これは、フィラメントトレースに役立ちます。
- マルチROIでクラスを選択し、右クリックしてROIとしてクラスを抽出します。上のプロパティパネルで、新しいROIを選択し、右クリック|エクスポート |バイナリとしてのROI(すべての画像を1つのファイルにエクスポートするオプションが選択されていることを確認します)。
8. ROIからのサブトモグラム平均化のための座標の生成
- クラスを抽出します。
- 平均化に使用するクラスを右クリック |クラスをROIとして抽出します。クラスROIを右クリック |接続されたコンポーネント |新しいマルチROI(26が接続されています)。
- 座標を生成します。
- 新しいマルチROI |スカラージェネレータ。データセットを使用した基本計測値の展開 | [重み付き重心 X、Y、Z ] をオンにします。データセットを選択して計算します。右クリック マルチROI |スカラー値をエクスポートします。[すべてのスカラー スロットを選択]、[OK] の順にオンにして、マルチ ROI の各クラスの重心ワールド座標を CSV ファイルとして生成します。
注: パーティクル同士が接近していて、セグメンテーションが接触している場合は、分水界変換を実行してコンポーネントをマルチ ROI に分離する必要があります。
- 新しいマルチROI |スカラージェネレータ。データセットを使用した基本計測値の展開 | [重み付き重心 X、Y、Z ] をオンにします。データセットを選択して計算します。右クリック マルチROI |スカラー値をエクスポートします。[すべてのスカラー スロットを選択]、[OK] の順にオンにして、マルチ ROI の各クラスの重心ワールド座標を CSV ファイルとして生成します。
9. 分水界変換
- マルチROIでクラスを右クリックしてクラスを抽出し、平均化に使用する |クラスをROIとして抽出します。この ROI 流域マスクという名前を付けます。
- (オプション) 穴を閉じます。
- セグメント化されたパーティクルに穴や開口部がある場合は、集水域のためにこれらを閉じます。[データ プロパティ] で ROI をクリックします。[セグメンテーション]タブ(左側)で、[形態学的操作]に移動し、必要な拡張、侵食、および閉じるの組み合わせを使用して、穴のないソリッドセグメンテーションを実現します。
- ROIをクリックしてROIを反転する |選択したオブジェクトをコピー します( [データ プロパティ] の下)。コピーした ROIを選択し、左側の [セグメンテーション ]タブで[ 反転]をクリックします。
- 逆 ROI を右クリックして距離マップを作成する |マッピングの作成 |距離マップ。後で使用するために、距離マップのコピーを作成して反転します(右クリック|修正と変換 |値の反転 |適用する)。この反転マップに 「ランドスケープ」という名前を付けます。
- シード ポイントを作成します。
- ROIを非表示にして、 距離マップを表示します。 [セグメンテーション ] タブで、[範囲の定義] をクリックし、各ポイントの中心にある数ピクセルのみが強調表示され、別のポイントに接続されなくなるまで 範囲 を縮小します。[ 範囲 ] セクションの下部にある [ 新規に追加] をクリックします。この新しい ROI シードポイントという名前を付けます。
- 流域変換を実行します。
- シードポイントROIを右クリック |接続されたコンポーネント |新しいマルチROI(26が接続されています)。新しく生成されたマルチROI |分水嶺変換。[横] という名前の距離マップを選択し、[OK] をクリックします。[Watershed Mask]という名前の ROI を選択し、[OK]をクリックして、各シード ポイントから分水界変換を計算し、個々のパーティクルをマルチ ROI の個別のクラスに分割します。手順 8.2 のように座標を生成します。
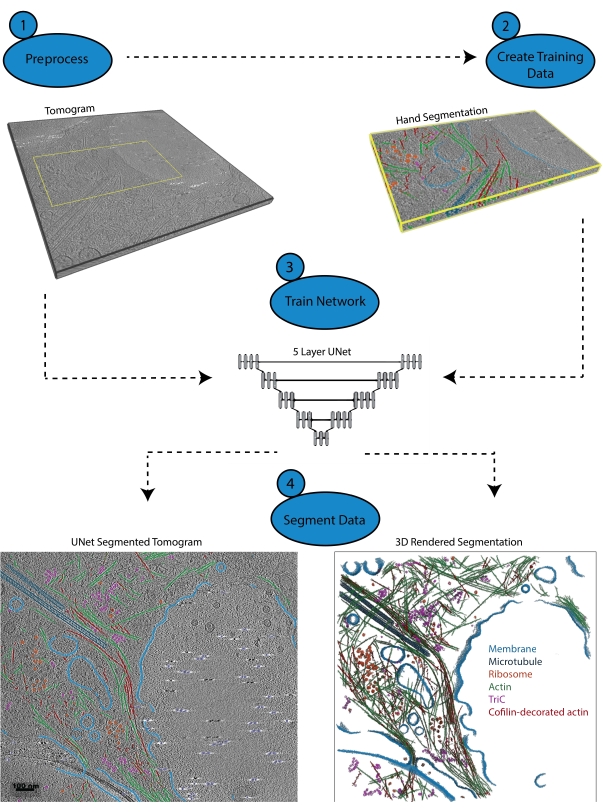
図1:ワークフロー。 1) 強度スケールをキャリブレーションし、データセットをフィルタリングすることにより、トレーニング断層撮影を前処理します。 2) トモグラムのごく一部を、ユーザーが識別したいすべての適切なラベルで手作業でセグメント化することにより、トレーニングデータを作成します。 3) フィルタリングされたトモグラムを入力として使用し、ハンドセグメンテーションをトレーニング出力として使用して、セグメンテーションウィザードで5層のマルチスライスU-Netをトレーニングします。 4) 訓練されたネットワークをフルトモグラムに適用して注釈を付け、セグメント化された各クラスから3Dレンダリングを生成できます。 この図の拡大版を表示するには、ここをクリックしてください。
結果
プロトコルに従って、5スライスのU-Netを1つの断層撮影(図2A)でトレーニングし、メンブレン、微小管、アクチン、フィデューシャルマーカー、バックグラウンドの5つのクラスを特定しました。ネットワークを合計3回繰り返しトレーニングした後、トモグラムに適用して完全にセグメント化して注釈を付けました(図2B、C)。最小限のクリーンアップは、手順 7.1 と 7.2 を使用して実行されました。関心のある次の3つの断層撮影(図2D、G、J)を前処理のためにソフトウェアにロードしました。画像をインポートする前に、トモグラムの1つ(図2J)は、わずかに異なる倍率で別の顕微鏡で収集されたため、ピクセルサイズを17.22 Å/pxから13.3 Å/pxに調整する必要がありました。IMODプログラム squeezevol は、次のコマンドでサイズ変更に使用されました。
'squeezevol -f 0.772 inputfile.mrc outputfile.mrc'
このコマンドの -f は、ピクセル サイズを変更する係数 (この場合は 13.3/17.22) を指します。インポート後、3 つの推論ターゲットすべてがステップ 3.2 および 3.3 に従って前処理され、5 スライスの U-Net が適用されました。最小限のクリーンアップが再度実行されました。最終的なセグメンテーションを 図 2 に示します。
各トモグラムからの微小管セグメンテーションは、バイナリ(ステップ7.4)TIFファイルとしてエクスポートされ、MRC(IMOD tif2mrc プログラム)に変換され、シリンダー相関とフィラメントトレースに使用されました。フィラメントのバイナリセグメンテーションは、断層撮影でのトレースよりもはるかに堅牢なフィラメントトレースをもたらします。フィラメントトレースの座標マップ(図3)は、微小管の向きを決定するための単一フィラメントに沿った最近傍測定(フィラメントパッキング)やヘリカルサブトモグラム平均化などのさらなる分析に使用されます。
失敗したネットワークやトレーニングが不十分なネットワークは簡単に判断できます。障害が発生したネットワークは構造をまったくセグメント化できませんが、トレーニングが不十分なネットワークは通常、一部の構造を正しくセグメント化し、誤検知と偽陰性の数が多数あります。これらのネットワークを修正し、繰り返しトレーニングしてパフォーマンスを向上させることができます。セグメンテーションウィザードは、トレーニング後にモデルの サイコロ類似度係数 (SegWizでは スコア と呼ばれる)を自動的に計算します。この統計は、トレーニングデータとU-Netセグメンテーションの類似性の推定値を提供します。Dragonfly 2022.1には、インターフェイスの上部にある [人工知能 ]タブからアクセスできるモデルのパフォーマンスを評価するための組み込みツールもあります(使用方法についてはドキュメントを参照してください)。
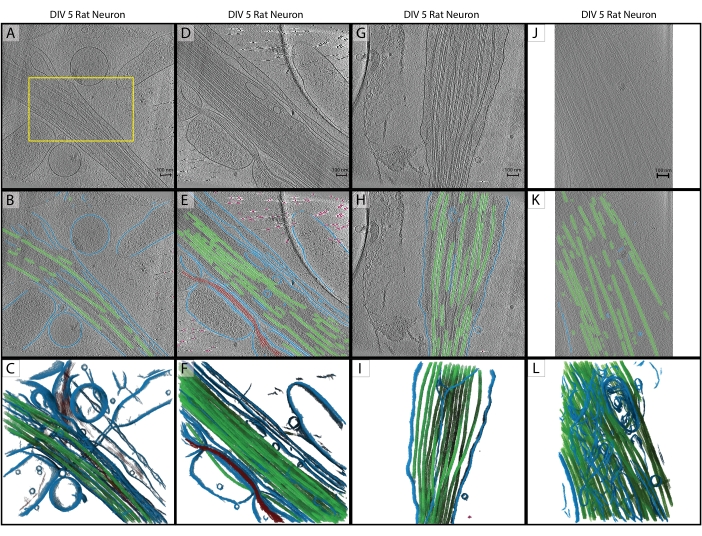
図 2: 推論。 (A-C)2019年にタイタンクリオスで収集されたDIV 5海馬ラットニューロンの元のトレーニング断層撮影。これは、IMODのCTF補正を使用したバックプロジェクション再構成です。(A)黄色のボックスは、トレーニング入力のためにハンドセグメンテーションが実行された領域を表します。(B)トレーニング完了後のU-Netからの2Dセグメンテーション。(C)膜(青)、微小管(緑)、アクチン(赤)を示すセグメント領域の3Dレンダリング。(D-F)トレーニング断層撮影と同じセッションからのDIV 5海馬ラットニューロン。(E)追加のトレーニングや迅速なクリーンアップなしでのU-Netからの2Dセグメンテーション。膜(青)、微小管(緑)、アクチン(赤)、基準(ピンク)。(F)セグメント化された領域の3Dレンダリング。(G-I)2019年のセッションからのDIV 5海馬ラットニューロン。(H)クイッククリーンアップによるU-Netからの2Dセグメンテーションと(I)3Dレンダリング。(J-L)DIV 5海馬ラットニューロン、2021年に異なる倍率で異なるタイタンクリオスで収集。IMODプログラムsqueezevolでピクセルサイズをトレーニング断層撮影に合わせて変更しました。(K)迅速なクリーンアップによるU-Netからの2Dセグメンテーション、適切な前処理によるデータセット間の堅牢な推論の実証、および(L)セグメンテーションの3Dレンダリング。スケールバー= 100 nm。略語:DIV =インビトロでの日数;CTF = コントラスト伝達関数。この図の拡大版を表示するには、ここをクリックしてください。
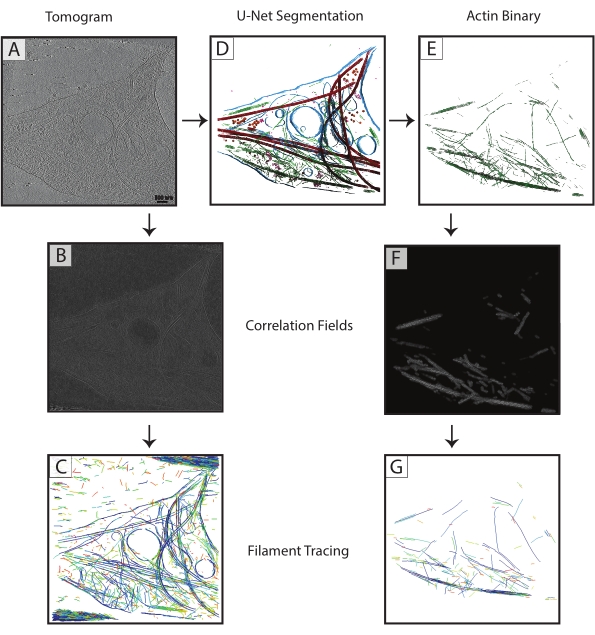
図3:フィラメントトレースの改善 。 (A)タイタンクリオスで収集されたDIV 4ラット海馬ニューロンの断層撮影。(B)アクチンフィラメント上の円柱相関から生成された相関マップ。(C)相関マップ内のアクチンフィラメントの強度を使用してパラメータを定義するアクチンのフィラメントトレース。トレースは、アクチンだけを追跡しようとしている間、膜と微小管、およびノイズをキャプチャします。(D)断層撮影のU-Netセグメンテーション。膜は青色、微小管は赤色、リボソームは青色、triCは紫色、アクチンは緑色で強調表示されています。(E)フィラメントトレース用のバイナリマスクとして抽出されたアクチンセグメンテーション。(F)(B)からの同一パラメータとの円柱相関から生成された相関マップ。(G)断層撮影からのアクチンフィラメントのみのフィラメントトレースを大幅に改善しました。略語:DIV = インビトロでの日数。 この図の拡大版を表示するには、ここをクリックしてください。
補足ファイル1:このプロトコルで使用されるトモグラムと、トレーニング入力として生成されたマルチROIは、バンドルされたデータセット(Training.ORSObject)として含まれています。 https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct を参照してください。
ディスカッション
このプロトコルでは、Dragonfly 2022.1 ソフトウェアを使用して 1 つのトモグラムから多クラス U-Net をトレーニングする手順と、同じデータセットからのものである必要のない他のトモグラムに対してそのネットワークを推測する方法について説明します。トレーニングは比較的速く(トレーニングされているネットワークと使用されているハードウェアによって、エポックあたり3〜5分、または数時間遅くなる可能性があります)、学習を改善するためにネットワークを再トレーニングすることは直感的です。前処理ステップがすべての断層像に対して実行される限り、推論は通常堅牢です。
一貫性のある前処理は、ディープラーニング推論の最も重要なステップです。ソフトウェアには多くのイメージングフィルターがあり、ユーザーは実験して、特定のデータセットに最適なフィルターを判断できます。トレーニング断層撮影で使用されるフィルタリングは、推論断層撮影にも同じ方法で適用する必要があることに注意してください。また、ネットワークに正確で十分なトレーニング情報を提供するように注意する必要があります。トレーニングスライス内でセグメント化されたすべての特徴を可能な限り慎重かつ正確にセグメント化することが重要です。
画像のセグメンテーションは、洗練された商用グレードのユーザーインターフェイスによって促進されます。ハンドセグメンテーションに必要なすべてのツールを提供し、トレーニングと再トレーニングの前に、あるクラスから別のクラスにボクセルを簡単に再割り当てできます。ユーザーは、トモグラムのコンテキスト全体内でボクセルを手動でセグメント化することができ、複数のビューとボリュームを自由に回転させる機能が与えられます。さらに、このソフトウェアは、複数のシングルクラスネットワークでセグメント化するよりも優れた16 を実行し、高速であるマルチクラスネットワークを使用する機能を提供します。
もちろん、ニューラルネットワークの機能には制限があります。クライオETデータは、本質的に非常にノイズが多く、角度サンプリングが制限されているため、同一のオブジェクト21に向き固有の歪みが生じます。トレーニングは、構造を正確に手作業でセグメント化するために専門家に依存しており、成功したネットワークは、与えられたトレーニングデータと同じくらい良い(または悪い)だけです。信号をブーストするための画像フィルタリングはトレーナーに役立ちますが、特定の構造のすべてのピクセルを正確に識別することが困難な場合がまだたくさんあります。したがって、トレーニングセグメンテーションを作成する際には、ネットワークがトレーニング中に学習できる最良の情報を持つように細心の注意を払うことが重要です。
このワークフローは、各ユーザーの好みに合わせて簡単に変更できます。すべての断層撮影をまったく同じ方法で前処理することが不可欠ですが、プロトコルで使用される正確なフィルタを使用する必要はありません。ソフトウェアには多数の画像フィルタリングオプションがあり、多くの断層撮影にまたがる大規模なセグメンテーションプロジェクトに着手する前に、ユーザーの特定のデータに合わせてこれらを最適化することをお勧めします。また、このラボのデータにはマルチスライス U-Net が最適に機能することがわかっていますが、別のユーザーは別のアーキテクチャ (3D U-Net や Sensor 3D など) の方が適していると感じるかもしれません。セグメンテーションウィザードは、同じトレーニングデータを使用して複数のネットワークのパフォーマンスを比較するための便利なインターフェイスを提供します。
ここで紹介するようなツールは、完全な断層撮影の手のセグメンテーションを過去のタスクにします。十分に訓練されたニューラルネットワークが堅牢に推測できるため、顕微鏡が収集できる限り迅速に断層撮影データを再構築、処理、および完全にセグメント化するワークフローを作成することは完全に可能です。
開示事項
このプロトコルのオープンアクセスライセンスは、Object Research Systemsによって支払われました。
謝辞
この研究は、ペンシルベニア州立医科大学と生化学分子生物学部、およびタバコ決済基金(TSF)の助成金4100079742-EXTの支援を受けました。このプロジェクトで使用されたCryoEMおよびCryoETコア(RRID:SCR_021178)のサービスと機器は、ペンシルベニア州立大学医学部から研究および大学院生の副学部長のオフィス および ペンシルベニア州保健省を通じて、タバコ決済基金(CURE)を使用して資金提供されました。内容は著者の責任であり、必ずしも大学または医学部の公式見解を表すものではありません。ペンシルベニア州保健局は、分析、解釈、または結論に対する責任を明確に否認します。
資料
| Name | Company | Catalog Number | Comments |
| Dragonfly 2022.1 | Object Research Systems | https://www.theobjects.com/dragonfly/index.html | |
| E18 Rat Dissociated Hippocampus | Transnetyx Tissue | KTSDEDHP | https://tissue.transnetyx.com/faqs |
| IMOD | University of Colorado | https://bio3d.colorado.edu/imod/ | |
| Intel® Xeon® Gold 6124 CPU 3.2GHz | Intel | https://www.intel.com/content/www/us/en/products/sku/120493/intel-xeon-gold-6134-processor-24-75m-cache-3-20-ghz/specifications.html | |
| NVIDIA Quadro P4000 | NVIDIA | https://www.nvidia.com/content/dam/en-zz/Solutions/design-visualization/productspage/quadro/quadro-desktop/quadro-pascal-p4000-data-sheet-a4-nvidia-704358-r2-web.pdf | |
| Windows 10 Enterprise 2016 | Microsoft | https://www.microsoft.com/en-us/evalcenter/evaluate-windows-10-enterprise | |
| Workstation Minimum Requirements | https://theobjects.com/dragonfly/system-requirements.html |
参考文献
- Bai, X. -. C., Mcmullan, G., Scheres, S. H. W. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- de Oliveira, T. M., van Beek, L., Shilliday, F., Debreczeni, J., Phillips, C. Cryo-EM: The resolution revolution and drug discovery. SLAS Discovery. 26 (1), 17-31 (2021).
- Danev, R., Yanagisawa, H., Kikkawa, M. Cryo-EM performance testing of hardware and data acquisition strategies. Microscopy. 70 (6), 487-497 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- . Tomography 5 and Tomo Live Software User-friendly batch acquisition for and on-the-fly reconstruction for cryo-electron tomography Datasheet Available from: https://assets.thermofisher.com/TFS-Assets/MSD/Datasheets/tomography-5-software-ds0362.pdf (2022)
- Danev, R., Baumeister, W. Expanding the boundaries of cryo-EM with phase plates. Current Opinion in Structural Biology. 46, 87-94 (2017).
- Hylton, R. K., Swulius, M. T. Challenges and triumphs in cryo-electron tomography. iScience. 24 (9), (2021).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Oikonomou, C. M., Jensen, G. J. Cellular electron cryotomography: Toward structural biology in situ. Annual Review of Biochemistry. 86, 873-896 (2017).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-the cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Lam, V., Villa, E. Practical approaches for Cryo-FIB milling and applications for cellular cryo-electron tomography. Methods in Molecular Biology. 2215, 49-82 (2021).
- Chreifi, G., Chen, S., Metskas, L. A., Kaplan, M., Jensen, G. J. Rapid tilt-series acquisition for electron cryotomography. Journal of Structural Biology. 205 (2), 163-169 (2019).
- Eisenstein, F., Danev, R., Pilhofer, M. Improved applicability and robustness of fast cryo-electron tomography data acquisition. Journal of Structural Biology. 208 (2), 107-114 (2019).
- Esteva, A., et al. Deep learning-enabled medical computer vision. npj Digital Medicine. 4 (1), (2021).
- Liu, Y. -. T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nature Methods. 14 (10), 983-985 (2017).
- Ronneberger, O., Fischer, P., Brox, T. U-net: Convolutional networks for biomedical image segmentation. Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 9351, 234-241 (2015).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iancu, C. V., et al. A "flip-flop" rotation stage for routine dual-axis electron cryotomography. Journal of Structural Biology. 151 (3), 288-297 (2005).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved