Method Article
Segmentação Baseada em Deep Learning de Tomogramas de Crio-Elétrons
Neste Artigo
Resumo
Este é um método para treinar um U-Net multi-fatia para segmentação multiclasse de tomogramas crio-elétrons usando uma porção de um tomograma como entrada de treinamento. Descrevemos como inferir essa rede para outros tomogramas e como extrair segmentações para análises posteriores, como a média do subtomograma e o rastreamento de filamentos.
Resumo
A tomografia crio-eletrônica (crio-ET) permite que os pesquisadores visualizem células em seu estado nativo e hidratado na mais alta resolução atualmente possível. A técnica tem várias limitações, no entanto, que tornam a análise dos dados que gera demorada e difícil. A segmentação manual de um único tomograma pode levar de horas a dias, mas um microscópio pode facilmente gerar 50 ou mais tomogramas por dia. Os atuais programas de segmentação de aprendizado profundo para crio-ET existem, mas estão limitados a segmentar uma estrutura de cada vez. Aqui, as redes neurais convolucionais U-Net multi-fatia são treinadas e aplicadas para segmentar automaticamente várias estruturas simultaneamente dentro de crio-tomogramas. Com o pré-processamento adequado, essas redes podem ser inferidas de forma robusta para muitos tomogramas sem a necessidade de treinar redes individuais para cada tomograma. Esse fluxo de trabalho melhora drasticamente a velocidade com que os tomogramas crio-elétrons podem ser analisados, reduzindo o tempo de segmentação para menos de 30 minutos na maioria dos casos. Além disso, as segmentações podem ser usadas para melhorar a precisão do rastreamento de filamentos dentro de um contexto celular e para extrair rapidamente coordenadas para a média do subtomograma.
Introdução
Os desenvolvimentos de hardware e software na última década resultaram em uma "revolução de resolução" para a microscopia crio-eletrônica (crio-EM)1,2. Comdetectores 3 melhores e mais rápidos, software para automatizar a coletade dados 4,5 e avanços de aumento de sinal, como placas de fase6, a coleta de grandes quantidades de dados crio-EM de alta resolução é relativamente simples.
O Cryo-ET fornece uma visão sem precedentes da ultraestrutura celular em um estado nativo e hidratado 7,8,9,10. A principal limitação é a espessura da amostra, mas com a adoção de métodos como a fresagem por feixe de íons focalizados (FIB), onde amostras celulares e teciduais espessas são diluídas para tomografia11, o horizonte para o que pode ser fotografado com crio-ET está em constante expansão. Os microscópios mais novos são capazes de produzir bem mais de 50 tomogramas por dia, e essa taxa só deve aumentar devido ao desenvolvimento de esquemas rápidos de coleta de dados12,13. Analisar as vastas quantidades de dados produzidos pela crio-ET continua a ser um gargalo para esta modalidade de imagem.
A análise quantitativa da informação tomográfica requer que ela seja anotada primeiro. Tradicionalmente, isso requer segmentação manual por um especialista, o que é demorado; dependendo da complexidade molecular contida no crio-tomograma, pode levar horas a dias de atenção dedicada. As redes neurais artificiais são uma solução atraente para esse problema, uma vez que podem ser treinadas para fazer a maior parte do trabalho de segmentação em uma fração do tempo. As redes neurais convolucionais (CNNs) são especialmente adequadas para tarefas de visão computacional14 e foram recentemente adaptadas para a análise de tomogramas crio-eletrônicos15,16,17.
As CNNs tradicionais exigem muitos milhares de amostras de treinamento anotadas, o que muitas vezes não é possível para tarefas de análise de imagens biológicas. Assim, a arquitetura U-Net tem se destacado nesse espaço18 porque depende do aumento de dados para treinar com sucesso a rede, minimizando a dependência de grandes conjuntos de treinamento. Por exemplo, uma arquitetura U-Net pode ser treinada com apenas algumas fatias de um único tomograma (quatro ou cinco fatias) e inferida robustamente para outros tomogramas sem reciclagem. Este protocolo fornece um guia passo a passo para treinar arquiteturas de redes neurais U-Net para segmentar crio-tomogramas de elétrons dentro do Dragonfly 2022.119.
O Dragonfly é um software desenvolvido comercialmente usado para segmentação e análise de imagens 3D por modelos de aprendizado profundo, e está disponível gratuitamente para uso acadêmico (algumas restrições geográficas se aplicam). Ele tem uma interface gráfica avançada que permite que um não-especialista aproveite ao máximo os poderes do aprendizado profundo tanto para segmentação semântica quanto para denoising. Este protocolo demonstra como pré-processar e anotar tomogramas crio-elétrons dentro do Dragonfly para treinar redes neurais artificiais, que podem então ser inferidas para segmentar rapidamente grandes conjuntos de dados. Ele ainda discute e demonstra brevemente como usar dados segmentados para análises posteriores, como rastreamento de filamentos e extração de coordenadas para a média de subtomogramas.
Protocolo
NOTA: O Dragonfly 2022.1 requer uma estação de trabalho de alto desempenho. As recomendações do sistema estão incluídas na Tabela de Materiais , juntamente com o hardware da estação de trabalho usada para este protocolo. Todos os tomogramas usados neste protocolo são agrupados 4x de um tamanho de pixel de 3,3 a 13,2 ang/pix. As amostras utilizadas nos resultados representativos foram obtidas de uma empresa (ver Tabela de Materiais) que segue diretrizes de cuidados com animais que se alinham aos padrões éticos desta instituição. O tomograma usado neste protocolo e o multi-ROI que foi gerado como entrada de treinamento foram incluídos como um conjunto de dados empacotado no Arquivo Suplementar 1 (que pode ser encontrado em https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct) para que o usuário possa acompanhar os mesmos dados, caso deseje. O Dragonfly também hospeda um banco de dados de acesso aberto chamado Infinite Toolbox, onde os usuários podem compartilhar redes treinadas.
1. Configuração
- Alterando o espaço de trabalho padrão:
- Para alterar a área de trabalho para espelhar a usada neste protocolo, no lado esquerdo do painel Principal, role para baixo até a seção Propriedades de exibição da cena e desmarque Mostrar legendas. Role para baixo até a seção Layout e selecione a exibição Cena única e Quatro Modos de Exibição Iguais .
- Para atualizar a unidade padrão, vá para Arquivo | Preferências. Na janela que se abre, altere a unidade Padrão de milímetros para nanômetros.
- Associações de teclas padrão úteis:
- Pressione Esc para exibir a mira nas visualizações 2D e permitir a rotação do volume 3D na visualização 3D. Pressione X para ocultar a mira nas visualizações 2D e permitir a tradução 2D e a tradução de volume 3D na visualização 3D.
- Passe o mouse sobre a mira para ver pequenas setas que podem ser clicadas e arrastadas para alterar o ângulo do plano de visualização nas outras visualizações 2D.
- Pressione Z para entrar no estado de zoom em ambas as exibições, permitindo que os usuários cliquem e arrastem para qualquer lugar para aumentar e diminuir o zoom.
- Clique duas vezes em uma exibição na cena Quatro Visualizações para focar apenas essa exibição; clique duas vezes novamente para retornar a todos os quatro modos de exibição.
- Salve periodicamente o progresso exportando tudo na guia Propriedades como um objeto ORS para facilitar a importação. Selecione todos os objetos na lista e clique com o botão direito do mouse em Exportar | Como objeto ORS. Nomeie o arquivo e salve. Como alternativa, vá para Arquivo | Salvar sessão. Para usar o recurso de salvamento automático no software, habilite-o por meio de Arquivo | Preferências | Salvamento automático.
2. Importação de imagens
- Para importação de imagens, vá para Arquivo | Importe arquivos de imagem. Clique em Adicionar, navegue até o arquivo de imagem e clique em Abrir | Seguinte | Acabamento.
Observação : O software não reconhece arquivos .rec. Todos os tomogramas devem ter o sufixo .mrc. Se estiver usando os dados fornecidos, vá para Arquivo | Importar objeto(s). Navegue até o arquivo Training.ORSObject e clique em Abrir e, em seguida, clique em OK.
3. Pré-processamento (Figura 1.1)
- Crie uma escala de intensidade personalizada (usada para calibrar intensidades de imagem entre conjuntos de dados). Ir para Utilitários | Gerente de Unidade de Dimensão. No canto inferior esquerdo, clique em + para criar uma nova Unidade de Dimensão.
- Escolha um recurso de alta intensidade (brilhante) e baixa intensidade (escuro) que esteja em todos os tomogramas de interesse. Dê à unidade um nome e uma abreviatura (por exemplo, para esta escala, defina as contas fiduciais como 0,0 Intensidade padrão e o fundo como 100,0). Salve a unidade de dimensão personalizada.
NOTA: Uma escala de intensidade personalizada é uma escala arbitrária que é criada e aplicada aos dados para garantir que todos os dados estejam na mesma escala de intensidade, apesar de serem coletados em momentos diferentes ou em equipamentos diferentes. Escolha recursos claros e escuros que melhor representem o alcance em que o sinal se enquadra. Se não houver fiduciais nos dados, basta escolher a característica mais escura que será segmentada (a região mais escura da proteína, por exemplo). - Para calibrar imagens para a escala de intensidade personalizada, clique com o botão direito do mouse no conjunto de dados na coluna Propriedades do lado direito da tela e selecione Calibrar escala de intensidade. Na guia Principal do lado esquerdo da tela, role para baixo até a seção Sonda . Usando a ferramenta de sonda circular com um diâmetro apropriado, clique em alguns lugares na região de fundo do tomograma e registre o número médio na coluna Intensidade bruta ; repita para marcadores fiduciais e clique em Calibrar. Se necessário, ajuste o contraste para tornar as estruturas visíveis novamente com a ferramenta Área na seção Nivelamento de janela da guia Principal.
- Filtragem de imagem:
NOTA: A filtragem de imagem pode reduzir o ruído e aumentar o sinal. Este protocolo usa três filtros que são incorporados no software, pois funcionam melhor para esses dados, mas existem muitos filtros disponíveis. Uma vez estabelecido um protocolo de filtragem de imagem para os dados de interesse, será necessário aplicar exatamente o mesmo protocolo a todos os tomogramas antes da segmentação.- Na guia principal do lado esquerdo, role para baixo até o Painel de Processamento de Imagens. Clique em Avançado e aguarde a abertura de uma nova janela. No painel Propriedades , selecione o conjunto de dados a ser filtrado e torne-o visível clicando no ícone de olho à esquerda do conjunto de dados.
- No painel Operações , use o menu suspenso para selecionar Equalização de histograma (na seção Contraste ) para a primeira operação. Selecione Adicionar operação | Gaussiano (na seção Suavização ). Altere a dimensão do kernel para 3D.
- Adicionar uma terceira operação; em seguida, selecione Unsharp (na seção Sharpensing ). Deixe a saída para este. Aplique a todas as fatias e deixe a filtragem ser executada e, em seguida, feche a janela Processamento de imagem para retornar à interface principal.
4. Criar dados de treinamento (Figura 1.2)
- Identifique a área de treinamento ocultando primeiro o conjunto de dados não filtrado clicando no ícone de olho à esquerda dele no painel Propriedades de dados . Em seguida, mostre o conjunto de dados recém-filtrado (que será automaticamente chamado DataSet-HistEq-Gauss-Unsharp). Usando o conjunto de dados filtrado, identifique uma sub-região do tomograma que contenha todos os recursos de interesse.
- Para criar uma caixa ao redor da região de interesse, no lado esquerdo, na guia principal , role para baixo até a categoria Formas e selecione Criar uma Caixa. Enquanto estiver no painel Quatro Visualizações , use os diferentes planos 2D para ajudar a guiar/arrastar as bordas da caixa para incluir apenas a região de interesse em todas as dimensões. Na lista de dados, selecione a região Caixa e altere a cor da borda para facilitar a visualização clicando no quadrado cinza ao lado do símbolo do olho.
NOTA: O menor tamanho de patch para um U-Net 2D é de 32 x 32 pixels; 400 x 400 x 50 pixels é um tamanho de caixa razoável para começar. - Para criar um ROI múltiplo, no lado esquerdo, selecione a guia Segmentação | Novo e marque Criar como Multi-ROI. Certifique-se de que o número de classes corresponde ao número de recursos de interesse + uma classe em segundo plano. Nomeie os Dados de Treinamento de vários ROIs e verifique se a geometria corresponde ao conjunto de dados antes de clicar em OK.
- Segmentando os dados de treinamento
- Role pelos dados até dentro dos limites da região em caixa. Selecione o Multi-ROI no menu de propriedades à direita. Clique duas vezes no primeiro nome de classe em branco no multi-ROI para nomeá-lo.
- Pinte com o pincel 2D. Na guia de segmentação à esquerda, role para baixo até Ferramentas 2D e selecione um pincel circular. Em seguida, selecione Gaussian adaptável ou OTSU local no menu suspenso. Para pintar, mantenha pressionada a tecla ctrl esquerdo e clique. Para apagar, mantenha pressionada a tecla Shift esquerda e clique.
Observação : O pincel refletirá a cor da classe selecionada no momento. - Repita a etapa anterior para cada classe de objeto no multi-ROI. Certifique-se de que todas as estruturas dentro da região in a box estejam totalmente segmentadas ou serão consideradas em segundo plano pela rede.
- Quando todas as estruturas tiverem sido rotuladas, clique com o botão direito do mouse na classe Plano de fundo no Multi-ROI e selecione Adicionar todos os Voxels não rotulados à classe.
- Crie um novo ROI de classe única chamado Mask. Verifique se a geometria está definida como o conjunto de dados filtrado e clique em aplicar. Na guia de propriedades à direita, clique com o botão direito do mouse na caixa e selecione Adicionar ao ROI. Adicione-o ao ROI da máscara.
- Para cortar os dados de treinamento usando a Máscara, na guia Propriedades , selecione o ROI múltiplo de Dados de Treinamento e o ROI de Máscara mantendo pressionada a tecla Ctrl e clicando em cada um. Em seguida, clique em Intercalar abaixo da lista de propriedades de dados na seção rotulada como operações booleanas. Nomeie o novo conjunto de dados Trimmed Training Input e verifique se a geometria corresponde ao conjunto de dados filtrado antes de clicar em OK.
5. Usando o assistente de segmentação para treinamento iterativo (Figura 1.3)
- Importe os dados de treinamento para o assistente de segmentação clicando primeiro com o botão direito do mouse no conjunto de dados filtrados na guia Propriedades e, em seguida, selecionando a opção Assistente de Segmentação . Quando uma nova janela for aberta, procure a guia de entrada no lado direito. Clique em Importar quadros de um multi-ROI e selecione a entrada de treinamento aparada.
- (Opcional) Crie um Visual Feedback Frame para monitorar o progresso do treinamento em tempo real.
- Selecione um quadro dos dados que não esteja segmentado e clique em + para adicioná-lo como um novo quadro. Clique duas vezes no rótulo misto à direita do quadro e altere-o para Monitoramento.
- Para gerar um Novo Modelo de Rede Neural, no lado direito da guia Modelos, clique no botão + para gerar um novo modelo. Selecione U-Net na lista e, em seguida, para a dimensão de entrada, selecione fatias 2.5D e 5 e clique em Gerar.
- Para treinar a Rede, clique em Treinar no canto inferior direito da janela do SegWiz .
NOTA: O treinamento pode ser interrompido mais cedo sem perder o progresso. - Para usar a rede treinada para segmentar novos quadros, quando o treinamento U-Net estiver concluído, crie um novo quadro e clique em Prever (canto inferior direito). Em seguida, clique na seta para cima no canto superior direito do quadro previsto para transferir a segmentação para o quadro real.
- Para corrigir a previsão, clique com a tecla Ctrl pressionada em duas classes para alterar os pixels segmentados de uma para a outra. Selecione ambas as classes e pinte com o pincel para pintar apenas pixels pertencentes a qualquer uma das classes. Corrija a segmentação em pelo menos cinco novos quadros.
NOTA: Pintar com a ferramenta pincel enquanto ambas as classes estão selecionadas significa que, em vez de apagar com a tecla Shift pressionada, como normalmente acontece, ela converterá pixels da primeira classe para a segunda. Ctrl-clique realizará o inverso. - Para treinamento iterativo, clique no botão Treinar novamente e permita que a rede treine mais para mais 30-40 épocas, momento em que pare o treinamento e repita as etapas 4.5 e 4.6 para outra rodada de treinamento.
Observação : dessa forma, um modelo pode ser iterativamente treinado e aprimorado usando um único conjunto de dados. - Para publicar a rede, quando estiver satisfeito com seu desempenho, saia do Assistente de Segmentação. Na caixa de diálogo que aparece automaticamente perguntando quais modelos publicar (salvar), selecione a rede bem-sucedida, nomeie-a e publique-a para disponibilizar a rede para uso fora do assistente de segmentação.
6. Aplicar a rede (Figura 1.4)
- Para se aplicar primeiro ao tomograma de treinamento, selecione o conjunto de dados filtrado no painel Propriedades . No painel Segmentação à esquerda, role para baixo até a seção Segmento com IA . Verifique se o conjunto de dados correto está selecionado, escolha o modelo que acabou de ser publicado no menu suspenso e clique em Segmento | Todas as fatias. Como alternativa, selecione Visualizar para exibir uma visualização de uma fatia da segmentação.
- Para aplicar a um conjunto de dados de inferência, importe o novo tomograma. Pré-processo de acordo com a Etapa 3 (Figura 1.1). No painel Segmentação, vá para a seção Segmento com IA. Verifique se o tomograma recém-filtrado é o conjunto de dados selecionado, escolha o modelo treinado anteriormente e clique em Segmento | Todas as fatias.
7. Manipulação e limpeza de segmentação
- Limpe rapidamente o ruído escolhendo primeiro uma das classes que tem ruído segmentado e o recurso de interesse. Clique com o botão direito do mouse | Ilhas de Processo | Remover por Voxel Count | Selecione um tamanho de voxel. Comece pequeno (~ 200) e aumente gradualmente a contagem para remover a maior parte do ruído.
- Para correção de segmentação, clique com a tecla Ctrl pressionada em duas classes para pintar apenas os pixels pertencentes a essas classes. Clique com a tecla Ctrl pressionada + arraste com as ferramentas de segmentação para alterar os pixels da segunda classe para a primeira e clique com a tecla Shift pressionada + arraste para realizar o oposto. Continue fazendo isso para corrigir rapidamente pixels rotulados incorretamente.
- Separe os componentes conectados.
- Escolha uma classe. Clique com o botão direito do mouse em uma classe em Multi-ROI | Separe os componentes conectados para criar uma nova classe para cada componente que não está conectado a outro componente da mesma classe. Use os botões abaixo do Multi-ROI para mesclar facilmente as classes.
- Exporte o ROI como binário/TIFF.
- Escolha uma classe no Multi-ROI, clique com o botão direito do mouse e Extrair classe como um ROI. No painel de propriedades acima, selecione o novo ROI, clique com o botão direito do mouse | Exportação | ROI como binário (certifique-se de que a opção para exportar todas as imagens em um arquivo esteja selecionada).
NOTA: Os usuários podem facilmente converter do formato tiff para mrc usando o programa IMOD tif2mrc20. Isso é útil para o rastreamento de filamentos.
- Escolha uma classe no Multi-ROI, clique com o botão direito do mouse e Extrair classe como um ROI. No painel de propriedades acima, selecione o novo ROI, clique com o botão direito do mouse | Exportação | ROI como binário (certifique-se de que a opção para exportar todas as imagens em um arquivo esteja selecionada).
8. Gerando coordenadas para sub-tomograma em média a partir do ROI
- Extraia uma classe.
- Clique com o botão direito do mouse em Classe a ser usada para média | Extrair classe como ROI. ROI da classe com o botão direito do mouse | Componentes conectados | Novo Multi-ROI (26 conectados).
- Gerar coordenadas.
- Clique com o botão direito do mouse no novo Multi-ROI | Gerador escalar. Expanda Medições Básicas com Conjunto de Dados | verifique Centro Ponderado de Massa X, Y e Z. Selecione o conjunto de dados e a computação. Clique com o botão direito do mouse em Multi-ROI | Exportar valores escalares. Marque Selecionar todos os slots escalares e, em seguida, OK para gerar coordenadas do mundo do centroide para cada classe no multi-ROI como um arquivo CSV.
NOTA: Se as partículas estiverem próximas umas das outras e as segmentações estiverem se tocando, pode ser necessário realizar uma transformação de bacia hidrográfica para separar os componentes em um multi-ROI.
- Clique com o botão direito do mouse no novo Multi-ROI | Gerador escalar. Expanda Medições Básicas com Conjunto de Dados | verifique Centro Ponderado de Massa X, Y e Z. Selecione o conjunto de dados e a computação. Clique com o botão direito do mouse em Multi-ROI | Exportar valores escalares. Marque Selecionar todos os slots escalares e, em seguida, OK para gerar coordenadas do mundo do centroide para cada classe no multi-ROI como um arquivo CSV.
9. Transformação de bacias hidrográficas
- Extraia a classe clicando com o botão direito do mouse na classe Multi-ROI a ser usada para a média | Extrair classe como ROI. Nomeie esta máscara de bacia hidrográfica de ROI.
- (Opcional) Feche buracos.
- Se as partículas segmentadas tiverem orifícios ou aberturas, feche-os para a bacia hidrográfica. Clique no ROI em Propriedades de Dados. Na guia Segmentação (à esquerda), vá para Operações Morfológicas e use qualquer combinação de Dilato, Erode e Fechar necessária para obter segmentações sólidas sem furos.
- Inverta o ROI clicando no ROI | Copie o objeto selecionado (abaixo de Propriedades de dados). Selecione o ROI copiado e, no lado esquerdo, na guia Segmentação , clique em Inverter.
- Crie um mapa de distância clicando com o botão direito do mouse no ROI invertido | Criar mapeamento de | Mapa de Distância. Para uso posterior, faça uma cópia do mapa de distância e inverta-o (clique com o botão direito do mouse | Modificar e transformar | Inverter valores | Inscreva-se). Nomeie este mapa invertido de Paisagem.
- Crie pontos de propagação.
- Oculte o ROI e exiba o Mapa de Distância. Na guia Segmentação , clique em Definir Intervalo e diminua o intervalo até que apenas alguns pixels no centro de cada ponto sejam realçados e nenhum seja conectado a outro ponto. Na parte inferior da seção Intervalo , clique em Adicionar a Novo. Nomeie esses novos pontos de semente de ROI.
- Realizar transformação de bacias hidrográficas.
- ROI de pontos de propagação com o botão direito do mouse | Componentes conectados | Novo Multi-ROI (26 conectados). Clique com o botão direito do mouse no Multi-ROI recém-gerado | Transformada de bacias hidrográficas. Selecione o mapa de distância chamado Paisagem e clique em OK; selecione o ROI chamado Máscara de Bacias Hidrográficas e clique em OK para calcular uma transformação de bacia hidrográfica de cada ponto de semente e separar partículas individuais em classes separadas no multi-ROI. Gere coordenadas como na etapa 8.2.
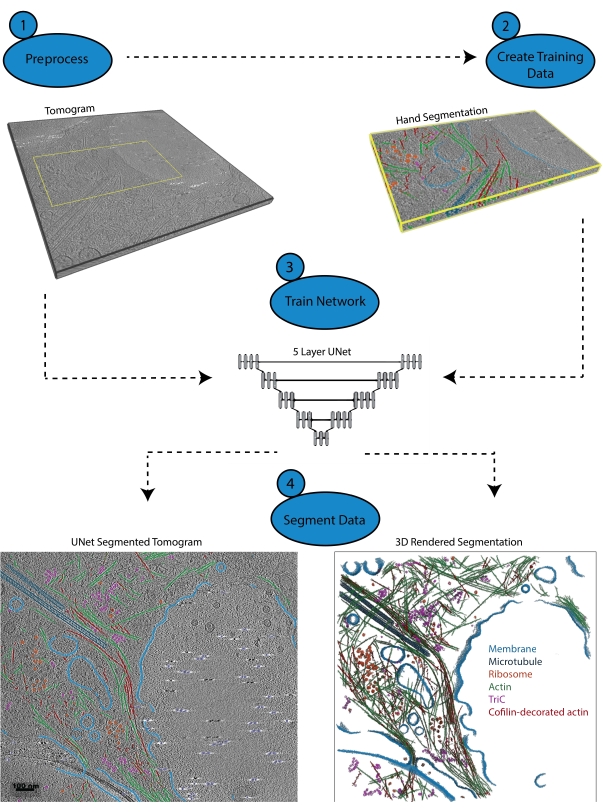
Figura 1: Fluxo de trabalho. 1) Pré-processe o tomograma de treinamento calibrando a escala de intensidade e filtrando o conjunto de dados. 2) Crie os dados de treinamento segmentando manualmente uma pequena porção de um tomograma com todos os rótulos apropriados que o usuário deseja identificar. 3) Usando o tomograma filtrado como entrada e a segmentação de mão como saída de treinamento, um U-Net de cinco camadas e várias fatias é treinado no assistente de segmentação. 4) A rede treinada pode ser aplicada ao tomograma completo para anotá-lo e uma renderização 3D pode ser gerada a partir de cada classe segmentada. Clique aqui para ver uma versão maior desta figura.
Resultados
Seguindo o protocolo, uma U-Net de cinco fatias foi treinada em um único tomograma (Figura 2A) para identificar cinco classes: Membrana, Microtúbulos, Actina, Marcadores fiduciais e Fundo. A rede foi treinada iterativamente um total de três vezes e, em seguida, aplicada ao tomograma para segmentá-lo completamente e anotá-lo (Figura 2B,C). A limpeza mínima foi realizada usando as etapas 7.1 e 7.2. Os próximos três tomogramas de interesse (Figura 2D,G,J) foram carregados no software para pré-processamento. Antes da importação da imagem, um dos tomogramas (Figura 2J) exigia ajuste do tamanho do pixel de 17,22 Å/px para 13,3 Å/px, pois era coletado em um microscópio diferente em uma ampliação ligeiramente diferente. O programa IMOD squeezevol foi usado para redimensionamento com o seguinte comando:
'squeezevol -f 0.772 inputfile.mrc outputfile.mrc'
Neste comando, -f refere-se ao fator pelo qual alterar o tamanho do pixel (neste caso: 13.3/17.22). Após a importação, todos os três alvos de inferência foram pré-processados de acordo com as etapas 3.2 e 3.3 e, em seguida, aplicou-se o U-Net de cinco fatias. A limpeza mínima foi novamente realizada. As segmentações finais são exibidas na Figura 2.
As segmentações de microtúbulos de cada tomograma foram exportadas como arquivos TIF binários (etapa 7.4), convertidos para MRC (programa IMOD tif2mrc ) e, em seguida, utilizados para correlação de cilindros e rastreamento de filamentos. Segmentações binárias de filamentos resultam em um rastreamento de filamentos muito mais robusto do que o rastreamento sobre tomogramas. Mapas de coordenadas do traçado de filamentos (Figura 3) serão usados para análises posteriores, como medições do vizinho mais próximo (empacotamento de filamentos) e subtomograma helicoidal com média ao longo de filamentos únicos para determinar a orientação dos microtúbulos.
Redes malsucedidas ou mal treinadas são fáceis de determinar. Uma rede com falha será incapaz de segmentar quaisquer estruturas, enquanto uma rede inadequadamente treinada normalmente segmentará algumas estruturas corretamente e terá um número significativo de falsos positivos e falsos negativos. Essas redes podem ser corrigidas e treinadas iterativamente para melhorar seu desempenho. O assistente de segmentação calcula automaticamente o coeficiente de similaridade Dice de um modelo (chamado de pontuação no SegWiz) depois que ele é treinado. Esta estatística fornece uma estimativa da semelhança entre os dados de treinamento e a segmentação U-Net. O Dragonfly 2022.1 também possui uma ferramenta integrada para avaliar o desempenho de um modelo que pode ser acessada na guia Inteligência Artificial na parte superior da interface (consulte a documentação para uso).
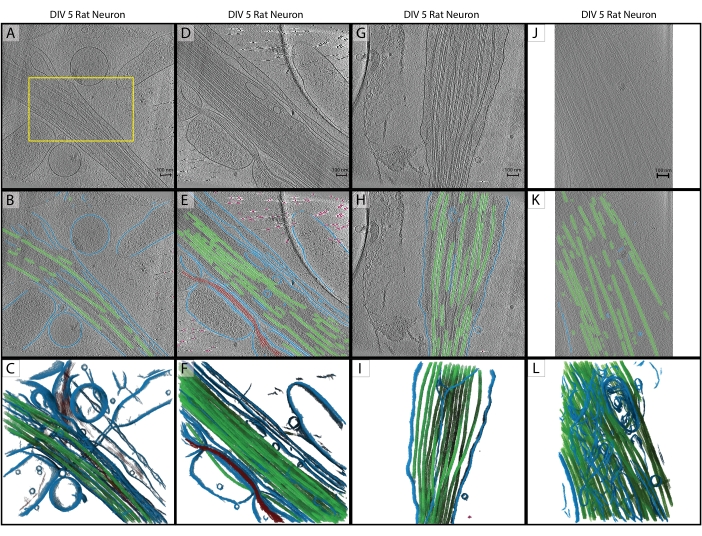
Figura 2: Inferência. (A-C) Tomograma de treinamento original de um neurônio de rato hipocampal DIV 5, coletado em 2019 em um Titan Krios. Trata-se de uma reconstrução retroprojetada com correção de CTF no IMOD. (A) A caixa amarela representa a região onde a segmentação manual foi realizada para a entrada do treinamento. (B) Segmentação 2D da U-Net após a conclusão do treinamento. (C) renderização 3D das regiões segmentadas mostrando membrana (azul), microtúbulos (verde) e actina (vermelho). (D-F) DIV 5 neurônio de rato hipocampal da mesma sessão que o tomograma de treinamento. (E) segmentação 2D da U-Net sem treinamento adicional e limpeza rápida. Membrana (azul), microtúbulos (verde), actina (vermelho), fiduciais (rosa). (F) Renderização 3D das regiões segmentadas. (G-I) DIV 5 neurônio de rato hipocampal da sessão de 2019. (H) Segmentação 2D a partir da U-Net com limpeza rápida e (I) renderização 3D. (J-L) Neurônio de rato hipocampal DIV 5, coletado em 2021 em um Titan Krios diferente em uma ampliação diferente. O tamanho do pixel foi alterado com o programa IMOD squeezevol para corresponder ao tomograma de treinamento. (K) segmentação 2D da U-Net com limpeza rápida, demonstrando inferência robusta em conjuntos de dados com pré-processamento adequado e (L) renderização 3D de segmentação. Barras de escala = 100 nm. Abreviaturas: DIV = dias in vitro; CTF = função de transferência de contraste. Por favor, clique aqui para ver uma versão maior desta figura.
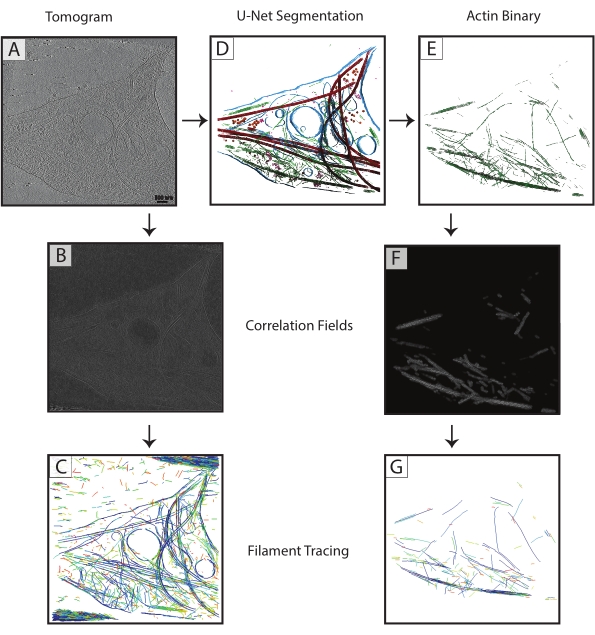
Figura 3: Melhora do traçado de filamentos . (A) Tomograma de um neurônio hipocampal de rato DIV 4, coletado em um Titan Krios. (B) Mapa de correlação gerado a partir da correlação do cilindro sobre filamentos de actina. (C) Traçado de filamentos de actina utilizando as intensidades dos filamentos de actina no mapa de correlação para definir parâmetros. O rastreamento captura a membrana e os microtúbulos, bem como o ruído, ao tentar rastrear apenas a actina. (D) Segmentação U-Net do tomograma. Membrana destacada em azul, microtúbulos em vermelho, ribossomos em laranja, triC em roxo e actina em verde. (E) Segmentação de actina extraída como uma máscara binária para rastreamento de filamentos. (F) Mapa de correlação gerado a partir da correlação do cilindro com os mesmos parâmetros de (B). (G) Rastreio de filamentos significativamente melhorado de filamentos de actina apenas do tomograma. Abreviação: DIV = days in vitro. Por favor, clique aqui para ver uma versão maior desta figura.
Arquivo suplementar 1: O tomograma usado neste protocolo e o multi-ROI que foi gerado como entrada de treinamento são incluídos como um conjunto de dados empacotado (Training.ORSObject). Veja https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct.
Discussão
Este protocolo estabelece um procedimento para usar o software Dragonfly 2022.1 para treinar um U-Net de várias classes a partir de um único tomograma e como inferir essa rede para outros tomogramas que não precisam ser do mesmo conjunto de dados. O treinamento é relativamente rápido (pode ser tão rápido quanto 3-5 min por época ou tão lento quanto algumas horas, dependendo inteiramente da rede que está sendo treinada e do hardware usado), e treinar uma rede para melhorar seu aprendizado é intuitivo. Contanto que as etapas de pré-processamento sejam realizadas para cada tomograma, a inferência é tipicamente robusta.
O pré-processamento consistente é a etapa mais crítica para a inferência de aprendizado profundo. Existem muitos filtros de imagem no software e o usuário pode experimentar para determinar quais filtros funcionam melhor para conjuntos de dados específicos; note que qualquer filtragem usada no tomograma de treinamento deve ser aplicada da mesma maneira aos tomogramas de inferência. Deve-se também ter o cuidado de fornecer à rede informações de treinamento precisas e suficientes. É vital que todos os recursos segmentados dentro das fatias de treinamento sejam segmentados com o maior cuidado e precisão possível.
A segmentação de imagens é facilitada por uma sofisticada interface de usuário de nível comercial. Ele fornece todas as ferramentas necessárias para a segmentação das mãos e permite a simples redesignação de voxels de qualquer classe para outra antes do treinamento e reciclagem. O usuário tem permissão para segmentar voxels manualmente dentro de todo o contexto do tomograma, e eles recebem várias visualizações e a capacidade de girar o volume livremente. Além disso, o software fornece a capacidade de usar redes multiclasse, que tendem a ter um desempenho melhor16 e são mais rápidas do que segmentar com várias redes de classe única.
Há, é claro, limitações para as capacidades de uma rede neural. Os dados crio-ET são, por natureza, muito ruidosos e limitados na amostragem angular, o que leva a distorções específicas de orientação em objetos idênticos21. O treinamento depende de um especialista para segmentar manualmente as estruturas com precisão, e uma rede bem-sucedida é tão boa (ou tão ruim) quanto os dados de treinamento que lhe são fornecidos. A filtragem de imagem para aumentar o sinal é útil para o treinador, mas ainda há muitos casos em que a identificação precisa de todos os pixels de uma determinada estrutura é difícil. Por isso, é importante que se tome muito cuidado na hora de criar a segmentação de treinamentos para que a rede tenha as melhores informações possíveis de aprender durante o treinamento.
Esse fluxo de trabalho pode ser facilmente modificado de acordo com a preferência de cada usuário. Embora seja essencial que todos os tomogramas sejam pré-processados exatamente da mesma maneira, não é necessário usar os filtros exatos usados no protocolo. O software tem inúmeras opções de filtragem de imagem, e recomenda-se otimizá-las para os dados específicos do usuário antes de iniciar um grande projeto de segmentação que abrange muitos tomogramas. Há também algumas arquiteturas de rede disponíveis para uso: descobriu-se que uma U-Net multi-fatia funciona melhor para os dados deste laboratório, mas outro usuário pode achar que outra arquitetura (como uma U-Net 3D ou um Sensor 3D) funciona melhor. O assistente de segmentação fornece uma interface conveniente para comparar o desempenho de várias redes usando os mesmos dados de treinamento.
Ferramentas como as apresentadas aqui tornarão a segmentação manual de tomogramas completos uma tarefa do passado. Com redes neurais bem treinadas que são robustamente inferíveis, é totalmente viável criar um fluxo de trabalho onde os dados tomográficos são reconstruídos, processados e totalmente segmentados tão rapidamente quanto o microscópio pode coletá-los.
Divulgações
A licença de acesso aberto para este protocolo foi paga pela Object Research Systems.
Agradecimentos
Este estudo foi apoiado pelo Penn State College of Medicine e pelo Departamento de Bioquímica e Biologia Molecular, bem como pelo subsídio 4100079742-EXT do Tobacco Settlement Fund (TSF). Os serviços e instrumentos CryoEM e CryoET Core (RRID:SCR_021178) utilizados neste projeto foram financiados, em parte, pela Faculdade de Medicina da Universidade Estadual da Pensilvânia através do Gabinete do Vice-Decano de Pesquisa e Estudantes de Pós-Graduação e do Departamento de Saúde da Pensilvânia usando Fundos de Liquidação do Tabaco (CURE). O conteúdo é de responsabilidade exclusiva dos autores e não representa necessariamente as opiniões oficiais da Universidade ou Faculdade de Medicina. O Departamento de Saúde da Pensilvânia especificamente se isenta de responsabilidade por quaisquer análises, interpretações ou conclusões.
Materiais
| Name | Company | Catalog Number | Comments |
| Dragonfly 2022.1 | Object Research Systems | https://www.theobjects.com/dragonfly/index.html | |
| E18 Rat Dissociated Hippocampus | Transnetyx Tissue | KTSDEDHP | https://tissue.transnetyx.com/faqs |
| IMOD | University of Colorado | https://bio3d.colorado.edu/imod/ | |
| Intel® Xeon® Gold 6124 CPU 3.2GHz | Intel | https://www.intel.com/content/www/us/en/products/sku/120493/intel-xeon-gold-6134-processor-24-75m-cache-3-20-ghz/specifications.html | |
| NVIDIA Quadro P4000 | NVIDIA | https://www.nvidia.com/content/dam/en-zz/Solutions/design-visualization/productspage/quadro/quadro-desktop/quadro-pascal-p4000-data-sheet-a4-nvidia-704358-r2-web.pdf | |
| Windows 10 Enterprise 2016 | Microsoft | https://www.microsoft.com/en-us/evalcenter/evaluate-windows-10-enterprise | |
| Workstation Minimum Requirements | https://theobjects.com/dragonfly/system-requirements.html |
Referências
- Bai, X. -. C., Mcmullan, G., Scheres, S. H. W. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- de Oliveira, T. M., van Beek, L., Shilliday, F., Debreczeni, J., Phillips, C. Cryo-EM: The resolution revolution and drug discovery. SLAS Discovery. 26 (1), 17-31 (2021).
- Danev, R., Yanagisawa, H., Kikkawa, M. Cryo-EM performance testing of hardware and data acquisition strategies. Microscopy. 70 (6), 487-497 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- . Tomography 5 and Tomo Live Software User-friendly batch acquisition for and on-the-fly reconstruction for cryo-electron tomography Datasheet Available from: https://assets.thermofisher.com/TFS-Assets/MSD/Datasheets/tomography-5-software-ds0362.pdf (2022)
- Danev, R., Baumeister, W. Expanding the boundaries of cryo-EM with phase plates. Current Opinion in Structural Biology. 46, 87-94 (2017).
- Hylton, R. K., Swulius, M. T. Challenges and triumphs in cryo-electron tomography. iScience. 24 (9), (2021).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Oikonomou, C. M., Jensen, G. J. Cellular electron cryotomography: Toward structural biology in situ. Annual Review of Biochemistry. 86, 873-896 (2017).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-the cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Lam, V., Villa, E. Practical approaches for Cryo-FIB milling and applications for cellular cryo-electron tomography. Methods in Molecular Biology. 2215, 49-82 (2021).
- Chreifi, G., Chen, S., Metskas, L. A., Kaplan, M., Jensen, G. J. Rapid tilt-series acquisition for electron cryotomography. Journal of Structural Biology. 205 (2), 163-169 (2019).
- Eisenstein, F., Danev, R., Pilhofer, M. Improved applicability and robustness of fast cryo-electron tomography data acquisition. Journal of Structural Biology. 208 (2), 107-114 (2019).
- Esteva, A., et al. Deep learning-enabled medical computer vision. npj Digital Medicine. 4 (1), (2021).
- Liu, Y. -. T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nature Methods. 14 (10), 983-985 (2017).
- Ronneberger, O., Fischer, P., Brox, T. U-net: Convolutional networks for biomedical image segmentation. Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 9351, 234-241 (2015).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iancu, C. V., et al. A "flip-flop" rotation stage for routine dual-axis electron cryotomography. Journal of Structural Biology. 151 (3), 288-297 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados