Method Article
안녕하세요 - C : Genomes의 3 차원 구조를 공부하는 방법.
* 이 저자들은 동등하게 기여했습니다
Erratum Notice
요약
히 - C 방식은 염색질 상호 작용 (1) 편견, 게놈 차원의 식별이 가능합니다. 히 - C의 커플 근접 결합 및 대규모 병렬 시퀀싱합니다. 그 결과 데이터가 여러 스케일에서 게놈 구조를 연구하는 데 사용할 수 있습니다 : 초기 결과는 같은 염색체 지역, 개방적이고 닫힌 염색질의 분리 및 megabase 규모의 염색질 구조와 같은 기능을 확인.
초록
염색체의 3 차원 접는는 게놈을 compartmentalizes하고 닫습니다 공간적 근접 2-6에 이러한 발기인 및 강화로 멀리 기능 요소를, 가져올 수 있습니다. 염색체 조직과 게놈 활동 사이의 관계를 파악하는 것은 전사 및 복제와 같은 게놈 프로세스를 이해하는 데 도움이됩니다. 그러나, 약간은 염색체가 접는 방법에 대해 잘 알려져 있습니다. 현미경은 loci 동시에 또는 높은 해상도에서 다수의 구별 수 없습니다. 날짜하려면, 염색체 형태 캡처 (3C) 및 그 후속 adaptations를 사용하여 염색체의 상호 작용 감지 게놈 - 광범위한 연구를 불가능 7-10 만들기, 대상 loci의 집합의 선택이 필요합니다.
우리는 하이 - C, 편견, 게놈 차원의 방식으로 원거리 상호 작용을 식별하는 능력이 3C의 확장을 개발했습니다. 안녕하세요 - C에서는 전지가 상호 작용하는 loci가 공유 결합 DNA - 단백질 상호 링크에 의해 서로 바운드되는 원인, 포름 알데히드와 함께 고정됩니다. DNA가 후 제한 효소와 조각되면 이러한 loci가 연결된 상태로 유지됩니다. 5 'overhangs가 다음 날이 엔드 내고이 교차 연결된 DNA 조각 사이의 결합 이벤트를 선호 희석 조건 하에서 수행 채워 있으므로 biotinylated 잔여물이 통합됩니다. 핵 서로 가까이에서 원래되었습니다 조각 쌍에 대응하는 결합 제품의 게놈 전체의 도서관에서이 결과를. 각 내고 제품은 접합의 사이트에서 비오틴과 함께 표시됩니다. 도서관은 가지각색이고, 분기점은 streptavidin 구슬로 다운 뽑아 있습니다. 정화 분기점은 이후 상호 작용하는 조각의 카탈로그 결과, 높은 처리량 시퀀서를 사용하여 분석하실 수 있습니다.
결과 연락처 행렬의 직접적인 분석은 염색체 지역의 존재와 작은 유전자 풍부한 염색체의 특혜 협회, 게놈 조직의 여러 기능을 보여줍니다. 상관 관계 분석은 인간 게놈 두 구획으로 차별 것을 보여주는, 연락처 행렬에 적용할 수 있습니다 : 오픈 액세스 및 활성 염색질과보다 치밀한 구획을 포함하는 적은 밀도 포장 구역은 폐쇄, 액세스할 수 및 비활성 염색질 지역을 포함. 마지막으로, 이론 및 전산 시뮬레이션 derivations와 결합 연락처 모체의 앙상블 분석, megabase 규모에 하이 C가 프랙탈 소구의 형태와 일치하는 기능을 보여줍 것으로 나타났다.
프로토콜
이 방법에 보고된 연구에 사용된 Lieberman - 에이든 외., 과학 326, 289-293 (2009) .
I.의 Crosslinking, 소화, DNA 엔드의 마킹과 블런트 엔드 내고
- 안녕하세요 - C는 모든 3C 기반 방법 중에서 일반적으로 스레드는 세포의 crosslinking 시작합니다. 시작하려면, 자기편하거나 중지하고, crosslink 세포 중, 2 X 10 7 및 2.5 X 10 7 포유 동물 세포 사이의 성장. (세포의 crosslinking에 대한 자세한 내용은 참조하시기 바랍니다 : 11
- 균질 화기를 사용하여 (500 μl 10 MM 트리스 - HCL 산도 8.0, 10 MM NaCl, 0.2 % Igepal CA - 630와 50 μl 테아제 억제제) 550 μl lysisbuffer에있는 세포를 Lyse. 5,000 RPM에서 염색질을 스핀 500 μl 1X NEBuffer 2 회 펠렛 씻으십시오.
- 5 번호 튜브에 1X NEBuffer 2 나누어지는의 염색질을 Resuspend와 362 μl의 최종 볼륨 1X NEBuffer 2를 추가합니다. 38 μl 1퍼센트 SDS 추가, 10 분 동안 65 ° C에서 신중하게 믹스와 부화. 바로 부화 후 다시 얼음에 튜브를 삽입하십시오.
- 44 μl 트리톤 X - 100 조심스럽게 혼합을 추가하여 SDS를 끄다. 회전하는 동안 ° C 야간 37 HindIII와 부화 400 유닛을 추가하여 염색질을 소화.
- 다음 단계는 하이 - C 특정하고 DNA를 표시하는 비오틴과 가교 조각의 무뚝뚝한 엔드 내고 수행과 끝이 있습니다. 이 단계는 결합의 분기점이 나중에 정화 수 있도록합니다. 튜브 1 biotinylation 단계를 받아야하지 말아야하는 대신 별도의 보관해야하고 소화를 위해 3C 제어 역할을하고, 결합 조건이 최적되었습니다.
- 제한 단편 overhangs에 기입하여 DNA가 남아있는 네 튜브에 비오틴로 끝나는 마르크, 1.5 μl 10 MM dATP, 1.5 μl 10 MM dGTP, 1.5 μl 10 MM dTTP, 37.5 μl 0.4 MM 비오틴 - 14 - dCTP를 추가하고 튜브 2-5 10 μl 5U/μl Klenow. 37 신중하게 섞어서 45 분 동안 품어 ° C.
- 얼음 튜브를 놓습니다. 효소를 inactivate하려면, 튜브 1-5 86 μl 10% SDS를 추가합니다. ° C 정확히 30 분 65 튜브를 품어 즉시 나중에 얼음을 놓으십시오.
- 내고는 가교 조각 사이의 결합 이벤트를 선호하기 위해 매우 희석 조건 하에서 수행됩니다. 얼음 작업, 7.61 ML의 결합 믹스 [745 μl 10% 트리톤 X - 100, 745 μl 10X 내고 버퍼 (500 MM 트리스 - HCL 산도 7.5, 100 MM MgCl 2, 100 MM DTT), 80 μl 10 MG / ML BSA를 추가 80 μl 100 MM ATP와 5.96 ML 물] 다섯 각각 15 ML 튜브를 번호. 해당 15 ML 튜브 각 소화 염색질 혼합물을 전송합니다.
- 일반 3C 내고 들어, 튜브 1-10 μl 1U/μl T4 DNA ligase의를 추가합니다. 무딘 엔드 하이 C의 결합을위한, 튜브 2-5 50 μl 1U/μl T4 DNA ligase의를 추가합니다. 반전 튜브로 믹스와 16에서 4 시간 동안 모든 5 튜브를 품어 ° C.
- Crosslinks는 반전과 단백질 튜브 당 50 μl 10 MG / ML proteinase K를 추가 65 ° C.에서 하룻밤 튜브를 잠복기에 의해 타락 아르 다음날 튜브 당 추가 50 μl 10 MG / ML proteinase K를 추가하고 65에서 배양을 계속 ° C를 다른 2 시간.
- 실내 온도에 반응 혼합물을 쿨 다섯 50 ML 원뿔 튜브에게 전송할 수 있습니다. 페놀 추출을 수행하여 이러한 튜브의 DNA 정화. 10 ML 페놀 산도 8.0 2 분 와동을 추가합니다. 3,500 rpm으로 10 분 동안 튜브를 스핀 조심스럽게 새로운 50 ML 튜브에 많은 수성 단계의 가능한 양도할 수 없습니다.
- 페놀 산도 8.0을 사용하여 추출 반복 : 클로로포름 (1:1)을하고 에탄올을 사용하여 DNA를 침전. (DNA의 정화에 대한 자세한 내용을 참조하시기 바랍니다 : 11
- 에탄올의 원심 분리는 DNA를 시켰던 후, 450 μl 1X TE 각 DNA 펠릿 (10 MM 트리스 - HCL 산도 8.0, 1 ㎜ EDTA (에틸렌 다이아 민 테트라 초산)) 디졸브. 1.7 ML의 원심 관에 DNA 혼합물을 전송합니다.
- 클로로포름 extractions : 정화의 또 다른 라운드 2 페놀을 수행하여 수행됩니다. 클로로포름 (1:1)과 1 분 와동 : 500 μl 페놀 산도 8.0을 추가합니다. 14,000 rpm으로 5 분 튜브를 원심 새로운 튜브로 수성 단계를 전송합니다. 두 번째 추출 후, NaOAc의 0.1x 볼륨, -80에서 100 % 에탄올과 부화 30 분 2X 볼륨 ° C.를 추가하여 DNA를 침전
- 시켰던 DNA를 회전 후 70 % 에탄올 각 DNA 펠렛을 씻어 25 μl 1X TE 각 DNA 펠렛을 resuspend. 1 μl 1 MG / ML RNAse마다 튜브를 추가하고 37 30 분 튜브를 잠복기가 존재 수있는 RNA ° C.이 저하될 풀 튜브 2-5의 하이 C의 내용은 여전히 3C 제어로 튜브 1 별도의 유지.
- 이제 안녕 - C 표시하고 결합의 효율성을 시험하는 좋은 기회입니다. 이러한 컨트롤은 하이 - C 라이브러리는 성공할 수 있는지 여부 우수 지표입니다.
- 라이브러리의 품질과 수량을 확인하려면, 2 μl와 0.8 % 아가로 오스 겔에서 3C 및 하이 C 라이브러리 모두에서 1시 10분 dilutions 6 μl aliquots를 실행합니다. (그림 2A 참조)
- 안녕하세요 - C 마킹 및 하이 C의 결합 효율은 PCR 다이제스트 분석에 의해 확인됩니다. 성공적인 보충과 HindIII 사이트의 결합 (AAGCTT) 제한 효소 NheI (GCTAGC)에 대한 사이트 만듭니다. 근접한 두개의 제한 조각의 형성 특정 내고 제품은 PCR (3C 11 템플릿으로 각 도서관의 0.2 μl를 사용하여 마찬가지로 증폭됩니다. PCR 제품은 이후 HindIII, NheI 또는 모두 소화하고 있습니다. 2 %의 겔에서 샘플을 실행 후 , 3C 및 하이 C의 결합 이벤트의 상대적인 숫자는 절단과 포경 밴드의 강도를 (그림 2B 참조) quantifying로 예상하실 수 있습니다.
- 일부 파편은 출혈도 잡았되지 않았을 것입니다, 나중에 그들을 잡아당기는 방지 T4의 DNA 중합 효소의 exonuclease 활동을 사용하여 이러한 unligated 끝에서 비오틴을 제거합니다.
- 비 출혈도 잡았 DNA의 끝에 비오틴 - 14 - dCTP는 T4의 DNA 중합 효소의 exonuclease 활동과 함께 제거됩니다. 1 μl 10 MG / ML BSA, 10 μl 10X NEBuffer 2, 1 μl 10 MM dATP, 1 μl 10 MM의 dGTP, 5 단위 100 μl의 총 볼륨에서 T4의 DNA 중합 효소와 함께 하이 - C 라이브러리의 5 μg을 믹스하고을 품어 12 혼합 ° C 2 시간. 가능하면 여러 5 μg 반응이 수행됩니다.
- 반응은 2 μl 0.5 M EDTA (에틸렌 다이아 민 테트라 초산)에게 산도 8.0을 추가하여 중지됩니다.
- 클로로포름 (1:1) 추출은 에탄올 침전에 의해 다음 과정을 수행한다 : DNA, 페놀 산도 8.0을 정화합니다.
- 뜨는은 삭제되고 DNA의 알약 100 μl 물의 총 볼륨 resuspended 및 풀링됩니다.
II. 전단 및 사이즈 선택
- 높은 처리량 시퀀싱에 적합 biotinylated DNA를 만들기 위해 DNA는 Covaris S2 악기와 함께 300-500 basepairs (듀티 사이클 5, 강도 5 사이클 / 200 시간 4 사이클 60 초를 버스트)의 크기에 빠졌어해야합니다 .
- 가지각색 DNA의 종료를 복구하려면, 14 μl 10X 내고 버퍼, 14 μl 2.5 MM dNTP 믹스, 5 μl T4의 DNA 중합 효소, 5 μl T4 폴리 뉴클레오 타이드의 키나제, 1 μl Klenow DNA의 중합 효소 1 μl 물을 추가합니다. 실온에서 30 분 알을 품다.
- 부화 후, 제조 업체의 권고에 따라 DNA를 정화하기 위해 Qiagen의 MinElute 칼럼을 사용합니다. 15 μl 1X 트리스 - 낮은 EDTA (에틸렌 다이아 민 테트라 초산) (: 10 MM 트리스 산도 8.0, 0.1 MM EDTA (에틸렌 다이아 민 테트라 초산) TLE)로 두 번 DNA를 Elute. 그런 다음, 10 μl 1 ㎜ dATP, 2 μl 물 3 μl Klenow (엑소 -)를 5 μl 10X NEBuffer2을 추가하여 최종 수리 DNA의 3 '끝까지 dATP를 첨부합니다. 37 30 분 동안 반응을 품어 ° C.
- Klenow의 조각을 inactivate하려면 65 20 분 동안 반응을 품어 ° C와 이후 얼음에 반응을 냉각. speedvac 사용하여 20 μl로 반응 볼륨을 줄일 수 있습니다.
- 다음 1X 태와 1.5 % 아가로 오스 겔에서 DNA를로드 80 - 90V에서 3.5 시간 동안 실행됩니다. SYBR 녹색으로 젤 얼룩 후 DarkReader에서 DNA를 시각화. 300 500 기본 쌍 사이 엑사이스 DNA 조각이 젤의 무게에 따라 2-4 열을 사용하여 Qiagen의 겔 추출 키트를 정화. 50 μl 1X TLE와 DNA를 Elute.
- Qiaquick 컬럼에서 eluates를 결합하고 1X TLE 300 μl에 최종 볼륨을 가져와. 마지막으로 QuBit fluorometer를 사용하여 퀀트 - IT 분석과 DNA 농도를 결정하고 DNA의 총 금액을 계산합니다.
III. 비오틴은 풀다운 및 이점 엔드 시퀀싱
- 프로토콜의이 섹션에서는 결합의 분기점은 이점 엔드 시퀀싱에 의해 염색질 조각을 상호 작용의 효율적인 식별 수 있도록 DNA 수영장에서 정화 수 있습니다. DNA의 LoBind 튜브의 모든 후속 단계를 수행합니다.
- 풀다운 μl 150 400 μl 트윈 버퍼 (: 5 MM 트리스 - HCL 산도 8.0, 0.5 MM EDTA (에틸렌 다이아 민 테트라 초산), 1 M NaCl, 0.05 % 트윈 TB)로 두 번 자기 streptavidin 비즈를 resuspended 세탁하여 비오틴을 위해 구슬을 준비합니다.
이러한 미래의 세척은 다섯 단계로 구성되어 있습니다 :- 구슬로 버퍼를 추가
- 새로운 튜브에 혼합 전송
- 상온에서 3 분 동안 샘플을 회전
- 자기 입자 농축기를 사용하여 비즈를 복원
- 뜨는을 제거
- 300 μl 2X 없음 트윈 버퍼 (2X NTB : 10 MM 트리스 - HCL 산도 8.0, 1 ㎜ EDTA (에틸렌 다이아 민 테트라 초산), 2 M NaCl)에 비즈를 Resuspend 300 μl 하이 C의 DNA와 결합. 안녕하세요 - C DNA라는 비오틴은 회전 15 분 동안 상온에서 혼합 잠복기하여 streptavidin 비즈에 바인딩을 허용합니다.
- 자기 입자 concentr와 streptavidin 비즈를 묶고있는 DNA를 복원애터, 그리고 표면에 뜨는를 제거합니다. 100 μl 1X 내고 버퍼 다음에 400 μl 1X NTB (5 MM 트리스 - HCL 산도 8.0, 0.5 MM EDTA (에틸렌 다이아 민 테트라 초산), 1 M NaCl)에있는 구슬을 씻으십시오. 50 μl 1X 내고 버퍼에있는 구슬을 Resuspend 새로운 튜브로 혼합물을 전송하기만하면됩니다.
- Illumina 이점 엔드 시퀀싱을위한 DNA를 준비하려면 이전 단계 2.6 계산 비오틴 풀다운을위한 입력으로 사용되는 DNA의 총량을하고있다 하이 C의 DNA의 양을 추정 20로 나누면 아래로 끌어 내고 사용할 수 있습니다되었습니다. 내고 사용할 수 하이 C의 DNA의 μg 당 Illumina 이점 최종 어댑터 6 picomoles를 추가합니다. DNA에 어댑터를 ligate에 1,200 유닛에게 T4 DNA Ligase의를 사용하십시오. 상온에서 2 시간 동안 부화.
- 히 - C DNA 바운드 구슬을 하자는 400 μl 1X 결핵과 함께 두 번 구슬을 세탁하여 비 출혈도 잡았 이점 엔드 어댑터를 제거합니다.
- 200 μl 후 50 μl 1X NEBuffer 2 다음 200 μl의 1X NTB와 구슬을 씻으십시오. 마지막 세척 후, 50 μl 1X NEBuffer 2의 구슬을 resuspend 새로운 튜브로 전송할 수 있습니다.
- 시퀀싱 충분히 PCR 제품을 생성하는 데 필요한 사이클의 수를 확인하려면, 6, 9, 12 또는 15주기와 네 곳의 시험 PCR 반응을 설정할 수 있습니다. (PCR의 증폭에 대한 자세한 내용은을 참조하십시오 : 12, 5 % polyacrylamide 젤과 Sybr 그린과 얼룩에 PCR 반응을 실행 가짜 밴드 400 사이의 얼룩의 존재의 부재를함으로써 최적의 사이클 번호를 확인 -. 어댑터 내고 후 가지각색 제품의 길이는 600 기본 쌍.
- PCR주기의 최적의 번호와 대규모 PCR에 하이 C 라이브러리 - 바운드 streptavidin 비즈의 나머지 부분을 확대. 별도의 우물에서 수영장 PCR 제품과 구슬을 확보. 젤에서 실행하고 1.8x 볼륨 Ampure 비즈는 제조 업체의 권장 사항에 따라 함께 PCR 제품의 나머지를 정화하기 위해 대규모 PCR 제품의 1 %가 별도로 보관.
- 50 μl 1X TLE 버퍼로 DNA를 Elute과 5 % polyacrylamide 젤에서 원본 PCR 제품의 1 % 나누어지는에 Ampure 비드 정화 PCR 제품의 1 %를 비교, PCR primers의 성공적인 제거를 보장.
- 또한 복제 1 히 - C 라이브러리의 μl와 생거 시퀀싱을 사용하여 약 100 클론의 제품을 결정하는 것이 좋습니다. 이것은 하이 - C는 PCR 혼합물에서 읽습니다 alignable의 상대 번호를 평가할 수 있도록합니다. 전형적인 결과를 얻으려면 그림 3B를 참조하십시오.
- Illumina 이점 엔드 시퀀싱과 하이 - C 라이브러리 순서. 독립적으로 상호 작용하는 염색질 조각을 식별하는 Maq (http://maq.sourceforge.net/)를 사용하여 각 끝을 맞춥니다.
IV. 대표 히 - C 결과
- 히 - C 프로토콜 품질 관리 기준을 기술적으로 잘 수행하고 있다고 생각할 수 있는데 때 다음과 같은 결과가 예상된다.
- 품질 관리 단계를 모두 3C 및 하이 C 라이브러리 10킬로바이트보다 큰로서 오히려 꽉 밴드를 실행하는 것을 공개한다. DNA의 얼룩이 좋지 내고 효율을 나타냅니다. 일반적으로 결합 효율 3C 템플릿 (그림 2A 참조)에 비해 하이 C 라이브러리에 약간 낮습니다.
- 안녕하세요 - C 표시 및 결합 효율 3C primers를 사용하여 생성된 PCR 제품의 소화에 의해 예상하실 수 있습니다. 3C의 분기점은 NheI 아닌 HindIII로 잘라 수 있습니다. 반대는 하이 - C 분기점에 대한 사실입니다. 이 PCR 다이제스트 분석은 효율적인 (그림 2B 참조) 결합의 분기점의 마킹을 확인, 하이 - C의 amplicons의 70 %가 NheI가 아니라 HindIII에 의해 절단되는 보여줍니다.
- 합성 읽은의 분석은 파란색과 빨간색 선으로 표시 intrachromosomal과 interchromosomal 모두 상호 작용에서 읽는 것을 표시해야 임의로 생성된에 비해 HindIII 제한 사이트에 훨씬 더 가까이 (그림 3A 참조), 녹색으로 표시된 읽습니다 맞춥니다.
- 성공적인 실험에서 alignable 읽기 쌍 55%는 interchromosomal 상호 작용을 나타냅니다. 15 % 떨어져 파편 미만 20킬로바이트 30 % 사이 intrachromosomal 상호 작용을 나타내는 이상 20킬로바이트는 (그림 3B 참조) 떨어져 intrachromosomal 읽기 쌍이있다. 이 배포판은 품질 관리의 한 형태로, 전에 높은 처리량 시퀀싱으로 샘플링 할 수 있으며, 복제와 약 100 클론의 생거 시퀀싱은 일반적으로 충분합니다.
- 염색질 상호 작용은 X와 Y - 축이 게놈 순서 loci를 대표하고 각 픽셀은 그들 사이의 상호 작용 관찰의 수를 나타냅니다 히트맵로 시각 수 있습니다. 일반적으로, 매우 선형 게놈에서 서로 가까이 DNA 조각의 경향이 서로 자주 교류해야합니다. 이것은 눈에 잘 띄는 대각선으로 intrachromosomal heatmaps (그림 4A 참조)에서 볼 수 있습니다.
- 다음 결과는 게놈 조직의 다양한 수준을 나타내기 위해 데이터를 분석의 다른 방법을 보여줍니다. 연락처 probabilit를 계획하고Y 대 게놈의 거리 (그림 5A 참조)에 문의의 확률 결국 고원에 도달, 게놈 거리의 함수로 감소 보여줍니다. 모든 거리에서 실선으로 표시된 intrachromosomal 상호 작용은, 점선 라인으로 표현, interchromosomal 상호 작용에 상대적으로 풍부한 있습니다. 이것은 직접 염색체 지역의 존재를 의미합니다.
- 염색체의 쌍 사이에 interchromosomal 연락처 관찰 / 예상 번호를 계산하는 것은 특정 염색체 쌍 사이의 특혜 협회 보여줍니다. 작은 유전자 풍부한 염색체는 우선적으로 밝은 붉은 색 (그림 5B 참조)로 표시, 서로 상호 작용할 수 있습니다.
- 개인 염색체도 검사를하실 수 있습니다. 원시 히트맵는 관찰 / 기대 히트맵 그 결과, loci의 쌍 사이의 게놈 거리에 대한 계정으로 예상 히트맵을 사용하여 조정할 수 있습니다. 그렇다면, 상관 행렬은 관찰 / 기대 heatmaps의 행과 열을 연관하여 생산 수 있습니다. 상관 관계 분석을 사용, 그것은 증명 두 구획으로 인간 게놈 segregates. 이것은 상관 heatmaps에 격자 무늬 패턴 (그림 4A - D 참조)에 의해 그림입니다. (히 - C 데이터 분석에 대한 자세한 내용은을 참조하십시오 : 1.
- 안녕하세요 - C 데이터를 사용하여 새로운 통찰력은 megabase 규모 염색질 접는로 얻고 있었다. 고분자 결로의 고전적인 모델은 평형 소구에 해당 염색질 팩을 제안합니다. 거리의 함수로하려하고 연락처 확률이 기울기 (그림 6A 참조) 약 -1입니다 게놈 거리와 전원 법으로 연락 확률 비늘을 보여줍니다. 이것은 평형 소구의 동작과 일치 아니지만, 프랙탈 소구 (그림 6B 참조)로 알려진 다른 구조와 일치하는 기대를 않습니다.
- 여기 두 개의 구형 구조가 표시됩니다. 착색 파란색에서 시안, 녹색, 황색, 오렌지 및 빨강 (그림 6C 상단 참조)에 이르기까지 하나의 종점에서 거리에 해당합니다. 평형 globules과는 달리, 프랙탈 globules는 entanglements 부족합니다. 프랙탈 소구에서 등고선을 따라 근처에 있습니다 loci (그림 6C 중반 참조) 단색 블록의 존재로 이어지는 3 차원에 가까운 경향이 있습니다. 이러한 블록 평형 소구 (그림 6C 하단 참조)에서 찾을 수 없습니다.
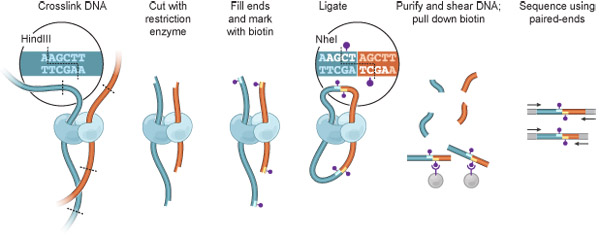
그림 1. 안녕 - C 개요. 전지는 공간 인접 염색질 세그먼트 (; 단백질, 이러한 상호 작용을 중재 수 있습니다 밝은 파란색과 시안에 표시됩니다 빨간색 진한 파란색, DNA 조각) 사이에 공유 결합 링크 결과, 포름 알데히드와 상호 연결되어 있습니다. 염색질은 제한 효소와 소화 (여기서, HindIII, 제한 사이트 : 점선은, 삽입된 페이지 참조). 그 결과 끈적 끝나 biotinylated 하나있는 세포핵 (보라색 점)으로 채워지게된다. 내고는 분자내 결합 이벤트를 좋아하는 매우 희석 조건 하에서 수행하며 HindIII 사이트가 손실되고 NheI 사이트는 (삽입된 페이지)가 생성됩니다. DNA가 정제와 가지각색, 그리고 biotinylated 분기점은 streptavidin 비즈를 사용하여 격리가있다는거다. 상호 작용하는 조각은 이점 엔드 시퀀싱에 의해 식별됩니다.

그림 2. 하이 C 라이브러리 품질 제어합니다. (A) 3C 제어 및 하이 C 라이브러리의 증가 금액은 0.8 % 아가로 오스 겔에 해결이되었습니다. 두 도서관은 10킬로바이트보다 큰 오히려 꽉 밴드로 실행됩니다. 히 - C 라이브러리에서 일반적인 결합 효율 3C 템플릿에서 관찰 무엇보다 약간 낮은 그리고 하이 C의 차선의 얼룩로 표시됩니다. (B) PCR 다이제스트 제어할 수 있습니다. 근접한 두개의 조각으로 구성된 내고 접합은 표준 3C PCR 조건을 사용하여 증폭됩니다. 히 - C의 결합 제품은 내고 사이트의 소화에 의해 종래의 3C에서 생산과는 구별하실 수 있습니다. 히 - C의 분기점은 NheI 아닌 HindIII에 의해 절단되며, 반대는 3C 분기점에 대한 사실입니다. 안녕하세요 - C amplicons의 70 %가 내고 접합의 마킹 효율적인 확인, NheI에 의해 절단되었습니다. 두 복제는 신뢰할 부량을 보장하기 위해 수행되었다.

그림 3. 안녕 - C는 품질 제어를 참조하십시오. (A) 두 intrachromosomal (파란색)과 interchromosomal (적색) 상호 작용은 무작위로 생성 (녹색) 읽기에 비해 HindIII 제한 사이트에 상당히 가까이 정렬에 해당하는 조각에서 읽습니다. intrachromosomal가 읽고 모두와 interchromosomal는 대지가 ~ 500 BP의 거리에 도달할 때까지 곡선 빠르게 HindIII 사이트 증가에서 거리로 감소 읽습니다. 이것은 시퀀싱에 사용되는 최대 조각 크기에 해당합니다. (B) 일반적으로 alignable 읽기 쌍 55%는 interchromosomal 상호 작용을 나타냅니다. 15 %는 파편 미만 20킬로바이트 사이 intrachromosomal 상호 작용을 나타냅니다떨어져 30 % 더 이상 20킬로바이트가 떨어져 intrachromosomal 읽을 쌍 있습니다. 이 배포판은 품질 관리의 한 형태로, 전에 높은 처리량 시퀀싱으로 샘플링 할 수 있으며, 복제와 약 100 클론의 생거 시퀀싱은 일반적으로 충분합니다.

그림 4. 상관 관계 분석은 핵 두 구획으로 시위는 것을 보여줍니다. (A) 염색체 14 intrachromosomal 상호 작용에 해당 히트맵. 각 픽셀은 1 MB 로커스와 다른 1 - MB의 로커스 사이의 모든 상호 작용을 나타냅니다; 강도는 읽기의 총 수 (범위 : 0-200 읽습니다)에 해당합니다. 틱 자국은 매 10 MB를 나타납니다. 히트맵는 강렬한 대각선의 형태로 대형 블록의 별자리에 기초 공사를 전시하고 있습니다. (염색체 14 acrocentric이며. 짧은 팔은 표시되지 않습니다) 주어진 게놈 거리에서 loci 한 쌍의 평균 연락처 확률을 계산하기 위해 하이 C의 데이터 세트를 사용하여, 기대 행렬이 될 무엇 (B) 해당을 생산 더 장기 구조가 없다면 관찰했다. 두 매트릭스의 지수가 관찰 / 기대 매트릭스이다 (C) 고갈이 파란색과 빨간색으로 농축 [범위 : 0.2 (블루) 5 (적색)에]의 표시입니다. 블록 패턴은 더 분명됩니다. 염색체 14을 따라 loci의 모든 쌍 intrachromosomal 상호 작용 프로필을 사이에 상관 관계 행렬 (D)는 상관 관계 [-1 (파란색) 1 (빨간색) 범위]을 보여줍니다. 인상적인 격자 무늬 패턴은 염색체 내에있는 두 구획의 존재를 나타냅니다.

그림 5. 존재와 염색체 지역의 조직입니다. (A)로 문의 확률 결국 ~ 90Mb (파란색)에서 고원에 도달, 염색체 1 게놈 거리의 함수로 줄어 듭니다. interchromosomal 접촉의 수준 (블랙 대시)는 염색체의 여러 쌍을 다른, 염색체 1 loci는 염색체 21 loci (적색 대시)와 상호 작용 가능성이 염색체 10 loci (그린 대시)와 최소 상호 작용 가능성이 가장 높은 수 있습니다. Interchromosomal 상호 작용은 intrachromosomal 상호 작용에 상대적으로 고갈됩니다. (B) 관찰된 / 염색체의 쌍 사이에 interchromosomal 연락처 수를 예상. 레드 농축을 표시하고, 파란색 고갈 나타냅니다 [범위 : 2 (빨강)로 0.5 (파랑)]. 작은 유전자 풍부한 염색체는 서로 더 많은 상호 작용하는 경향이 있습니다.

그림 6. 염색질의 현지 포장은 프랙탈 소구의 동작과 일치합니다. (A) 게놈 거리의 함수로 문의 확률은 게놈 (파란색) 전체 평균. 저명한 전력 법률 스케일링은 500킬로바이트와 -1.08의 경사 (시안에 표시된 적합한)와 (과 음영 지역) 7MB 사이에 볼 수있다. (B) 평형 (빨간색)와 프랙탈 (파란색) globules에 대한 거리의 함수로 연락 확률에 대한 시뮬레이션 결과. 프랙탈 소구에 대한 사면은 우리의 새로운 이론적 예측 1 확인 (시안) 매우 거의 -1입니다. 평형 소구에 대한 사면 전에 이론적 기대와 일치하는, -3 / 2입니다. 평형 소구에 대한 경사는 히 - C 데이터에 보는 반면 프랙탈 소구에 대한 경사가 밀접하게, 하이 - C 결과의 관찰 경사 유사합니다. (C) 톱 : 펼쳐 폴리머 체인, 4000 단량체 오래. 착색은 파란색에서 시안, 녹색, 황색, 오렌지 및 빨강에 이르기까지 하나의 종점에서 거리에 해당하는 중간 :. 우리 앙상블에서 그린 프랙탈 소구의 전형. 프랙탈 globules는 entanglements 부족합니다. . 평형 소구 : 등고선을 따라 근처에 있습니다 Loci의 표면과 단면 바닥에 뚜렷한 아르 큰 단색 블록의 존재로 이어지는 3 차원에 가까운 경향이 있습니다. 구조는 매우 얽혀 버린이며 등고선 (유사한 컬러)를 따라 인근에 있습니다 loci는 3D에 근처에있을 필요가 없습니다.
토론
우리는 편견, 게놈 - 광범위한 방식으로 염색질 상호 작용을 매핑하여 게놈의 3 차원 구조를 공부하는 방법을 제시. 분리 이전의 작품에서이 기술을 설정 뭐가 가장 중요한 실험 단계 - 무딘 엔드 내고 전에 가교 조각의 끝을 제한에 biotinylated 세포핵의 결합입니다. 이 단계를 수행하면 성공적으로 깊은 모든 결합의 분기점의 시퀀싱 수 있으며 하이 - C에게 그 범위와 능력을 제공합니다.
읽은의 개수는 궁극적으로 상호 작용지도의 해상도를 결정합니다. 여기에서 인간 게놈에 대한 1 MB 상호 작용지도는 ~ 30000000 alignable 읽기를 사용하여 제공됩니다. N의 요소에 의해 '모든 목적을'해상도를 향상시키기 위해, 읽기의 숫자는 N 2의 요인에 의해 증가한다.
히 - C 기술은 쉽게 이러한 하이브리드 라이브러리 생성 후에 캡처 (게놈의 특정 부분을 대상으로)와 결합 후 염색질의 immunoprecipitation (특정 단백질과 연관된 영역의 염색질 환경을 검토) 같은 다른 기술과 결합 수 있습니다.
공개
감사의 말
AP 에이든, XR 바오, M. 브레너, D. Galas, W. 고스퍼, A. Jaffer, A. 멜린 니 코브 감독, A. Miele, G. Giannoukos, C. Nusbaum, AJM Walhout, 우리는 토론과 코드 A. Kosmrlj 감사 , L. 우드, 그리고 K. Zeldovich 토론, 시각화에 도움과 L. 개프니 및 B. 웡.
패니와 존 허츠 재단 대학원 교제, 국방 과학 대학원 친목, NSF 대학원 친교, 국립 우주 바이오 메디컬 연구소, 그리고 노을 부여하여 지원. 국가 인간 게놈 연구소 (NHGRI) (EL)에서 T32 HG002295; i2b2 (생물 및 예의상 통합을위한 정보학), 브리검 여성 병원의 (LAM)에서 바이오 메디컬 컴퓨팅에 대한 NIH 지원 센터, 부여도 없습니다. NHGRI에서 HG003143하고 켁 재단 뛰어난 젊은 학자 수상 (JD). 원료 및 매핑 하이 - C 시퀀스 데이터는 GEO 데이터베이스 (에 입금되지 않은 www.ncbi.nlm.nih.gov/geo/ 가입도). GSE18199. 추가 시각화는에서 구할 수 있습니다 http://hic.umassmed.edu .
자료
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
참고문헌
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유