JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
랜덤 코니컬 틸트 메서드를 사용하여 Exosome 단지의 단일 입자의 전자 현미경의 재건
요약
이 문서는 부정적인 얼룩의 전자 현미경을 (EM)을 사용하여 생물 학적 macromolecules의 3 차원 (3D) 재건을하는 표준 방법을 설명합니다. 이 프로토콜에서는, 우리는 임의의 원뿔 기울기 재건 방법 (RCT)을 사용하여 중간 해상도 Saccharomyces cerevisiae의 exosome의 복합의 3D 구조를 얻는 방법을 설명합니다.
초록
단일 입자 전자 현미경 (EM)이 재건 최근 대형 macromolecular의 단지의 3 차원 (3D) 구조를 얻을 수있는 인기있는 도구가되었습니다. X - 선 crystallography에 비해 몇 가지 독특한 장점이 있습니다. 첫째, 단일 입자 EM 재건 특히 대형 macromolecular의 단지에 대해, X - 레이 crystallography에 병목있는 단백질 샘플을 구체화 필요가 없습니다. 둘째, 그것은 단백질 샘플을 다량 필요하지 않습니다. 결정에 필요한 단백질의 밀리그램에 비해, 단일 입자 EM 재건은 부정적인 얼룩 EM 방법을 사용하여 나노 어금니 농도에서 단백질 솔루션의 몇 마이크로 리터 필요합니다. 그러나, 높은 대칭과 함께 몇 macromolecular 어셈블리에도 불구하고, 하나의 입자는 EM은 특히 이러한 대칭없이 많은 표본에 대한 (하단보다 1 나노미터 해상도) 상대적으로 낮은 해상도로 제한됩니다. 이 방법도 연구에서 분자의 크기에 의해 제한, 일반적으로 냉동 수산화 표본에 대한 부정적인 스테인드 표본 300 KDA에 대한 즉, 100 KDA.
알 수없는 구조의 새로운 샘플의 경우, 우리는 일반적으로 부정적인 얼룩으로 분자를 포함하는 중금속 솔루션을 사용합니다. 표본은 다음 분자의 2 차원 (2D) micrographs을 위해 전송 전자 현미경으로 검사합니다. 이상적으로, 단백질 분자가 단일 차원 구조를 가지고 있지만 micrographs 다른 방향을 나타냅니다. 이러한 micrographs은 "단일 입자"로 디지털 및 컴퓨터에서 처리됩니다. 2 차원 배열과 분류 기법을 사용하여 같은 전망에 균일한 분자는 클래스로 클러스터된 수 있습니다. 그들의 평균 분자의 2 차원 형태의 신호를 향상시킬 수 있습니다. 우리가 적절한 상호 표정 (오일러 각도)와 입자를 지정 후, 우리는 3 차원 가상 볼륨에 2D 입자 이미지를 재구 성할 수있게 될 것입니다.
단일 입자 3 차원 재건에 필수적인 단계가 제대로 각 단일 입자의 적절한 방향을 지정하는 것입니다. 각도 reconstitution 1 임의의 원뿔 기울기 (RCT) 방법 2를 포함하여, 각 입자에 대한보기를 할당하는 방법은 여러 가지가 있습니다. 이 프로토콜에서는, 우리는 부정적인 얼룩 EM와 RCT를 사용하여 효모 exosome 복합 3D 재건을 얻는 우리의 연습을 설명합니다. 이것은 전자 현미경 및 이미지 프로세싱 우리의 프로토콜 RCT의 기본 원칙을 다음 있지만 방법을 수행할 수있는 유일한 방법은 아니라는 것을 주목하여야한다. 우리가 먼저 지속적인 얇은 탄소 필름 층으로 덮여 홀리 탄소 격자를 사용하여 단백질의 크기에 비해 두께 Uranyl - 편대의 계층에 단백질 샘플을 포함하는 방법에 대해 설명합니다. 그러면 표본을 수집 전송 전자 현미경에 삽입 untilted (0 급) 및 처리와 효모 exosome의 초기 3D 모델을 얻기 위해 나중에 사용됩니다 micrographs의 기울어진 (55 급) 쌍. 이를 위해, 우리는 RCT을 수행하고 다음 상세 검색 방법 3 일치 투영을 사용하여 초기 3D 모델을 수정하십시오.
프로토콜
1. 랜덤 코니컬 틸트 방법의 원리
- 임의의 원뿔 틸트 방식의 원리는 전자 현미경 내부의 표본 같은 지역의 micrographs 한 쌍을 데리고가 필요합니다. 한 사진은 untilted 위치 (그림 1A)의 시편을 찍은이고 다른 사진은 50-70도 (우리의 경우에, 우리는 55도 사용) 사이의 각도로 기울어져 시편을 찍은 것입니다. (그림 1B)
- 선택된 컴퓨터를 사용하여 디지털 현미경 쌍 같은 입자의 측면과 이미지의 측면을 넣어 것입니다. (그림 1C)
- 입체 좌표에서 untilted 입자와 그 기울어져 파트너의 이미지는 틸트 축과 기울기 각도의 방향으로 서로 상호 있습니다. (그림 1D)
- untilted 입자 이미지 정렬 해당 azimuthal 위치에 기울어져 입자의 이미지를 제공합니다. (그림 1E)
- azimuthal 공간을 채우는 기울어져 입자의 여러 이미지를 사용하여 분자의 3 차원 구조는 다시 - 투영 알고리즘을 사용하여 복원하실 수 있습니다. (그림 1 층)
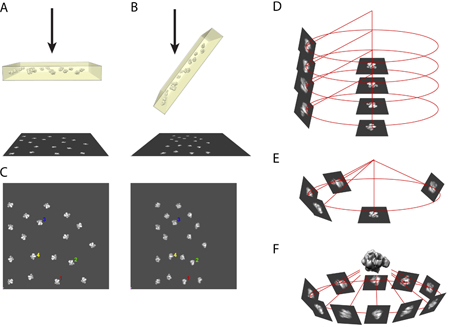
그림 1. RCT 재건을위한 원칙의 그림.
2. 얇은 탄소와 대상 홀리 탄소의 그리드 준비
이유 : 우리는 임의의 원뿔 틸트 재건을위한 단백질 샘플을 수정하는 부정적인 얼룩 방법을 사용합니다. 너무 건조 중에 평평화없이 macromolecules을 보존하기 위해, 우리는 단백질 4 차원에 대한 두께 깊은 얼룩에있는 단백질 분자를 포함하려고합니다. 일반적으로 연속적인 탄소는 부정적인 스테인드 표본 제작에 사용됩니다. 탄소 이러한 종류의 그러나, 단백질 입자 주위에 얼룩 두께를 제어하기가 어렵습니다. 우리는 따라서 사용하는 집에서 만든 스테인드 부정적인 표본을 만들기 위해 탄소 필름 (~ 5 나노미터 두께)의 얇은 층으로 덮여 홀리 탄소 격자합니다. 그것이 최적의 얼룩 두께 단백질을 포함하는 훨씬 더 쉽게되도록 구멍에 의해 형성된 작은 우물은 단백질 솔루션과 그리드에 얼룩 솔루션을 유지하실 수 있습니다. 또한, 구멍을 통해 탄소의 얇은 층 크게 배경 소음을 줄여줍니다.
- 0.5 % Formvar 솔루션을 준비합니다. 펌 후드에서 100 ML 유리 비이커에 0.45 g의 폴리비닐 공식 수지 90 ML의 클로로포름을 추가합니다. 비커를 커버하기 위해 알루미늄 호일을 사용하고, 자기 활동가에 대한 공식적인 수지를 용해 수 있도록 작은 저어 막대를 사용합니다. 그것은 수지를 해산하기 위해 약 15 분 정도 소요됩니다.
- Formvar의 해산 동안 깨끗한 현미경 유리는 메탄올의 슬라이드와 Kimwipes와 건조 닦으십시오.
- Formvar 수지가 완전히 클로로포름에 용해되면 용액의 표면에 1 ML 50 % 글리세롤을 추가합니다. 추가 글리세롤의 볼륨을 조절하면 홀리 탄소에있는 구멍의 밀도에 영향을 미칩니다. 약 1 인치 깊이의 솔루션에 ultrasonicator의 팁을 찍어 Formvar 솔루션에 글리세롤의 방울의 감광 유제를 만들기 위해 1 분 sonicate 최대 전력을 사용합니다. 이 솔루션은이 단계 이후 은하됩니다. 긴 sonication은 홀리 탄소에 구멍을 작은 크기를 일으 킵니다.
- 즉시 sonication 후, 1 초 동안 감광 유제에 수직으로 청소 유리 슬라이드를 찍어 그들을 데리고 슬라이드의 표면 위에 얇은 플라스틱 필름을 형성 필터 용지를 사용하여 슬라이드 '하의는 점이 되리. choloroform 증발 후, 가벼운 현미경 필름에있는 구멍의 밀도와 크기를 확인합니다. 필요에 따라 준비 상태를 조정합니다. 조건이 여기에 설명된로, 우리는 일반적으로 3 ~ 4 마이크로 미터 400 메쉬 그리드의 각 광장에서 10 ~ 20 구멍의 직경 구멍을 구하십시오.
- 유리 슬라이드는 건조 후, 슬라이드 표면에 플라스틱 필름의 가장자리를 잘라. 증류수의 표면에 필름을 플로트. 물 표면에 박막은 빛의 반사에 대한 반짝이기는하지만 각도로 볼 수 있습니다. 영화 다운 직면하고있는 격자 '매끄러운 표면 하나씩, 일에 400 메쉬 구리 격자.
- 종이를 사용하여 그것에 격자와 플라스틱 필름을 선택하십시오. 종이 뒤집기를하고는 배양 접시에 말리면. 구멍에 잔류 글리세롤을 제거하고 공기에있는 종이가 건조하도록 메탄올의 종이를 적시게.
- 탄소 증발기에서 ~ 20 나노미터의 두께와 탄소의 레이어와 코트의 격자. 두께는 탄소의 회색에 의해 결정하실 수 있습니다.
- Formvar를 제거 반 시간 동안 클로로포름에 탄소와 그리드를 적시게. 격자가 건조되면, 우리는 얻은 집에서 만든 홀리 탄소 격자합니다.
- 갓 죽습 운모의 표면에 두께 약 5 나노미터와 탄소의 얇은 레이어를 증발.
- 조심스럽게 증류수 아래 홀리 탄소 격자를 넣어. 물 표면에 운모 표면에서 얇은 탄소 플로트와 홀리 탄소에 그것을 입금천천히 격자. 펌 후드에 그리드를 건조시킵니다.
3. Exosome 단지의 부정적인 스테 이닝
이유 : uranyl 아세테이트, uranyl의 편대, phosphotungstic 산성 암모늄 molybdate, 그리고 다른 사람을 포함하여 부정 얼룩의 EM을 위해 사용할 수있는 몇몇 중금속 얼룩 솔루션이 있습니다. 다른 얼룩 솔루션은 서로 다른 독특한 특성이 있습니다. 예를 들어, uranyl 아세테이트는 입자의 높은 콘트라스트를 제공하지만 산성 환경을 좋아하지 않아 단백질 단지 충돌을 수 있습니다. 그 샘플, 중성 pH를 산성에서 phosphotungestic 좋은 얼룩 솔루션이 될 수 있습니다. 우리는 좋은 단위와 분자 단위로 높은 침투 능력으로 인해 포화 Uranyl의 편대 (UF) 솔루션을 선택합니다.
- 1 분 위해 증류수를 끓여. 상온으로 천천히 시원한 시간을 보내세요. 이 단계는 물에서 용존산소를 제거하는 것입니다.
- 신선한 2 % Uranyl의 편대 (UF) 솔루션을 확인하십시오. 1.5 ML 튜브 1 ML 물 20 MG의 UF를 섞습니다. 10 분 와동.
- 10 M의 수산화 칼륨 2 microliter를 추가하여 5.0 산도 값을 조정합니다. 즉시 섞는다. 솔루션 색깔은 더 노란색해야합니다. 솔루션의 산도가 너무 높은해서는 안됩니다, 그렇지 않으면 얼룩이 precipitates.
- 또 다른 10 분 vortexer에 튜브를 넣어.
- 10 분에 대해 최대 속도로 데스크탑 원심 분리기에 대한 솔루션을 스핀.
- 0.2 마이크로 미터 PVDF 막 통해 솔루션을 필터링합니다. 이것은 신선한 UF 솔루션입니다. 조명을 방지하기 위해 알루미늄 호일의 조각에 솔루션 튜브를 커버. 이 솔루션은 같은 날 사용할 수 있습니다.
- 글로우 방전 한 얇은 탄소 - 오버 - 홀리 전류 25mA에서 30 초 동안 글로우 방전 장치를 사용하여 탄소 격자.
- 벤치에 깨끗한 parafilm 한 조각을 넣어. parafilm 상단 50 microliter의 UF 얼룩 솔루션 3 방울을 넣어.
- 희석 버퍼 (25 MM 트리스 - HCL 산도 7.5, 100 MM NaCl, 2 MM DTT)를 사용하여 50 ~ 100 nm의의 농도에 exosome 복잡한을 희석. 형광 배출 격자에 희석 단백질 4 microliter 놔. 샘플 한 분간 그리드에가요. (참고 :. 분자의 이러한 최종 농도는 일반적으로 부정적인 스테인드 그리드에 잘 분산 입자의 최적 밀도를 제공합니다 uranyl의 편대 또는 uranyl의 acecate 얼룩 솔루션의 경우, 일반적으로 인산염이나 소금의 높은 농도는 (이상 0.5 M)입니다 좋은 얼룩 결과를 얻을 수 좋지 않아. 우리의 경험 Hepes 또는 파이프는 uranyl 얼룩 솔루션으로 잘 작동하고 있음을 나타냅니다.)
- 그리드의 가장자리에서 잔여 솔루션을 얼룩과 얼룩 방울 위에 즉시 격자를 플립 각 비말에 약 10 초 격자를 씻어 필터 종이를 사용하십시오.
- 마지막 헹굼 후 다른 1 분을위한 그리드에 얼룩 체류를하게 다음 필터 종이로 떨어져 얼룩 얼룩. 좋은 깊은 얼룩 결과를 얻을 수 있도록 그리드 표면에 얼룩이 솔루션의 얇은 레이어를 유지. 그리드 퓸 후드에 신속하게 건조 보자.
4. Exosome 단지의 전자 현미경
이유 : 틸팅 단계로 모든 전송 전자 현미경은 RCT 재건을위한 시편의 기울기 쌍을 수집하는 데 사용할 수 있습니다. 이론적으로, 높은 각도 표본은 더 나은 데이터를 수집 기울어져 수 있습니다. 연습에서, 표본 홀더와 그리드의 형상의 디자인으로 인해, 최대 실행할 각도는 50-70도 정도에서 제한됩니다. 이 프로토콜에서는, 우리는 황비홍 Tecnai - 12 전자 현미경을 사용하여 절차를 설명합니다. 현미경의 다른 모델의 경우 작업은 프로젝트와 악기의 속성의 요구에 따라 조정해야합니다.
- 샘플 홀더에있는 샘플 격자를 넣고 다음 페이 Tecnai -12 전자 현미경에 홀더를 넣어. 현미경은 120 KV에서 운영하고 있습니다. 우리는 사진 촬영을 Gatan Ultrascan4000 CCD 카메라를 사용합니다. 디지털 현미경 인터페이스의 '구성 카메라'대화 상자에서 '수직 축을 중심으로 대칭'이 올바른 handedness 결정을 보장하기 위해 선택 해제되어 있는지 확인합니다. (참고 :. 독자가 3D 모델을 얻을 스파이더 RCT 재건 절차에 의존하고 특히이 중요)
- 최고의 스테인드 사각형을 찾을 낮은 배율에서 샘플 격자를 확인하십시오. 사각형의 이러한 종류의 약 1의 크기와 구멍의 십여 ~ 2 마이크로 미터와 그들의 어두운 얼룩 영역이 있어야합니다. (그림 2)

그림 2. 좋고 나쁜 얼룩과 사각형을 보여주는 그리드의 낮은 배율 현미경. - 페이 사용자 인터페이스의 저용량 모드를 설정하고 검색을 정렬, 초점 및 저용량 모드에서 위치를 노출. 우리는 노출과 검색을위한 회절에서 1.5 미터 카메라 초점 길이, 52000를위한 150,000의 배율을 사용합니다. 노출 시간은 1 두 번째 조정됩니다. 거리 노출 POS에서 초점 순위가 2 마이크로 미터를 설정틸팅 축을 따라 ition.
- 검색 모드에서 좋은 얼룩으로 구멍을 찾아 위치를 저장합니다. 좋은 구멍은 일반적으로 그들의 어두운 얼룩 그라디언트가 검색 모드에서 관찰했습니다. 광장의 CCD 영상을 찍으세요. 55 도까지 표본을 기울이기 다른 사진을 찍었어요. 두 사진을 비교, 두 micrographs에 결합하여 구멍을 식별합니다. (그림 3)
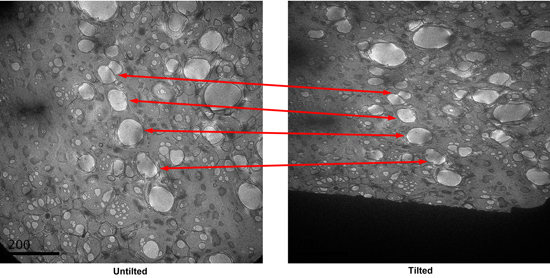
그림 3. 검색 모드에서 광장의 micrographs의 기울기 쌍. 해당 이점 구멍이 표시됩니다. - 다시 0 정도 무대를 기울이기. 높은 배율에서 검색 모드에서 확인된 각 구멍의 사진을 취할 수있는 낮은 복용량 키트를 사용합니다. 사용 defocus은 약 -0.7 마이크로 미터이다.
- 모든 구멍은 사진을 촬영한 후에 55 도까지 무대를 기울. -1.2에 대한 마이크로 미터의 defocus로 untilted 사람과 같은 배율에서 기울어져 표본의 micrographs를 가져가라. 낮은 배율 micrographs의 패턴에 따라 micrographs의 해당 기울어져 쌍을 식별합니다. 집에서 만든 홀리 탄소 격자의 불규칙한 패턴은 상관 관계를하는 데 도움이됩니다. micrographs의 기울기 쌍 검사와 같은 얕은 스테인드 분야 (입자가 기울어진 상태에 따라 그 주위에 후광을 가지고 나타납니다)로 나쁜 얼룩과 micrographs를 제거합니다. (그림 4)
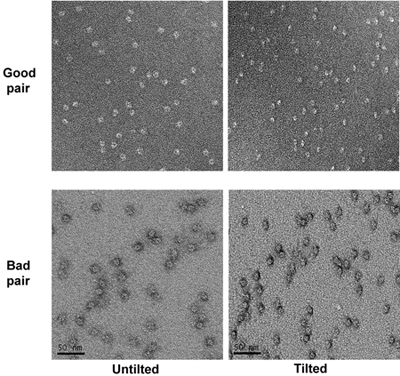
그림 4. 높은 배율에서 시료의 micrographs 두 기울기 쌍. 좋고 나쁜 micrographs가 표시됩니다.
5. 데이터의 이미지 처리
이유 : 컴퓨터에 RCT의 재건을 수행하기 위해 다양한 옵션과 소프트웨어 패키지가 있습니다. 가장 일반적으로 사용되는 스파이더 5. 거미의 RCT을 수행하는 기본적인 프로토콜은 웹 페이지에서 찾을 수 있습니다 http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . 거미의 RCT을 수행하기 위해 상세한 프로토콜 쉐이크 외하여 문서에 설명되어 있습니다. 6의 프로토콜에서는, 우리는 IMAGIC - 5 7 프로토콜의 비디오 버전의 스파이더의 조합을 사용합니다. 우리는 또한 전적으로 프로토콜의 텍스트 버전에서 거미를 사용하는 대체 절차를 제공합니다.
- 프로그램을 설치합니다. 우리는 Gatan 디지털 이미지에서 거미 이미지 형식으로 이미지 포맷을 변경하려면 EMAN 8 패키지에 proc2d을 사용합니다. IMAGIC - 5는 2 차원 배열을하는 데 사용됩니다. 거미는 3D 재건 및 개선을하는 데 사용됩니다.
하위 섹션 1 : 입자의 기울기 쌍을 잡았습니다.
- EMAN에 proc2d 명령을 사용하여 거미 이미지 형식으로 *. dm3 Gatan 디지털 이미지를 변환합니다. 기울여서 untilted 쌍은 각각 *** t.spi 및 *** u.spi 같은 패턴으로 지정됩니다.
- 스파이더 프로그램 패키지에 배포 '웹'프로그램을 사용하여 입자 쌍을 선택하십시오. 의 지시를 따르십시오 좌표 automaticly 각각 untilted 및 기울어져 입자 DCU ***. SPI 및 DCT ***. SPI에 저장됩니다. 세타는이 없기 때문에 기울기 쌍 사이 WEB에서 조립하는 동안 세 각도는 최종 재건의 올바른 handedness을 보증하지 않습니다 :. 다른 문서 DCB ***. SPI는 기울여서 untilted 입자 사이의 기울기 각도 정보 (주를 포함 기호 (양수 값)와 파이와 감마는 그들에게 설정 초기 값에 의존하고 있습니다. 기울어져 현미경에 defocus 그라데이션의 방향을 검토 것이 올바른를하기 위해 피팅 전에 파이와 감마의 올바른 초기 값을 설정하는 데 도움 재건. 그림 5의 handedness은 올바른 규칙을 보여줍니다.)
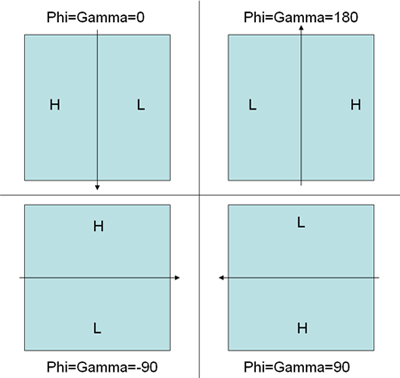
그림 5. defocus 기울기에 의해 결정 맞는 모드를 따기 WEB 경사 쌍의 입자의 각도 결정의 규칙을 설명하기 위해 그림. L은 낮은 defocus 지역을 대표하면서 H는 높은 defocus 영역을 나타냅니다. 화살표는 틸팅 축을 나타냅니다. PHI와 감마에 해당하는 올바른 초기 각도는 각 스키마에 대해 표시됩니다. - 로 웹페이지에 표시된 거미 스크립트의 수정된 버전을 사용하는 모든 고른 입자 아웃 박스 http://www.wadsworth.org/spider_doc/spider/docs/partpick.html을 . 스크립트가 u.spi 및 t.spi로 untilted 및 기울어져 입자 스택을 저장합니다. 입자 출신 현미경의 번호는 particle_list.spi에 저장해야합니다. (참고 : 이것은에 대한 올바른 오일러 각도 파일을 생성하는 것이 매우 중요합니다RCT.)
하위 섹션 2 : untilted 입자 이미지의 2 차원 배열과 분류.
- untilted 입자 Imagic - 5 패키지로 em2em 프로그램을 사용하여 IMAGIC - 5 형식으로 변환합니다. 정렬과 반복 IMAGIC - 5 프로그램 (부록 A)를 사용하여 단일 클래스로 입자를 분류. 수업 우리가 imagic_classes.lis으로 저장 입자의 테이블을 조회 생성 IMAGIC - 5에서 MSA - 이름 수준의 명령을 사용합니다. (참고 :. 각 클래스의 분류 및 정렬은 동일한 모양과 입자의 수를 증가뿐만 아니라 수업에 변화를 줄일 수있다 분산지도 수업의 품질에 대한 정보를 제공할 수 있습니다.)
- IMAGIC - 5 헤더 명령을 사용하여 각 입자의 정렬의 번역 및 회전 값을 플롯 파일 (비디오 데모에 ali_50.plt) 생성.
- 스파이더 문서 파일 base_file ***. SPI로 수업이 룩업 테이블에서 배포 펄 스크립트 lis2spi.pl 사용하여 변환 http://cryoem.berkeley.edu을 .
- 배포 스크립트를 사용 plt2spi.pl 거미 문서 파일 ali_50.spi에 단계 5.6에서 번역 및 회전 값을 생성 줄거리 파일에서 각 입자의 정렬의 번역 및 회전 값을 변환 http://cryoem.berkeley.edu을 .
그것은 우리 손 안에이 직업에서 최고의 성능을 제공하기 때문에 우리는 2D 정렬 및 분류에 대한 IMAGIC - 5를 사용합니다. 2 차원 배열과 분류에 대한 거미의 대체 전략에서 찾을 수 있습니다 http://www.wadsworth.org/spider_doc/spider/docs/align.html . 우리는 또한 exosome의 untilted 입자에 2D 분석을 수행하기 위해 스파이더를 사용했습니다. 다음은 간단한 절차입니다.
에서 설명한대로 대체 5.5) 참조 - 무료 정렬을 사용하여 이미지를 맞춥니다. 두 가지 간단한 스크립트를 찾을 수 있습니다 회전과 문서 파일 angular_file.spi에있는 모든 입자의 변화를 저장합니다.
대체 5.6)가로 설명한 같은 전망과 함께 그룹으로 정렬된 입자를 분류 우리는 분류를위한 K - 수단 방법을 사용했습니다. 생성 base_file ***. SPI는 분류에 따라.
하위 섹션 3 : 기울어져 입자 이미지를 사용하여 입체 재건.
- 대역 통과 필터, 마스크 센터 단지 IMAGIC - 5에 untilted 입자로 기울어져 입자. 라는 입자에 대한 새 데이터 집합을 생성합니다. (참고 :이 단계는 선택 사항입니다 그것은 거미에서 할 수 있습니다.).
- 클래스 룩업 테이블 문서 ali_50.spi 단계 5.3 및 부록 B.처럼 거미 스크립트를 사용 단계 5.4에서 입자 목록 파일 partile_list.spi에 웹에 의해 생성되는 DCB ***. SPI 파일에서 anglular 문서 파일 만들기
- 에서 설명한대로 각 클래스의 재건을 할 수 스파이더에서 재건축 스크립트를 사용 http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . 입자의 모든 클래스를 하나의 3 차원 재건 볼륨에 기여하고 있습니다. 3D 모델은 UCSF - 치메라 9 검사하실 수 있습니다. 오일러 각도 (0,0,0)에서 3D 모델의 2 차원 투영은 재건의 품질을 확인 untilted 입자에서 해당 2D 클래스 평균 비교할 수 있습니다. 유사한 볼륨을 찾기, 정렬 및 프로 시저 다음과 같은 초기 볼륨 생성하기 위해 그들을 병합 http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html을 .
하위 섹션 4 : untilted 입자 이미지를 사용하여 3D 재건 다듬 는다.
- 높은 해상도 볼럼를 얻기 위해 모든 untilted 입자에 대한 병합 초기 볼륨의 투영 검색 상세 검색을 할에서 설명한대로 거미 스크립트를 사용하여 누락 콘과 평평화 유물없이 E http://www.wadsworth.org/spider_doc/spider/docs/techs/recon/refine.html .
6. 대표 결과 :
RCT 방법을 사용, 우리는 5000 틸트 쌍 총 (그림 6)에서 exosome의 약 50 reconstructions를 획득했습니다. 50 3D 모델에서, 우리는 주로 두 직교 경관을 감상할 수있는 격자의 복잡한 앉아 여러 방향을 볼 수 있습니다. 평평화 유물은 탄소 표면에 직각 방향에있는 볼륨의 많은에서도 감지할 수있다. 우리는 정렬을 수행하고 직교 전망에 두 초기 볼륨을 생성하는 3D 볼륨 병합. 5000 untilted 입자 이미지를 사용하여, 우리는 모두 초기 모델 (그림 7)에서 약 18 앵스트롬 해상도에서 exosome의 동일한 최종 3D 재건을 획득했습니다. 구조는 효모 exosome의 아키텍처를 공개하고 RNA 기판 모집 경로 10에 통찰력을 제공했습니다.

그림 6. RCT 재건축하여 exosome 복잡한 3D 모델 50.
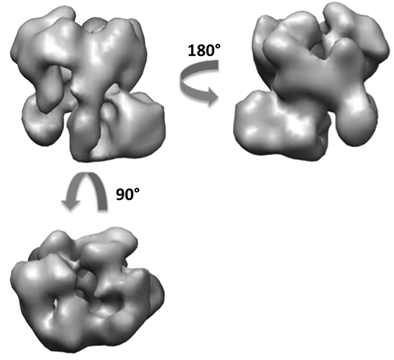
그림 7. 정제 후 exosome 복합의 3D 재건.
부록 :
IMAGIC - 5 2D 정렬 및 분류에 대한 부록 A.이 스크립트 파일.
파일 : auto_align_i.sh
파일을 보시려면 여기를 클릭하세요
부록 B. 스파이더에서 3D 재건 각도 파일을 생성하기위한 스크립트 파일.
파일 : generate_angular_file.spi
파일을 보시려면 여기를 클릭하세요
토론
이 문서에서 우리는 샘플 준비 상세한 프로토콜과 부정적인 얼룩의 전자 현미경을 사용하여 exosome 복잡한 세 차원 재건을 제시. 이 방법을 사용하여, 우리는 구조의 어떤 사전 지식없이 임의의 원뿔 기울기 방법을 사용하여 3D 재건을 획득하였습니다. 랜덤 원뿔 틸트 방식은 반드시 단일 샘플을 필요로하지 않습니다 그러나 다음과 같은 투영 일치 상세 검색 단계는 높은 해상도를 달성하기 위해서...
공개
감사의 말
저자는 전체 프로토콜을 확립하기 위해 도움 예일 대학에서 초기 프로토콜과 왕 실험실의 구성원 확립 도움 UC 버클리에서 노갈레즈 실험실의 구성원을 감사드립니다. 우리는 또한 그들의 지원에 대한 의학의 예일 학교 cryo - EM 시설 및 고성능 계산 센터에서 직원을 인정합니다. HW는 스미스 가족 Awardee입니다.
자료
| Name | Company | Catalog Number | Comments |
| Polyvinyl Formal Resin | Electron Microscopy Sciences | 63450-15-7 | |
| Uranyl Formate | Electron Microscopy Sciences | 22451 | |
| Superfrost Microscope Slides | Thermo Fisher Scientific, Inc. | 4951F-001 | |
| 400 mesh grid regular | SPI Supplies | 3040C | |
| Carbon coater Auto 306 | Edwards Lifesciences | ||
| Tecnai-12 Electron Microscope | FEI | ||
| Glow Discharger | BAL-TEC | Sputter Coater SCD 005 |
참고문헌
- van Heel, M. Angular reconstitution: a posteriori assignment of projection directions for 3D reconstruction. Ultramicroscopy. 21, 111-123 (1987).
- Radermacher, M. Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J Electron Microsc Tech. 9, 359-394 (1988).
- Penczek, P. A., Grassucci, R. A., Frank, J. The ribosome at improved resolution: new techniques for merging and orientation refinement in 3D cryo-electron microscopy of biological particles. Ultramicroscopy. 53, 251-270 (1994).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative staining and image classification - powerful tools in modern electron microscopy. Biol Proced Online. 6, 23-34 (2004).
- Frank, J., Radermacher, M., Penczek, P., Zhu, J., Li, Y., Ladjadj, M., Leith, A. SPIDER and WEB: processing and visualization of images in 3D electron microscopy and related fields. J Struct Biol. 116, 190-199 (1996).
- Shaikh, T. R., Gao, H., Baxter, W. T., Asturias, F. J., Boisset, N., Leith, A., Frank, J. SPIDER image processing for single particle reconstruction of biological macromolecules from electron micrographs. Nat Protoc. 3, 1941-1974 (2008).
- Heel, M. v. a. n., Harauz, G., Orlova, E. V., Schmidt, R., Schatz, M. A new generation of the IMAGIC image processing system. J Struct Biol. 116, 17-24 (1996).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: semiautomated software for high-resolution single-particle reconstructions. J Struct Biol. 128, 82-97 (1999).
- Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C., Ferrin, T. E. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25, 1605-1612 (2004).
- Wang, H. W., Wang, J., Ding, F., Callahan, K., Bratkowski, M. A., Buttler, J. S., Nogales, E., Ke, A. Architecture of the yeast Rrp44 exosome complex suggests routes of RNA recruitment for 3' end processing. Proc Natl Acad Sci USA. 104, 16844-16849 (2007).
- Scheres, S. H., Nunez-Ramirez, R., Sorzano, C. O., Carazo, J. M., Marabini, R. Image processing for electron microscopy single-particle analysis using Xmipp. Nat Protoc. 3, 977-990 (2008).
- Yoshioka, C., Pulokas, J., Fellmann, D., Potter, C. S., Milligan, R. A., Carragher, B. Automation of random conical tilt and orthogonal tilt data collection using feature-based correlation. J Struct Biol. 159, 335-346 (2007).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유