JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
포유류의 링커 - 히스톤 Subtypes의 표현 분석
요약
우리는 H1의 링커 histones의 표현 수준을 분석 assays 집합을 설명합니다. H1의 histones의 단백질 양을 정함은 HPLC 분석에 의해 달성되는 반면, 개별 H1 유전자의 mRNA는 정량적 실시간 PCR 다음에 임의의 프라이머 기반 역방향 전사로 측정됩니다.
초록
링커 히스톤 H1 높은 순서 구조로 염색질의 접이식 촉진, nucleosome 핵심 입자와 링커 DNA에 바인딩합니다. H1은 포유류의 개발 1 필수적이며, 생체내 2-4 특정 유전자 발현을 조절. 고도 보존 히스톤 단백질 중에서 H1의 링커 histones의 가족은 가장 이질적인 그룹입니다. differentially 개발되는 동안 서로 다른 세포 유형의 규제 포유류 11 H1의 subtypes가 있습니다. 이러한 H1의 subtypes 5 체세포 H1s (H1a-E), 교체 H1 0, 4 배아 세포 특정 H1의 subtypes 및 H1x 5 포함되어 있습니다. DNA와 결합 친화성 및 염색질 압축 능력 6-9에 차이가 여러 H1의 subtypes의 존재는 염색질 기능의 변조의 추가 레벨을 제공합니다. 따라서 mRNA와 단백질의 두 개별 H1의 subtypes의 양적 표현 분석은, 이상의 규정의 이해에 필요한주문 염색질 구조와 기능.
여기 개별 H1의 subtypes의 표현 수준 (그림 1) 분석을 위해 설계 assays 집합을 설명합니다. 다양한 H1 변종의 유전자의 mRNA 발현은보다 정확한 빠르고, 그리고 북부 얼룩 분석의 대체 접근법에 비해 훨씬 적은 샘플을 필요로 매우 민감하고 정량 역방향 전사-PCR (qRT-PCR) assays의 집합에 의해 측정됩니다. 대부분의 다른 세포 mRNA 메시지와는 달리, H1 유전자의 대부분을 포함하여 대부분의 히스톤 유전자, 대한 mRNAs는 오랜 polyA 꼬리 부족하지만, 3 '번역되지 않은 지역 (UTR) 10시 줄기 루프 구조를 포함합니다. 따라서 cDNAs 대신 oligo-DT의 primers의 무작위 primers를 사용하여 역방향 전사하여 총 RNA의 준비가되어 있습니다. 각 H1의 subtypes (표 1) 특정 primers와 실시간 PCR의 assays는 개별 H1를 나눠 봤는데요의 mRNA 수준의 고도의 양적 측정을 얻기 위해 수행되는의. 가사 유전자 표현은 정상화를위한 컨트롤로 분석된다.
각 H1를 나눠 봤는데요 및 핵심 histones의 단백질의 상대적인 풍요로움은 11-13 포유 동물 세포에서 추출한 총 histones의 반대 위상 고성능 액체 크로마 토그래피 (RP-HPLC) 분석을 통해 얻어진다. HPLC 방법 및 용출 조건은 여기 마우스 H1의 subtypes의 최적 분판을주는 설명했다. HPLC 프로필을 quantifying함으로써, 우리는뿐만 아니라 세포의 nucleosome 비율 H1을 결정으로, H1 가정 내에서 개별 H1의 subtypes의 상대적 비율을 계산.
프로토콜
1. 샘플 준비 및 RNA 추출
- RNA 추출하기 전에 모든 작업 표면과 pipettes은 70 % 에탄올로 닦아되어야 등의 RNase 보내겠 등의 RNase의 오염 제거 솔루션과 치료. 이러한 행위는 RNase의 오염 및 RNA 저하의 가능성을 줄여줍니다. 모든 절차를 위해 장갑을 착용한다.
- 마우스 조직에서 RNA를 추출하려면 euthanized 마우스에서 관심 장기를 해부하고, 얼음 추위 인산에 조직을 씻어 (PBS : 0.13 M NaCl, 5 밀리미터 인산 나트륨 이염의 heptahydrate, 5 밀리미터 나트륨 dihydrogen 인산의 heptahydrate, 산도 7.4) 염분 버퍼 . 단계 1.4에서 추출 RNA 즉시 진행합니다. 신선한 조직은 RNA 추출을 위해 처리될 수없는 경우 조직 샘플은 즉시 액화 질소에 냉동 스냅이어야하며 ° C에서 나중에 사용하기 위해 -80에 저장됩니다.
- RNA는 자기편 세포 배양에서 추출하고자한다면, 문화 미디어를 대기음 PBS의 충분한 양의와 린스, 그리고 (Invitro Trizol 시약을 추가플레이트쪽으로 세대)와 1.4 단계로 진행합니다. 정학 재배 세포의 경우 원심 분리하여 세포 및 펠렛 세포를 수확. 미디어를 삭제하시겠습니까 PBS와 함께 간략하게 알약을 린스, 그리고 펠렛 원심 분리로 세포. Trizol 시약을 추가하고 1.4 단계로 진행합니다.
- 충분한 Trizol 시약은 고품질의 RNA를 얻기 위해 필요합니다. 100 MG 조직, 5 - - 10 × 10 6 셀 (서스펜션 문화) 또는 3.5 ㎝ 판 당을 (자기편 문화) 50에서 RNA를 추출할 수 있도록 한 ML Trizol 시약을 사용합니다. Polytron PT2100 균 질기 (또는 동급)과 Trizol 시약의 조직을 Homogenize. Trizol 시약에 대한 제조 업체의 설명서 (Invitrogen)에 따라 조직 샘플이나 세포로부터 RNA를 추출할 수 있도록 진행합니다.
- RNA 농도는 Nanodrop 1000 (써모 과학)와 RNA 품질은 겔 전기 영동으로 분석됩니다를 사용하여 측정됩니다. 전형적인 항복 1 X 10 6 교양 세포 당 조직 혹은 RNA의 5-15 μg의 MG 당 RNA 중 1-10 μg의 범위. 를 제거하려면게놈 DNA의 흔적에서 RNA의 잠재적인 오염은 RNA 샘플은 제조 업체의 지시에 따라 RNase없는 DNase (시그마 AMP-D1)로 처리됩니다. 이 치료에서 RNA 전혀 저하를 보장 없다고 RNA 농도 결정 및 겔 전기 영동을 반복합니다. 저장소가 -80에서 RNA를 추출 ° C.
* 참고 : RNA는 또한 키트 설명서에 따라 RNAeasy 키트 (Qiagen)를 사용하여 추출되거나 DNA와 RNA 모두 원하는 경우의 DNA / RNA 키트 (Qiagen)에 의해.
2. 양적 역방향 스크립트 작성 PCR (qRT-PCR)
- 총 RNA는 반대로 cDNA를 사용하여 윗첨자 III 첫째 - 스트랜드 합성 시스템 (Invitrogen)에 베꼈는데됩니다. 대부분의 H1 유전자의 mRNA는 폴리-A-꼬리 부족하므로, cDNA 합성을위한 primers로 대신 oligo-DT의 임의 hexamers를 사용하는 것이 중요합니다. 그러나, 임의 hexamers 및 oligo-DT 수오의 낮은 레벨에서 polyadenylated 메시지와 유전자 표현 분석도 원하는 경우, 혼합물주민 등록증은 polyadenylated mRNAs의 역방향 전송 효율을 향상시키는 반전 전사 (RT) 반응에 사용되는.
- 제조 업체의 설명서에 따라 RT 반응을 수행합니다. 간단히,
- 0.5 ML PCR 튜브에 전체 RNA 5 μg, 50 NG / μl 무작위 hexamers 1 μl, 10 MM dNTP 믹스 1 μl을 결합, 10 μl 등 총 반응 부피를 만들기 위해 DEPC-처리된 H 2 O를 추가합니다. 잘 섞어서 65시 5 분 품어 ° 얼음 1 분 인큐베이션 다음 C를.
- cDNA 합성 믹스의 10 μl 준비 : 10xRT 버퍼, 25 MM 4 μl의 2 μl를 MgCl 2, 0.1M DTT, RNaseOut 1 μl (40 U / μl), 윗첨자 III RT (200 U / 1 μl 2 μl μl)하고 / 프라이머 혼합물을 RNA 추가.
- 25 10 분 동안 품어 50 ° C시 50 분 다음 5 분간 85 ° C에서 반응을 종료 ° C,.
- 각 반응은 일반적으로 100-250 NG / cDNA 제품의 μl를 얻을. -20에서 cDNA 제품을 저장° C 또는 실시간 정량 PCR (qPCR)를 즉시 수행합니다.
- qPCR 정확하게 높은 효율성과 재현성 14과 대상 시퀀스 복사본을 수량하실 수 있습니다. 우리는 qPCR은 이중 좌초된 DNA (dsDNA)로 intercalates에서만 형광 신호를 제공합니다 SYBR 녹색 염료에 의해 측정한 선택하십시오. Taqman 검정 14만큼 특정 아니지만,이 방법보다 비용 효과적이고, 쉽게 연구실에서 채택되는이며, qPCR에 더 많은 융통성을 제공합니다. 따라서 반응 효율과 특이성을 보장하기 위해 증폭 줄거리 (그림 2A) 및 qPCR 제품의 파생 녹는 곡선 (그림 2B)을 검토하는 것이 중요합니다.
| 히스톤 subtypes | 마우스 히스톤 명명법 | 인간 히스톤 명명법 | ||
| GENE 이름 | 가입 번호. | 유전자 이름 | 가입 번호. | |
| 히스톤 H1a | Hist1h1a | NM_030609 | HIST1H1A (H1.1) | NM_005325 |
| 히스톤 H1b | Hist1h1b | NM_020034 | HIST1H1B (H1.5) | NM_005322 |
| 히스톤 H1c | Hist1h1c | NM_015786 | HIST1H1C (H1.2) | NM_005319 |
| 히스톤 H1d | Hist1h1d | NM_145713 | HIST1H1D (H1.3) | NM_005320 |
| 히스톤 H1e | Hist1h1e | NM_015787 | HIST1H1E (H1.4) | NM_005321 |
| 히스톤 H1 ° | H1f0 | NM_008197 | H1F0 | NM_005318 |
| 히스톤 H1oo | H1foo | NM_183811 | H1FOO | NM_153833 |
| 히스톤 H1t | Hist1h1t | NM_010377 | HIST1H1T | NM_005323 |
| 히스톤 H1t2 | H1fnt | NM_027304 | H1FNT | NM_181788 |
| 히스톤 H1x | H1fx | NM_198622 | H1FX | NM_006026 |
| 히스톤 Hils1 | Hils1 | NM_081792 | HILS1 | AY286318 |
표 1. 마우스와 인간에서 히스톤 H1의 나눠 봤는데요의 명명법.
- 앞으로 디자인 및 각 H1 유전자 (표 1) 특정 PCR primers의 역방향. 특히 중앙 구형 도메인에 해당하는 지역의 체세포 H1s 사이에서 높은 서열 유사성으로 인해, 그것은 특정 H1를 나눠 봤는데요위한 primers는 O와 일치하지 않도록 중요군터 H1 유전자, 또는 교차 다른 H1의 subtypes를 증폭. 그것은 대부분의 H1 유전자가 introns가 포함되지 않도록주의하는 것도 중요하다. 따라서 일반적으로 게놈 오염을 피하기 위해 RT-PCR을 위해 채택 인트론 - 스패닝 primers 사용할 수 없습니다. 대신 RNA 샘플은 게놈 오염의 흔적을 제거하는 DNase (1.5 참조)으로 미리 다루어야하는 경우도 있습니다. 또한, RT (-)-qPCR은 cDNA 샘플의 게놈 오염의 부족을 검증하기 위해 병렬로 진행해야합니다.
- 표현 표본 간의 변경되지 않은 내부 레퍼런스 유전자에 대한 또한 디자인 primers. 종종 이러한 glyceraldehyde-3-인산염 탈수소 효소 (GAPDH)와 베타-actin 유전자와 같은 가사 유전자는, 기준 유전자로 선택됩니다. 가사 유전자 qPCR 신호는 정규화 컨트롤 역할을합니다.
- 2X IQ SYBR 녹색 Supermix (바이오 방사선) (포함하는 dNTPs, 50 U / ML iTaq 제품의 DNA 중합 효소, 6 밀리미터 MgCl 2, SYBR 녹색 나는 20의 12.5 μl : 다음과 같은 각각의 PCR 반응 (총 부피 25 μl)을 준비NM 플루오레신), 10 nm의 순방향 / 역방향 프라이머 믹스의 4, cDNA μl / NG 1.25 μl의 2 μl와 ddH 2 O의 9.25 μl, 그리고 Microseal 96 - 웰 PCR 플레이트에 잘 섞는다. 판 덮개 판을 밀봉되도록 Microseal 'B'접착제 씰 (바이오 방사선)를 사용합니다. 누르거나 짧게 와동 PCR 판, 그리고 짧은 원심 분리하여 반응 혼합물을 돌리다. qPCR에 대한 MyIQ 단일 색상 실시간 PCR 감지 시스템 (바이오 방사선)에 접시를 놓습니다.
- 30 초 동안 95 ° C 3 분 동안, 10 초 동안 95 ° C의 40주기에 이어, 60 ° C에서 20 초간, 72 ° C에서 : 우리는 다음 qPCR 조건을 사용합니다. PCR 효율과 CT (주기의 임계값) 값을 증폭 곡선 (그림 2A) 확인합니다. 임계값 라인이 자동으로 IQ5 광학 시스템 소프트웨어 버전 2.0으로 설정할 수 있습니다.
- 필요한 프라이머 효율성과 최적의 cDNA 농도는 표준 곡선 분석으로 테스트할 수있는 게놈 DNA의 직렬 희석 전qPCR와 CT 값에 사용되는 S '는 템플릿의 DNA 금액의 로그 역모를 꾸몄다됩니다. 높은 특이성과 효율성의 primers와 최적화된 qPCR 분석은 결정 계수 (R 2)> 0.98로, 선형 표준 곡선을 제공합니다. 가난한 증폭 효율을 가지고하는 경향이 200 BP, 이상 amplicon 길이 primers을 피하십시오.
- SYBR 녹색 어떤 dsDNA를 감지하므로 원하는 amplicon 아니지만 프라이머의 dimers이나 오염 물질이, 증폭과 감지 않도록 qPCR에 따라 용융 곡선 실행을 수행하는 것이 중요합니다. 용해 - 곡선 분석, 프로그램 내용은 qPCR 장비는 ° C ~ 95 ° C ~ 0.5 ° C 단위의 데이터 수집있는 55에서 샘플을 가열합니다. MyIQ (바이오 방사선)에 용해 곡선 분석의 기본 설정은 악기는 다음과 같습니다 95 ° C에서 1 분, 55 ° 설정점 55에서 10 초 81주기에 이어 1 분, C에 대한 ° C, 커브를 용융 + 임시직 0.5 ° C (카메라가 각 사이클에서 데이터를 수집.)
- 이후 dsDNA의 용융 온도 (TM)는 amplicon의 길이와 GC 함량에 따라 좌우가 다를 amplicons 다른 TM (들)을해야합니다. 원하는 amplicons의 특정 녹는 온도뿐만 아니라 소음 amplicon 봉우리 (그림 2B)의 부족을 확인하는 qPCR 제품의 파생 녹는 곡선을 검사합니다.
- 통계 분석을 위해 각 분석에 대한 반응을 복제하거나 세중의를 준비합니다. 이러한 RT와 같은 qRT-PCR에 대한 부정적인 컨트롤을 포함 (-)-qPCR (역방향 전사없이 템플릿으로 RNA와 qPCR)없이 cDNA, RNA 또는 PCR 믹스에 추가 DNA를 원본과 qPCR. RT (-) (- 그림 2 및 3) RT () - qPCR는 잠재적인 게놈 DNA의 오염에 대한 제어 역할을하실 수 있습니다.
- IQ5 광학 시스템 소프트웨어 버전 2.0 (바이오 방사선)로 qPCR 데이터를 분석. H1 유전자의 상대적인 표현 수준을 얻을 수있는 가사 유전자 (예 : GAPDH, SS-actin, HPRT)의 표현과 함께 H1 이소형 유전자의 표현 가치를 정상화.
* 모든 절차는 얼음 또는 4 ° C.에서 수행되어야합니다
- 마우스 조직을 해부하고 얼음 차가운 PBS로 헹굼. (즉시 추출을 진행하지 않으면 동결 스냅과 스텝 1.2에 설명된대로 샘플을 저장할 수 있습니다.) 면도날과 조각으로 조직을 말하다. 다운스 호모 지 나이저 (B 유봉)으로 말하다 전송합니다. 10 ML의 자당 버퍼를 (0.3 M의 자당, 15 MM NaCl, 10 밀리미터 HEPES [산도 7.9]은, 2 밀리미터 EDTA (에틸렌 다이아 민 테트라 초산), 0.5 MM PMSF, 완벽한 미니 프로 테아제 억제제 칵테일 정제는, 신선한 추가) 조직의 당 그램 추가합니다. 10-15 스트로크와 함께 조직을 Homogenize.
- 15 ML 튜브 30 초 (원심 분리기 모델 : Eppendorf 5810R) 500 rpm으로 스핀에 homogenates 전송, 신중하게 5 분 동안 2,000 rpm으로 새 튜브 (펠릿 - 조직 파편을 버리고)에 뜨는을 전송하고, 원심 분리기, 펠렛 세포 있습니다. 3.4 단계로 진행합니다.
- histones과 염색질은 단일층 재배 세포에서 추출하는 경우, 린스PBS와 함께 문화 접시에 PBS를 추가하고 원심 분리하여 세포 스크레이퍼, 그리고 펠렛 세포를 사용하여 세포를 수확. 원심 분리에 의한 정지 재배 세포 들어, 펠렛은 세포.
- 0.5 % NP-40 (g 조직 금액 또는 10 (8) 세포를 당)로 보충 10 ML의 자당 버퍼에 세포 펠렛을 Resuspend. 다운스 호모 지 나이저 (B 유봉)로 샘플을 전송 20 분 보육 내에 10 스트로크를 dounce. 이 시점에서 핵을 얻을 수 있습니다. 현미경 하에서 핵의 품질을 검사합니다. 5 분간 2,000 rpm으로 원심 분리하여 펠렛은 핵. 뜨는 폐기하십시오.
- 한 조직의 G 또는 10 (8) 셀 당 - 3 ML 높은 소금 버퍼 (각 사용하기 전에 신선한를 추가할 0.35 M KCl, 10 MM 트리스는 [산도 7.2], 5 밀리미터 MgCl 2, 0.5 MM PMSF가)에 핵 펠렛을 Resuspend. 작은 다운스 호모 지 나이저 (B 유봉)로 샘플을 전송하고 5-10 뇌졸중으로 homogenize.
- 이어서 20 분 동안 얼음에 품어 3 Eppendorf 튜브 (1 ML 각),로 나누어지는 현탁액을펠렛 염색질 ~ 10 분간 14,000 rpm으로 원심 분리. 뜨는 폐기하십시오.
- 각 염색질 펠렛으로 0.8 ML 0.2 NH 2 SO 4를 추가합니다. 펠렛 완전히 dissociated 때까지 잘 펠렛을 갈기 위해 eppendorf 튜브 유봉의 dounce을 사용합니다. 4 ° C에서 하룻밤 사이에 회전 플랫폼에서 샘플을 품어. 총 histones는 산성 치료의 단계로 압축이 풀립니다.
- 10 분 동안 14,000 rpm으로 원심 분리기. 이 Eppendorf 튜브 (400 μl / 튜브)에 뜨는 (히스톤 추출물)를 전송합니다. 펠렛 폐기하십시오. 각 튜브에 얼음 차가운 에탄올 2.5 권 (1 ML)를 추가 ... 샘플은 -20 ° C에서 하룻밤 사이에 유지.
- 펠렛 총 histones ~ 10 분 동안 14,000 rpm으로 원심 분리기, 뜨는 버리고. 70 % EtOH로 펠렛 세 번 씻어, 건조 공기하기 20-30분 동안 벤치에 둡니다. -80 ° C에서 말린 단백질을 저장하거나 ddH 2 O에서 그들을 해산하고 즉시 HPLC 분석을 수행합니다. 마른 단백질은 저장될 수-80 ° C에서 최대 1 년 간입니다.
4. 링커 Histones의 HPLC 분석
- 리버스 상 칼럼과 HPLC 기기의 용량에 따라 ddH 2 O의 권장 볼륨에 히스톤 펠렛을 Resuspend. 우리는 HPLC 분석에 C18 역방향 위상 칼럼 250 X 4.6 mm (Vydac)와 Äktapurifier UPC 900 악기 (GE 헬스케어)을 사용합니다. 우리는 일반적으로 분석을위한 ddH 2 0 100 μl에 총 histones의 50-100μg를 해산.
- 불용성 잔류물을 제거하기 위해 5 분간 14,000 rpm으로 원심 분리기. 브래드 포드 단백질 분석은 컬럼에 주입되는 단백질의 양을 결정하는 데 사용됩니다. HPLC 시스템에서 역방향 위상 열에 총 단백질 50-100 μg을 주사. 단백질 로딩 초과 금액은 칼럼의 막힘을 방지하기 위해 피해야한다.
- 분류 증가 acetonitrile 기울기와 링커 histones 및 핵심 histones는 표 2에 나열.
표 2. 시간이 지남에 acetonitrile 기울기 증가.
- 유출물은 (Figu 214 NM에서 모니터링하고, HPLC 프로파일된다다시 4) 기록 및 유니콘 5.11 소프트웨어 (GE 헬스케어)로 Äktapurifier UPC 900 (GE 헬스케어)을 사용하여 분석하고 있습니다. 자세한 분석을 위해, 예를 들어 SDS-PAGE와 질량 분석법 - 단백질 분수는 분수 자동 수집기 (GE Frac-920)로 회수 할 수 있습니다.
- 각 H1를 나눠 봤는데요 및 H2B의 봉우리 214 값은 각각 대응하는 히스톤 단백질의 펩티드 채권의 숫자로 정규화됩니다. 개별 H1 히스톤 H1 가족 내에서 subtypes뿐만 아니라, nucleosome 코어 입자에 H1의 subtypes의 비율의 상대적인 비율은 (그림 5) 214 값이 표준으로부터 계산하실 수 있습니다.
5. 대표 결과
포유류의 H1의 subtypes, 전반적인 흐름도 및 개별 히스톤 H1 유전자의 표현 분석의 대표적인 결과의 목록은 각각 표 1, 그림 1과 그림 2-5에 표시됩니다.그림 2A를 사용 H1a qPCR 반응의 전형적인 증폭 곡선을 보여줍니다 cDNA 그림 2B는 해당 amplicons의 파생 녹는 곡선을 보여주는 반면, 마우스 간 및 mESCs에서 준비했습니다. 용해 곡선 86시 용융 온도 (TM)에서 하나의 특징적인 피크 ° H1a PCR amplicon에 대한 C를 표시하고 H1a qPCR 분석의 높은 특이성을 제안 아닌 구체적인 배경 봉우리가 부족한 형편입니다. 증폭 줄거리 (그림 2A)의 평가는 각 시료의 세중의 제품 qPCR 반응이 높은 재현성을 제안, 거의 동일한 CT 값으로 일관된 신호를 준 것으로 나타났습니다. 빌드 - 업 RT에서 amplicons의 부족 (-)-qPCR 반응은 게놈 DNA의 오염이 존재하거나 최소한 아니었 나타냅니다. 같은 GAPDH 같은 H1 유전자와 가사 유전자의 CT 값을 활용하여 각 H1 유전자의 상대 RNA 표현 수준이 계산되었다. H1 ° 및 H1a의 유전자에 대한 계산 결과의 예는 다음이 표시됩니다 그림 3. H1 ° 표현 mESCs보다 간장에 훨씬 더 높습니다 반면 H1a의 mRNA의 상대적인 표현 수준은, 마우스 간 비해 mESCs의 상위입니다.
mESC 대 성인 마우스 간장의 H1a 또는 H1 °의 표현의 차이는 히스톤 단백질의 HPLC 프로파일 (그림 4)에서 나타난다. H1 °, 차별화 특정 H1은 성인 간 총 H1 (그림 5A)의 27.2 %를 차지하고, 성숙한 조직에 다량으로 축적됩니다. 반대로, H1 ° 단백질 undifferentiated mESCs (그림 4B)에서 거의 결석입니다. 한편, H1a는 높은 mESCs (그림 3 & 4B)에서 mRNA의 성적 증명서 및 단백질에 둘 다 표시됩니다. HPLC 프로파일에서 H1 봉우리의 부량 통해 H1 가족 내의 개별 H1를 나눠 봤는데요의 상대적 비중이 (그림 5A) 결정됩니다. 또한, 개별 H1를 나눠 봤는데요의 값 (또는nucleosome 당 총 H1)는 H2B위한 214 값 (그림 5B) 표준의 절반에 해당하는 H1의 subtypes (또는 총 H1의 합계)의 표준 214 피크 값의 비율을 계산하실 수 있습니다.
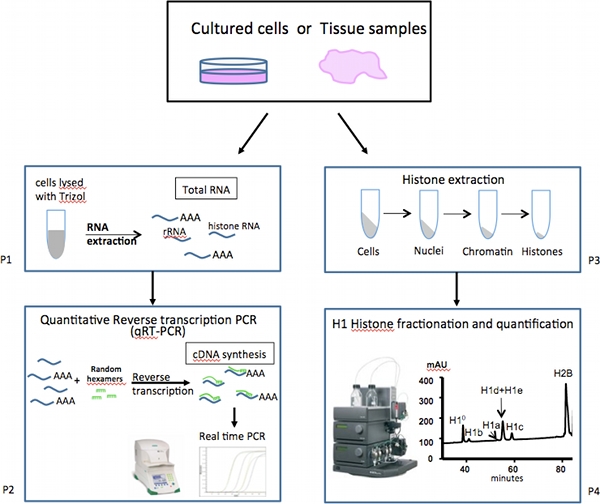
그림 1. 포유류 링커 - 히스톤 subtypes의 표현 분석의 전체 구조.
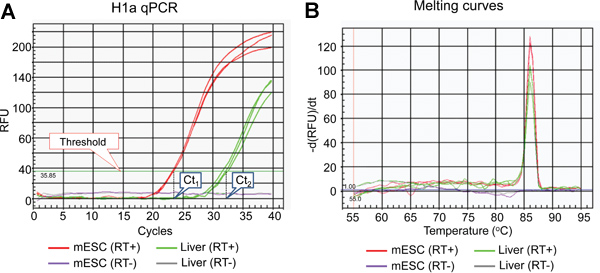
그림 2. H1a qPCR 분석의 대표 결과입니다. H1a qPCR 분석의 (A) 증폭 플롯. 임계값 라인과 IQ5 광학 시스템 소프트웨어에 의해 설정 CT 값이 표시됩니다. (B) qPCR 제품의 파생 녹기-곡선은 ()에 표시됩니다.
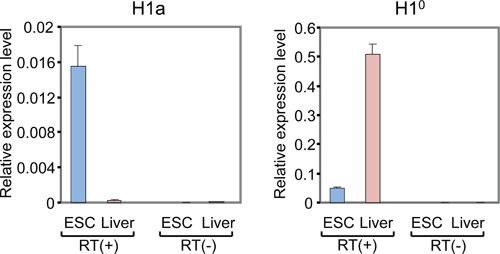
mESCs 성인 손에 H1a과 H1 °의 mRNA 수준의 그림 3. qRT-PCR 분석간을 ouse. Y 축은 참조 유전자 GAPDH의에 H1 유전자의 상대적인 표현 수준을 나타냅니다. (-) RT와 qPCR 샘플 (역방향 전사없이 RNA) 최소 없거나 전혀없는 신호를 보여줍니다.
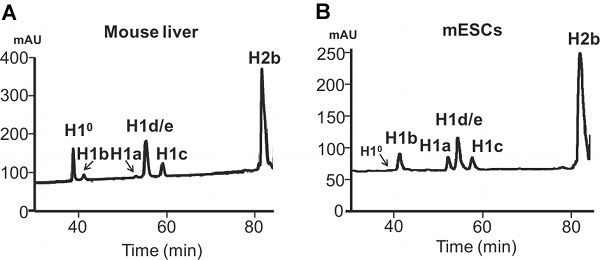
포유류 세포에서 추출한 histones의 그림 4. HPLC 분석. 성인용 마우스 간 ()와 마우스 ESCs (B)에서 추출한 100 μg 총 histones의 리버스 상 HPLC 분석. X 축 : 용리 시간. Y 축 : mAU, 밀리 흡수 장치.
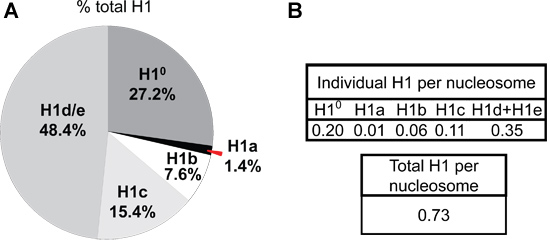
그림 5. 성인 마우스 간장의 nucleosome 비율 당 H1를 나눠 봤는데요 조성 및 H1. 각 H1 이소형 및 H2B에 대한 피크 면적의 214 값을 유니콘 5.11 소프트웨어 (GE 헬스케어)을 사용하여 계산하고, 해당 히스톤 단백질에 존재하는 펩티드 채권의 숫자로 정규화됩니다. 정규의 합계모든 H1의 subtypes의 214 값이 총 H1에 대한 값으로 얻어진다. 각 H1를 나눠 봤는데요에 대한 전체 H1의 비율 ()뿐만 아니라, nucleosome에 H1의 비율은 (H2B의 214 값의 표준의 절반으로 표시) (B) 성인 마우스 간에 표시된 HPLC 프로파일로부터 계산 그림 4A 인치
토론
assays의 집합은 포유 동물 링커 히스톤 subtypes의 표현 수준의 포괄적인 분석을 가능하게 여기 제시. 적절히 설계된 qRT-PCR의 assays는 어떤 포유 동물 히스톤 H1 유전자의 RNA 메시지 중 매우 민감하고 정확한 측정을 제공합니다. 링커 히스톤 나눠 봤는데요의 유전자에 대해 qRT-PCR assays의 중요한 부분은 반대로 전사를 바탕으로 임의의 프라이머를 사용하여 cDNA의 준비입니다. 대부분의 H1 유전자를 포함?...
공개
관심의 어떠한 충돌 선언 없습니다.
감사의 말
이 작품은 NIH 교부금 GM085261와 조지아 암 연합 내외 학술 상 (YF까지)에 의해 지원됩니다.
자료
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 | |
| RNase 보내겠 | 응용 Biosystems | AM9780 | |
| Trizol 시약 | Invitrogen | 15596-018 | |
| SuperScriptIII | Invitrogen | 18080-051 | |
| 절대 에탄올 | 피셔 과학 | BP2818-4 | |
| IQ SYBR 녹색 | 바이오 방사선 | 170-8880 | |
| RNeasy 미니 키트 | Qiagen | 74,104 | |
| Deoxyribonuclease 전 | 시그마 | AMP-D1 | |
| Microseal 96 - 웰 PCR 플레이트 | 바이오 방사선 | MSP-9605 | |
| Microseal 'B'씰 접착제 | 바이오 방사선 | MSB-1001 | |
| 자당 | Agros의 생명체 | AC40594 | |
| 인산 나트륨 이염의 heptahydrate (나 2 HPO 4 · 7H 2 O) | 피셔 과학 | BP332 | |
| 나트륨 염화물 (NaCl) | Bioanalytical 미국 | AB01915 | |
| 나트륨 dihydrogen 인산의 heptahydrate (아니 2 PO 4 · 7H2O) | 피셔 과학 | BP-330 | |
| HEPES | 피셔 과학 | BP310 | |
| 전체 미니 proteinase 억제제 칵테일 정제 | 로슈 응용 과학 | 11836153001 | |
| EDTA (에틸렌 다이아 민 테트라 초산) | 시그마 | E-5134 | |
| Phenylmethanesulfonyl 플루오르 (PMSF) | Bioanalytical 미국 | AB01620 | |
| Nonidet-40 (NP-40) | Bioanalytical 미국 | AB01425 | |
| 염화칼륨 (KCl) | 피셔 과학 | BP366 | |
| 트리스는 [히드록시 메틸 aminomethane] | Bioanalytical 미국 | AB02000 | |
| 마그네슘 염화물 (MgCl2) | 피셔 과학 | BP214 | |
| 황산 (H2SO4) | VWR | VW3648-3 | |
| 암모늄 수산화물 (NH 4 OH) | Agros의 생명체 | AC42330 | |
| 브래드 포드 단백질 분석 | 바이오 방사선 | 500-0001 | |
| Acetonitrile | EMD | AX0145-1 | |
| Trifluoroacetic 산성 (TFA) | JTBaker | 9470-01 |
참고문헌
- Fan, Y. H1 linker histones are essential for mouse development and affect nucleosome spacing in vivo. Mol. Cell Biol. 23, 4559-4572 (2003).
- Woodcock, C. L., Skoultchi, A. I., Fan, Y. Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17-25 (2006).
- Shen, X., Gorovsky, M. A. Linker histone H1 regulates specific gene expression but not global transcription in vivo. Cell. 86, 475-483 (1996).
- Alami, R. Mammalian linker-histone subtypes differentially affect gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 100, 5920-5925 (2003).
- Happel, N., Doenecke, D. Histone H1 and its isoforms: contribution to chromatin structure and function. Gene. 431, 1-12 (2009).
- Clausell, J., Happel, N., Hale, T. K., Doenecke, D., Beato, M. Histone H1 subtypes differentially modulate chromatin condensation without preventing ATP-dependent remodeling by SWI/SNF or NURF. PLoS One. 4, 0007243-0007243 (2009).
- Khadake, J. R., Rao, M. R. DNA- chromatin-condensing properties of rat testes H1a and H1t compared to those of rat liver H1bdec; H1t is a poor condenser of chromatin. Biochemistry. 34, 15792-15801 (1995).
- Orrego, M. Differential affinity of mammalian histone H1 somatic subtypes for DNA and chromatin. BMC Biol. 5, 22-22 (2007).
- Th'ng, J. P., Sung, R., Ye, M., Hendzel, M. J. H1 family histones in the nucleus. Control of binding and localization by the C-terminal domain. J. Biol. Chem. 280, 27809-27814 (2005).
- Wang, Z. F., Sirotkin, A. M., Buchold, G. M., Skoultchi, A. I., Marzluff, W. F. The mouse histone H1 genes: gene organization and differential regulation. J. Mol. Biol. 271, 124-138 (1997).
- Brown, D. T., Sittman, D. B. Identification through overexpression and tagging of the variant type of the mouse H1e and H1c genes. J. Biol. Chem. 268, 713-718 (1993).
- Fan, Y., Sirotkin, A., Russell, R. G., Ayala, J., Skoultchi, A. I. Individual somatic H1 subtypes are dispensable for mouse development even in mice lacking the H1(0) replacement subtype. Mol. Cell. Biol. 21, 7933-7943 (2001).
- Fan, Y., Skoultchi, A. I. Genetic analysis of H1 linker histone subtypes and their functions in mice. Methods Enzymol. 377, 85-107 (2004).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Res. 6, 986-994 (1996).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유