JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
단일 Oocyte의 Bisulfite의 돌연변이 유발
요약
Bisulfite의 돌연변이 유발이의 DNA methylation 분석을위한 황금 표준입니다. 우리 수정된 프로토콜은 단일 세포 수준에서 DNA의 methylation 분석을 허용하고 특별히 개별 oocytes를 위해 설계되었습니다. 또한 절단 단 배아 사용할 수 있습니다.
초록
Epigenetics는 모든 물려 전할 수있는 포괄하고 가역 개조 따라서 신 유전자 접근하고,이 유전자 전사 1 규제의 기본 메커니즘임을 염색질합니다. DNA의 methylation은 억압 마르크로 주로 역할을 epigenetic 변형입니다. CPG dinucleotides의 cytosines 향해 메틸 그룹의 공유 결합 외에도 통해 그것은 염색질을 응축하고 유전자에게 2를 입을 관련된 프로세스를 시작하기 위해 추가적인 억압 단백질과 히스톤 수정을 모집할 수 있습니다. 그것이 발달 프로그래밍, 세포 분화, retroviral 요소의 억압, X 염색체 불활 성화 및 게놈 각인에 중요한 역할을 담당으로 DNA의 methylation은 정상 개발을 위해 필수적입니다.
의 DNA methylation 분석을위한 가장 강력한 방법 중 하나는 bisulfite의 돌연변이 유발됩니다. 나트륨 bisulfite은 uracils로 cytosines을 deaminates의 DNA mutagen입니다. PCR 증폭 및 seque 따라 ncing는 이러한 변환 이벤트가 thymines으로 감지됩니다. Methylated cytosines는 deamination로부터 보호하기 때문에 각각의 뉴클레오 티드 수준 3에서 DNA의 methylation의 식별을 가능하게, cytosines로 남아있다. bisulfite의 돌연변이 유발 분석의 개발은 원래보다 민감한 및 7 재현할 수있는 것들을 향해 4-6을보고 그에서 고급했다. 하나의 주요 발전은이를 거친 bisulfite 처리 8 일부터 DNA를 보호, 아가로 오스 비드에 DNA의 작은 금액을 삽입했습니다. 이 활성화된 methylation 분석은 oocytes와 blastocyst 단계의 배아 9 수영장에서 수행해야합니다. 날짜까지 가장 정교한 bisulfite의 돌연변이 유발 프로토콜은 개별 blastocyst 단계의 배아 10입니다. blastocysts은 평균 64 셀 (게놈의 DNA 120-720 PG를 포함하는)을 가지고 있기 때문에 그러나이 방법은 개별 oocytes 또는 절단 단 배아의 methylation 연구를위한 효과되지 않습니다. 십t "> oocytes 11 포함해 분의 DNA 양의 아가로 오스의 삽입에서 단서를 타고, 우리가 oocytes 직접 아가로 오스와 용해 용액 비드 후 즉시 검색하고 oocyte에서 조나 pellucida의 제거에 포함된 약자 방법을 제시한다. 이것은 우리에게 수 두 개의 단일 oocyte의 bisulfite의 돌연변이 유발의 주요 과제 우회 :. 열화로부터 DNA를 분 양을 보호하고, 다양한 프로토콜 단계 동안 후속 손실 데이터가 단일 oocytes에서 얻은 때 중요한 것은,, 수영장 이내 PCR 바이어스의 문제가 제거되고 더 나아가. 하나 이상 methylation 패턴과 아무런 샘플이 분석 12 대상에서 제외되므로 부주의 적운 세포 오염.이 방법에 의해 감지가이 프로토콜은 단일 세포 수준에서 DNA의 methylation의 성공과 재현성 분석을위한 향상된 방법을 제공하고 이상적으로 적합합니다 개별 oocytes뿐만 아니라 절단 - 무대 배아입니다.
프로토콜
1 일
이러한 GIBCO 물 등의 살균, 증류수로 oocyte 컬렉션 당일 신선한 다음과 같은 솔루션을 준비합니다. 유전자 오염의 가능성을 줄이기 위해 종종 장갑을 변경하고 필터 팁을 사용합니다. 언제 오픈 튜브 멀리 각도 유지하고, 사용하지 않을 때 모든 튜브를 요점을 되풀이하다. 우리는 솔루션 N +1로 만들어진 것이 좋습니다.
3퍼센트 LMP 아가로 오스
30 MG 낮은 융점 (LMP) 아가로 오스
최대 1 ML GIBCO H 2 O로
@ 70 용해 ° C
용해 용액
8 μl 용해 완충액
1 μl proteinase K
1 μl 10 % IGEPAL
사용할 준비까지 얼음 곳
2시 1분 아가로 오스 : 용해 용액은 (개인 oocyte 당 10 μl, 금액은 3 oocytes입니다)
20 μl 3퍼센트 LMP 아가로 오스
10 μl 용해 용액
@ 70 섞는다 ° C
SDS의 LY시스 버퍼 (개인 oocyte 당 501 μl)
| 1X TE 산도 7.5 | 450 μl |
| 10% SDS | 50 μl |
| Proteinase K | 1 μl |
| 501 μl |
1. Oocyte 컬렉션
- M2 미디어 해부 마우스 oviducts를 삽입하고, 적운 세포 단지를 추출 ampullae 찢어.
- 0.3 MG / M2 미디어 30 μl 방울의 ML hyaluronidase 용액을 사용하여 적운 전지 복잡한에서 oocytes를 분리합니다. 오직 한 긴 노출 그들을 손상시킬 수 있으므로 그것이, 적운 세포를 제거하는 데 걸리는로 솔루션 oocytes 보관하십시오. 주기적으로 적운 세포를 제거하는 M2 미디어의 30 μl 방울의 3 배 oocytes를 씻으십시오.
- 산성 tyrode의 솔루션을 사용하여 조나 pellucida를 제거합니다. 또 다른 30 전송할 처음엔 솔루션 중 하나를 30 μl 드롭에 oocytes를 삽입하고,μl 드롭으로 함께 수행하는 미디어는 산성을 희석하고 효율성을 감소시킵니다. 오직 한가 긴 노출 그들을 손상시킬 수 있으므로 조나을 제거하는 데 걸리는로 솔루션 oocytes 보관하십시오. 참고 : 인간 조나 pellucida가 마우스보다 산성 tyrode의 솔루션과 치료에 더 저항이기 때문에 산성 tyrode의 솔루션이나 pronase의 증가 농도는, 인간의 샘플에 사용할 수 있습니다.
- 한 번 더 M2 미디어 30 μl 드롭에 oocytes 씻으십시오.
2. 아가로 오스의 임베딩 및 용해
- 아가로 오스의 삽입을 수행하려면 70 ° C heatblock에 용해 용액을 넣으십시오. 용해 용액 : 2시 1분 아가로 오스를 생산 용해 용액에 preheated LMP 아가로 오스를 추가합니다.
- 최소한의 M2 미디어 깨끗한 유리 슬라이드를 향해 하나의 oocyte를 놓습니다. 피펫 팁에 용해 용액, 그리고 (현미경으로) 부드럽게 그것 미님와 혼합 수 있도록 유리 슬라이드에 (~ 1 μl 이하) 소량을 추방 : 아가로 오스의 10 μl를 타고알 미디어. 부드럽게 피펫 팁에 oocyte를 데리러와 구슬이 영역을 형성하므로 300 μl 광유와 Eppendorf 튜브에 모두 10 μl 넣어.
참고 :이 프로세스는 아가로 오스로 비교적 빨리 할 수 있어야합니다 단단해 온도는 빠르면 5와 ° C ~ 70 ° C. 아래로 떨어뜨리면 - 10 분 동안 얼음 튜브를 품어. 용해를 수행하려면 300 μl 미네랄 오일을 제거하고 SDS의 용해 완충액 500 μl를 추가합니다. 50 ° C의 물을 욕조에 밤새 품어.
참고 : 용해 용액 (표 1) 또한이 목적을 위해 사용될 수 있습니다.
제 2 일
bisulfite의 돌연변이 유발 당일 신선한 다음과 같은 솔루션을 준비합니다. 유전자 오염의 가능성을 줄이기 위해 종종 장갑을 변경하고 필터 팁을 사용합니다. 언제 오픈 튜브 멀리 각도 유지하고, 사용하지 않을 때 모든 튜브를 요점을 되풀이하다. 우리는 솔루션 N +1로 만들어진 것이 좋습니다.
2| 3 M NaOH | |
| 0.1 M NaOH | 14.5 ML autoclaved ddH 2 O의 3M 0.5 ML |
| 0.3 M NaOH | 13.5 ML autoclaved ddH 2 O에있는 3M의 1.5 ML |
2.5 M의 Bisulfite 솔루션
- 3.8 g의 나트륨 bisulfite
5.5 ML GIBCO 증류수 H 2 O
한 ML 3 M NaOH
@ 실내 온도를 해산 - 110 MG의 히드로퀴논
한 ML GIBCO 증류수 H 2 O
@ 90 ° C (오직 한 녹이신 수만 위해 정기적으로 믹스)를 용해
완전히 용해되면 용액 ()을 섞어 및 (b)
* 빛 * 저리
3. Bisulfite의 돌연변이 유발
- 완전 500 μl SDS의 용해 버퍼를 제거하고 300 μl 광유 (~ 20 시간) 추가합니다. 나머지 모든 용해 버퍼 일을 희석합니다E 아가로 오스가 가열되고 비드가 후속 단계에서 용해에 더 민감한 메시지가 나타납니다. 즉시 bisulfite의 돌연변이 유발을 진행, 또는 최대 5 일 대한 -20 ° C에서 저장합니다.
- 해당되는 경우, 냉동고에서 oocytes를 제거하고 (아가로 오스 비드가 상대적으로 반투명에만 때까지) 해동 봅시다. 10 분간 얼음에있는 품어 따라 90 ° C 열 구역 2.5 분 동안 품어.
참고 : 혼합이나 볶음, 이상 2.5 분 연장하거나, 온도 변동하지 마십시오. - 변성을 수행하려면 미네랄 오일을 제거하고 각 튜브, 영화 1 ML 0.1M NaOH를 추가하고 5-6 시간을 바꾸란.
- 반전 37 ° C waterbath에서 15 분, 매 3-4분 동안 품어. 비드는 NaOH에 부유한다.
- bisulfite 처리를 수행하려면 부드럽게 튜브를 돌려서 다음, NaOH를 제거하고 300 μl 광유와 500 μl bisulfite 솔루션을 추가합니다. 50 ° C waterbath에서 3.5 시간 동안 튜브를 품어. * 빛 * 저리
참고 : 부화 기간은 경험적으로 관심의 유전자에 대해 결정 할 수 있습니다. - desulfonation를 수행하려면 3 분 동안 얼음에 품어, 그럼, 미네랄 오일과 bisulfite 용액을 제거 부드럽게 돌려서, 1 ML 0.3 M NaOH를 추가합니다. 톡하고 5-6 시간을 바꾸란.
- 반전 37 ° C waterbath에서 15 분, 매 3-4분 동안 품어. 비드는 NaOH에 부유한다.
- 샘플을 씻으 먼저 부드럽게 회전하여 다음 NaOH를 제거하고 1 ML 1X TE 산도 7.5를 추가합니다. 실온 (뿌리에서)에서 5~10분 동안 흔들어. 다시 부드럽게 돌려 다음, 1X TE를 제거합니다. 두번 세탁 과정을 반복합니다.
- 한 ML autoclaved ddH 2 O에 추가 실온 (뿌리에서)에서 5~10분 동안 흔들어. 부드럽게 돌려 후 H 2 O를 제거 두번 ddH 2 O 세척을 반복합니다.
- 뜨는의 산도를 확인, 그것은 산도 5.0이어야합니다. 그래도 너무 기본적인면, H 2 O를하고 다시 씻어 오직 아가로 오스 될 떠나 모든 뜨는 제거광고.
4. 1 일 및 2 차 라운드 PCR 증폭
- **을 씻는 동안에 1 일 왕복 PCR 믹스 ** 준비
| 앞으로 외부 10 μm의 프라이머 | 0.5 μl |
| 외부 10 μm의 프라이머의 후진 | 0.5 μl |
| 240 NG / ML tRNA | 1 μl |
| H 2 O | 13 μl |
Illustra 레디 - 투 - 이동 핫 스타트 PCR 비즈에 추가
조심스럽게 PCR 튜브 (~ 10 μl)로 고체 아가로 오스 비드 밀어
70 ° C와 믹스에 대한 열
25 μl 광유 추가
총 : 50 μl
- 증폭하다
참고 : 마우스 Snrpn위한 사이클링 조건 예제 94에서 2 분간 변성은 94에서 30 초 40주기에 이어 ° C, ° C, 50 ° C에서 1 분, 168시 분 ° C 및 68에서 마지막 십분 연신율 단계 ° C. 1 일 원형 마우스 H19을위한 PCR 및 Peg3 위해 온도를 어닐링하는 것은 50 ° C.이다 - 제 2 라운드 PCR 혼합물 준비
| 앞으로 내부 10 μm의 프라이머 | 0.5 μl |
| 내부 10 μm의 프라이머의 후진 | 0.5 μl |
| H 2 O | 19 μl |
Illustra 레디 - 투 - 이동 핫 스타트 PCR 비즈에 추가
템플릿으로 5 μl 제 1 라운드의 제품을 추가합니다. ° C에서 1 분 아가로 오스을 약화시키기 위해 70까지 1 일 왕복 제품을 가열. 미네랄 오일 층 아래 피펫해야합니다.
25 μl 광유 추가
총 : 50 μl
참고 : Snrpn, H19, 그리고 Peg3위한 중첩된 프라이머 시퀀스는 시장 Velker 외 10에서 찾을 수 있습니다,12.
- 증폭하다
참고 : 사이클링 조건 마우스 Snrpn위한 94에서 2 분간 변성있다 ° 94에서 30 초 40주기에 이어 C, ° C, 50 ° C에서 1 분, 그리고 68시 일분 ° C, 그리고 마지막 10 분 이격 68에서 단계 ° C에서 10. 마우스 H19과 Peg3이 필요하다 50 ° C 제 2 라운드 PCR을 위해 온도를 풀림. - 진단 테스트로 패하면서 샘플 methylation 또는 스트레인에만 제한 효소로 절단 할 수 있습니다.
| 제 2의 라운드 제품 | 4 μl |
| 제한 효소 | 1 μl |
| 버퍼 | 1 μl |
| H 2 O | 4 μl |
- 8 % 아크릴 아미드 겔에 소화 제품을 Electrophorese. 모든 이기종 밴드 하나 이상의 seque를 나타냅니다제정신.
5. TA 클로닝 및 콜로니 PCR
- 제 2 라운드 제품, 70 먼저 열을 복제 ° C에서 1 분 Promega pGEM-T 벡터 시스템 (피셔 과학 고양이 # A1360)를 사용하여 벡터로 다음 ligate 아가로 오스를 부드럽게합니다.
| 제 2 라운드 PCR | 1 μl |
| pGEMT 쉬운 벡터 | 1 μl |
| Ligase | 1 μl |
| H 2 O | 2 μl |
| 2X 내고 버퍼 | 5 μl |
하룻밤 @ 4 ° C에서 PCR 기계를 품어.
- 15 분 동안 얼음 유능한 E.coli 세포 (Zymo 리서치 사의 고양이 # T3009)를 녹여. 8 μl E.coli와 15 분 동안 얼음에 부화를 내고 3 μl 내고 반응을 추가합니다.
- 42 ° C waterbath 40 초간 열 충격,2 분 동안 얼음에 품어. 1 시간 (뿌리)를위한 37 ° C에서 60 μl SOC 매체와 부화를 추가합니다.
- 37 ° C 하룻밤에 LB / 한천 / IPTG / Xgal / 앰프 강판 및 품어 접시에 반응 혼합물을 모두 놓습니다.
- 식민지 PCR 믹스를 준비
| 20 μm의 M13 앞으로의 프라이머 | 0.7 μl |
| 20 μm의 M13 역방향 프라이머 | 0.7 μl |
| 5 배 그린 바둑 DNA 형성 촉매 버퍼 | 7.0 μl |
| 10 MM dNTP | 0.7 μl |
| DNA 형성 촉매 장치의 DNA 중합 효소 | 0.28 μl |
| H 2 O | 25.62 μl |
| 35 μl 총 |
PCR 튜브에 35 μl 콜로니 PCR 마스터 믹스를 추가합니다. 피펫 팁, 그리고 PCR 반응에 소용돌이를 함께 접시에서 하얀 세균성 식민지를 선택하십시오.
- 94시 10 분 변성으로 확대 ° 94 45 초 30주기에 이어 C, ° 57에서 C 30 초 ° C, 72시 일분 ° C, 72에서 마지막 10 분 연신율 단계 ° C. 1.5 % 아가로 오스 겔에 4 μl를 Electrophorese. 시퀀싱을위한 PCR 제품의 ~ 30 μl을 보냅니다.
참고 : oocytes의 경우, 5 콜로니 PCR 제품은 순서가있다. - 시퀀싱 결과가 획득되면, methylation 패턴을 읽을 수 있습니다. CG로 남아 모든 원본 CG는 methylated했다, 이제 TG 존재하는 어떤 원래 CG가 unmethylated되었다.
6. 대표 결과
우리 연구에서는 분석은 개별 oocytes과 배아 (그림 1)에서 methylation을 각인. bisulfite 변환 primers를 사용하여 중첩된 PCR 증폭에 따라 그것은 아가로 오스 겔 (그림 2)에서 올바른 조각 크기를 시각화하여 성공적인 전환을 확인할 수 있습니다. 개별 oocyte가한 부모의 allele을 대표하고, 이론적으로는, 각인 methylation 패턴을 가지고 있습니다. 따라서 패하면서 PCR 제품은 무심코 오염에 대한 검사를 할 수 있습니다. 의 DNA methylation (예 : HinfI 또는 DpnII 등)에 민감한 제한 효소는 그것이 methylated 또는 unmethylated allele (그림 3)가 포함되어 있는지 여부를 평가하기 위해 두 번째 라운드 PCR 제품을 소화하는 데 사용할 수 있습니다. T로 변환된다 unmethylated C가 더 이상 효소에 의해 인식되지 않으며 포경가되는 동안 효소 인식 시퀀스 내에서 methylated C는 죽습됩니다. 그것이 적운 세포 오염 (그림 3)는 지표이기 때문에 methylated 및 unmethylated 대립 유전자 모두를 포함하는 모든 MII의 oocyte 샘플은 폐기되어야한다. 결합 및 변형에 이어 성공적인 식민지 PCR 증폭은 올바른 제품 크기 샘플 (그림 4) 시퀀싱을위한 메일이 발송되었습니다 보장하기 위해 아가로 오스 겔에 시각이 될 수 있습니다. 마지막으로, 다섯 개인 CL의 순서MII의 oocyte에서 사람 다섯 동일한 methylation 패턴과 동일한 nonCpG 전환율 (그림 5A)을 생산한다. 하나 이상의 패턴을 포함하는 모든 샘플 (그림 5B) 폐기되어야합니다. ovulated MII의 oocytes 두 염색체 사본 또는 첨부 극지 몸을 갖고 있기 때문에 두 개의 유사한 시퀀스 패턴 (그림 5c)를 취득 가능성이 있습니다. 우리는 적운 셀 오염을 배제할 수 없다 때문에 고도 ...와 비슷하지 않은 methylation 패턴을 가지고 oocytes에서 폐기 데이터를 권해드립니다.

그림 1. 단일 Oocyte의 Bisulfite의 돌연변이 유발 분석의 도식.
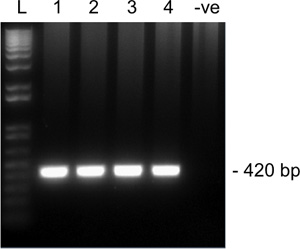
노래에서 Snrpn을위한 제 2 라운드 증폭의 그림 2. 대표 결과1.5 % 아가로 오스 겔에 르 MII의 oocyte. 레인 1-4 네 개의 싱글 MII의 oocytes이며 차선 5는 부정적인 컨트롤 (없음 oocyte)입니다. Snrpn에 대한 예상 amplicon 크기는 420 BP이다. L, 사다리.
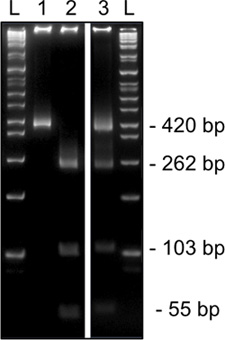
8 % 아크릴 아미드 겔의 한 MII의 oocyte에서 Snrpn을위한 제 2 라운드 methylation에 특정한 제한 소화의 그림 3. 대표 결과입니다. HinfI 진단 제한 소화 (420bp, 차선 1) 제한 사이트를 abolishes T를 비호하는 unmethylated DNA를 보여줍니다 또는 (잘라내기, 262, 103, 54 BP, 차선 2) 인식 사이트 내에서 C를 포함하고 methylated DNA. methylated 및 unmethylated 두 제한 효소 사이트를 (절단 및 포경 밴드, 차선 3) 보여주는 소화 적운 세포 오염의 지표. L, 사다리.
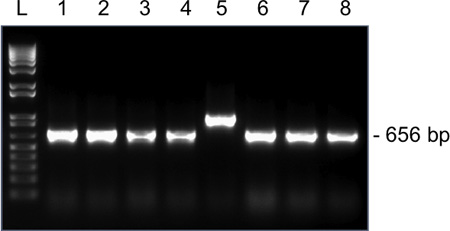
4 그림. 1.5 % 아가로 오스 겔에 대한 하나의 MII의 oocyte에서 Snrpn에 대한 식민지 PCR 증폭을위한 대표 결과입니다. 예상 amplicon 크기는 pGEM-T 쉬운 벡터로 Snrpn의 결합에 따라 앞으로 M13을 사용하고 역방향 primers 것은 656 BP이다. 레인 1-8, 클론 1-8에서 amplicons. 클론 5 잘못된 amplicon 크기를 가지고 있으며, 배열 전송해서는 안됩니다.

그림 5. 대표 단일 MII의 oocyte에서 Snrpn에 대한 결과를 순서. Snrpn가 oocytes에 methylated있다. 검은 동그라미 methylated CpGs를 나타냅니다. 흰색 동그라미는 unmethylated CpGs를 나타냅니다. CPG 번호와 위치는 B6 변형 암컷 마우스에 대한 대표적인 것입니다. ) 단일 MII의 oocyte에서 Snrpn에 대한 결과를 순서 예상. DNA의 단일 가닥은 모두 다섯 클론으로 증폭한다. 하나의 methylation 패턴과 동일한 비 CPG 변환 은어와 OocytesN은 (비 CpGs의 비율 변환 총 비 CPG cytosines의 비율로 thymine로 변환되지 않은 CPG cytosines의 숫자로 계산 오른쪽으로 표시됨) 분석에 포함되어야한다. 적운 세포 오염과 단일 MII의 oocyte에서 Snrpn위한 나) 장면 결과입니다. 여러 개의 스트랜드 증폭을 나타내는 methylation 상태 및 전환 패턴 사이의 차이를 확인합니다. C) 염색체 사본 또는 정반대의 신체를 포함 모두와 함께 단일 MII의 oocyte에서 Snrpn에 대한 결과를 배열.
Access restricted. Please log in or start a trial to view this content.
토론
이것은 하나의 oocyte 분석이 중요하고 특별한주의를 필요로 번호가 많은 단계가 있습니다. 첫 번째는 oocyte 세척이다. 그것은 가능한 많은 적운 세포로 제거하는 hyaluronidase 치료에 따라 drops 새로운 매체의 각 oocyte 여러 번 씻어 특히 중요합니다. 또한, 조나 pellucida 제거를위한 산성 tyrode의 솔루션으로 oocytes를 전송할 때하는 것은 매체를 둘러싼 것은 적운 세포의 명확한지 확인하십시오. oocyte는 조?...
Access restricted. Please log in or start a trial to view this content.
공개
저자가 공개하는 게 없다.
감사의 말
이 작품은 웨스턴 온타리오, 산과학 및 Gynaecology학과의 대학에 의해 지원되었다; 및 Ministryof 연구 및 혁신, 조기 연구원 상으로부터 보조금 ER06-02-188. MMD는 복제, 조기 개발 및 보건 (REDIH) 대학원 장학금에 미치는 영향에 CIHR 교육 프로그램에 의해 지원되었다.
Access restricted. Please log in or start a trial to view this content.
자료
특정 시약 및 장비의 표.
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 | 댓글 |
| Oocyte 컬렉션 | |||
| Hyaluronidase | 시그마 | H4272 | |
| 산성 Tyrode | 시그마 | T1788 | |
| Proteinase K | 시그마 | P5568 | |
| 10 % IGEPAL | Bioshop | NON999.500 | |
| 용해 용액 | |||
| 트리스 산도 7.5 | Bioshop | TRS001.5 | |
| LiCl | 시그마 | L9650 | & NBSP; |
| EDTA (에틸렌 다이아 민 테트라 초산) 산도 8.0 | 시그마 | E5134 | |
| 뚜껑 | Bioshop | LDS701.10 | |
| DTT | Invitrogen | P2325 | |
| SDS의 용해 완충액 | |||
| TE 산도 7.5 | Bioshop (트리스) 시그마 (EDTA (에틸렌 다이아 민 테트라 초산)) | TRS001.5 E5134 | |
| 10% SDS | Bioshop | SDS001.500 | |
| Bisulfite 전환 | |||
| 수산화 나트륨 | 시그마 | S8045 | |
| 나트륨 Hydrogensulfite (나트륨 Bisulfite) | 시그마 | 243,973 | ; |
| 히드로퀴논 | 시그마 | H9003 | |
| 낮은 녹는 점 (LMP) 아가로 오스 | 시그마 | A9414 | |
| 미네랄 오일 | 시그마 | M8410 | |
| M2 중간 | 시그마 | M7167 | |
| GIBCO 증류수 물을 | Invitrogen | 15230-196 | |
| Autoclaved 이중 증류 (DD) 물 | |||
| PCR | |||
| Illustra 핫 스타트 믹스 RTG | GE 헬스케어 | 28-9006-54 | |
| 240 NG / ML 효모 tRNA | Invitrogen | 15401-011 | |
| 5 배 그린 GoTaq 반응 버퍼 | 프로메가 | M7911 | |
| 내부 및 외부 중첩된 primers | 시그마 | ||
| 내고 | |||
| Promega pGEM-T 쉬운 벡터 | 피셔 과학 | A1360 | |
| TA 복제 | |||
| 유능한 E.coli 세포 | Zymo 리서치 주식 회사 | T3009 | |
| 장비 | |||
| 현미경 해부 | |||
| 70 ° C와 90 ° C 열 블록 | |||
| 37 ° C와 50 ° C Waterbaths (42 ° C에서 변환을위한) | |||
| 흔들리는 것 | |||
| PCR 기계 | |||
참고문헌
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. 33, 245-254 (2003).
- Rodenhiser, D., Mann, M. Epigenetics and human disease: translating basic biology into clinical applications. CMAJ. 174, 341-348 (2006).
- Frommer, M. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U.S.A. 89, 1827-1831 (1992).
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Feil, R., Charlton, J., Bird, A. P., Walter, J., Reik, W. Methylation analysis on individual chromosomes: improved protocol for bisulphite genomic sequencing. Nucleic Acids Res. 22, 695-696 (1994).
- Raizis, A. M., Schmitt, F., Jost, J. P. A bisulfite method of 5-methylcytosine mapping that minimizes template degradation. Anal. Biochem. 226, 161-166 (1995).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA Methylation: Bisulphite Modification and Analysis. J. Vis. Exp. (56), e3170(2011).
- Olek, A., Oswald, J., Walter, J. A modified and improved method for bisulphite based cytosine methylation analysis. Nucleic Acids Res. 24, 5064-5066 (1996).
- Mann, M. R. Selective loss of imprinting in the placenta following preimplantation development in culture. Development. 131, 3727-3735 (2004).
- Market-Velker, B. A., Zhang, L., Magri, L. S., Bonvissuto, A. C., Mann, M. R. Dual effects of superovulation: loss of maternal and paternal imprinted methylation in a dose-dependent manner. Hum. Mol. Genet. 19, 36-51 (2010).
- Meng, L. H., Xiao, S. Q., Huang, X. F., Zhou, Y., Xu, B. S. A study on bisulfite sequencing method for methylation status of imprinted genes in single human oocytes. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 25, 289-292 (2008).
- Denomme, M. M., Zhang, L., Mann, M. R. Embryonic imprinting perturbations do not originate from superovulation-induced defects in DNA methylation acquisition. Fertil. Steril. 96, 734-738 (2011).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Hajj, N. E. l Limiting dilution bisulfite (pyro)sequencing reveals parent-specific methylation patterns in single early mouse embryos and bovine oocytes. Epigenetics. 6, 1176-1188 (2011).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유