JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
고처리량/고함량 스크리닝에 적용된 세포내 진균의 정량화를 위한 현미경 표티픽 분석
요약
여기서는 작은 간섭 합성 RNA(siRNA), 화학 화합물 및 진균 결핵 돌연변이 라이브러리의 고처리량/고함량 스크린에 적용되는 표현성 분석을 설명합니다. 이 방법은 자동 공초점 현미경 검사를 사용하여 형광으로 표지된 숙주 세포 내의 형광표지 진균 결핵의 검출에 의존합니다.
초록
치료와 백신의 가용성에도 불구하고 결핵 (TB)은 세계에서 가장 치명적이고 광범위한 세균 감염 중 하나입니다. 수십 년 이후, 다중 및 광범위 하 게 약물 저항 하는 긴장의 갑작스런 버스트는 결핵의 제어에 대 한 심각한 위협. 따라서 결핵, 진균 결핵(Mtb)의 원인제에 중요한 새로운 표적과 경로를 식별하고 결핵 치료제가 될 수있는 새로운 화학물질을 찾는 것이 필수적이다. 한 가지 접근법은 건초 더미에서 바늘을 검색 할 수 있도록 대규모 라이브러리의 유전 및 화학 적 스크린에 적합한 방법을 설정하는 것입니다. 이를 위해, 우리는 자동 공초점 현미경 검사를 사용하여 형광 표지 숙주 세포 내에서 형광 표지 Mtb의 검출에 의존하는 phenotypic 분석법을 개발했습니다. 이러한 시험관 내 분석법은 Mtb의 식민지화 공정을 숙주로 정량화할 수 있으며, siRNA-, 화학 화합물 또는 Mtb 돌연변이 라이브러리의 스크린에 적합한 384웰 마이크로플레이트 포맷에 최적화되었다. 그런 다음 이미지는 다중 파라메트릭 분석을 위해 처리되며, 이는 숙주 세포 내의 Mtb의 발병기에서 추론을 제공합니다.
서문
지난 몇 년 동안 보고된 신흥 및 재발하는 전염하는 병원균 중, 진균 결핵(Mtb)은 2011년에 140만 명의 사망자와 870만 건의 새로운 감염을 담당하는 두드러진 장소를 보유하고 있습니다(글로벌 결핵 보고서 2012, www.who.int/topics/tuberculosis/en/). 다약물 치료의 가용성에도 불구하고, 감염된 사람들의 수는 여전히 증가하고 다중 약물 내성 (MDR) 뿐만 아니라 광범위하게 약물 내성 (XDR) Mtb는 빠르게 전 세계적으로확산1. 더욱이, Mtb 항원의 존재를 고려할 때, 전 세계 인구의 3분의 1이 Mtb에 의해 잠잠하게 감염된 것으로 간주된다는 것이 분명합니다. 따라서 Mtb과 싸우는 새로운 수단이 절실히 필요합니다. 이러한 맥락에서, 우리는 자동화된 공초점 형광 현미경3에의하여 숙주 세포로 Mtb 침입 및 곱셈을 감시에 의존하는 체외 시각적 표현체 분석법을 개발하였다. 384웰 마이크로티터 플레이트에서 자동 이미지 수집 및 분석과 결합하여 분석체의 적응은 화합물, siRNAs 및 세균 돌연변이의 중간 규모의 라이브러리의 고함량/고처리량 스크리닝(HC/HTS)을 허용했습니다. 이 표현성 분석에 게놈 넓은 RNAi 라이브러리의 검사는 따라서 Mtb 인신 매매 및 세포 내 복제에 관련된 주요 호스트 요인뿐만 아니라 결핵 바실러스에 의해 악용 호스트 경로의 용해를 가능하게했다. 이 특정 현상 분석의 또 다른 적응은 Mtb 내 phagosomal 지속성에 필수적인 세균인의 식별을 위한 것이었습니다. 예를 들어, phagosome 성숙의 체포는 대식세포에서 Mtb의 생존 그리고 복제를 용이하게 하는 주요 기계장치 의 한으로 간주됩니다. 형광 표지산산구획에서 Mtb 녹아웃 돌연변이체의 세포외 국소화 모니터링은 생존 과정에 관여하는 세균 유전자의 식별을허용4. 마지막으로, Mtb의 고함량 이미징은 세포내 세균 성장3과같은 다양한 현상을 억제하기 위한 약물 효율을 정량화하는 우수한 방법을 제공한다. 전부, 높은 처리량 표현분석의 이 모형은 결핵에 대하여 약 발견을 가속화하고 이러한 다른 접근에 의해 집합된 데이터는 Mtb에의해 발휘된 호스트 조작의 더 나은 이해에 기여합니다.
프로토콜
1. 높은 처리량 게놈 와이드 siRNA 스크리닝
인간 유형-II 폐렴구체 모델 A549 세포주에서 수행된 스크리닝은 Mtb H37Rv를 통해 바이러스 성형 단백질(GFP)을 발현하는 감염 시 수행하였다. 이 절차는 그림 1A에설명되어 있습니다.
- 4 μM의 농도에 도달하기 위해 1 x siRNA 버퍼로 어머니 판 (96 웰 플레이트)에 저장된 말린 siRNA 라이브러리를 다시 중단한 다음 혼합물의 10 μl을 384 웰 딸 플레이트 (딸 플레이트 1)로 옮길 수 있습니다.
- 딸 플레이트 1에 1x siRNA 버퍼 10 μl을 추가하여 siRNA를 2배 희석시 합니다. siRNA 재서스펜션 후, 플레이트는 벗겨질 수 있는 알루미늄 씰로 밀봉되어 -20°C이상 에서 최소 6개월 및 최대 2-3년까지 보관할 수 있지만, 저장 시간은 siRNA 라이브러리 제조업체의 권장 사항에 따라 달라질 수 있습니다.
- 딸 접시 1에 siRNAs를 희석하여 딸 플레이트 2에 넣고 500 nM의 농도에 도달하십시오. siRNA 재서스펜션 후, 플레이트는 벗겨질 수 있는 알루미늄 씰로 밀봉되어 -20°C이상 에서 최소 6개월 및 최대 2-3년까지 보관할 수 있지만, 저장 시간은 siRNA 라이브러리 제조업체의 권장 사항에 따라 달라질 수 있습니다.
- 사용하기 전에 실온에서 딸 플레이트 2를 해동하십시오.
- 딸 플레이트 2에서 siRNA2.5 μl을 가져 와서 384 웰 분석 플레이트에 넣습니다.
- 1.5단계에서 동일한 384-음분석 플레이트에서 각각의 우물에 2.5 μl 음수 및 양수 제어 siRNA를 추가합니다.
- 1x D-PBS에서 트랜스페션 시약을 희석하여 각 우물에서 0.1 μl 형질 전환 시약을 제공하고 5분 동안 실온에서 희석된 형질 방출 용액을 미리 배양할 수 있는 충분한 용액을 산출합니다.
- 형질 전환 시약/PBS 용액 혼합물의 7.5 μl을 분석 판에 각각 잘 넣고 실온에서 30분 동안 배양합니다.
- 10% 태아 소 혈청(FBS)으로 보충된 RPMI 1640 배지에서 중단된 A549 세포(1,500세포/웰)의 40μl을 추가합니다. 5%CO2를함유하는 대기에서 37°C에서 3일 배양 기간 동안 세포를 유지한다. 이 세포는 매 24 시간 마다 분할, 따라서 대략 12,000 세포는 경질 후에 3 일 우물에 있습니다.
- 2주 된 GFP 표현 Mtb H37Rv 문화를 D-PBS(MgCl2 및 CaCl2무료)로 씻어 내고 원심분리를 5분 동안 4,000 x g로 씻어 내고 세척을 폐기합니다. 이 단계를 3배 반복합니다(GFP-Mtb H37Rv 문화 조건의 경우 프로토콜 3참조).
- 세균성 골을 10 ml의 RPMI 1640 배지에 10% FBS로 보충하고 세균성 골체가 퇴적물을 허용하도록 실온에서 1 시간 동안 데캔트를 보충합니다.
- 세균성 상체를 수집하고 OD600(OD 600은 0.6-0.8 사이여야 함)과 GFP-형광(RFU 값) 및 마이크로플레이트 판독기를 사용하여 세균 농도를 결정한다. 동일한 조건에서 제조된 다른 배양에 대한 실험 전에 생성된 RFU 값 = f(CFU 값)를 표시하는 참조 회귀 선을 사용하여 서스펜션의 티터를 계산한다. 감염(MOI)의 복합성에 해당하는 2.4 x 106 박테리아/ml를 함유한 세균현탁액을 준비한다.
- 384-음 분석 플레이트의 배지를 제거하고 갓 준비된 세균 현탁액 25 μl을 추가합니다.
- 384-음분석 플레이트를 37°C에서 5%CO2를함유한 대기시 5시간 동안 배양한다.
- 매체를 제거하고 10% FBS 3x로 보충된 RPMI 배지로 세포를 부드럽게 세척합니다.
- 남은 세포외 세균을 죽이기 위해, 5%CO2를함유한 대기시 37°C에서 37°C에서 50 μg/ml를 함유한 신선한 RPMI-FBS 배지50 μl로 세포를 치료한다.
- 중간 함유 아미카신을 제거하고 10% FBS로 보충된 신선한 RPMI 배지 50 μl을 추가합니다. 384-잘 분석 플레이트를 37°C에서 5일 동안 5%CO2를함유한 대기권에서 배양한다.
- 이미지 수집 에 앞서 PBS (최종 농도 5 μg /ml)에 갓 준비 된 30 μg /ml의 10 μl을 추가하고 37 ° C에서 10 분 동안 배양하십시오.
- 플레이트를 자동 공초점 현미경으로 로드합니다.

큰 이미지를 보려면 여기를 클릭하십시오. - 노출 매개 변수를 설정합니다. 방출 필터 450 nm및 GFP 형광을 이용한 발산 레이저 405 nm를 사용하여 DAPI 형광을 기록하여 방출 필터 520 nm를 가진 엑아시 레이저 488 nm를 사용한다.
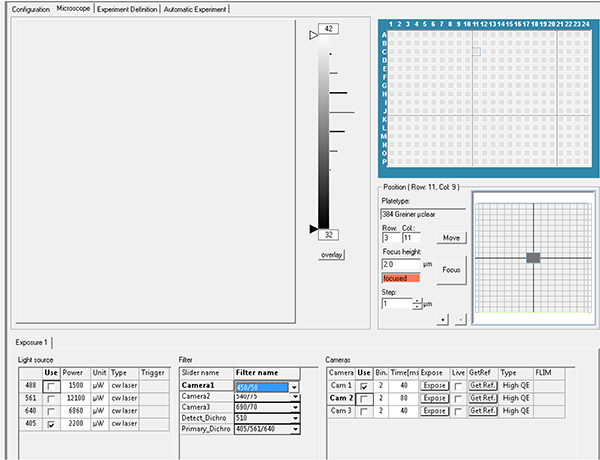
큰 이미지를 보려면 여기를 클릭하십시오.
큰 이미지를 보려면 여기를 클릭하십시오. - 획득할 각 웰의 우물과 필드를 선택하여 레이아웃 및 하위 레이아웃 매개 변수라고 합니다.
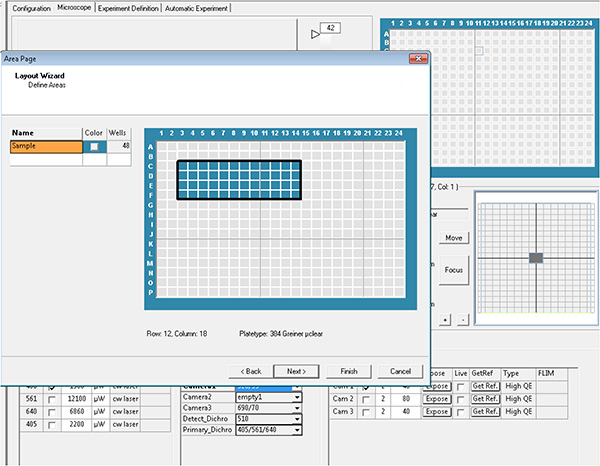
큰 이미지를 보려면 여기를 클릭하십시오.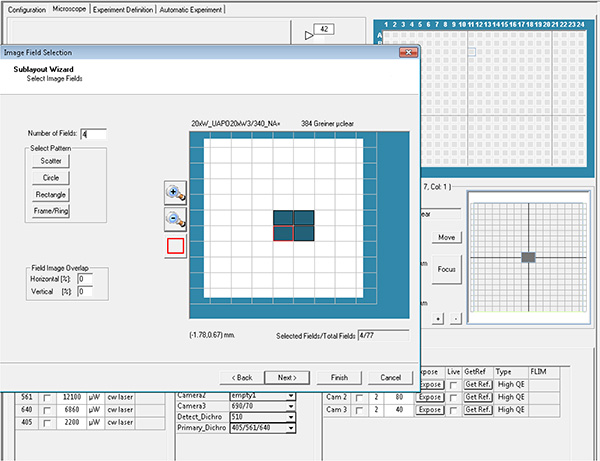
큰 이미지를 보려면 여기를 클릭하십시오. - 1.20 및 1.21 단계의 매개 변수를 사용하여 실험 파일을 생성하고 자동 수집을 실행합니다.
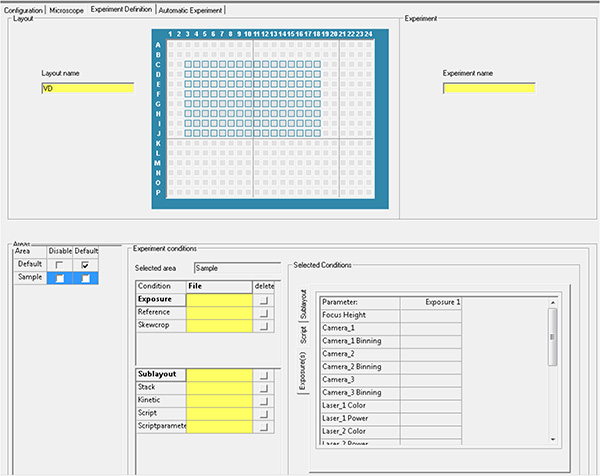
큰 이미지를 보려면 여기를 클릭하십시오.
큰 이미지를 보려면 여기를 클릭하십시오. - 이미지를 원격 서버로 전송합니다.
큰 이미지를 보려면 여기를 클릭하십시오. - 이미지 분석 소프트웨어를 사용하여 이미지를 평가합니다. 픽셀 강도 특성알고리즘(도 4A)을사용하여 GFP 채널로부터 핵 검출 알고리즘 및 세균 영역을 사용하여 DAPI 채널에서 세포 핵을 검출한다.
참고: 이 프로토콜은 세포 내 Mtb 성장에 대한 유전자 침묵의 효과를 연구하도록 최적화되어 있습니다. Mtb는 최적의 조건에서 매 20 시간마다 분할 느린 성장 박테리아입니다. 감염 후 5 일 후 세포 외 Mtb의 양은 세포 용해가 없는 상태에서 여전히 낮으며 분석의 품질에 영향을 미치지 않았습니다. 이 프로토콜은 광범위하게 방출되고 새로운 세포를 감염시킬 수 있는 Mycobacteria smegmatis 및 Escherichia coli 같이 급속한 성장 박테리아를 사용하여 siRNA 스크린에 적응하기 위하여 항생제 처리 및 잠복기 시간의 기간에, 최적화되어야 합니다.
2. 높은 처리량 화합물 스크리닝
Mtb H37Rv 감염 호스트 세포에 수행 된 스크리닝. 이 절차는 그림 1B에설명되어 있습니다.
- DMSO 100%에서 용해된 화합물 라이브러리를 포함하는 384웰의 어머니 판을 해동한다. 10% FBS로 보충된 RPMI 1640 배지의 10 μl을 포함하는 384웰 딸 플레이트에서 화합물의 0.5 μl을 전송한다.
- 2주 된 GFP 표현 Mtb H37Rv 문화를 D-PBS(MgCl2 및 CaCl2무료)로 씻어 내고 원심분리를 5분 동안 4,000 x g로 씻어 내고 세척을 폐기합니다. 이 단계를 3배 반복합니다(GFP-Mtb H37Rv 문화 조건의 경우 프로토콜 3참조).
- 세균성 골을 10 ml의 RPMI 1640 배지에 10% FBS로 보충하고 세균성 골체가 퇴적물을 허용하도록 실온에서 1 시간 동안 데캔트를 보충합니다.
- 세균상수체를 수집하고 마이크로플레이트 판독기를 사용하여 OD600(OD 600 은 0.6-0.8 사이여야 함)과 GFP 형광(RFU 값) 측정한다. 실험 전에 생성된 RFU 값 = f(CFU 값)를 표시하는 참조 회귀 선을 사용하여 서스펜션의 티터를 계산합니다. 일반적인 농도는 1 x 108 박테리아/ml입니다.
- 수확 6 일 된 기본 인간 대식세포에서 4 x 105 세포/ml RPMI 1640 배지10% 태아 소 혈청 (FBS) 및 50 ng/ml 재조합 인간 M-CSF (인간 말초 혈액 모노사이클 세포 정화 및 대식세포 분화의 경우 프로토콜 4 참조).
- 1-5에 이르는 다른 MOI에서 원액1차 세포를 37°C에서 2시간 동안 90rpm에서 가벼운 흔들림으로 현탁액하여 다른 MOI에서 바실리로 배양한다.
- 감염된 세포를 350 x g에서 원심분리로 세척하여 세포외 박테리아를 제거합니다. 각 원심 분리 단계 후, 10% FBS로 보충된 RPMI 1640 배지에서 펠릿을 다시 놓습니다. 이 단계를 2배 반복합니다.
- 50 μg/ml에서 10% FBS 및 아키카신으로 보충된 RPMI 1640 배지에서 감염된 세포를 중단하고 37°C에서 1시간 동안 가벼운 흔들림으로 현탁액을 배양한다.
- 350 x g에서 원심분리에 의해 아키카신을 함유하는 세포 배양 배지를 제거하고 10% FBS 및 50 ng/ml 재조합 인간 M-CSF로 보충된 완전한 RPMI 1640 배지로 감염된 세포를 세척한다. 한 번 반복합니다.
- 감염된 대식세포 현탁액의 40 μl을 2.1단계에서 동일한 분석 플레이트에 추가하여 이미 화합물 희석제의 10 μl을 함유하고 있습니다. 각 우물에서 DMSO의 최종 농도는 이제 1 %에 도달했습니다.
- 5%CO2를함유한 대기에서 37°C에서 5일간 분석플레이트를 배양한다.
- 세포가 구형극홍형염으로 살아있는 세포를 얼룩지게 합니다.
- 플레이트를 자동 공초점 현미경으로 로드합니다.

큰 이미지를 보려면 여기를 클릭하십시오. - 노출 매개 변수를 설정합니다. 방출 필터 690 nm와 GFP 형광을 사용하여 방출 필터 640 nm와 GFP 형광을 사용하여 광적 형광을 기록 520 nm.

큰 이미지를 보려면 여기를 클릭하십시오.
큰 이미지를 보려면 여기를 클릭하십시오. - 획득할 각 웰의 우물과 필드를 선택하여 레이아웃 및 하위 레이아웃 매개 변수라고 합니다.

큰 이미지를 보려면 여기를 클릭하십시오.
큰 이미지를 보려면 여기를 클릭하십시오. - 2.14 및 2.15 단계의 매개 변수를 사용하여 실험 파일을 생성하고 자동 수집을 실행합니다.

큰 이미지를 보려면 여기를 클릭하십시오.
큰 이미지를 보려면 여기를 클릭하십시오. - 이미지를 원격 서버로 전송합니다.

큰 이미지를 보려면 여기를 클릭하십시오. - 이미지 분석 소프트웨어를 사용하여 이미지를 평가합니다. 픽셀 강도 속성알고리즘(그림 4B)을사용하여 GFP 채널에서 픽셀 강도 특성 알고리즘 및 세균 영역을 사용하여 극적 채널에서 셀 영역을 감지합니다.
참고: 이 프로토콜은 형광 단백질(1개 우물/1 돌연변이)을 표현하는 돌연변이체에 의한 화합물을 대체하여 Mtb 돌연변이 라이브러리 스크리닝에 적용될 수있다(도 1C,브로딘 외 참조) 4).형광 돌연변이는 우물에서 먼저 시드된다 (잘 당 세균 현탁액의 20 μl). 박테리아는 그 때 세포 현탁액의 30 μl에 의해 복구됩니다. 원심분리후 350 x g에서 1분 동안, 플레이트는 5%CO2를함유하는 대기에서 37°C로 배양된다. 인큐베이션 시간 및 MOI는 분석에 의존합니다. 예를 들어, phagosome 산성화와 같은 초기 세포 이벤트의 시각화를 위해, 세포는 1-20에서 배열하는 MOI를 가진 2 시간 동안 감염될 수 있다. 리소솜은 리소트래커 염료를 2μM에서 1.5시간 동안 37°C에서 37°C로염색한 후 10% 포름알린 또는 4%의 파라포름데히드(PFA)로 고정된다. 공초점 이미지는 리소좀 검출 및 세포극 지역화4에적합한 알고리즘을 특징으로 하는 이미지 분석 스크립트를 사용하여 획득및 최종 적으로 분석된다.
3. 진균 결핵 H37Rv (GFP-H37Rv) 문화 조건을 표현하는 녹색 형광 단백질
장기 보관을 위해 GFP-H37Rv는 D-PBS(유리병당 약 1 x 108 균주)로 동결되었습니다.
- 미들브룩 OADC 농축 10%, 글리세롤 0.5%, 트웬-80 0.05%, 하이그로마이신 B(50 μg/ml)로 보충된 7H9 국물 중형 50ml를 함유한 Erlenmeyer 플라스크에서 GFP-H37Rv의 냉동 육수 바이알 1개를 재차 중단합니다.
- 37 °C에서 8 일 동안 배양하십시오.
- GFP-H37Rv 배양의 OD600을 측정합니다.
- GFP-H37Rv 배양을 희석하여 미들브룩 OADC 농축10%, 글리세롤 0.5%, 트위엔-80 0.05%, 하이그로미신 B(50 μg/ml)로 보충된 신선한 7H9 국물 배지에서 OD 600 = 0.1을 얻습니다.
- GFP-H37Rv를 37°C에서 37°C에서 8일 더 배양한 후 분석에 사용한다.
4. 전신 혈액 또는 버피 코트 준비에서 인간의 말초 혈액 단화 세포 정화
- 1%의 FBS를 함유한 1x D-PBS(MgCl 2 및 CaCl2에서 무료)로 혈액 파우치2x를희석시합니다.
- Ficoll 밀도 그라데이션 원심분리에 의해 단핵구를 400 x g에서 20분 동안 분리합니다.
- 격리된 단핵구를 수집합니다.
- 1x D-PBS(MgCl2 및 CaCl2에서무료)로 모노사이클 3x를 세척하여 실온에서 10분 동안 400 x g에서 원심분리로 1% FBS를 함유하고 있습니다.
- 세포를 최대 1 x 107 셀/ml로 농축합니다.
- 제조업체의 프로토콜에 따라 CD14 자성 구슬을 사용하여 단백구를 정화합니다(재료 참조).
- CD14-단핵구 정제 후, RPMI 1640에서 1.5 x 106 셀/ml의 세포를 종자 10% FBS와 40 ng/ml의 인간 대식세포 콜로니 자극 인자(hM-CSF)를 보완하고 5%CO2를함유한 대기 중 37°C에서 4일 동안 배양하였다.
- 4일 후 신선한 RPMI 1640으로 배지를 10% FBS와 40 ng/mlhM-CSF로 보완하고 5%CO2를함유한 대기 중 37°C에서 2일간 배양하였다.
- 6 일 후, 세포는 분석에 사용할 수 있습니다.
결과
높은 처리량 게놈 와이드 siRNA 스크리닝
Mtb는 체외뿐만 아니라 여러 가지 다른 폐 상피 세포에서 면역 세포를 식민지화 할 수 있습니다. 예를 들어, Mtb는인간 형 II 폐렴구균5-7의모델로 일반적으로 사용되는 A549 상피 세포를 감염시키고 손상시킬 수 있다. Dectin-1은 A549 세포에서 세포 내 진균 성장에 Mtb uptake, 프로염증 반응 및 항균 효과에 관여?...
토론
우리는 여기에 GFP 표현 Mtb H37Rv 균주를 사용하여 표현 형광 숙주 세포를 감염시키는 표현적 분석에 필요한 방법을 설명, 이는 높은 함량 / 높은 처리량 스크린에 적합하게. 이 프로토콜은 광범위한 화합물, 형광 프로브 및 Mtb 돌연변이체에 적용될 수 있습니다. 위에서 설명한 각 프로토콜에 대해 이미지 수집 전에 고정 및 면역 라벨링 단계를 수행할 수 있습니다. 우리는 이미지를 얻기...
공개
이해 상충이 선언되지 않았습니다.
감사의 말
이 작업에 대한 재정 지원은 유럽 공동체 (ERC-STG INTRACELLTB 그랜트 n ° 260901, MM4TB 그랜트 n ° 260872), 기관 국립 드 레체쉬, 페더 (12001407 (D-AL) Equipex Imaginex BioMed), 및 지역 노르드 파스 칼레에 의해 제공되었다. 우리는 플랫폼 BICeL에서 가스파드 딜로이슨, 엘리자베스 베르크마이스터, 안토니노 본지오반니, 프랭크 라폰트의 기술적 도움을 감사하게 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| µclear-plate black, 384-well | Greiner Bio-One | 781091 | 127.8/86/15 MM with Lid, TC treated |
| CellCarrier 384-well plate | PerkinElmer | 6007550 | Black, Clear Bottom, with Lid, TC treated |
| V-bottom white, 384-well plate | Greiner Bio-One | 781280 | |
| sealing tape, breathable, sterile | Corning | 3345 | |
| Lipofectamine RNAiMax | Life Technologies | 13778150 | Transfection reagent |
| Dimethyl sulfoxide | Sigma-Aldrich | 34943 | |
| RPMI 1640 + GlutaMAX-I | Life Technologies | 61870-010 | Cell culture medium |
| D-PBS 1x [-]MgCl2/[-]CaCl2 | Life Technologies | 14190-094 | Dulbecco's Phosphate Salin Buffer |
| D-PBS 1x [+]MgCl2/[+]CaCl2 | Life Technologies | 14190-091 | Dulbecco's Phosphate Salin Buffer |
| Fetal bovine serum | Life Technologies | 2610040-79 | |
| Ficoll Paque PLUS | Dutscher | 17-1440-03 | Ficoll for Peripherical Blood Monocyte Cells purification |
| CD14 MicroBeads, human | Miltenyi | 130-050-201 | Purification of CD14+ Monocytes |
| Human M-CSF, premium gr. (1000 μg) | Miltenyi | 130-096-493 | Macrophage Colony Stimulating Factor |
| LS Columns | Miltenyi | 130-042-401 | Columns for CD14+ Monocytes isolation |
| Tween 80 | Euromedex | 2002-A | Mycobacteria culture |
| Glycerol high purity | Euromedex | 50405-EX | Mycobacteria culture |
| Middlebrook OADC enrichment | Becton-Dickinson | 211886 | Mycobacteria culture |
| 7H9 | Becton-Dickinson | W1701P | Mycobacteria culture |
| Versene 1x | Life Technologies | 15040033 | Nonenzymatic cell dissociation solution |
| DAPI | Life Technologies | D1306 | Nuclei dye |
| Hoechst 33342 | Life Technologies | H3570 | Nuclei dye |
| Syto60 | Life Technologies | S11342 | Nuclei/cytoplasm dye |
| Formalin | Sigma-Aldrich | HT5014 | Cell fixation solution |
| siRNA targeting Dectin-1 | Santa-Cruz | sc-63276 | |
| *siGenome* Nontargeted siRNA pool | Dharmacon | D-001206-14 | |
| Rifampicin | Sigma-Aldrich | R3501 | antibiotic |
| Isoniazid (INH) | Sigma-Aldrich | I3377-50G | antibiotic |
| Hygromycin B | Life Technologies | 10687-010 | antibiotic |
| Amikacin | Sigma-Aldrich | A1774 | antibiotic |
| Automated Confocal Microscope OPERA | PerkinElmer | Image acquisition | |
| Columbus 2.3.1 Server Database | PerkinElmer | Data transfer, storage, and analysis | |
| Acapella 2.6 software | PerkinElmer | Image-based analysis | |
| GraphPad Prism5 software | GraphPad | Statistical analysis | |
| Excel 2010 | Microsoft | Statistical analysis |
참고문헌
- Gandhi, N. R., et al. Multidrug-resistant and extensively drug-resistant tuberculosis: a threat to global control of tuberculosis. Lancet. 375, 1830-1843 (2010).
- Barry, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat. Rev. Microbiol. 7, 845-855 (2009).
- Christophe, T., Ewann, F., Jeon, H. K., Cechetto, J., Brodin, P. High-content imaging of Mycobacterium tuberculosis-infected macrophages: an in vitro model for tuberculosis drug discovery. Future Med. Chem. 2, 1283-1293 (2010).
- Brodin, P., et al. High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. PLoS Pathog. 6, (2010).
- Bermudez, L. E., Goodman, J. Mycobacterium tuberculosis invades and replicates within type II alveolar cells. Infect. Immun. 64, 1400-1406 (1996).
- Dobos, K. M., Spotts, E. A., Quinn, F. D., King, C. H. Necrosis of lung epithelial cells during infection with Mycobacterium tuberculosis is preceded by cell permeation. Infect. Immun. 68, 6300-6310 (2000).
- McDonough, K. A., Kress, Y. Cytotoxicity for lung epithelial cells is a virulence-associated phenotype of Mycobacterium tuberculosis. Infect. Immun. 63, 4802-4811 (1995).
- Lee, H. M., Yuk, J. M., Shin, D. M., Jo, E. K. Dectin-1 is inducible and plays an essential role for mycobacteria-induced innate immune responses in airway epithelial cells. J. Clin. Immunol. 29, 795-805 (2009).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J. Biomol. Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat. Methods. 6, 569-575 (2009).
- Carralot, J. P., et al. Automated high-throughput siRNA transfection in raw 264.7 macrophages: a case study for optimization procedure. J. Biomol. Screen. 14, 151-160 (2009).
- Zhang, X. D. Illustration of SSMD, z score, SSMD*, z* score, and t statistic for hit selection in RNAi high-throughput screens. J. Biomol. Screen. 16, 775-785 (2011).
- Song, O. R., et al. Confocal-based method for quantification of diffusion kinetics in microwell plates and its application for identifying a rapid mixing method for high-content/throughput screening. J. Biomol. Screen. 15, 138-147 (2010).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유