JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
단일 분자 형광 현미경에 의한 수용체 역학의 고해상도 시공간 분석
요약
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
초록
단일 분자 현미경은 그 부분 (예를 들면., 역학, 다른 주와 인구, 과도 상호 작용의 공존) 일반적으로 앙상블 측정에 숨겨진,, 등에 관한 특히, 신호 분자의 행동을 분석 할 수있는 강력한 방법으로 대두되고 표준 생화학 적 또는 현미경 방법으로 얻은. 따라서, 이러한 수용체 - 수용체 상호 작용과 같은 동적 이벤트는, 높은 시공간 해상도를 가진 살아있는 세포에서 실시간으로 따라야 할 수있다. 이 프로토콜은 작고 밝은 유기 형광체와 직접 살아있는 세포의 표면에 단일 수용체를 시각화하는 전반사 형광 (TIRF) 현미경으로 라벨에 기반 방법을 설명한다. 이러한 접근 방식은 하나가 정확하게 수용체를 지역화 수용체 복합체의 크기를 측정하고, 이러한 과도 수용체 - 수용체 상호 작용과 같은 동적 이벤트를 캡처 할 수있다. 이 프로토콜은 퍼가기하는 방법에 대한 자세한 설명을 제공샘플 준비, 이미지 수집 및 이미지 분석을 포함하는 단일 분자 실험을 rform. 예로서, 본 방법의 애플리케이션이 G-단백질 결합 수용체를 분석하기 위해, 즉., 2 - 아드레날린 및 γ-아미노 낙산 타입 B (GABA의 B) 수용체 β,보고된다. 프로토콜은 다른 막 단백질 및 다양한 셀 모델, 형질 감염 방법 및 표지 전략을 적용 할 수있다.
서문
세포 표면에있는 수용체는 세포 외 환경을 감지하고 취기 제, 이온, 작은 신경 전달 물질 및 대형 단백질 호르몬 자극 등 다양한 반응한다. 세포막의 유체 특성은 수용체와 다른 막 단백질의 운동을 할 수 있습니다. 이것은 단백질 복합체와 같은 기능 유닛으로 조립하고 세포 내부에 신호를 형질 도입하는 수용체에 의해 사용되는 것과 같은 과도 단백질 - 단백질 상호 작용의 발생의 형성에 필수적이다. 예를 들어, 세포 표면 수용체 1의 가장 큰 가족을 구성하는 G-단백질 결합 수용체 (GPCR에)는, 신호 전달의 미세 조정 조절에 관여하는 것으로 나타나고 di-/oligomers를 형성하기 위해 제안되었다 중요한 생리 및 약리학 적 결과 2-5가있을 수 있습니다.
단일 분자 현미경 직접 높은 spatiotemp과 시각화의 큰 잠재력을 가지고경구 해상도 그들의 조합을 포함하여 살아있는 세포의 표면에 위치한 각각의 수용체의 동적 거동은 다이머 및 고차 분자 복합체 6-10을 형성한다. 이것은 일반적으로 분자의 수천 또는 수백만의 평균 행동을보고 표준 생화학 적 및 현미경 방법에 비해 여러 가지 장점을 제공합니다.
충분히 밝고 광 안정성과 형광 단백질 라벨은 단일 분자 현미경을 위해 필수적이다. 이 프로토콜은 공유 세포 표면의 수용체에 작고 밝은 유기 형광 물질을 부착 최근에 도입 된 SNAP 태그 (11)을 이용합니다. SNAP는 돌이킬 형광 - 복합 benzylguanine (형광-BG) 파생 상품으로 표시 할 수있는 인간의 DNA 수리 효소 O 6 alkylguanine-DNA의 alkyltransferase에서 파생 된 20 kD의 단백질 태그입니다. CLIP, SNAP에서 파생 된 추가 설계 태그 대신 형광-C로 표시 할 수 있습니다onjugated benzylcytosine 유도체 12.
이 논문에보고 된 프로토콜은 형질 및 레이블 작은 유기 형광 11 수용체를 SNAP-태그와 살아있는 세포 (10)의 표면에 하나의 수용체 나 수용체 복합체를 시각화하기 위해 총 내부 반사 형광 (TIRF) 현미경을 사용하는 방법에 대해 설명합니다. 세포 외 SNAP-태그 세포 표면 단백질 10> 90 %의 표지 효율이보고 된 프로토콜 결과. 수용체 복합체의 크기와 이동성을 분석 할뿐만 아니라 과도 수용체 - 수용체 상호 작용을 캡쳐하는 단일 분자 데이터를 사용하는 방법에 대한 추가 정보가 제공된다. 전체 프로토콜의 개략적 인 흐름은도 1에 제시되어있다. 예로 중국 햄스터 난소 SNAP-태그 된 G-단백질 결합 수용체 (GPCR에)와 (CHO) 세포의 형질 감염이 같은 형광 물질 BG 유도체로 라벨링 하였다 잘 자사의 응용 프로그램이 quantif하기Y와 모니터 수용체 di-/oligomerization가 설명되어 있습니다. 이 프로토콜은 다른 세포 표면 단백질 및 형광 태그 (예., CLIP), 등 형질 및 표지 방법으로 확장 될 수있다.
프로토콜
1. 샘플 준비
- 커버 슬립 청소
참고 : 흄 후드에서 작업 할 수 있습니다.- 개별 커버 슬립을 분리하는 커버 슬립 홀더에 유리 커버 슬립 (24mm 직경)를 배치하는 깨끗한 핀셋을 사용합니다.
- 비이커에 커버 슬립으로 홀더를 넣고, 커버 슬립이 덮여 때까지 클로로포름을 추가합니다. RT에서 1 시간 동안 욕조 초음파 처리기에서 증발 및 초음파 처리를 줄이기 위하여 알루미늄 호일로 비이커 커버. 비커 밖으로 커버 슬립 홀더를 가지고 coverslips를 건조 할 수 있습니다.
- 대신 클로로포름 5M NaOH 용액으로 단계 1.1.2를 반복합니다.
- 새로운 비커에 커버 슬립 홀더를 가지고 증류수로 3 회 세척한다. 100 % 에탄올로 가득 유리 세포 배양 접시에서 청소 커버 슬립을 넣어.
- 교정 샘플의 제조
- 적당한 용매에 형광 염료를 녹여.
- 까지 형광 염료의 1:10 시리얼 희석을 준비부터 1 pm 1 nm의 필터 (0.22 μm의) 물을 살균.
- 100 % 에탄올에 저장 청소 커버 슬립을 가지고 필터 멸균 수로 세척. 별도의 청소 커버 슬립의 각 형광 염료 희석 자리 20 μL. 커버 슬립이 멸균 후드 건조시킵니다. 사용할 때까지 빛과 먼지 커버 슬립을 보호합니다. 단일 형광 분자의 강도 (3 단계 참조)을 추정하기 위해이 샘플을 사용합니다.
- 형질
- 1시 1분 둘 베코 변형 이글 배지 / 영양 혼합물 문화 CHO 세포 F-12 10 % 소 태아 혈청 (FBS), 100 U / ㎖ 페니실린, 37 ° C에서 100 ㎍ / ml의 스트렙토 마이신,에 보충 (DMEM/F12) . 5 % CO 2 주 : 형광도를 최소화하기 위해 실험을하는 동안 페놀 레드 무료 용지를 사용하십시오.
- , 100 % 에탄올 용액에서 청소 커버 슬립을 멸균 인산 완충 생리 식염수 (PBS)로 세척하고, 6 웰 세포 배양 (PL)의 각 웰에 한 커버 슬립을 배치먹었다.
- 를 Trypsinize는 세고 3 × 105 세포 / 웰 커버 슬립을 함유하는 6 - 웰 세포 배양 접시의 밀도로 시드 CHO 세포. 세포가 약 달성하기 위해 24 시간 동안 (37 ° C에서 5 % CO 2) 배양기에서 성장하자. 형질 전환을위한 최적의 세포 밀도 80 % 자랄.
- 각 웰의 경우, (예를 들면., 2 - 아드레날린 수용체 β SNAP-태그) 원하는 플라스미드 DNA의 2 μg을 희석 500 μL의 티멤 매체를 포함하는 두 개의 튜브에서 6 μL의 리포 펙 타민 2000. 실온에서 5 분 동안 품어.
- 하나의 튜브에 단계 1.3.4에서 솔루션을 결합하고 형질 전환 혼합물을 얻기 위해 혼합한다. 20 분 동안 RT에서 형질 감염 혼합물을 배양한다.
- 배양 (1.3.5) 동안, CHO 세포를 가지고 예열 (37 ℃) PBS로 두 번 세척한다. 1 ㎖ / 10 % FBS와 보충 페놀 레드 무료 DMEM/F12 매체의 잘하지만 항생제로 PBS를 교체합니다.
- 전체 형질 추가부드럽게 이온 혼합물 아니라 각 단계 1.3.5 적하에서 (1 ㎖), 그리고 완전한 혼합을 보장하기 위해 앞뒤 판 바위.
- 5 % CO 2, 37 ℃에서 2 ~ 4 시간 동안 배양하고 다음 단계로, 그 후 즉시 진행 참고 :.이 형질 전환 조건에 대한 단일 적합한 수용체 밀도 <0.45 입자 / μm의 2를 달성하기 위해 최적화 된 분자 영상. 다른 세포 구조 나 시약을 사용하는 경우 조정이 필요할 수 있습니다.
- 단백질 라벨
- 1 μM의 최종 농도를 얻기 위해 10 % FBS로 보충 된 1 ㎖ DMEM/F12 배지에서 형광 물질 BG 스톡 용액의 1 ㎕의 희석. 인큐베이터에서 형질 전환 세포를 가지고 예비 가온 (37 ° C) PBS로 두 번 세척한다. 1 μM 형광-BG 용액 1 ㎖의 PBS를 교체하고 인큐베이터에서 37 ° C 5 % CO 2에서 20 분 동안 품어.
- 배양 후, 세포를 세 씻어10 % FBS와 보충 DMEM/F12 매체와 시간마다 37 ℃에서 5 분 인큐베이션 핀셋 (표시 셀) 커버 슬립을 가지고 이미징 챔버에 배치합니다.
- 300 ㎕의 영상 버퍼로 두 번 씻으십시오. 신선한 영상 버퍼 300 μl를 추가하고 영상 (2 부)로 바로 이동합니다.
2. 이미지 획득
NOTE : 전반사 형광 오일 침지 높은 개구 수 대물 (. 예 100X magnification/1.46 개구), 최적의 레이저 (예를 들어, 405 나노 미터, 488 나노 미터, 561 ㎚, 645 탑재 (TIRF) 현미경을 사용 nm의 다이오드 레이저), 전자 곱한 전하 결합 소자 (EMCCD) 카메라, 인큐베이터 및 단일 형광 분자를 시각화하는 온도 제어.
- 원하는 현미경 매개 변수, 예를 설정합니다., 레이저 라인, TIRF 각도 (이 매개 변수에 침투를 제어사라져가는 필드의 기의 깊이), 노출 시간, 프레임 속도 및 영화 (10) 당 이미지 수. 온도 감도는 수분 응축을 방지하기 위해 항상 히터 / 보육 및 온도 제어를 유지합니다.
- 현미경의 100X 목적에 침지 기름 한 방울을 넣어. 현미경의 시편 홀더에 표시된 세포 이미징 챔버를 배치하고 시야 조명을 사용하여 초점 세포를 가져옵니다.
- TIRF 조명으로 전환합니다. 그러나 동시에, 원하는 셀에 대한 검색 광표백을 최소화하도록 가능한 한 낮은 레이저 출력을 유지한다.
- 원하는 셀을 선택하고 정밀한 초점을 조정합니다. 단일 형광의 시각화를 허용 레벨로 레이저 파워를 설정한다. 이미지 시퀀스를 획득. TIFF로 RAW 이미지 시퀀스 파일을 저장합니다.
3. 교정 (유리 및 단량체 / 이량 체 수용체 컨트롤에 단일 형광)
- 각 교정 SAMPL 조립촬상 챔버에서 1.2에 설명 된대로 전자 제조. 현미경으로 각 샘플을 놓고 한 단계 참고로 표백 잘 분리 회절 제한 장소를 포함하는 샘플을 선택합니다.이 반점은 형광 염료의 분자 하나를 나타냅니다.
- 2 단계 설명 TIRF 이미지 시퀀스를 획득 중요. 동일한 촬상 파라미터 보정 것들을 포함한 모든 실험에 사용되어야한다.
- 4.2 - 검색 및 4.1에 설명 된대로 추적 분석을 수행합니다. 4.2.6에 설명 된대로 각 입자의 강도를 추출합니다. 이 데이터에서 평균 (μ)와 하나의 형광 강도의 표준 편차 (σ)를 계산합니다.
- 선택적 : 단량체 세포 표면 수용체 (. 예, CD86)로 형질 감염된 세포에서 동일한 분석을 수행, N-말단은 SNAP (10)의 하나 또는 두 개의 카피로 태그 및 형광 BG-유도체로 표지. FOLLO유리에 하나의 형광 위에서 설명한 절차 w. Calebiro, D. 등의 설명에 따라 라벨 효율을 예상하고있다. 10
4. 이미지 분석
- 이미지 시퀀스 준비
- 이미지를 자르려면 이미지 처리 소프트웨어 (예., ImageJ에)를 사용합니다.
- 별도의 개별 프레임을 저장합니다. TIFF 이미지를 새 폴더에, 각 이미지 프레임 번호를 나타낸다.
- 셀의 형상을 따라 그 (ROI)의 영역을 드로잉하고 ImageJ에있는 공구 측정 또는 다른 소프트웨어에서 유사한 공구를 사용하여 셀 면적을 측정한다. 세포 표면 영역에 의해 동영상의 시작 부분에 입자의 총 개수로 나눔으로써 입자 밀도를 계산하기 위해이 값을 사용한다.
- 입자 검출 및 추적
참고 : 매트랩 작업 등 U-추적 13로 비 상업용 소프트웨어를 사용하여자동으로 감지하고 하나의 수용체 입자를 추적 할 수있는 환경.
참고 : U-트랙 알고리즘은 다중 가설 추적 방식을 기반으로합니다. 이 방법은 한 프레임에 주어진 입자, 다음 프레임에 주어진 입자에 해당하는 표시, 사라 / 또는 다른 입자 /로 분할 병합 개별 확률이 할당되는 비용 행렬을 구축하여 프레임 사이의 입자를 연결합니다. 전 세계적으로 비용을 최소화하는 솔루션은, 즉., 확률이 가장 높은 사람은 마지막으로 선택됩니다. 이는 또한 일시적으로 사라지는 입자, 형광 깜박임 기인 전형적인 현상을 추적한다. U-트랙 (2.1.0)의 최신 버전은 이러한 분석의 실행을 촉진 그래픽 사용자 인터페이스를 가지고 있습니다.- 영화 선택 인터페이스를 열기 위해 Matlab을 명령 프롬프트에서 다음을 입력합니다 "movieSelectorGUI". 을 만드는 지침에 따라이전에 저장 한 별도의 이미지부터 새 영화 데이터베이스 (4.1.2 참조).
- 입자 검출 및 추적에 필요한 화소 크기에서 나노 초 시간 간격, 개구, 카메라 비트 깊이와 형광 물질의 발광 파장을 제공한다. 영화 데이터베이스를 저장합니다.
- 영화 선택 인터페이스에서 객체의 유형으로 "단일 입자"를 선택, 분석을 실행합니다. 입자 탐지 및 추적에 사용되는 매개 변수를 정의 할 수있는 새 창이 나타납니다. 기본 매개 변수로 시작합니다. . 감지 및 / 또는 추적의 품질이 선택 (예를 들어, 일부 입자가 검출이나 트랙이 단편화되지 않습니다) 만족하지 않은 경우 나중에 이러한 매개 변수를 조정합니다 추적 설정에서 저장하는 "매트릭스 형식으로 내보낼 추적 결과를"확인 "trackedFeaturedInfo라는 단일 매트릭스 (필드의 좌표와 모든 입자의 진폭"). 이러한 매개 변수에 대한 자세한 내용은 U-트랙 문서를 참조하십시오.
- 검출 알고리즘을 실행합니다. 이 알고리즘은 자동으로 각 회절 한정 스폿 로컬 강도 맥시마 주위 현미경의 점 분포 함수와 동일한 표준 편차 이차원 가우스 함수 피팅에 의해 (즉. 단일 수용체 / 수용체 복합체)의 배경 위에 위치 및 세기를 결정 . 그 후, 추적 알고리즘을 실행. . 매트 파일의 분석 결과를 저장합니다.
- 트랙을 시각화하고 탐지 및 추적의 품질을 확인하기 위해 U-트랙 패키지에 포함 된 "movieViewer"루틴, 또는 유사한 사용자 지정 것들을 사용합니다.
- 각 프레임에서 추적 입자의 위치와 진폭 (예., 강도)을 볼 수 있도록. 매트 파일을 엽니 다. 4.2.4 단계에서 생성 된 데이터는 tracksFinal 아칸소의 tracksCoordAmpCG 필드에 포함되어 있습니다선 및 / 또는 trackedFeaturedInfo합니다. 입자의 총 개수에서 4.1.3에서 측정 셀 표면적으로 나눈 값이 입자 밀도를 산출 검출.
- 선택적 : 입자가 수용체 입자의 움직임을 분석하는 (4.2.6 참조) 시간의 경과에 위치 사용. Matlab을 유사한 소프트웨어를 사용하여 평균 제곱 변위 (MSD) 및 확산 계수 (D)를 계산한다. 각 입자마다 시간 간격 (Δ의 t)를 고려, 다음과 같은 공식을 사용하여 평균 제곱 변위 (MSD)를 계산 :
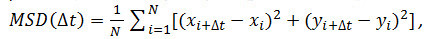
Δ의 t 프레임의 시간 간격이고, N 분석 단계의 수이고, x 및 y는 입자의 X 및 Y는 인덱스에 의해 지시 된 프레임에 위치한다. 선형 관계 : 주어진 입자의 운동 종류를 평가하는 시간 플롯 위에 MSD를 사용자유 확산 (예., 브라운 운동), 양의 곡률을 나타냅니다 (즉., 곡선이 포물선 모양) 감독 운동을 제안, 음의 곡률이 제한 운동 (14)을 나타낸다. 자유롭게 확산 입자의 경우, 다음의 식으로 얻어 MSD 데이터 피트에 의해 각 입자의 확산 계수 (D)를 계산한다 :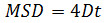
- 가우스 피팅 방법으로 입자 크기의 계산
NOTE : 보정 샘플 (유리 및 / 또는 형광 표지 된 단량체 성 수용체에 단일 형광체)의 강도 분포를 알면, 수용체 복합체의 크기를 결정하기 위해 이미지 시퀀스의 시작 부분에서의 입자 농도의 분포에 혼합 가우스 피팅을 수행 (예. 입자 당 수용체의 수) 10. Matlab을 또는 s를 사용하여 이러한 분석을 수행imilar 소프트웨어.- 강도의 변화가 제 (photobleaching에 의한 대부분의 경우에 저하)가 발생하기 전에 프레임의 첫 번째 프레임에서의 입자의 강도를 평균하여 각 입자의 강도를 계산한다.
- 다음 식에 따라 혼합 가우시안 피팅을 수행합니다 :
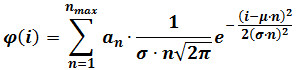
φ (i)는 입자가 나는 강도를 갖는 주파수이고, n은 구성 요소의 수입니다, α N 구성 요소 n의 높이에 기여하는 매개 변수이고, μ와 σ는 평균과 참조 하나의 형광 강도의 표준 편차입니다 . (3 단계에서 설명 된대로 계산) 주 : T를 결정F-검정에 의해 판단으로 그는 점차 구성 요소를 추가 할 때까지 N 최대 증가하여 각 이미지 시퀀스에 대한 구성 요소 (N 최대)의 최대 수는 더 이상 통계적으로 더 나은 피팅을 생산하지 않습니다. - 선택 사항 : (. 예를 들면, 400 프레임 시퀀스의 마지막 60 프레임) 동영상의 마지막 프레임에서 얻어진 강도 분포에 혼합 가우시안 적합을 수행합니다. 이러한 매개 변수에 대한 정교한 예측을 제공하는이 피팅 후의 값, 단계를 반복 4.3.2와 μ와 σ를 교체합니다.
- 혼합 가우스 적당한 각 성분의 곡선 (AUC) 아래의 면적을 계산한다. 다른 크기 분포 전체의 AUC에 의해 각 구성 요소의 AUC 값을 나눔으로써 (즉, 단량체, 이량 체, 삼량 체 등)의 수용체 입자의 상대적인 풍부함을 계산한다.
- 선택 사항 : 다른 세포와 다른 크기와 입자의 분포는 입자 밀도 (10)와 상관 관계 플롯을 생성하는 (4.2.6에 설명 된대로 계산) 해당 입자 밀도에서 사용 데이터.
- 단계 피팅 방법으로 입자 크기의 계산
NOTE : 수용체 복합체 (10)의 크기를 결정하기 위해 다른 방법으로 스텝 - 피팅 분석을 사용한다. N의 형광을 포함하여 입자 점진적으로 표백제 N 단계를 강도 프로파일을 생성 할 것으로 예상된다 -이 분석의 기초는 순간 실종에 하나의 형광 결과의 빛에 의한 파괴 (photobleaching에)가 있다는 것입니다.- U-트랙 또는 유사한 탐지 / 추적 소프트웨어 (4.2.6 참조)에 의해 생성 된. 매트 파일에서 각 입자의 강도 프로파일을 추출합니다.
- 이러한 제시된 하나로서, 스텝 피팅 알고리즘을 사용심판의. 10, 각 입자에 대한 표백 단계의 수를 계산.
- 선택적 : (4.3 참조) 다른 크기의 수용체 입자의 상대적인 풍부함을 나타내는 분포를 생성하고 이전에 혼합 가우스 피팅의 결과에 대하여 기술 된 바와 같이 입자 밀도로 그들을 연관시키는 결과를 사용한다.
결과
기술 된 프로토콜은 서로 다른 막 단백질의 다양한 적용 할 수있다. 예로서, β-2 아드레날린 성 및 GABA B 수용체로 얻어진 대표적인 결과 (10)를보고한다. 하나의 분자에서 형광 신호가 미약하기 때문에, 배경 형광의 최소화는 성공적인 결과에 대한 첫 번째 중요한 단계입니다. 따라서, 광범위하게 세정 커버 슬립 (도 2A)를 사용하는 것뿐만 아니라, 시료의 autofluo...
토론
설명 프로토콜은 단일 분자 수준에서 공간 배열, 이동성과 세포 표면 수용체 복합체의 크기를 분석 할 수 있습니다. 형광 단백질의 사용에 비해 더 밝고 광 안정성이다 작은 유기 형광체와 라벨은 단일 수용체 입자의 확장 된 시각화를 허용하는 장점을 갖는다. 매우 낮은 발현 수준이 달성되기 때문에 (<0.45 수용체 입자 / 2 ㎛)을, 수용체 및 기타 막 단백질의 생리 학적 특성은 사람을 초?...
공개
The authors declare that they have no competing financial interests.
감사의 말
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
자료
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
참고문헌
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유