JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
동심 젤 시스템은 3 차원 세포의 이동에 매트릭스 미세 환경의 생물 물리학 역할을 연구하는
요약
기계적 특성 및 세포 외 매트릭스의 미세 강하게 세포의 3 차원 이동에 영향을 미칩니다. 시험 관내 방법은 개별 세포 집단 수준에서 모두 biophysically 변수 환경에서 시공간 세포 이동 동작을 연구하기 위해, 설명한다.
초록
이동하는 세포의 능력은 암 및 암전이에에서 배아 발생 및 상처 치유 수명에 걸쳐 세포 기능의 다양한 중요하다. 강렬한 연구 노력에도 불구하고, 세포 이동의 기본 생화학 및 생물 물리학 적 원리는 아직도 완전히 특히 생리 관련 3 차원 (3D) 미세 환경에서 이해되지 않습니다. 여기서는 3D 세포 이동 동작의 정량적 검사가 가능하도록 설계 시험 관내 분석을 기술한다. 이 방법은 이전에 빈 세포 외 기질 (ECM)로 마이그레이션하는 세포의 mechanosensing 능력과 성향을 이용한다. 우리 모델 시스템으로서 콜라겐 겔에서 고도로 침윤성 유방암 세포의 침윤, MDA-MB-231을 사용한다. 주 동안 배양 세포 집단의 확산 및 개별 세포의 이주 역학 라이브 세포 이미징을 사용하여 모니터링 및 시공간 - 분해 된 데이터를 추출하기 위해 분석 될 수있다. 게다가상기 방법에 따라서 세포 이동에 생물 물리학 적 미세 인자의 역할을 조사하기 간단하지만 강력한 방법을 제공하고, 다양한 세포 외 기질을위한 용이하게 적응할 수있다.
서문
세포의 이주는 배아 발달, 지혈, 면역 응답으로서뿐만 아니라, 혈관 질환, 염증, 암과 같은 병리학 한 공정에서 다양한 생리 학적 반응에서 중요한 역할을한다. 세포 이동의 기초 생화학 및 생물 물리학 적 요인을 해부하여 세포 기능의 기본 원리를 이해뿐만 아니라, 조직 공학, 안티 전이 및 항 염증성 약물 개발 등 다양한 생물 의학 응용 프로그램을 전진뿐만 따라서 근본적으로 중요하다. 생체 관측 기술적 도전이기 때문에, 많은 노력은 세포 이동의 시험관 재현부에 집중되어왔다.
세포의 이동을 연구하는 체외 방법에서 크게 특히, 두 개의 차원 (2D) 표면 분석을위한 스크래치를 설계 또는 분석이 상처 치유되었습니다. 이러한 분석은 간단한 실험 장치를 제공, 쉬운 가축세포 이미징 및 세포 이동의 기초가 다양한 생화학 적 메커니즘에 유용한 통찰력을 제공한다. 그러나 이러한 분석은 생체 마이그레이션에 대한 이해의 중요한 측면이다 세포 외 기질 (ECM) 건축과 리모델링을 고려하지 않습니다. 최근에는 점차 3D 배양 모델은 종종 콜라겐 계 매트릭스 3에서, 더 나은 상황 생체 유사한 플랫폼을 제공하는 것이 이해되었다. 실제로, 세포는 특히 인해 환경 (4)의 다른 차원을 2 차원 표면에 나타나는 것과 구별되는 migrational 동성을 나타낸다. 더욱이, 행렬의 생물 리 학적 및 기계적 특성을 민감하게 종양 세포 침윤 (6)의 컨텍스트에 포함하여, 세포 이동 (5)에 영향을 미친다.
여기서, 우리는 제조 조건에서 쉽게 변할 수 생물 리 학적 특성을 갖는 ECM 차원 셀 마이그레이션 동작을 연구하는 방법을 제시한다. 세포는 아르"내부 젤"의 시드와로 새어 처음에 무 세포 "외부 젤을"침략 수 있습니다. 상기 방법은 세포가 밀접 7 mechanosensing 연결되어 외측 겔에서 무 세포 지역으로 이동하는 세포의 존재를 인식 할 수있는 능력, 및 성향에 의존한다. 이 연구에서 우리는 매우 침습적 유방암 세포, MD-MBA-231에 의해 침략 ECM의 콜라겐 네트워크를 사용합니다. 기계적 특성 모두 내측 및 외측 겔의 미세 조정 8 및 생리 학적으로 관련된 조건을 달성하기 위해 구를 특징으로 할 수있다. 셀 트랙의 재건 및 분석은 인구 수준 및 개별 셀 레벨 모두에서 시공간 마이그레이션 동작의 상세한 정량적 검사를 할 수 있습니다. 중요한 것은, 동심 젤 시스템의 설치, 따라서 중요한 통찰력을 제공하고, 특히 암 세포에 침입, 세포 마이그레이션 직면 한 생체 조직 토폴로지를 모방세포 이동 및 전이의 물리적 메커니즘.
프로토콜
1. 세포 수확
- 37 ° C, 5 % CO 2 배양기에서 MD-MBA-231 세포를 얻습니다. 0.5 % 트립신 - EDTA 용액을 이용하여 조직 배양 접시에서 세포를 분리합니다. T25 플라스크에서 배양 세포 트립신 EDTA 용액 1 ㎖를 사용합니다.
- 4 분의 200 × g에서 원심 분리하여 15ml의 원뿔형 튜브 펠렛의 세포 상등액을 흡인하고, 세포를 재현 탁 배양액 5ml에.
- 혈구를 사용하여, ρ, 셀 밀도를 계산.
주 : 셀 시드 내측 겔을 제조하기 위해, 세포 현탁액을 이후 10 × 최종 세포 파종 밀도에 도달하기 위해 희석된다. 따라서, 10 × 세포 현탁액 농축이 요구된다. - 10 × 세포 농도를 달성하는 데 필요한 유체의 체적을 계산한다 :
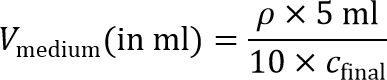
참고 : 마지막 셀 시드 밀도를 최종 C, 약 2의(15); 10 6 세포 / ㎖의 MD-MBA-231 세포에 권장되며,이 프로토콜에 사용된다. 기타 파종 밀도는 다른 세포 유형에 대해 탐구 할 수있다. - 펠렛의 세포 하나 더 4 분 동안 200 × g에서 원심 분리하여 15 ML 원뿔 튜브 시간, 뜨는을 대기음.
- 세포 응집을 최소화하기 위해 충분히 세포의 무 혈청 배지 (단계 1.3에서 계산 V 배지)에 필요한 양의 세포를 재현 탁.
주 : 페놀 레드가 자동 형광, 형광 / 반사 영상을 방해 할 수 있습니다. 페놀 레드없는 배지의 사용은 최상의 이미지 품질을 달성하기 위해 고려 될 수있다.
콜라겐 솔루션 2. 준비
- 주식 콜라겐 솔루션, 10 × PBS 버퍼, 밀리-Q의 H 2 O, 0.1 M의 NaOH, 여러 마이크로 원심 튜브를 얻습니다. 조기 콜라겐 중합을 방지하기 위해 얼음을 모두 유지하고 무균 상태를 유지한다.
- 멸균 유리 - 평형을37 ° C 배양기에서 사전 온난화로 바닥 접시.
참고 :이 프로토콜의 모든 볼륨이 잘 12mm 유리 바닥 접시에 최적화되었습니다. 다른 접시 형이 사용되는 경우, 대응하여 볼륨을 조절한다. - 콜라겐 스톡 농도에 기초하여 상기 내측 겔 (용액 I) 2.4 ㎎ / ㎖의 콜라겐 용액 50 μL를 준비하기 위해 필요한 요구되는 부피를 계산한다.
주 : 겔 내측 기타 콜라겐 농도도 사용될 수있다. - 멸균 환경 (일반적으로 바이오 안전성 후드)에서, 천천히 부드러운 소용돌이와 (단계 2.3에서 계산) 콜라겐 원액 필요한 양의 10 × PBS 버퍼의 5 μl를 추가합니다. 공기 방울 형성을 방지하기 위해주의하십시오.
- 교정의 pH 미터를 사용하여 0.1 M의 NaOH를 이용하여 7.4로 혼합물의 pH를 조정한다. 대략적인 기준으로, (양이 주식 농도와 산도에 따라 달라집니다) 7.4에 가까운 pH를 가지고 약 5 μl를 사용합니다.
참고 : 볼륨이에 관여합니다단계는 표준 pH 미터를 사용하기에는 너무 작다. 다음 트릭 중 하나를 사용하여- 여러 샘플 콜라겐 솔루션을 준비합니다. 표준 pH 측정기를 사용하여 대량으로 pH를 조정하고 샘플에 걸쳐 콜라겐 솔루션을 배포 할 수 있습니다.
- 대안 적으로, 더 많은 양의 콜라겐 용액의 pH를 조정 (Ⅰ. 예., 표준 pH 미터의 사용을 허용 양). 최종 pH로 pH를 가져 필요한 양의 NaOH를 참고. 볼륨을 확장하고 실험에 NaOH를 적절한 양을 사용합니다. 리트머스 종이를 사용하여 pH 값을 확인합니다.
- 그렇지 않으면, 더 정확하게 소량의 pH를 조정하기 위해 마이크로 pH 전극을 사용한다.
- O. H 2 μL를 사용하여 45의 부피로 용액을 가져와 조기 콜라겐 중합을 방지하기 위해 얼음의 모든 단계를 수행합니다.
동심 젤 문화 3. 형성
- 예열 된 유리 바닥 접시를 타고 일부터 (단계 2.2 참조)전자 인큐베이터.
- I. 용액에 재현 탁하고 (단계 1.5에서 제조) 10 × 농축 된 세포 현탁액의 5 μL를 추가 철저. 공기 방울 형성을 방지하기 위해주의하십시오. 혼합물을 50 μL의 현재 볼륨 및 (최종 = 2 × 106 세포 / ㎖ c) 최종 농도 콜라겐 (2.4 ㎎ / ㎖), 셀 밀도를 포함한다.
- 이 돔 형상의 액적 (도 1a)을 형성하도록 I는 웰의 중심으로 서서히 세포 - 함유 용액 20 μL를 추가한다. 이 단계에서 거품 형성을 방지하기 위해주의하십시오. 거품 형태의 경우, 신중하지만 빨리 시도 중 하나를 파열되거나 피펫 팁을 사용하여 빨아. 조심스럽게 내부 겔을 45 분 동안 중합 수 있도록 인큐베이터에서 접시를 놓습니다.
- (외부, 무 세포 콜라겐 젤) 솔루션 O이 배양 단계가 끝나기 전에 약 15 분을 준비합니다.
참고 : 외부 젤 상태는 콜라겐 농도와 중합의 pH 조건에 변경 될 수 있습니다다른 마이크로 (10)과 네트워크를 구하십시오. 7.4의 pH에서 중합 4.0 ㎎ / ㎖ 콜라겐 젤 -이 프로토콜에서 1.5에 초점을 맞 춥니 다.- 콜라겐 스톡 농도에 기초하여, 최종 농도 콜라겐 용액 O 200 μL를 준비하는 데 필요한 요구 체적을 계산한다.
- 10 × PBS의 20 μL는 부드러운 소용돌이와 (3.4 단계에서 계산) 콜라겐 원액의 필요한 양을 천천히 버퍼에 추가합니다. 교정의 pH 미터를 이용하여, 0.1 M의 NaOH를 사용하여 최종 pH로 혼합물의 pH를 조정한다. pH 조정에 대한 2.5 단계로주의를 참조하십시오.
- O. H 2를 사용하여 200 μL의 최종 부피로 용액을 가져와 조기 콜라겐 중합을 방지하기 위해 얼음의 모든 단계를 수행합니다.
- 내부 겔의 45 분의 중합 후 인큐베이터에서 접시를 타고 (단계 3.3 참조). 이 솔루션은 완전히 내부를 덮도록 조심스럽게, 내부 젤 위에 솔루션 O 180 μl를 추가젤과 잘 (그림 1B)를 채 웁니다.
- 외부 젤 비 균일 한 섬유 방향으로 이어질 수있는 솔루션을 교반하지 않고 신중하게이 단계를 수행합니다. 피펫 팁과 내부 젤에 손이 닿지 않도록주의하고, 기포 나 공기 주머니의 형성을 방지하기 위해. 거품 형태의 경우, 신중하지만 빨리 시도 중 하나를 파열되거나 피펫 팁을 사용하여 빨아. 부드럽게 외부 겔 중합을 수 있도록 인큐베이터에서 접시를 다시 배치합니다.
- 중합 45 분 후 인큐베이터에서 접시를 가져 가라. 거칠게 취급하면 여전히 바닥면으로부터 분리 할 수 있지만, 이미 상당히 겔,이 시점에서 고화한다.
- 부드럽게 접시 (도 1C)로 가온 세포 배양 배지 2 ㎖를 붓는다. 젤이 완전히 매체에 빠져들되어 있는지 확인합니다. 문화의 기간에 걸쳐 3 일 - 매 2 매체를 새로 고칩니다.
4. 라이브 셀 이미징
- 장기 라이브 세포 이미징 기능을 갖춘 반전 공 초점 현미경을 사용하여 이미지를 수행합니다. 포함 내장 된 온도 (37 ° C) 및 CO 2 (5 %) 제어와 배양 챔버. 현미경에 전환하고 실험을 시작하기 전에 단계를 적어도 1 시간을 따뜻하게.
참고 : 긴 작동 거리와 사용 대물 렌즈는 3D 젤 세포의 관찰 및 현지화를 최적화 할 수 있습니다. - 3 차원 시스템에서 세포의 정확한 위치 파악을 할 수 있도록 30 분 동안 형광 세포 추적기 염료 5 μl를 함유하는 배지에서 배양 한 겔. 그 후, 1 × PBS로 세척하여 세 번 결합되지 않은 염료를 제거합니다. 그 후, 접시에 세포 배양 배지를 추가한다.
- 인큐베이터에서 접시를 타고 현미경 스테이지 (그림 1D)에 놓습니다.
참고 : 라이브 세포 이미징 바로 외부 겔의 중합 후 원칙적으로 시작할 수 있습니다. 그러나이 시점에서, 세포를 전n은 내부 젤은 아직 확산되지 않았다. 외측 겔, 라이브 셀 이미징 배양 개시 후 (셀 타입에 따라) 24 시간을 시작할 수 침범 제 세포의 이주를 조사. 대략적인 기준으로, 약 12 - 문화의 14 일이 외부 젤을 입력 내부 겔에 세포의 대부분이 필요하다. - 내부 젤을 둘러싼 외부 젤 지역에서보기 (VOV의)의 볼륨을 선택. 24 시간의 배양이 세포 집단 확산 것이다 후 내부와 외부 사이의 계면 겔을 교차하고, 외측 겔 침투하기 시작했다.
- 보브의 경우, 바로 옆에있는 외부 젤 (7)의 외곽에 가까운 겔 인터페이스, 중간 지역, 지역에 젤 영역을 포함한다. 가능한 에지 효과를 피하기 위하여, 겔의 상단에서뿐만 아니라, 가까운 아래쪽 및 측면 표면으로부터 50㎛의 영역보다 제외. 각 보브는 일반적으로 X에서 (647 × 647 × 100 μm의 3을 측정Z의 -stack 5 μm의 간격이 각각, Y, Z 방향).
- 확인 촬상 모드, 채널 / 필터, 노출 시간, 이미지 해상도가 올바르게 선택된다. 콜라겐 네트워크의 라벨없는 이미징의 경우, 시간 경과 라이브 셀 이미징 동안 동시에 반사율 공 촛점 현미경을 사용한다.
- 샘플 이미지를 가지고 강도 막대 그래프를 플롯과 충분한 신호를 관찰하고 히스토그램이 제로 최대 강도 사이에 위치하도록함으로써 포화를 방지하기 위해 이득 및 오프셋을 조정합니다. 실험 기간에 걸쳐 더 이상 이러한 설정을 변경하지 마십시오.
- 8 시간 (또는 필요 이상) 10 분, 시간 간격, Δ의 t으로, 선택한 보브 년대의 시간 경과 이미지를 가져 가라.
5. 셀 추적 및 데이터 분석
- appropria를 사용하여 Z -stack 이미지에서 정량적 이미지 포스트 - 프로세싱을 수행 테 이미지 처리 소프트웨어.
- 세그먼트 시간 경과 이미지는 자동으로 값 (x, y, z, t)의 3D 셀 위치를 선택합니다.
- 모든 프레임의 경우, 수동으로 현지화 정확성을 확인하고 세포 착각되었을 수 세포 파편 및 세포 돌기 때문에 오탐 (false positive)를 제거합니다. 다른 오브젝트로 분석 및 분할 겹치거나 연결된 세포의 증식 세포를 제거합니다.
- 시계열로 각 셀의 위치를 연결하여 이전 단계에서 얻어진 셀 좌표 (x, y, z, t)로부터 3D 경과 셀 트랙을 생성한다.
- 임계 트랙 길이 (보통 20 분)보다 짧은 트랙을 제거하여 임의의 시스템 노이즈를 제거합니다.
- 필요한 경우, 트랙에 의한 전체적인 순 변위를 감산함으로써 샘플 드리프트 보정.
- 셀의 변위를 계산세틸 / ftp_upload / 52735 / 52735eq2.jpg "폭 ="80 "/>, 및 세포 이동 거리,
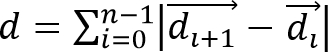 관찰 된 셀 궤적으로부터, 여기서시 셀의 3 차원 위치를 나타내는 벡터이고, 시점의 총 수이다.
관찰 된 셀 궤적으로부터, 여기서시 셀의 3 차원 위치를 나타내는 벡터이고, 시점의 총 수이다. - Δ (t)가 프레임 간의 시간 간격, S = D / (n 개 •의 Δ의 t)와 같은 세포 속도를 계산한다. P = Δd 및 / D를 사용하여 세포 이동의 방향성 (또는 지속성)을 계산합니다. 지속성이 간단한 측정은 P = 0 순 변위가 0이고 P = 1 궤적이 직선 방향 라인이 있음을 의미한다.
결과
여기에 제시된 동심 겔 분석 고도로 침윤성 유방암 세포를 사용하여 수행하고, MDA-MB-231, 2.4 ㎎ / ml의 내측 콜라겐 젤, 예로서 = 2 × 106 세포 / ml의 세포 시딩 밀도와. 도 2에 도시 된 바와 같이, 통상적으로 배양 몇 일 후, 세포 내외 겔 인터페이스를 위반하고 외측 겔 침투하기 시작했다. 셀 인구는 반경 방향 바깥쪽으로 주로 확산.
외측 겔 중합 조건은 세포...
토론
In this protocol we describe an in vitro assay to study the 3D migrational behavior of cells in matrix environments that topologically resemble ECMs encountered in vivo. There are three main strengths of this assay as compared to other currently available methods. First, this assay allows one to simultaneously examine the cell migration mechanisms at both population level and individual cell level. This opens up possibilities of studying collective cell migration13, which has to date been lar...
공개
저자가 공개하는 게 없다.
감사의 말
저자는 중요한 토론 W. 일 및 K. 젠슨 감사, 싱가포르 국립 대학의 나노 역학 연구소의 지원을 인정합니다. NAK는 마리 퀴리 IIF 원정대에 의해 지원을 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| Cell culture incubator | Fisher Scientific Pte Ltd | Model: 371, S/No 318854-6055 | |
| Confocal microscope | Nikon A1R | Inverted confocal laser scanning microscope equipped with incubator chamber | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Life Technologies | 11965-092 | |
| Fetal Bovine Serum (FBS) | Life Technologies | 10082-147 | |
| Fluorescent CellTracker dye CMTMR | Life Technologies | C2927 | |
| Glass-bottom dish | IWAKI Cell Biology | 3931-035 | 35 mm diameter dish with 12 mm diameter glass-bottom well |
| Hemocytometer | iN CYTO | DHC-N01 (Neubauer Improved) | |
| Microprocessor pH meter | Hanna Instruments | pH 211 | |
| Nutragen Collagen | Advanced BioMatrix | #5010-D | Acid-solubilized bovine collagen type I (stock pH ~ 2) |
| Objective lens | Nikon | CFI Super Plan Fluor ELWD ADM 20XC, W.D. 8.2-6.9mm, NA 0.45. | |
| Penicillin-Streptomycin | Life Technologies | 15140-122 | |
| pH meter | Sartorius | S/No 29153352 | Basic pH Meter PB-11 |
| Trypsin-EDTA | Life Technologies | 15400-054 |
참고문헌
- Horwitz, R., Webb, D. Cell migration. Curr Biol. 13 (19), R756-R759 (2003).
- Liang, C. C., Park, A. Y., Guan, J. L. In vitro scratch assay: a convenient and inexpensive method for analysis of cell migration in vitro. Nat. Protoc. 2 (2), 329-333 (2007).
- Provenzano, P. P., Eliceiri, K. W., Inman, D. R., Keely, P. J. Engineering three-dimensional collagen matrices to provide contact guidance during 3D cell migration. Curr. Prot. Cell Biol. 10, 10-17 (2010).
- Friedl, P., Sahai, E., Weiss, S., Yamada, K. M. New dimensions in cell migration. Nat. Rev. Mol. Cell Biol. 13 (11), 743-747 (2012).
- Grinnell, F., Petroll, W. M. Cell motility and mechanics in three-dimensional collagen matrices. Annu. Rev. Cell. Dev. Biol. 26, 335-361 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nat. Rev. Cancer. 11 (7), 512-522 (2011).
- Sun, W., Kurniawan, N. A., Kumar, A. P., Rajagopalan, R., Lim, C. T. Effects of migrating cell-induced matrix reorganization on 3D cancer cell migration. Cell. Mol. Bioeng. 7 (2), 205-217 (2014).
- Achilli, M., Mantovani, D. Tailoring mechanical properties of collagen-based scaffolds for vascular tissue engineering: the effects of pH, temperature and ionic strength on gelation. Polymers. 2 (4), 664-680 (2010).
- Kurniawan, N. A., Wong, L. H., Rajagopalan, R. Early stiffening and softening of collagen: interplay of deformation mechanisms in biopolymer networks. Biomacromolecules. 13 (3), 691-698 (2012).
- Sun, W., Lim, C. T., Kurniawan, N. A. Mechanistic adaptability of cancer cells strongly affects anti-migratory drug efficacy. J. R. Soc. Interface. 11 (99), 20140638 (2014).
- Guzman, A., Ziperstein, M. J., Kaufman, L. J. The effect of fibrillar matrix architecture on tumor cell invasion of physically challenging environments. Biomaterials. 35 (25), 6954-6963 (2014).
- Wolf, K., et al. Physical limits of cell migration: control by ECM space and nuclear deformation and tuning by proteolysis and traction force. J. Cell Biol. 201 (7), 1069-1084 (2013).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol. Cell Biol. 10 (7), 445-457 (2009).
- Vedula, S. R. K., Ravasio, A., Lim, C. T., Ladoux, B. Collective cell migration: a mechanistic perspective. Physiology. 28 (6), 370-379 (2013).
- Petrie, R. J., Doyle, A. D., Yamada, K. M. Random versus directionally persistent cell migration. Nat. Rev. Mol. Cell Biol. 10 (8), 538-549 (2009).
- Miron-Mendoza, M., Seemann, J., Grinnell, F. The differential regulation of cell motile activity through matrix stiffness and porosity in three dimensional collagen matrices. Biomaterials. 31 (25), 6425-6435 (2010).
- Mouw, J. K., et al. Tissue mechanics modulate microRNA-dependent PTEN expression to regulate malignant progression. Nat. Med. 20 (4), 360-367 (2014).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin. Cell Dev. Biol. 20 (8), 931-941 (2009).
- Wong, L. H., Kurniawan, N. A., Too, H. -. P., Rajagopalan, R. Spatially resolved microrheology of heterogeneous biopolymer hydrogels using covalently bound microspheres. Biomech. Model. Mechanobiol. 13 (4), 839-849 (2014).
- Gupton, S. L., Waterman-Storer, C. M. Spatiotemporal feedback between actomyosin and focal-adhesion systems optimizes rapid cell migration. Cell. 125 (7), 1361-1374 (2006).
- Provenzano, P. P., Inman, D. R., Eliceiri, K. W., Trier, S. M., Keely, P. J. Contact guidance mediated three-dimensional cell migration is regulated by Rho/ROCK-dependent matrix reorganization. Biophys. J. 95 (11), 5374-5384 (2008).
- Wolf, K., et al. Multi-step pericellular proteolysis controls the transition from individual to collective cancer cell invasion. Nat. Cell Biol. 9 (8), 893-904 (2007).
- Jansen, K. A., Bacabac, R. G., Piechocka, I. K., Koenderink, G. H. Cells actively stiffen fibrin networks by generating contractile stress. Biophys. J. 105 (10), 2240-2251 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유